Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista mexicana de fitopatología
versão On-line ISSN 2007-8080versão impressa ISSN 0185-3309
Rev. mex. fitopatol vol.41 no.2 Texcoco Mai. 2023 Epub 11-Ago-2023
https://doi.org/10.18781/r.mex.fit.2210-4
Artículos de revisión
Efectores: actores claves en la fitopatología
1 Unidad de Biotecnología, Centro de Investigación Científica de Yucatán, A.C., Mérida, Yucatán, México, C.P. 97205.
Los efectores son moléculas pequeñas, mayormente proteínas, producidas por los microorganismos que las utilizan para interaccionar con sus hospederos. En el caso de los hospederos vegetales, los efectores suprimen la inmunidad vegetal interfiriendo en la percepción del microorganismo, la señalización y la biosíntesis de fitorreguladores, entre otros procesos. En los últimos años ha crecido el interés de los efectores en la fitopatología debido a su contribución en la virulencia de los fitopatógenos y, por ende, en el impacto de éstos en la producción agrícola. Sin embargo, los efectores son complejos. Por un lado, estas moléculas son secretadas para el beneficio del fitopatógeno y suelen desencadenar susceptibilidad a la enfermedad. Sin embargo, las plantas han desarrollado receptores que reconocen a algunos efectores y este reconocimiento desencadena resistencia a la enfermedad. Es decir, algunos efectores resultan en salud vegetal, mientras otros determinan el desarrollo de la enfermedad. Esta revisión se enfoca en los efectores de los fitopatógenos y sus funciones, así como los mecanismos que muchos usan para vencer la inmunidad innata vegetal, por lo que son actores claves en la fitopatología. Por último, se describen los potenciales usos de los efectores en el sector agrícola y los retos asociados con su aplicación.
Palabras claves: Efectores proteicos; efectorómica; interacción planta-patógeno; proteínas R; cognados; protección vegetal; fitopatología
Effectors are small molecules, mostly proteins, produced by microorganisms that use them to interact with their hosts. Regarding plant hosts, effectors suppress plant immunity by interfering with microorganism perception, signaling, and biosynthesis of phytoregulators, among other processes. In recent years, interest in effectors in phytopathology has grown due to their contribution to phytopathogen virulence and, by extension, their impact on agricultural production. However, effector molecules are complex. On one hand, these molecules are secreted for the benefit of the phytopathogen and often trigger disease susceptibility. However, plants have evolved receptors that recognize some effectors, and this recognition can trigger disease resistance. Essentially, some effectors safeguard plant health, while others promote disease development. This review focuses on the effectors of phytopathogens and their functions, as well as the mechanisms that many of them use to overcome plant innate immunity, making them key players in phytopathology. Finally, the potential uses of effectors in the agricultural sector and the challenges associated with their application are described.
Key words: Protein effectors; effectoromics; plant-pathogen interaction; R proteins; cognates; crop protection; phytopathology
Todas las plantas, incluyendo las de mayor importancia agrícola en el mundo, como el arroz, el maíz, la soya y el trigo se ven afectados por severas infecciones causadas por fitopatógenos como bacterias, hongos, oomicetos y virus (Nazarov et al., 2020; Velásquez et al., 2018). Las infecciones causadas por Magnaporthe oryzae en arroz, Puccinia spp. en trigo y Fusarium spp. en todos los cereales, por ejemplo, contribuyen a las pérdidas en rendimiento y calidad de la cosecha de manera global (Almeida et al., 2019). Se estiman que las pérdidas agronómicas causadas por plagas y enfermedades abarcan hasta 40% de la producción global anual (FAO, 2017). Las incidencias de las enfermedades vegetales reducen la producción de alimentos y aumentan el costo de producción, haciéndoles menos accesibles al consumidor (Ristaino et al., 2021; Savary et al., 2019) Además, reducen la diversidad de las especies, por ejemplo, de insectos y microbios benéficos (Gupta et al., 2022; van der Sluijs, 2020), y presentan un riesgo a la salud humana debido al uso indiscriminado de pesticidas para controlar las poblaciones de los fitopatógenos (Rani et al., 2021).
Centrar esfuerzos en el estudio de factores claves que impiden o determinan el desarrollo de la enfermedad vegetal puede permitir el desarrollo de métodos de control eco-amigables que reduzcan el control químico (Thakur et al., 2020; Baker et al., 2020). El objetivo de esta revisión es mostrar la importancia de los efectores en la fitopatología y proponer sus potenciales usos para la protección vegetal. Para esta revisión se buscaron artículos con palabras clave como “efectores proteicos”, “efectores en interacciones planta-patógeno”, “aplicaciones de efectores” y “efectoromica en hongos patogénicos” en las bases de datos de Google Scholar y PubMed. Se seleccionaron artículos de los últimos 15 años, con mayor énfasis en los últimos cinco años; también se incluyeron algunos artículos más antiguos que son referentes en el área de los efectores.
Muchos efectores desempeñan un papel en la supresión de las respuestas inmunitarias de la planta hospedera; algunos efectores son enzimas hidrolíticas, otros son inhibidores de enzimas, otros juegan un papel en modular el microbioma del hospedero o protegen físicamente al patógeno contra lisis enzimática (Rocafort et al., 2020; Schreiber et al., 2021; Snelders et al., 2022; Zhang et al., 2022).
Los efectores forman parte del arsenal molecular de los microorganismos, tanto benéficos como patogénicos, que interactúan con la planta hospedera o con otros microorganismos (Castillo-Sanmiguel et al., 2020; Todd et al., 2022a). Sin embargo, estas moléculas se han estudiado mayormente en el contexto de los fitopatógenos, ya que son herramientas clave empleadas por el fitopatógeno para lograr una infección y obtener los nutrientes del hospedero. Se han encontrado en fitopatógenos fúngicos (Carreón-Anguiano et al., 2020; Sperschneider et al., 2018), bacterianos (Rufián et al. 2021), virales (Huang, 2021) hasta organismos más grandes como los insectos (Chen et al. 2019; Ray y Casteel, 2022) y nematodos (Figura 1A) (Verhoeven et al., 2023; Vieira y Gleason, 2019). El desarrollo de las tecnologías ómicas (genómica, transcriptómica y proteómica) así como de programas bioinformáticos, han permitido la identificación e investigación de amplios catálogos de efectores contenidos en los genomas de los organismos (Carreón-Anguiano et al., 2022; Chen et al., 2021; Huang et al., 2022).

Figura 1 Interacción patógeno-hospedero. A) Efectores, las armas moleculares de fitopatógenos y plagas. Las bacterias, hongos, oomicetos, virus, insectos y nematodos son capaces de secretar una plétora de efectores hacia el hospedero, que les permiten impedir su reconocimiento por la planta, bloquear la respuesta inmune y favorecer su propio desarrollo. B) Por su parte, las plantas, en la primera línea de defensa, reconocen “patrones moleculares asociados a microorganismos” (MAMP, por sus siglas en inglés), que son moléculas conservadas, como la quitina de los hongos y activa el mecanismo de defensa MTI. En otra línea de defensa vegetal, las proteínas de resistencia (R) reconocen los efectores Avr del patógeno, lo que induce la respuesta de hipersensibilidad y la muerte localizada de las células del hospedero para contener la infección (ETI). Cuando las proteínas R son incapaces de reconocer al efector, se desarrolla la susceptibilidad mediada por el efector y se establece la enfermedad.
La identificación y caracterización de los efectores son actividades sumamente importantes, porque dan la pauta para comprender mejor cómo los fitopatógenos infectan a sus hospederos, causando reducciones masivas en el rendimiento de los cultivos y amenazando la seguridad alimentaria a nivel mundial. El estudio de los efectores está abriendo un camino para su aplicación en el sector agrícola, lo cual involucra la creación de nuevos métodos para controlar las poblaciones de fitopatógenos en los sistemas de manejo integrado.
Los efectores y sus características
Los efectores se definen como pequeñas moléculas, generalmente secretadas, que manipulan la estructura y función de la célula hospedera, permitiendo que el microorganismo establezca una interacción con el hospedero (Fabro, 2022; Langin et al., 2020). Estas moléculas producen cambios físicos y fisiológicos en los organismos diana (los organismos sobre los cuales actúan) y, en algunos casos, en los propios microrganismos que los producen (Cai et al., 2023; Figueroa et al., 2021; He et al., 2020). La mayoría de los efectores conocidos hoy en día son de naturaleza proteica (Carreón-Anguiano et al., 2020; Kanja y Hammond-Kosack, 2020; Sperschneider y Dodds, 2022), aunque también se han descrito metabolitos secundarios (Rangel y Bolton, 2022) y ARN pequeños (Yamankurt et al., 2020).
La mayoría de los efectores conocidos no están conservados entre diferentes organismos, por lo que los enfoques de identificación in silico (predicción) se han basado en: a) criterios estructurales relativamente amplios, principalmente la longitud del número de aminoácidos (usualmente menos de 400 aminoácidos); b) presencia de señal de secreción, que aumenta la probabilidad de la salida de la proteína de las células del fitopatógeno (Carreón-Anguiano et al., 2020; Sperschneider et al., 2018; Sperschneider y Dodds, 2022); c) el número o porcentaje del aminoácido cisteína, ya que los efectores son ricos en este aminoácido; d) y también se seleccionan candidatos basados en la ausencia de dominios transmembranales (Carreón-Anguiano et al., 2020). Existen otras características que ayudan a refinar su identificación, como es el incremento de su expresión durante la interacción del fitopatógeno con su hospedero (Tao et al., 2020; Toruño et al., 2016). Asimismo, en las secuencias de aminoácidos de los efectores proteicos se pueden identificar pequeñas regiones conservadas llamadas motivos, por ejemplo, los motivos RxLR, CHXC, o LFLAK, frecuentes en los efectores de los oomicetos (Fabro et al., 2022).
Algunos efectores están codificados por genes que se encuentran en cromosomas dispensables (llamados así porque no están presentes en todas las cepas de un microorganismo; contrario de los indispensables) o en regiones cromosómicas ricas en repeticiones y con escasa densidad de genes (Peng et al., 2019). Noar y Daub (2016) analizaron en el hongo Pseudocercospora fijiensis la distribución de los genes relacionados con la virulencia, encontrando que la mayoría se localiza en regiones genómicas “dispensables”, y mediante análisis transcriptómico se observó que estos genes se expresan durante la infección del banano (Musa acuminata). Mediante el empleo de EffHunter, un algoritmo que integra las características canónicas de los efectores (Carreón-Anguiano et al., 2020) se predijeron 136 efectores en P. fijiensis, y éstos se distribuyen tanto en las regiones genómicas dispensables como también en las regiones genómicas presentes en todas las cepas, conocido como genoma “core”.
Los genes que codifican proteínas efectoras están bajo alta presión evolutiva, lo que lleva a una tasa de mutación más elevada que en otras familias de genes. En consecuencia, es común observar polimorfismos en las secuencias de efectores entre los aislados de una especie, cambios asociados con la adaptación y virulencia sobre el hospedero (Kanja y Hammond-Kosack 2020; Padilla-Ramos et al., 2018). Con respecto al nivel de conservación, algunos efectores están presentes en microorganismos relacionados, y algunos entre organismos filogenéticamente distantes. En estos casos, las proteínas ortólogas (es decir proteínas homólogas en secuencia, que tienen la misma función en organismos diferentes y provienen de un ancestro común) presentan poca similitud de secuencia. Un claro ejemplo es el efector Avr4, ya que es un efector compartido entre especies de la misma clase de Dotidiomicetos; el Avr4 de P. fijiensis solo presenta 50.5% de identidad con su proteína ortóloga en el hongo Cladosporium fulvum (Hurlburt et al., 2018).
A pesar de que el porcentaje de identidad entre los miembros de una familia de efectores suele ser baja, los análisis ómicos realizados actualmente evidencian en los efectores la presencia de dominios y motivos proteicos conservados. Entre los dominios más frecuentemente identificados están LysM, ceratoplatanina, ARNasa, inductor de necrosis (NPP1 o NEP), CFEM, entre otros (Carreón-Anguiano et al., 2022; Outram et al., 2021; Zhao et al., 2020). Investigaciones recientes revelan que cada microorganismo puede poseer cientos de efectores (Carreón-Anguiano et al., 2020; Noar y Daub, 2016; Sperschneider et al., 2018), con diferentes funciones, que se expresan en diferentes momentos (Noar y Daub, 2016; Toruño et al., 2016). La mayoría interfiere con funciones de señalización en la planta, la síntesis de fitorreguladores o en los mecanismos de defensa (Fabro 2022; Han y Kahmann, 2019; Padilla-Ramos et al., 2018; Plett et al., 2020).
Efectores y la inmunidad vegetal (enfermedad o salud)
Las plantas tienen un sistema inmunológico innato que responde a la presencia de los fitopatógenos (Chang et al., 2022; Jones y Dangl 2006; Thordal-Christensen, 2020). Las plantas reconocen moléculas conservadas llamadas “patrones moleculares asociados a microorganismos” (MAMP, por sus siglas en inglés), y el reconocimiento desencadena la respuesta inmune primaria (defensa basal). La inmunidad activada por estos MAMP involucra la participación en la planta de receptores que reconocen esos patrones moleculares (PRR), y disparan la respuesta de inmunidad (MTI, por “MAMP-Triggered Immunity”). Entre las moléculas MAMP se encuentran la quitina y el glucano de los hongos, la flagelina bacteriana, entre otros (Alhoraibi et al., 2019; Zhou y Zhang, 2020). Esta detección ocurre en el apoplasto de las células vegetales y la planta se defiende produciendo especies reactivas de oxígeno (ROS), compuestos antimicrobianos y enzimas hidrolíticas. Los fitopatógenos responden secretando efectores, que le permiten inactivar la respuesta de inmunidad o superar los efectos de los mecanismos de defensa del hospedero. En su coevolución, las plantas han desarrollado una segunda línea de defensa, la inmunidad desencadenada por efectores (ETI, por sus siglas en ingles), que involucra la detección de los efectores de avirulencia (efectores Avr) (Figura 1B). Las proteínas que reconocen a los efectores Avr son receptores intracelulares conocidas como proteínas de resistencia (R), también llamados cognados, y juegan un papel muy importante en los programas de mejoramiento genético (Ghislain et al., 2019; Li et al., 2020; Thordal-Christensen, 2020). Todas las plantas tienen una amplia familia de genes de resistencia; por ejemplo, en el genoma de Arabidopsis thaliana se predicen más de 200 genes que codifican receptores tipo quinasa con motivos repetidos ricos en leucina (LRR-RLK), una clase de genes R (Wu et al., 2016).
La relación entre los factores de avirulencia con la proteína R, es una interacción ubicua en la naturaleza, pero fue descrita primero en la interacción entre el hongo biótrofo Melampsora lini y la planta Linum usitatissimum (Flor, 1942). En ausencia de la proteína R o cognado, o en presencia de una proteína R incapaz de reconocer al efector, no se enciende el mecanismo ETI de protección y defensa y el efector promueve la virulencia del fitopatógeno (Jones y Dangl 2006; Todd et al., 2022a). Esto conlleva a la susceptibilidad desencadenada por efectores (ETS, por sus siglas en ingles); la expresión de los genes efectores Avr típicamente alcanza su punto máximo en las primeras etapas de la infección. Cuando la proteína Avr del fitopatógeno es reconocida, se desencadena la respuesta de hipersensibilidad (RH), un fenómeno importante de la ETI. La RH es desfavorable para el fitopatógeno debido a la muerte celular localizada del hospedero, que se produce en el sitio de infección y evita la invasión, manteniendo a la planta sana. Esta interacción molecular es clave en la interacción incompatible, que involucra un hospedero resistente y un fitopatógeno cuya virulencia no es suficiente para contrarrestar la defensa vegetal. Por el contrario, la asociación del fitopatógeno con el hospedante susceptible resulta en una interacción compatible a través del ETS (He et al., 2020; Thordal-Christensen, 2020; Todd et al., 2022a).
Caracterización de los efectores
La búsqueda in silico de efectores en los genomas microbianos resulta en decenas o cientos de candidatos a efectores (Carreón-Anguiano et al., 2022; Sperschneider et al., 2018); los cuales se deben validar experimentalmente para comprobar si son efectores verdaderos y en consecuencia estudiar su función.
Nicotiana benthamiana se ha utilizado ampliamente como planta modelo para expresar a los efectores de manera transitoria y estudiar el fenotipo que resulta de dicha expresión; su éxito como planta modelo es debido a su tamaño pequeño, crecimiento rápido y facilidad de manipulación genética. El método más empleado para el análisis funcional es el de “agroinfiltración”, el cual involucra la transformación de Agrobacterium tumefaciens con un vector de expresión que contenga la región codificante completa del efector de interés; posteriormente la bacteria transformada se infiltra en hojas de N. benthamiana (Figura 2). Los efectores verdaderos son identificados porque generan un fenotipo particular como resultado de la RH. La mayoría produce lesiones características entre cloróticas y necróticas en la hoja, debido a que su presencia activa la reacción sistémica adquirida (Jones y Dangl 2006; Porras et al., 2022). En cambio, otros efectores, por ejemplo, de fitopatógenos biotróficos, se identifican por impedir la activación de la reacción sistémica adquirida y la RH (Zhang et al., 2022). El éxito de N. benthamiana o de Arabidopsis thaliana como plantas modelo para identificar efectores de los fitopatógenos que no la infectan naturalmente se ha descrito como resistencia inespecífica (NHR, “non-host resistance”). Esta forma de resistencia se refiere a la resistencia exhibida por una especie vegetal contra todas las variantes genéticas de una especie de fitopatógeno no adaptado (Wu et al., 2023).
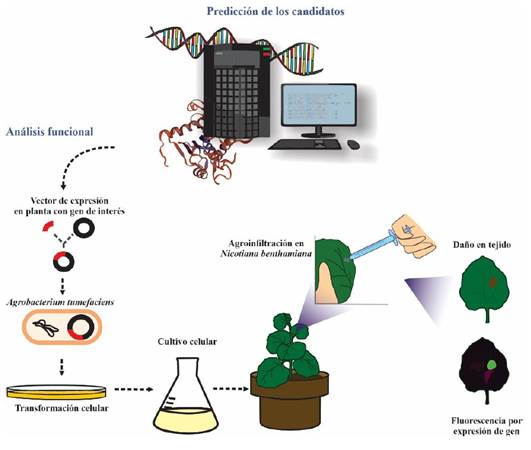
Figura 2 Flujo de investigación en la efectorómica. Los efectores son identificados primero in silico mediante bioinformática y aquellos seleccionados son evaluados mediante análisis funcional. La secuencia completa de la región codificante es clonada en Agrobacterium tumefasciens, y luego inoculada en las hojas de la planta modelo Nicotiana benthamiana. Los efectores que son reconocidos por alguno de los receptores de la planta generan lesiones en las hojas o, por el contrario, en el caso de los efectores biotróficos éstos se reconocen porque pueden suprimir la respuesta hipersensible en plantas en las que se haya clonado una proteína R determinada y se co-agroinfiltre el efector que reconoce esa proteína cognada, junto con el candidato en evaluación que se sospecha es efector asociado a un organismo biotrófico.
Actualmente, existen varias técnicas para elucidar la función de los efectores. Las técnicas más comunes interfieren con su expresión, como la generación de mutantes “knock out” en la que se elimina el gen del genoma del patógeno, el “knock down” o silenciamiento génico utilizando ARN de interferencia (ARNi) que no elimina el gen, pero interfiere pos-transcripcionalmente sobre el ARNm, impidiendo su correcta traducción, y la edición génica con CRISPR/Cas9 (Kanja y Hammond-Kosack, 2020). Al interrumpir la función del gen, se evalúa si la virulencia sobre un hospedero susceptible se pierde (o se reduce) para comprobar que el candidato es un efector verdadero, y una posible proteína de interés en la fitopatología (Kanja y Hammond-Kosack, 2020).
Otros análisis para caracterizar al efector es su clonación como quimera, fusionando la región codificante del efector con una proteína fluorescente. Esto permite definir su localización subcelular, es decir, si es apoplástica o si la proteína se dirige a los organelos intracelulares, lo que ayuda a buscar posteriormente sus proteínas diana (Camborde et al., 2022; Tang et al., 2022). Identificar y caracterizar efectores continúan siendo tareas desafiantes. A pesar de que comenzaron a estudiarse hace 80 años (Flor, 1942), en 2020 sólo se conocían las funciones de 150 efectores verdaderos fúngicos (Carreón-Anguiano et al., 2020), pero el número se ha incrementado significativamente y hoy se conocen más de 300 efectores (Nur et al., 2021; Sperschneider y Dodds, 2022).
Funciones de los efectores
Los efectores participan en todas las interacciones planta-microbio, tanto en las interacciones negativas con los fitopatógenos, como en las interacciones entre plantas y microorganismos benéficos, como las micorrizas (Plett et al., 2020; Kang et al., 2020), y recientemente fueron descubiertos en las interacciones microbio-microbio (Snelders et al., 2020; Snelders et al., 2021). Con respecto a los fitopatógenos, las funciones de los efectores reflejan su estilo de vida; por ejemplo, los fitopatógenos biotróficos requieren un hospedero vivo para completar su ciclo de infección mientras que los fitopatógenos necrotróficos requieren tejido muerto. Por otro lado, los fitopatógenos hemibiotróficos son una combinación de los dos anteriores, obteniendo su nutrición primero de tejido vivo y al final de su ciclo, de tejido muerto. Mientras que los efectores de los organismos biotróficos a menudo funcionan bloqueando la inmunidad del hospedero, los efectores asociados a necrótrofos encienden las respuestas de defensa del hospedero, pero de una manera descontrolada, intensa y no localizada, induciendo la muerte del hospedero. Los fitopatógenos hemibiotróficos inicialmente producen efectores que suprimen la respuesta inmune y la muerte celular, pero luego, en la fase necrotrófica, secretan efectores que causan la muerte de la célula hospedera (Castillo-Sanmiguel et al., 2022; Jones y Dangl 2006; Thordal-Christensen, 2020; Todd et al., 2022a) (Figura 3). Por ejemplo, en Botrytis cinerea el efector BcNEP1 se expresa fuertemente al inicio de la infección, mientras que los efectores BcSSP2 y BcNEP2 se expresan de manera tardía (Zhu et al., 2022). Similarmente, en Colletotrichum spp., género de hongos hemibiótrofos, hay efectores que participan de manera específica en la etapa de biotrofía, mientras otros facilitan la necrotrofía, induciendo la muerte celular (Ono et al., 2020; Tsushima et al., 2021).

Figura 3 Funcionalidad biológica de efectores según los hábitos tróficos de los diferentes tipos de fitopatógenos. A) en los biótrofos, los efectores bloquean la respuesta hipersensible y suprimen la respuesta de defensa vegetal. Los efectores biotróficos mantienen vivas las células del hospedero. Ejemplos de organismos biotróficos son los hongos Ustilago maydis, Blumeria graminis y Puccinia triticina. B) los necrótrofos liberan efectores que causan la muerte celular a través de necrosis, fagocitosis, etc. Ejemplos de organismos necrotróficos son los hongos Botrytis cinerea y Alternaria brassicicola, y la bacteria Rhizobium radiobacter. C) Los hemibiótrofos poseen ambos tipos de efectores. En el inicio de la interacción emplean efectores expresados en la fase biotrófica que evitan la muerte celular por RH; al final de su ciclo de vida, emplean los efectores asociados a la fase necrotrófica, que provocan la muerte de su hospedero. Ejemplos de organismos hemibiotróficos son los hongos Colletotrichum graminicola, Cladosporium fulvum, el oomiceto, Phytophthora infestans y la bacteria Pseudomonas syringae.
Los efectores ampliamente estudiados son los conocidos como apoplásticos (extracelulares); hasta la última década era parte de la definición de los efectores (Carreón-Anguiano et al., 2020) pues se creía que todos los efectores eran extracelulares; no obstante, se han descrito efectores intracelulares que actúan en el citoplasma y en los organelos y van ganando terreno en la efectorómica (Sperschneider y Dodds, 2022; Tariqjaveed et al., 2021; Todd et al., 2022b). Los efectores apoplásticos suelen ser proteínas pequeñas con actividad enzimática que degradan la pared celular, o expansinas que la relajan; otros son inhibidores de proteasas o inhibidores del reconocimiento del fitopatógeno por la planta (Fabro, 2022; He et al., 2020; Langin et al., 2020), entre otras funciones. Dentro de las células hospederas, los efectores intracelulares varían en localización y funciones biológicas; la mayoría de sus dianas en el hospedero son proteínas con funciones importantes en la inmunidad vegetal (Thordal-Christensen, 2020). Las dianas en el hospedero suelen ser proteasas, componentes del sistema ubiquitina-proteasoma, proteínas involucradas en la transcripción, receptores y las proteínas de las vías de biosíntesis y señalización de los fitorreguladores que regulan la defensa vegetal (Fabro 2022; Han y Kahmann, 2019). El Cuadro 1, presenta algunos ejemplos de efectores caracterizados. El lector puede ampliar el catálogo de efectores en las revisiones de Kanja y Hammond-Kosack, (2020), Todd et al., (2022a, 2022b) y Zhang y colaboradores (2022), entre otros.
Cuadro 1 Ejemplos de efectores caracterizados y sus funciones en el hospedero.
| Efector | Organismo | Actividad Biológica | Localización de la proteína | Rol de virulencia/Patogenicidad | Diana vegetal | Referencia |
|---|---|---|---|---|---|---|
| AVR 2 | Cladosporium fulvum | Induce RH; inhibidor de proteasa | Apoplasto | Inhibe Rcr3 y otras proteasas | Cf-2 | Ali y Bakkeren 2011y Selin et al., 2016z |
| AVR 4 | C. fulvum | Induce RH; Unión a quitina | Apoplasto;célula fúngica, pared de quitina | Protege contra quitinasas | Cf-4 | Ali y Bakkeren 2011y Selin et al., 2016z |
| AVR 9 | C. fulvum | Induce RH; Inhibidor de carboxypeptidasa | Apoplasto | Desconocido | Cf.9 | Ali y Bakkeren 2011y Selin et al., 2016z |
| BcSSP2 | Botrytis cinerea | Efector citotóxico; Induce muerte celular | Apoplasto | No esencial para la patogenicidad | Desconocido | Zhu et al., 2022 |
| PaMissP10b | Pisolithus albus | Interactua con la S-adenosylmetionina Descarboxilasa | Citoplasma | Modifica la ruta de biosintesis de poliaminas | Desconocido | Plett et al., 2020 |
| PWL 1 | Magnaporthe oryzae | Proteína hidrofilica rica en glicina | Complejo interfacial biotrófico | Desconocido | Desconocido | Ali y Bakkeren 2011y Selin et al., 2016z |
| PWL 2 | M. oryzae | Proteína hidrofilica rica en glicina | Citoplasma | Desconocido | Desconocido | Ali y Bakkeren 2011y Selin et al., 2016z |
| PWL 3 | M. oryzae | Proteína hidrofilica rica en glicina | Probablemente apoplasto | No funcional | Desconocido | Ali y Bakkeren 2011y |
| AVR 3 (SIX 1) | Fusarium oxysporum f. sp. lycopersici | Desconocido | Xilema | Requerido para la total virulencia | I-3 | Ali y Bakkeren 2011y Selin et al., 2016z |
| AVR 4 (SIX2) | F. oxysporum f. sp. lycopersici | Desconocido | Xilema | Requerido para la total virulencia | Desconocido | Ali y Bakkeren 2011y Selin et al., 2016z |
| MfAVR4 | Pseudocercospora fijiensis | Peritrofina-A con unión a quitina, inducción de RH | Probablemente apoplasto | Protección del hongo contra quitinasas | Cf-4 y Hcr9 | Ali y Bakkeren 2011y Selin et al., 2016z |
| RxLR30 | Phytophthora brassicae | Efector de la familia RXLR | Probablemente apoplasto | Inhibe la secreción de antimicrobianos mediado por vesículas | RABA GTPase | Tomczynska et al., 2018 |
| MoCDIP6 | M. oryzae | Induce Muerte Celular | No reportado | Induce necrosis en hojas | Relacionados con Patogénesis (PR): OsCHT1, OsCHT3, OsNac4, OsPR1B | Guo et al., 2019 |
| PstCEP1 | Puccinia striiformis | Induce HR / muerte celular programada | Citoplasma | Responde a altas temperaturas | Desconocido | Tao et al., 2020 |
| PTTG08198 (CFEM) | P. triticina | Aumenta muerte celular | No reportado | Promueve acumulación de ROS | Desconocido | Zhao et al., 2020 |
| BLN08 | Bremia lactucae | Efector de la familia WY | Mitocondria | Induce muerte cellular en lechuga | Desconocido | Wood et al., 2020 |
| VdAMP3 | Verticillium dahliae | Suprime la respuesta inmune / Induce necrosis y senescencia | Xilema | Manipulación del microbioma | Desconocido | Snelders et al., 2021 |
| XopL | Xanthomonas oryzae | Contrarresta la autofagia en el hospedero | Citoplasma | Se une y degrada al componente SH3P2 de la vía de autofagia | SH3P2 | Leong et al., 2022 |
| MeTCTP | Meloidogyne enterolobii | Suprime la inmunidad vegetal | Citoplasma | Se une al calico e impide su aumento en el citosol | Unión directa al calcio | Guo et al., 2022 |
| MiMSP32 | M. incognita | Interactúa con una enzima involucrada en la síntesis de jasmonato; suprime la inmunidad vegetal | Citoplasma | Promueve la susceptibilidad, contribuye a la virulencia | 12-oxofitodienoato reductasa 2 (OPR2) | Verhoeven et al., 2022 |
| Al6 | Apolygus lucorum | Suprime la inmunidad vegetal y permite que el insecto se alimente | Apoplasto, citoplasma | Usa la glutation peroxidasa para evitar acumulación de especies reactivas de oxígeno | Glutation peroxidasa | Dong et al., 2023 |
| βC1 | Virus del rizado amarillo de la hoja del tomate | Reduce la actividad de terpeno sintasa. | Núcleo | Disminuye producción de volátiles, provoca mayor atracción a la planta del insecto Bemisia tabaci y mejora el desempeño de éste | PIF y MYC2 | Ray y Casteel, 2022 |
y, z trabajo de revisión.
A través de sus efectores, los microorganismos suelen manipular en su beneficio la síntesis de los fitorreguladores jasmonato (JA), salicilato (SA) y etileno (ET) (Alhoraibi et al., 2019; Chini et al., 2018; Langin et al., 2020). Por ejemplo, el efector Cmu del hongo Erysiphe quercicola tiene actividad de corismato mutasa, enzima que inhibe la síntesis del ácido salicílico en el hospedero (He et al., 2021). El efector VdIsc1, de Verticillium dahliae tiene actividad de isocorismatasa que también interfiere en la síntesis del ácido salicílico (Zhu et al., 2017), mientras el efector RipAB de Ralstonia solanacearum interfiere con la señalización regulada por el ácido salicílico (Qi et al., 2022), Estos ejemplos destacan la importancia de inhibir la síntesis de este fitorregulador, el cual participa en la señalización y en la defensa vegetal. En la micorriza Laccaria bicolor, el efector MiSSP7 interactúa con las proteínas represoras PtJAZ5 y PtJAZ6 de la ruta de señalización del ácido jasmónico, evitando la degradación de estas proteínas represoras y en consecuencia evita la transcripción de genes de defensa regulados por el ácido jasmónico, lo que permite que se establezca el mutualismo entre la micorriza y el hospedero (Plett et al., 2014). Otros efectores afectan la fisiología del hospedero para crear un ambiente propicio para la colonización; el efector AvrE de Pseudomonas syringae regula los niveles del ácido abscísico en las células para inducir el cierre estomático y así aumentar los niveles de agua en el tejido vegetal (Hu et al., 2022).
En otras ocasiones, los microorganismos secretan efectores que promueven la síntesis o mimetizan a los fitorreguladores. Por ejemplo, el fitopatógeno necrotrófico Lasiodiplodia mediterranea produce un análogo del ácido jasmónico, el éster lasiojasmonato A (LasA). LasA se puede convertir en jasmonil-isoleucina (JA-Ile), un potente activador de la señalización del ácido jasmónico e inductor de la muerte celular, facilitando la necrotrofía de este patógeno (Chini et al., 2018).
Perspectivas de la efectorómica en la agricultura
El interés en la efectorómica y su importancia en la agrobiotecnología ha crecido de manera importante en la última década, y actualmente es un área prioritaria en las investigaciones de las interacciones fitopatógeno-hospedero. Algunos reportes han mostrado que algunos efectores pudieran en el futuro ser utilizados como bioproductos por sí mismos, para inducir respuestas de defensa vegetal. Por ejemplo, el efector MSP1 del hongo hemibiótrofo Magnaporthe oryzae se expresó de manera heteróloga en la bacteria Escherichia coli y se aplicó 0.1μM de la proteína recombinante en las hojas de plántulas de arroz, lo que potenció la respuesta de defensa y evitó su infección (Wang et al., 2016). Recientemente, de este mismo hongo, se reportaron los efectores MoCDIP6 y MoCDIP7; siguiendo un proceso similar, se observó que las plantas tratadas no mostraron síntomas de necrosis o marchitez, y comenzaron a expresar genes relacionados a la resistencia; cuando se inoculó una cepa virulenta de M. oryzae, las plantas desarrollaron menos lesiones y más pequeñas en comparación con las plantas control (Guo et al., 2019). En Fusarium oxysporum, en interacción con la planta de tabaco, el efector FocCP1 indujo la expresión de genes relacionados con la señalización por ácido salicílico. Cuando se aplicó FocCP1 en plantas de tabaco y se inoculó con el virus del mosaico del tabaco, las plantas tratadas desarrollaron menos síntomas que en el control (Li et al., 2019). Esta es una prometedora línea de investigación, ya que ofrece una perspectiva eco-amigable en comparación a los pesticidas comerciales, pero actualmente hay pocos trabajos dirigidos a explorar el aprovechamiento agrobiotecnológico de los efectores. La inmensa mayoría de los trabajos se enfocan a aspectos básicos de su estructura, función o localización celular.
En otra línea de aplicación, Vleeshouwers y colaboradores (2011) impulsaron el uso de los efectores para seleccionar germoplasma resistente para programas de mejoramiento genético. Se han empleado efectores de Phytophthora infestans para seleccionar plantas de papa, dónde se identificaron un conjunto de genes de resistencia que fueron útiles para desarrollar variedades mejoradas. Hoy en día, introducir genes de resistencia en germoplasma susceptible, es una de las aplicaciones más promisorias en programas de mejoramiento genético (Chen et al., 2022; Ji et al., 2022; Ochola et al., 2020). También se han usado proteínas efectoras recombinantes para la identificación de plantas susceptibles. Se hipotetiza que, al mutar los genes de susceptibilidad, las plantas tendrán resistencia más duradera en comparación con la resistencia mediada por genes de resistencia (R) (Campos et al., 2021; Garcia-Ruiz et al., 2021; Koseoglou et al., 2022; Ribeiro et al., 2022).
Debido a la falta de conservación de los efectores, el desarrollo de la efectorómica ha sido lenta y difícil; sin embargo, con los avances en análisis masivos, actualmente la efectorómica está en pleno desarrollo y tiene mucho que ofrecer a la agricultura (Li et al., 2021; Van de Wouw y Idnurm, 2019). En consecuencia, es necesario contar con métodos robustos de predicción, así como protocolos de caracterización masiva de efectores, que permitan identificar efectores con funciones indispensables en la infección y que sean capaces de conferir protección a la planta al menos contra el fitopatógeno que lo produce, o preferentemente protegerla contra múltiples fitopatógenos a la vez.
Panaroma actual de la efectorómica en México
La efectorómica es un campo de desarrollo incipiente en México. Los primeros trabajos en los que participaron investigadores mexicanos se enfocaron a la identificación de efectores de los oomicetos P. infestans durante la infección de jitomate y de Phytophthora capsici en interacción de tipo no-hospedera en Nicotiana spp. (Zuluaga et al., 2015; Vega-Arreguín et al., 2017). En estos trabajos identificaron diversidad de efectores, entre ellos, encontraron a los efectores IpiO y SNE1 durante la fase biotrófica, y a PiNPP1.1 durante la fase necrotrófica. Las familias de efectores RXLR, CRN, y NPP, comunes en oomicetos, fueron identificadas también (Zuluaga et al., 2015). Por su parte, las especies de Nicotiana mostraron resistencia contra P. capsici. El análisis identificó que la resistencia es mediada por el gen I2R que reconoce en el fitopatógeno al efector proteico PcAvr3a1 (Vega-Arreguín et al., 2017).
En hongos fitopatógenos, se está abordando la identificación de efectores en P. fijiensis, el hongo causante de la Sigatoka negra en bananos y plátanos. En ese primer análisis se identificaron 136 efectores canónicos, es decir, que cumplen con todas las características clásicas de los efectores (secretados, pequeño tamaño, alto contenido de cisteínas) (Carreón-Anguiano et al., 2020). Con el objetivo de contribuir a la efectorómica fúngica en el mundo, Carreón-Anguiano et al. (2022) crearon un algoritmo, WideEffHunter, que es capaz de identificar efectores no-canónicos, encontrando que los efectores canónicos comprenden aproximadamente 10% de los efectoromas en hongos y en oomicetos. Se espera que la identificación de efectoromas globales permita identificar nuevas familias de efectores, mayor número de efectores que en diferentes organismos comparten homología, y nuevos motivos y dominios en los efectores proteicos (Carreón- Anguiano et al., 2022; Todd et al., 2022b).
Otros trabajos mexicanos se han enfocado en organismos no fitopatógenos. Guzmán-Guzmán et al. (2017) identificaron bioinformáticamente 233 efectores al unir los proteomas de Trichoderma virens, T. atroviride y T. reesei, donde seleccionaron 16 efectores de T. virens y T. atroviride y los caracterizaron, encontrando que unos se expresan cuando estos hongos colonizan A. thaliana, mientras otros se expresan cuando se enfrentan al hongo fitopatógeno Rhizoctonia solani. Entre los efectores de Trichoderma se han encontrado hidrolasas, hidrofobinas, cerato-plataninas, y efectores con dominios CFEM, o dominios LysM (Ramírez-Valdespino et al., 2019; Romero-Contreras et al., 2019). Interesantemente, una hidrofobina de clase II, tvhydii1, se sobre-expresa en T. virens en presencia del fitopatógeno R. solani; las mutantes que pierden la función de tvhydii1 pierden parte de su capacidad de colonizar las raíces de las plantas, mientras que su sobre-expresión la incrementa (Guzmán et al., 2017).
Recientemente, Báez-Astorga et al. (2022) reportaron el mecanismo de acción del agente de biocontrol Bacillus cereus, que es capaz de inhibir in vitro a Fusarium verticillioides, un fitopatógeno que causa pudrición de la mazorca y las raíces del maíz. F. verticillioides secreta el efector Fv-cmp con actividad de proteasa que digiere las quitinasas tipo A y B de la planta. Por su parte, B. cereus secreta los efectores ChiAy ChiB con actividad quitinasa que in vitro actúan sobre los conidios de F. verticillioides e impiden que germinen y se desarrollen en hifas, particularmente ChiB que posee el dominio CBM 2, el cual le permite unirse con más fuerza a la pared del hongo y tener mayor actividad que ChiA. Aunque aún son muy pocos los trabajos desarrollados en México en el área de la efectorómica, estas investigaciones muestran resultados prometedores en cuanto a aplicaciones biotecnológicas. Es deseable que en México se impulse también el estudio de los efectores en las bacterias fitopatógenas, e insectos y nematodos plagas. Cabe mencionar que el interés en estas áreas ha crecido en los últimos años a nivel mundial, pero el número de grupos de investigación aún es limitado por lo que representa un nicho de oportunidad para contribuir con investigaciones mexicanas.
Conclusiones
Los efectores son sumamente importantes para el establecimiento de interacciones biológicas; dentro del rango de interacciones en que se encuentran, la interacción planta-patógeno es la más estudiada. El primer acercamiento hacia los efectores fue en la interacción L. usitatissimum- M. lini en los años 40 y aunque en la actualidad se tiene grandes avances en la efectorómica, el conocimiento es aún limitado. En consecuencia, es necesario agilizar la priorización de efectores para su caracterización pues se identifican cientos de ellos para cada organismo durante el análisis in silico.
La identificación y caracterización de los efectores indispensables para la virulencia de los fitopatógenos podría ser clave para el desarrollo de nuevos métodos de manejo de enfermedades en la agricultura, basado en los efectores. La identificación de las proteínas dianas en el hospedero se encuentra rezagada; dentro de estas dianas hay posibles proteínas de resistencia cuyos genes pueden ser utilizados para la protección vegetal. Indudablemente, los efectores de los microrganismos representan nichos de oportunidad que se requieren comprender para poder aprovecharlos para el bien de la población y la seguridad alimentaria mundial.
Agradecimientos
Este trabajo fue financiado por el Consejo Nacional de Ciencia y Tecnología-México (CONACyT-México), con el Proyecto FOP16-2021-01 No. 320993, y las becas No. 291236 de JNA Todd, No. 644399 de OJ Couoh-Dzul y No. 700673 de KGCA.
REFERENCIAS
Alhoraibi H, Bigeard J, Rayapuram N, Colcombet J and Hirt H. 2019. Plant Immunity: The MTI-ETI Model and Beyond. Current issues in molecular biology 30: 39-58. https://doi.org/10.21775/cimb.030.039 [ Links ]
Ali, S. and Bakkeren, G. 2011. Fungal and oomycete effectors - strategies to subdue a host. Canadian Journal of Plant Pathology 33(4): 425-446. https://doi.org/10.1080/07060661.2011.625448 [ Links ]
Almeida F, Rodrigues ML and Coelho C. 2019. The Still Underestimated Problem of Fungal Diseases Worldwide. Frontiers in Microbiology 10: 214. https://doi.org/10.3389/fmicb.2019.00214 [ Links ]
Báez-Astorga PA, Cázares-Álvarez JE, Cruz-Mendívil A, Quiroz-Figueroa FR, Sánchez-ValleVI and Maldonado-Mendoza IM. 2022. Molecular and biochemical characterisation of antagonistic mechanisms of the biocontrol agent Bacillus cereus B25 inhibiting the growth of the phytopathogen Fusarium verticillioides P03 during their direct interaction in vitro, Biocontrol Science and Technology 32(9): 1074-1094. https://doi.org/10.1080/09583157.2022.2085662 [ Links ]
Baker BP, Green TA and Loker AJ. 2020. Biological control and integrated pest management in organic and conventional systems. Biological Control 140: 104095. https://doi.org/10.1016/j.biocontrol.2019.104095 [ Links ]
Cai J. Jiang Y, Ritchie ES, Macho AP, Yu F and Wu D. 2023. Manipulation of plant metabolism by pathogen effectors: More than just food. FEMS Microbiology Reviews fuad007. https://doi.org/10.1093/femsre/fuad007 [ Links ]
Camborde L, Kiselev A, Pel MJC, Le Ru A, Jauneau A, Pouzet C, Dumas B and Gaulin E. 2022. An oomycete effector targets a plant RNA helicase involved in root development and defense. New Phytologist 233(5): 2232-2248. https://doi.org/10.1111/nph.17918 [ Links ]
Campos MD, Félix MDR, Patanita M, Materatski P and Varanda C. 2021. High throughput sequencing unravels tomato-pathogen interactions towards a sustainable plant breeding. Horticulture Research 8(1): 171. https://doi.org/10.1038/s41438-021-00607-x [ Links ]
Carreón-Anguiano KG, Todd JNA, Chi-Manzanero BH, Couoh-Dzul OJ, Islas-Flores I, and Canto-Canché B. 2022. WideEffHunter: An Algorithm to Predict Canonical and Non-Canonical Effectors in Fungi and Oomycetes. International journal of molecular sciences 23(21): 13567. https://doi.org/10.3390/ijms232113567 [ Links ]
Carreón-Anguiano KG, Islas-Flores I, Vega-Arreguín J, Sáenz-Carbonell L and Canto-Canché B. 2020. EffHunter: A Tool for Prediction of Effector Protein Candidates in Fungal Proteomic Databases. Biomolecules 10(5): 712. https://doi.org/10.3390/biom10050712 [ Links ]
Castillo-Sanmiguel PA, Cortés-Sánchez LR and Acero-Godoy J. 2022. Molecular aspects of tomato (Solanum lycopersicum) vascular wilt by Fusarium oxysporum f. sp. lycopersici and antagonism by Trichoderma spp. Mexican Journal of Phytopathology 40(1): 82-102. https://doi.org/10.18781/R.MEX.FIT.2106-1 [ Links ]
Chang M, Chen H, Liu F and Fu ZQ. 2022. PTI and ETI: Convergent pathways with diverse elicitors. Trends in Plant Science 27(2): 113-115. https://doi.org/10.1016/j.tplants.2021.11.013 [ Links ]
Chen CY, Liu YQ, Song WM, Chen DY, Chen FY, Chen XY, Chen ZW, Ge SX, Wang CZ, Zhan S, Chen XY and Mao YB. 2019. An effector from cotton bollworm oral secretion impairs host plant defense signaling. Proceedings of National Academy of Sciences of the United States of America 116(28): 14331-14338. https://doi.org/10.1073/pnas.1905471116 [ Links ]
Chen L, Wang H, Yang J, Yang X, Zhang M, Zhao Z, Fan Y, Wang C and Wang J. 2021. Bioinformatics and Transcriptome Analysis of CFEM Proteins in Fusarium graminearum. Journal of Fungi 7(10): 871. https://doi.org/10.3390/jof7100871 [ Links ]
Chini, A., Cimmino, A., Masi, M., Reveglia, P., Nocera, P., Solano, R., & Evidente, A. (2018). The fungal phytotoxin lasiojasmonate A activates the plant jasmonic acid pathway. Journal of experimental botany 69(12): 3095-3102. https://doi.org/10.1093/jxb/ery114 [ Links ]
Dong Y, Zhang W, Jin Y, Shen D and Xia A. 2023. Apolygus lucorum effector Al6 promotes insect feeding performance on soybean plants: RNAi analysis and feeding behaviour study with electrical penetration graph. Insect molecular biology,32(1): 1-10. https://doi.org/10.1111/imb.12808 [ Links ]
Fabro G. 2022. Oomycete intracellular effectors: specialised weapons targeting strategic plant processes. The New Phytologist 233(3): 1074-1082. https://doi.org/10.1111/nph.17828 [ Links ]
FAO, 2017. The future of food and agriculture - trends and challenges. Recuperado en 12 de marzo de 2023, de http://www.fao.org/3/a-i6583e.pdf [ Links ]
Figueroa M, Ortiz D and Henningsen EC. 2021. Tactics of host manipulation by intracellular effectors from plant pathogenic fungi. Current Opinion in Plant Biology 62: 102054. https://doi.org/10.1016/j.pbi.2021.102054 [ Links ]
Flor HH. 1942. Inheritance of pathogenicity in Melampsora lini. Phytopathology 32: 653-669 [ Links ]
Garcia-Ruiz H, Szurek B and Van den Ackerveken G. 2021. Stop helping pathogens: engineering plant susceptibility genes for durable resistance. Current Opinion in Biotechnology 70: 187-195. https://doi.org/10.1016/j.copbio.2021.05.005 [ Links ]
Ghislain M, Byarugaba AA, Magembe E, Njoroge A, Rivera C, Román ML, Tovar JC, Gamboa S, Forbes GA, Kreuze JF, Barekye A and Kiggundu A. 2019. Stacking three late blight resistance genes from wild species directly into African highland potato varieties confers complete field resistance to local blight races. Plant Biotechnology Journal 17(6): 1119-1129. https://doi.org/10.1111/pbi.13042 [ Links ]
Guo B, Lin B, Huang Q, Li Z, Zhuo K and Liao J. 2022. A nematode effector inhibits plant immunity by preventing cytosolic free Ca2+ rise. Plant Cell Environment 45 (10): 3070-3085. https://doi.org/10.1111/pce.14406 [ Links ]
Guo X, Zhong D, Xie W, He Y, Zheng Y, Lin Y, Chen Z, Han Y, Tian D, Liu W, Wang F, Wang Z and Chen S. 2019. Functional identification of novel cell death-inducing effector proteins from Magnaporthe oryzae. Rice 12(1): 59. https://doi.org/10.1186/s12284-019-0312-z [ Links ]
Gupta A, Singh UB, Sahu PK, Paul S, Kumar A, Malviya D, Singh S, Kuppusamy P, Singh P, Paul D, Rai JP, Singh HV, Manna MC, Crusberg TC, Kumar A and Saxena AK. 2022. Linking Soil Microbial Diversity to Modern Agriculture Practices: A Review. International Journal of Environmental Research and Public Health 19(5): 3141. https://doi.org/10.3390/ijerph19053141 [ Links ]
Guzmán-Guzmán P, Porras-Troncoso MD, Olmedo-Monfil V and Herrera-Estrella A. 2019. Trichoderma Species: Versatile Plant Symbionts. Phytopathology 109(1): 6-16. https://doi.org/10.1094/PHYTO-07-18-0218-RVW [ Links ]
Han X and Kahmann R. 2019. Manipulation of phytohormone pathways by effectors of filamentous plant pathogens. Frontiers in Plant Science 10: 822. https://doi.org/10.3389/fpls.2019.00822 [ Links ]
He Q, Liu Y, Liang P, Liao X, Li X, Li X, Shi D, Liu W, Lin C, Zheng F and Miao W. 2021. A novel chorismate mutase from Erysiphe quercicola performs dual functions of synthesizing amino acids and inhibiting plant salicylic acid synthesis. Microbiological Research 242: 126599. https://doi.org/10.1016/j.micres.2020.126599 [ Links ]
He Q, McLellan H, Boevink PC and Birch P. 2020. All roads lead to susceptibility: the many modes of action of fungal and oomycete intracellular effectors. Plant communications 1(4): 100050. https://doi.org/10.1016/j.xplc.2020.100050 [ Links ]
Hu Y, Ding Y, Cai B, Qin X, Wu J, Yuan M, Wan S, Zhao Y and Xin XF. 2022. Bacterial effectors manipulate plant abscisic acid signaling for creation of an aqueous apoplast. Cell Host & Microbe 30(4): 518-529.e6. https://doi.org/10.1016/j.chom.2022.02.002 [ Links ]
Huang C. 2021. From Player to Pawn: Viral Avirulence Factors Involved in Plant Immunity. Viruses 13(4): 688. https://doi.org/10.3390/v13040688 [ Links ]
Huang Z, Li H, Zhou Y, Bao Y, Duan Z, Wang C, Powell CA., Chen B, Zhang M and Yao W. 2022. Predication of the Effector Proteins Secreted by Fusarium sacchari Using Genomic Analysis and Heterogenous Expression. Journal of Fungi 8(1): 59. https://doi.org/10.3390/jof8010059 [ Links ]
Hurlburt NK, Chen LH, Stergiopoulos I and Fisher AJ. 2018. Structure of the Cladosporium fulvum Avr4 effector in complex with (GlcNAc)6 reveals the ligand-binding mechanism and uncouples its intrinsic function from recognition by the Cf-4 resistance protein. PLoS Pathogens 14(8):e1007263. https://doi.org/10.1371/journal.ppat.1007263 [ Links ]
Ji Z, Guo W, Chen X, Wang C and Zhao K. 2022. Plant executor genes. International Journal of Molecular Sciences 23(3):1524. https://doi.org/10.3390/ijms23031524 [ Links ]
Jones JD and Dangl JL. 2006. The plant immune system. Nature 444(7117):323-329. https://doi.org/10.1038/nature05286 [ Links ]
Kang H, Chen X, Kemppainen M, Pardo AG, Veneault-Fourrey C, Kohler A and Martin FM. (2020). The small secreted effector protein MiSSP7.6 of Laccaria bicolor is required for the establishment of ectomycorrhizal symbiosis. Environmental Microbiology 22(4): 1435-1446. https://doi.org/10.1111/1462-2920.14959 [ Links ]
Kanja C and Hammond-Kosack KE. 2020. Proteinaceous effector discovery and characterization in filamentous plant pathogens. Molecular Plant Pathology 21(10):1353-1376. https://doi.org/10.1111/mpp.12980 [ Links ]
Koseoglou E, van der Wolf JM, Visser RGF and Bai Y. 2022. Susceptibility reversed: modified plant susceptibility genes for resistance to bacteria. Trends in Plant Science 27(1):69-79. https://doi.org/10.1016/j.tplants.2021.07.018 [ Links ]
Langin G, Gouguet P and Üstün S. 2020. Microbial effector proteins - a journey through the proteolytic landscape. Trends in Microbiology 28(7): 523-535. https://doi.org/10.1016/j.tim.2020.02.010 [ Links ]
Leong JX, Raffeiner M, Spinti D, Langin G, Franz-Wachtel M, Guzman AR, Kim JG, Pandey P, Minina AE, Macek B, Hafrén A, Bozkurt TO, Mudgett MB, Börnke F, Hofius D and Üstün S. 2022. A bacterial effector counteracts host autophagy by promoting degradation of an autophagy component. The EMBO Journal 41(13):e110352. https://doi.org/10.15252/embj.2021110352 [ Links ]
Li Q, Wang B, Yu J and Dou D. 2021. Pathogen-informed breeding for crop disease resistance. Journal of Integrative Plant Biology, 63(2): 305-311. https://doi.org/10.1111/jipb.13029 [ Links ]
Li S, Dong Y, Li L, Zhang Y, Yang X, Zeng H, Shi M, Pei X, Qiu D and Yuan Q. 2019. The Novel Cerato-Platanin-Like Protein FocCP1 from Fusarium oxysporum Triggers an Immune Response in Plants. International Journal of Molecular Sciences 20(11):2849. https://doi.org/10.3390/ijms20112849 [ Links ]
Li W, Deng Y, Ning Y, He Z and Wang GL. 2020. Exploiting Broad-Spectrum Disease Resistance in Crops: From Molecular Dissection to Breeding. Annual Review of Plant Biology, 71(1): 575-603. https://doi.org/10.1146/annurev-arplant-010720-022215 [ Links ]
Nazarov PA, Baleev DN, Ivanova MI, Sokolova LM and Karakozova MV. (2020). Infectious Plant Diseases: Etiology, Current Status, Problems and Prospects in Plant Protection. Acta Naturae 12(3): 46-59. https://doi.org/10.32607/actanaturae.11026 [ Links ]
Noar RD and Daub ME. 2016. Transcriptome sequencing of Mycosphaerella fijiensis during association with Musa acuminata reveals candidate pathogenicity genes. BMC Genomics 17(1):690. https://doi.org/10.1186/s12864-016-3031-5 [ Links ]
Nur M, Wood K and Michelmore R. 2021. EffectorO: motif-independent prediction of effectors in oomycete genomes using machine learning and lineage specificity. Preprint. bioRxiv. https://doi.org/10.1101/2021.03.19.436227 [ Links ]
Ochola S, Huang J, Ali H, Shu H, Shen D, Qiu M, Wang L, Li X, Chen H, Kange A, Qutob D and Dong S. 2020. Editing of an effector gene promoter sequence impacts plant-Phytophthora interaction. Journal Integrative Plant Biology 62(3):378-392. https://doi.org/10.1111/jipb.12883 [ Links ]
Ono E, Mise K and Takano Y. 2020. RLP23 is required for Arabidopsis immunity against the grey mould pathogen Botrytis cinerea. Scientific Reports 10(1):13798. https://doi.org/10.1038/s41598-020-70485-1 [ Links ]
Outram MA, Solomon PS and Williams SJ. 2021. Pro-domain processing of fungal effector proteins from plant pathogens. PLoS Pathogen 17(10):e1010000. https://doi.org/10.1371/journal.ppat.1010000 [ Links ]
Padilla-Ramos R, Salas-Muñoz S, Velásquez-Valle R and Reveles-Torres LR. 2018. A novel molecular approach in the study of parasite-host interaction. Revista Mexicana de Fitopatología 37(1): 95-114. https://doi.org/10.18781/R.MEX.FIT.1808-6 [ Links ]
Peng Z, Oliveira-Garcia E, Lin G, Hu Y, Dalby M, Migeon P, Tang H, Farman M, Cook D, White FF, Valent B and Liu S. 2019. Effector gene reshuffling involves dispensable mini-chromosomes in the wheat blast fungus. PLoS Genetics 15(9):e1008272. https://doi.org/10.1371/journal.pgen.1008272 [ Links ]
Plett JM, Plett KL, Wong-Bajracharya J, de Freitas Pereira M, Costa MD, Kohler A, Martin F and Anderson IC. 2020. Mycorrhizal effector PaMiSSP10b alters polyamine biosynthesis in Eucalyptus root cells and promotes root colonization. New Phytologist 228(2):712-727. https://doi.org/10.1111/nph.16759 [ Links ]
Plett JM, Daguerre Y, Wittulsky S, Vayssières A, Deveau A, Melton S J, Kohler A, Morrell-Falvey J L, Brun A, Veneault-Fourrey C and Martin F. 2014. Effector MiSSP7 of the mutualistic fungus Laccaria bicolor stabilizes the Populus JAZ6 protein and represses jasmonic acid (JA) responsive genes. Proceedings of the National Academy of Sciences of the United States of America 111(22): 8299-8304. https://doi.org/10.1073/pnas.1322671111 [ Links ]
Porras R, Miguel-Rojas C, Pérez-de-Luque A and Sillero JC. 2022. Macro- and Microscopic Characterization of Components of Resistance against Puccinia striiformis f. sp. tritici in a Collection of Spanish Bread Wheat Cultivars. Agronomy 12(5):1239. https://doi.org/10.3390/agronomy12051239 [ Links ]
Qi P, Huang M, Hu X, Zhang Y, Wang Y, Li P, Chen S, Zhang D, Cao S, Zhu W, Xie J, Cheng J, Fu Y, Jiang D, Yu X and Li B. 2022. A Ralstonia solanacearum effector targets TGA transcription factors to subvert salicylic acid signaling. The Plant Cell 34(5): 1666-1683. https://doi.org/10.1093/plcell/koac015 [ Links ]
Ramírez-Valdespino CA, Casas-Flores S and Olmedo-Monfil V. 2019. Trichoderma as a Model to Study Effector-Like Molecules. Frontiers in Microbiology 10:1030. https://doi.org/10.3389/fmicb.2019.01030 [ Links ]
Rangel LI and Bolton MD. 2022. The unsung roles of microbial secondary metabolite effectors in the plant disease cacophony. Current Opinion in Plant Biology 68:102233. https://doi.org/10.1016/j.pbi.2022.102233 [ Links ]
Rani L, Thapa K, Kanojia N, Sharma N, Singh S , Grewal AS, Srivastav A Land Kaushal J. 2021. An extensive review on the consequences of chemical pesticides on human health and environment. Journal of Cleaner Production 283: 124657. https://doi.org/10.1016/j.jclepro.2020.124657 [ Links ]
Romero-Contreras YJ, Ramírez-Valdespino CA, Guzmán-Guzmán P, Macías-Segoviano JI, Villagómez-Castro JC and Olmedo-Monfil V. (2019). Tal6 From Trichoderma atroviride Is a LysM Effector Involved in Mycoparasitism and Plant Association. Frontiers in Microbiology 10: 2231. https://doi.org/10.3389/fmicb.2019.02231 [ Links ]
Ray S and Casteel CL. 2022. Effector-mediated plant-virus-vector interactions. Plant Cell 34(5):1514-1531. https://doi.org/10.1093/plcell/koac058 [ Links ]
Ribeiro JA, Albuquerque A Materatski P, Patanita M, Varanda CMR, Félix MDR and Campos MD. 2022. Tomato response to Fusarium spp. infection under field conditions: study of potential genes involved. Horticulturae 8: 433. https://doi.org/10.3390/horticulturae8050433 [ Links ]
Ristaino JB, Anderson PK, Bebber DP, Brauman KA, Cunniffe NJ, Fedoroff NV, Finegold C, Garrett KA, Gilligan CA, Jones CM, Martin MD, MacDonald GK, Neenan P, Records A, Schmale D G, Tateosian L and Wei Q. 2021. The persistent threat of emerging plant disease pandemics to global food security. Proceedings of the National Academy of Sciences of the United States of America 118(23): e2022239118. https://doi.org/10.1073/pnas.2022239118 [ Links ]
Rocafort M, Fudal I and Mesarich C H. 2020. Apoplastic effector proteins of plant-associated fungi and oomycetes. Current Opinion in Plant Biology 56: 9-19. https://doi.org/10.1016/j.pbi.2020.02.004 [ Links ]
Rufián JS, Rueda-Blanco J, López-Márquez D, Macho AP, Beuzón CR and Ruiz-Albert J. 2021. The bacterial effector HopZ1a acetylates MKK7 to suppress plant immunity. New Phytologist 231(3):1138-1156. https://doi.org/10.1111/nph.17442 [ Links ]
Savary S, Willocquet L, Pethybridge SJ, Esker P, McRoberts N and Nelson A. 2019. The global burden of pathogens and pests on major food crops. Nature Ecology & Evolution 3(3): 430-439. https://doi.org/10.1038/s41559-018-0793-y [ Links ]
Schreiber KJ, Chau-Ly IJ and Lewis JD. 2021. What the Wild Things Do: Mechanisms of Plant Host Manipulation by Bacterial Type III-Secreted Effector Proteins. Microorganisms 9(5): 1029. https://doi.org/10.3390/microorganisms9051029 [ Links ]
Selin C, de Kievit TR, Belmonte MF and Fernando WG. 2016. Elucidating the role of effectors in plant-fungal interactions: progress and challenges. Frontiers in Microbiology 7:600. https://doi.org/10.3389/fmicb.2016.0060Snelders NC, Petti GC, van den Berg GCM, Seidl MF and Thomma BPHJ. 2021. An ancient antimicrobial protein co-opted by a fungal plant pathogen for in planta mycobiome manipulation. Proceedings of the National Academy of Sciences of the United States of America 118(49):e2110968118. https://doi.org/10.1073/pnas.2110968118 [ Links ]
Snelders NC, Rovenich H, Petti GC, Rocafort M, van den Berg GCM, Vorholt JA, Mesters JR, Seidl MF, Nijland R and Thomma BPHJ. 2020. Microbiome manipulation by a soil-borne fungal plant pathogen using effector proteins. Natural Plants 6(11):1365-1374. https://doi.org/10.1038/s41477-020-00799-5 [ Links ]
Snelders NC, Rovenich H and Thomma BPHJ. 2022. Microbiota manipulation through the secretion of effector proteins is fundamental to the wealth of lifestyles in the fungal kingdom. FEMS Microbiology Reviews 46(5): fuac022. https://doi.org/10.1093/femsre/fuac022 [ Links ]
Sperschneider J, Dodds PN, Gardiner DM, Singh KB and Taylor JM. 2018. Improved prediction of fungal effector proteins from secretomes with EffectorP 2.0. Molecular Plant Pathology 19(9):2094-2110. https://doi.org/10.1111/mpp.12682 [ Links ]
Sperschneider J and Dodds PN. 2022. EffectorP 3.0: prediction of apoplastic and cytoplasmic effectors in fungi and oomycetes. Molecular plant-microbe interactions 35(2): 146-156. https://doi.org/10.1094/MPMI-08-21-0201-R [ Links ]
Tang Z, Bernards MA and Wang A. 2022. Simultaneous determination and subcellular localization of protein-protein interactions in plant cells using bimolecular fluorescence complementation assay. Methods in Molecular Biology 2400:75-85. https://doi.org/10.1007/978-1-0716-1835-6_8 [ Links ]
Tao F, Hu Y, Su C, Li J, Guo L, Xu X, Chen X, Shang H and Hu X. 2020. Revealing differentially expressed genes and identifying effector proteins of Puccinia striiformis f. sp. tritici in response to high-temperature seedling plant resistance of wheat based on transcriptome sequencing. mSphere. 5(3):e00096-20. https://doi.org/10.1128/mSphere.00096-20 [ Links ]
Tariqjaveed M, Mateen A, Wang S, Qiu S, Zheng X, Zhang J, Bhadauria V and Sun W. 2021. Versatile effectors of phytopathogenic fungi target host immunity. Journal of Integrative Plant Biology, 63(11): 1856-1873. https://doi.org/10.1111/jipb.13162 [ Links ]
Thakur N, Kaur S, Tomar P, Thakur S and Yadav AN. 2020. Chapter 15 - Microbial biopesticides: Current status and advancement for sustainable agriculture and environment. In A. A. Rastegari, A. N. Yadav, & N. Yadav (Eds.), New and Future Developments in Microbial Biotechnology and Bioengineering (pp. 243-282). Elsevier. https://doi.org/10.1016/B978-0-12-820526-6.00016-6 [ Links ]
Thordal-Christensen H. A 2020. Holistic view on plant effector-triggered immunity presented as an iceberg model. Cell Molecular Life Science 77(20): 3963-3976. https://doi.org/10.1007/s00018-020-03515-w [ Links ]
Todd JNA, Carreón-Anguiano KG, Islas-Flores I and Canto-Canché B. (2022a). Microbial Effectors: Key Determinants in Plant Health and Disease. Microorganisms 10(10): 1980. https://doi.org/10.3390/microorganisms10101980 [ Links ]
Todd JNA, Carreón-Anguiano KG, Islas-Flores I and Canto-Canché B. (2022b). Fungal Effectoromics: A World in Constant Evolution. International journal of molecular sciences 23(21): 13433. https://doi.org/10.3390/ijms232113433 [ Links ]
Tomczynska I, Stumpe M and Mauch F. 2018. A conserved RxLR effector interacts with host RABA-type GTPases to inhibit vesicle-mediated secretion of antimicrobial proteins. The Plant journal: for cell and molecular biology 95(2):187-203. https://doi.org/10.1111/tpj.13928 [ Links ]
Toruño TY, Stergiopoulos I and Coaker G. 2016. Plant-pathogen effectors: cellular probes interfering with plant defenses in spatial and temporal manners. Annual Review in Phytopathology 54: 419-41. https://doi.org/10.1146/annurev-phyto-080615-100204 [ Links ]
Tsushima A, Narusaka M, Gan P, Kumakura N, Hiroyama R, Kato N, Takahashi S, Takano Y, Narusaka Y and Shirasu K. 2021. The Conserved Colletotrichum spp. Effector Candidate CEC3 Induces Nuclear Expansion and Cell Death in Plants. Frontiers in Microbiology, 12: 682155. https://doi.org/10.3389/fmicb.2021.682155 [ Links ]
van der Sluijs JP. 2020. Insect decline, an emerging global environmental risk. Current Opinion in Environmental Sustainability, 46: 39-42. https://doi.org/10.1016/j.cosust.2020.08.012 [ Links ]
Van de Wouw AP and Idnurm A. 2019. Biotechnological potential of engineering pathogen effector proteins for use in plant disease management. Biotechnology Advances, 37(6): 107387. https://doi.org/10.1016/j.biotechadv.2019.04.009 [ Links ]
Vega-Arreguín JC, Shimada-Beltrán H, Sevillano-Serrano J and Moffett P. 2017. Non-host Plant Resistance against Phytophthora capsici Is Mediated in Part by Members of the I2 R Gene Family in Nicotiana spp. Frontiers in plant science 8: 205. https://doi.org/10.3389/fpls.2017.00205 [ Links ]
Velásquez AC, Castroverde CDM and He SY. 2018. Plant-Pathogen Warfare under Changing Climate Conditions. Current Biology 28(10): R619-R634. https://doi.org/10.1016/j.cub.2018.03.054 [ Links ]
Verhoeven A, Finkers-Tomczak A, Prins P, Valkenburg-van Raaij DR, van Schaik CC, Overmars H, van Steenbrugge JJM, Tacken W, Varossieau K, Slootweg EJ, Kappers IF, Quentin M, Goverse A, Sterken MG and Smant G. 2023. The root-knot nematode effector MiMSP32 targets host 12-oxophytodienoate reductase 2 to regulate plant susceptibility. New Phytologist 237(6), 2360-2374. https://doi.org/10.1111/nph.18653 [ Links ]
Vieira P and Gleason C. 2019. Plant-parasitic nematode effectors - insights into their diversity and new tools for their identification. Current Opinion in Plant Biology 50:37-43. https://doi.org/10.1016/j.pbi.2019.02.007 [ Links ]
Vleeshouwers VG, Raffaele S, Vossen JH, Champouret N, Oliva R, Segretin ME, Rietman H, Cano LM, Lokossou A, Kessel G, Pel MA and Kamoun S. 2011. Understanding and exploiting late blight resistance in the age of effectors. Annual Review in Phytopathology 49:507-531. https://doi.org/10.1146/annurev-phyto-072910-095326 [ Links ]
Wang Y, Wu J, Kim SG, Tsuda K, Gupta R, Park SY, Kim ST and Kang KY. 2016. Magnaporthe oryzae-Secreted Protein MSP1 Induces Cell Death and Elicits Defense Responses in Rice. Molecular Plant-Microbe Interactions 29(4), 299-312. https://doi.org/10.1094/MPMI-12-15-0266-R [ Links ]
Wood KJ, Nur M, Gil J, Fletcher K, Lakeman K, Gann D, Gothberg A, Khuu T, Kopetzky J, Naqvi S, Pandya A, Zhang C, Maisonneuve B, Pel M and Michelmore R. 2020. Effector prediction and characterization in the oomycete pathogen Bremia lactucae reveal host-recognized WY domain proteins that lack the canonical RXLR motif. PLoS Pathogens 16(10):e1009012. https://doi.org/10.1371/journal.ppat.1009012 [ Links ]
Wu Y, Sexton W, Yang B and Xiao S. 2023. Genetic approaches to dissect plant nonhost resistance mechanisms. Molecular Plant Pathology 24(3), 272-283. https://doi.org/10.1111/mpp.13290 [ Links ]
Wu Y, Xun Q, Guo Y, Zhang J, Cheng K, Shi T, He K, Hou S, Gou X and Li J. 2016. Genome-Wide Expression Pattern Analyses of the Arabidopsis Leucine-Rich Repeat Receptor-Like Kinases. Molecular Plant 9(2): 289-300. https://doi.org/10.1016/j.molp.2015.12.011 [ Links ]
Yamankurt G, Stawicki RJ, Posadas DM, Nguyen JQ, Carthew RW and Mirkin CA. 2020. The effector mechanism of siRNA spherical nucleic acids. Proceedings of the National Academy of Sciences of the United States of America 117(3):1312-1320. https://doi.org/10.1073/pnas.1915907117 [ Links ]
Zhang S, Li C, Si J, Han Z and Chen D. 2022. Action Mechanisms of effectors in plant-pathogen interaction. International Journal of Molecular Science 23(12):6758. https://doi.org/10.3390/ijms23126758. [ Links ]
Zhao S, Shang X, Bi W, Yu X, Liu D, Kang Z, Wang X and Wang X. 2020. Genome-wide identification of effector candidates with conserved motifs from the wheat leaf rust fungus Puccinia triticina Frontiers in Microbiology 11:1188. https://doi.org/10.3389/fmicb.2020.01188 [ Links ]
Zhou JM and Zhang Y. (2020). Plant Immunity: Danger Perception and Signaling. Cell, 181(5), 978-989. https://doi.org/10.1016/j.cell.2020.04.028 [ Links ]
Zhu W, Yu M, Xu R, Bi K, Yu S, Xiong C, Liu Z, Sharon A, Jiang D, Wu M, Gu Q, Gong L, Chen W and Wei W. (2022). Botrytis cinerea BcSSP2 protein is a late infection phase, cytotoxic effector. Environmental Microbiology 24(8): 3420-3435. https://doi.org/10.1111/1462-2920.15919 [ Links ]
Zhu X, Soliman A, Islam MR, Adam LR and Daayf F. 2017. Verticillium dahliae’s isochorismatase hydrolase is a virulence factor that contributes to interference with potato’s salicylate and jasmonate defense signaling. Frontiers in Plant Science 8:399. https://doi.org/10.3389/fpls.2017.00399 [ Links ]
Zuluaga AP, Vega-Arreguín JC, Fei Z, Ponnala L, Lee SJ, Matas AJ, Patev S, Fry WE and Rose JK. 2016. Transcriptional dynamics of Phytophthora infestans during sequential stages of hemibiotrophic infection of tomato. Molecular plant pathology 17(1): 29-41. https://doi.org/10.1111/mpp.122630 [ Links ]
Recibido: 30 de Octubre de 2022; Aprobado: 22 de Marzo de 2023











 texto em
texto em 


