Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de fitopatología
versión On-line ISSN 2007-8080versión impresa ISSN 0185-3309
Rev. mex. fitopatol vol.40 no.3 Texcoco sep. 2022 Epub 14-Nov-2022
https://doi.org/10.18781/r.mex.fit.2206-6
Artículos científicos
Identificación preliminar y relación filogenética de begomovirus asociados con Capsicum spp. en la península de Yucatán, México
1 Unidad de Bioquímica y Biología Molecular de Plantas, Centro de Investigación Científica de Yucatán A.C., Mérida, Yucatán, México, C.P. 97205
2 Unidad de Ciencias del Agua, Centro de Investigación Científica de Yucatán A.C., México, C.P. 77500
Se colectaron plantas de chile con síntomas virales en la Península de Yucatán, México (YPM), con el objetivo de caracterizar la identidad parcial y las relaciones genéticas y filogenéticas de los begomovirus que infectan diferentes especies de chile, que incluyen Capsicum chinense, variedades locales de C. annuum, C. frutescens y C. annuum var. aviculare, así como algunas especies de maleza. Las muestras de hoja fueron colectadas durante dos temporadas de infestación severa de mosquita blanca. El ADN genómico de las plantas se extrajo y un fragmento de los begomovirus se amplificó utilizando cebadores universales. Los amplicones fueron secuenciados y utilizados para análisis genéticos y filogenéticos. Los resultados demostraron que el 90.1% de las plantas colectadas (151) estuvieron infectadas por begomovirus. Los datos mostraron que el Pepper golden mosaic virus (PepGMV) es el virus más frecuente en las plantas cultivadas, semicultivadas y silvestres que se analizaron en este trabajo. Además, las secuencias analizadas indicaron la presencia del Euphorbia mosaic virus-Yucatán Peninsula (EuMV-YP) en plantas de chile habanero, así como la predominancia del PepGMV en diferentes especies de maleza. La variación genética de las secuencias del fragmento del componente A de los begomovirus, mostró que los aislados del PepGMV analizados están estrechamente relacionados entre ellos y comparten un 95-99% de identidad. Los datos mostraron que los begomovirus que infectan a las plantas colectadas incluyen a las especies parcialmente identificadas como PepGMV, Pepper huasteco yellow vein virus (PHYVV), Tomato severe leaf curl virus (ToSLCV), Tomato yellow leaf curl virus (TYLCV) and EuMV-YP.
Palabras clave: razas silvestres de Capsicum; chile habanero; filogenia; maleza
Pepper plants with viral symptoms were collected in the Yucatan Peninsula, Mexico (YPM) with the objective to characterize the partial identity and the genetic and phylogenetic relationship of the begomoviruses infecting different pepper species, including Capsicum chinense, various C. annuum landraces, C. frutescens, C. a. var. aviculare, as well as some weeds. The leaf samples were collected during two severe whitefly infestation seasons. Total genomic DNA from the sampled plants was extracted and a fragment of the begomoviruses was amplified using universal primers. Amplicons obtained were sequenced and sequences were used for genetic and phylogenetic analysis. Our results demonstrate that 90.1 % of the total sampled plants (151) were infected with begomoviruses. The data identified Pepper golden mosaic virus (PepGMV) as the most frequent species found within cultivated, semi-cultivated, and wild-analyzed samples. In addition, sequence analysis indicates the presence of Euphorbia mosaic virus-Yucatán Peninsula (EuMV-YP) infecting a Habanero chili plant, as well as the predominance of PepGMV in different weed plant species. Genetic variation based on nucleotide distance analysis from partial DNA-A begomovirus sequences indicated that the PepGMV isolates were closely related among them sharing 95 to 99% nucleotide sequence identity. Data showed that begomovirus that infected the sampled plants included the partially identified species of PepGMV, Pepper huasteco yellow vein virus (PHYVV), Tomato severe leaf curl virus (ToSLCV), Tomato yellow leaf curl virus (TYLCV) and EuMV-YP.
Key words: Capsicum annuum landraces; habanero chili; phylogeny; weed plants
Los miembros de la familia Geminiviridae, que tienen genomas circulares de ADN monocatenario (ssDNA) encapsidados en partículas icosaédricas gemelas, forman la segunda familia más grande de virus de plantas. El género Begomovirus comprende virus con genomas monopartitas (un componente de ADN de ~2,9 kb) y/o en su mayoría bipartitas (dos componentes de ADN de ~2,6 kb, denominados ADN-A y ADN-B) (Brown et al., 1995; Fauquet y Stanley, 2005; Jones, 2003). Los begomovirus incluyen más de 440 especies, algunas de las cuales pueden infectar plantas dicotiledóneas. Las especies de begomovirus son transmitidas por mosquitas blancas del complejo de especies crípticas Bemisia tabaci (Fiallo-Olive et al., 2021). Se han documentado enfermedades devastadoras causadas por begomovirus en diferentes cultivos en América Central, México, la cuenca del Caribe, el sur de los Estados Unidos de América y América del Sur. Las pérdidas oscilan entre el 50 y el 90 % de la producción total (Ala-Poikela et al., 2005; Brown et al., 2005; Morales et al., 2005; Rausch et al., 2005; Torres-Pacheco et al., 1996). Reportes previos han confirmado la incidencia de begomovirus en una variedad de plantas cultivadas de la familia Solanácea y flora nativa de varias regiones de México (Ascencio-Ibañez et al., 1999; Garrido-Ramirez y Gilbertson, 1998; Gregorio-Jorge et al., 2010; Hernández-Zepeda et al., 2007a; Hernández-Zepeda et al., 2007b; Hernández-Zepeda et al., 2007c; Torres-Herrera et al., 2019; Torres-Pacheco et al., 1996). En particular, el PepGMV ha sido reportado en chile cultivado y silvestre en diferentes regiones de México como Baja California, Sinaloa, Sonora, la Comarca Lagunera, Campeche, Yucatán, San Luis Potosí y Michoacán, entre otros lugares (Hernandez-Espinal et al., 2018; Morales-Aguilar et al., 2019; Rodelo-Urrego et al., 2015; Rodriguez-Negrete et al., 2019).
Capsicum annuum (familia: Solanaceae), un cultivo de alto valor debido a sus frutos no picantes (pimiento dulce) y picantes (chile syn. hot pepper), es la especie de chile más cultivada en todo el mundo (Bosland y Votova, 2000; Park et al., 2021). Esta especie, junto con otras especies picantes y valiosas que se originaron y domesticaron en América tropical, como C. chinense (chile habanero), C. pubescens (chile manzano) y C. frutescens (chile tabasco o picopaloma), se cultivan y se consumen ampliamente en México (Cazares-Sánchez et al., 2005). Los problemas persistentes que afectan a estos cultivos incluyen una variedad de virus y sus vectores. Gran parte de las pérdidas de rendimiento se atribuyen a enfermedades virales causadas por begomovirus y al uso de genotipos de chile susceptibles (Anaya-Lopez et al., 2003; Torres-Pacheco et al., 1996). La diversidad del chile es un recurso esencial para la mejora genética y el manejo del cultivo. En combinación con la maleza, los chiles actúan como reservorio hospedero de virus que, junto con la mosquita blanca, contribuyen a la diversificación de patógenos en un proceso que debe investigarse más a fondo. En los estados de la Península de Yucatán en México (YPM), variaciones significativas en las razas nativas de C. annuum var. annuum (cuyos nombes mayas son Ya´ax iik, X´kat iik, Dulce y el chile silvestre C. annuum var. aviculare, llamado Maax iik), se producen en respuesta a cambios de estrés biótico y/o abiótico. Estas variedades locales, que se cultivan principalmente en huertos familiares con el sistema de Milpas (asociado a otros cultivos), o se cosechan in situ, se utilizan para el consumo local y como fuente de ingresos de las familias rurales (Cazares-Sánchez et al., 2005). A pesar de su importancia, existe información limitada sobre la distribución e incidencia de diferentes patógenos como los begomovirus. El chile habanero, un cultivo esencial tanto para uso local como para exportación, se cultiva en extensos campos agrícolas. Sin embargo, el mayor uso de agroquímicos no ha reducido las pérdidas de la cosecha. Más bien, la infestación de la mosquita blanca y las enfermedades virales todavía afectan al 90% del cultivo durante la estación seca (marzo a mayo de cada año; observaciones de agricultores locales y del autor). La severidad de estos problemas ha afectado el cultivo de chile habanero en campo. La producción de campo tradicional se ha desplazado hacia sistemas de cultivo alternativos como el acolchado, la agricultura en microtúnel y los invernaderos. El primer paso hacia el desarrollo de estrategias amplias de control es la identificación y caracterización genética de los patógenos (y sus vectores), que son comunes a las especies de chile silvestre y cultivado, así como a las especies de maleza que están asociadas con ellas. El presente estudio tuvo como objetivo caracterizar la identidad parcial y las relaciones genéticas y filogenéticas de los begomovirus que infectan diferentes chiles, incluyendo C. chinense cultivado, varias variedades locales de C. a. annuum y C. frutescens y la especie silvestre C. annuum var. aviculare (Maax iik), así como algunas plantas silvestres que crecen como maleza en diferentes localidades del estado de Yucatán y Campeche. Se utilizaron análisis genéticos y filogenéticos basados en secuencias de nucleótidos de la región central del gen de la proteína de la capside (Cp) (CPR) para explorar la diversidad genética de los begomovirus y determinar la posible relación con otros begomovirus bipartitas conocidos.
Materiales y métodos
Material vegetal y fuentes de virus
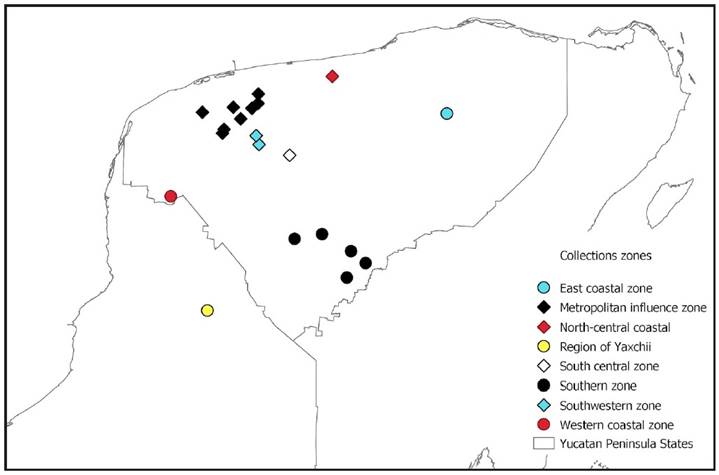
Se utilizaron un total de 151 muestras de plantas de chile con síntomas, incluidas las cultivadas de tipo habanero Capsicum chinense (45) y tipo Scotch Bonet (12); las variedades locales de C. annuum var. annuum identificadas regionalmente como Dulce (18); X´kat iik (12) y Ya´ax iik (8); plantas de C. annumm. var. aviculare conocida localmente como Maax iik (43) y plantas de C. frutescens (ocho) tipo Tabasco, que crecen silvestres o en huertos familiares. Adicionalmente, se recolectó maleza representativa de diferentes familias: Callicarpa spp., (Verbenaceae) (uno), Croton malvaviscifolius (Euphorbiaceae) (uno), Dicliptera sexangularis (Acanthaceae) (uno), Herissantia crispa (Malvaceae) (uno) y Nicotiana tabacum (Solanaceae) (uno). El material vegetal recolectó en diferentes localidades de los estados de Yucatán (YS) y Campeche (CS), ambos ubicados en la YPM (Cuadro 1, Figura 1). Se recolectaron muestras de hojas de maleza en sitios no agrícolas y en áreas de cultivo de chile, durante dos temporadas de infestación severa de mosquita blanca (de febrero a mayo y de julio a principios de octubre de 2017). Las muestras se almacenaron a -20 ºC en el laboratorio de virología molecular del CICY para utilizarlas como fuente del ADN viral. El ADN genómico total de las hojas de plantas de chile y de maleza se extrajo de acuerdo con los métodos de Dellaporta et al. (1983) y Echevarría-Machado et al. (2005), respectivamente. Las muestras de hojas se limpiaron con agua destilada estéril antes de la extracción de ADN para eliminar una posible contaminación por mosquita blanca.
Cuadro 1 Ubicaciones donde se recolectó el material biológico en los estados de Yucatán (YS) y Campeche (CS), México.
| State | Region/ zone | District |
|---|---|---|
| Yucatan | Metropolitan influence zone | Conkal (10)z, Cholul (8), Chicxulub Pueblo (8), Dzitya (5), Hunucma (6), Hunxectaman (6), Merida (6), Uman (8) |
| North-central coastal | Dzidzantun (6) | |
| East coastal zone | Espita (10) | |
| Western coastal zone | Halacho (6) | |
| South central zone | Cuzama (8) | |
| Southern zone | Catmis (6), Peto (10), Polinkin (6), Tixmehuac (6), Tekax (10) | |
| Southwestern zone | Timucuy (8), Subincancab (8) | |
| Campeche | Region of Yaxchii | Hopelchen (10) |
zLos números entre paréntesis representan el número de plantas recolectadas por localidad.
Amplificación, clonación, secuenciación y análisis de secuencias mediante la reacción en cadena de la polimerasa (PCR)
Se utilizó el par de cebadores prAV324/AC889 para la amplificación por PCR del fragmento génico de la región central del gen de la proteína de la capside viral (core Cp) de ~576 pb para el diagnóstico de los begomovirus (Brown et al., 2005), siguiendo el protocolo recomendado por los autores. Los productos de PCR específicos de begomovirus de ~576 pb se purificaron con un kit QIAEX II (QIAGEN) y se clonaron en el vector pGEM T-Easy (Promega, Madison, WI, EE. UU.) siguiendo las instrucciones del fabricante. Las secuencias de nucleótidos se determinaron con un secuenciador ABI 377 (Davis Sequencing Co., CA, EE. UU.). La mayoría de las clonas se secuenciaron en dirección directa e inversa. Para aumentar la probabilidad de detectar múltiples especies de begomovirus en muestras de campo con infecciones mixtas, se clonaron de tres a cinco productos de PCR y se analizaron sus secuencias de nucleótidos. Las secuencias de nucleótidos (120) de muestras clonadas seleccionadas al azar (52) se editaron manualmente para reducir la degeneración en los cebadores (EditSeq, DNASTAR versión 5.08, Madison, WI, EE. UU.), lo que resultó en una secuencia de 533 nt del core Cp. La identificación parcial de especies de los Begomovirus se realizó mediante la comparación con secuencias disponibles en la base de datos NCBI-GenBank (http://www.ncbi.nlm.nih.gov) por BLASTn (Altschul et al., 1997).
Análisis de datos de las secuencias
Los análisis genéticos y filogenéticos se realizaron sobre los datos de 25 secuencias parciales del core Cp de begomovirus identificadas en el presente estudio, 23 de las cuales se aislaron de especies y variedades locales de Capsicum y de dos diferentes especies de maleza. Además, se obtuvieron 36 secuencias de referencia estándar, editando el core Cp del genoma begomoviral completo de secuencias obtenidas de la base de datos GenBank (Tabla 2). Estas secuencias se incluyeron en el análisis filogenético. La alineación de secuencias múltiples de los taxones se realizó con CLUSTALW en MegAlign (DNASTAR versión 5.08, Madison, WI). El análisis de la matriz de distancia genética, la diversidad y el patrón de sustituciones de nucleótidos (total, sinónima y no sinónima) se realizó con 23 secuencias de nucleótidos del core Cp del PepGMV (Cuadro 2). Las secuencias del PepGMV seleccionadas incluyeron tres muestras de cada especie de Capsicum y una secuencia de cada maleza. La región genómica del core Cp (CPR) corresponde a la región del gen en las posiciones 147-679, de acuerdo a la secuencia del GenBank AF077025. Las pruebas evolutivas moleculares se realizaron utilizando los métodos de 2 parámetros de Kimura y Kumar. El análisis Bootstrap con 1000 repeticiones se realizó utilizando el software MEGA versión 11 (Tamura et al., 2021). Se realizó una prueba Z basada en codones, promediando secuencias generales, para probar la fuerza selectiva que opera en secuencias del core Cp, utilizando el método Kumar en MEGA 11. Se eliminaron todas las posiciones que contenían espacios y datos faltantes (opción de eliminación completa). Hubo un total de 177 posiciones en el conjunto de datos final. Para reconstruir el árbol filogenético, se analizaron múltiples secuencias alineadas por parsimonia múltiple usando MEGA 11, con 1000 iteraciones por réplica. El árbol de consenso estricto se construyó usando la configuración estándar de MEGA 11. El soporte estadístico para cada clado principal se calculó usando Bootstrap con 1000 iteraciones por réplica. El árbol se enraizó usando la secuencia del core Cp editada del virus del rayado del maíz (MSV-CP) del género Mastrevirus, familia Geminiviridae (Tabla 2).
Cuadro 2 Geminivirus utilizado en el alineamiento de nucleótidos y análisis filogenéticos, con el respectivo acrónimo del virus y número accesión de GenBank.
| Geminivirus | Acronym | GB Acc No |
|---|---|---|
| African cassava mosaic virus-[Nigeria] | ACMV-[NG] | X17095 |
| Abutilon mosaic virus | AbMV | X15983 |
| Bean calico mosaic virus | BCaMV | AF11018 |
| Bean golden mosaic virus-[Brazil] | BGMV-[BR] | M88686 |
| Bean golden yellow mosaic virus-[Mexico] | BGYMV-[MX] | AF17355 |
| Bean golden yellow mosaic virus-[Guatemala] | BGYMV-[GT] | M91604 |
| Cabbage leaf curl virus | CaLCuV | U65529 |
| Chino del tomate virus | CdTV | AF22666 |
| Corchorus yellow vein virus | CYVV-[Hoa] | AY72790 |
| Cotton leaf curl Gezira virus | CLCuGV | AY03600 |
| Cucurbit leaf crumple virus-[Arizona] | CuLCrV-[Ari] | AF25620 |
| Euphorbia mosaic virus-[Yucatan Peninsula] | EuMV-[YP] | DQ31893 |
| Euphorbia mosaic virusz | H6-EuM-YP (C.chinense cv. Habanero)z | EU15585 |
| Maize streak virus | MSV-CP | NC00134 |
| Melon chlorotic leaf curl virus-[Guatemala] | MCLCuV-[GT] | AF32549 |
| Pepper golden mosaic virus-[Chiapas] | PepGMV-[Chi] | AF07702 |
| Pepper golden mosaic virus-[Costa Rica] | PepGMV-[CR] | AF14922 |
| Pepper golden mosaic virus-[Distorter] | PepGMV-[D] | AY92851 |
| Pepper golden mosaic virus-[Mo] | PepGMV-[Mo] | AY92851 |
| Pepper golden mosaic virus-[Serrano] | PepGMV-[Ser] | AY92851 |
| Pepper golden mosaic virus-[Tamaulipas] | PepGMV-[Tam] | U57457 |
| Pepper golden mosaic virus-[Nicaragua] | PepGMV-[Ni] | AJ842140 |
| Pepper golden mosaic virusz | 1-3-PepGMV (D. sexangularis)z | EU155830 |
| 3-PepGMV (C. annuum cv Sweet pepper)z | EU155831 | |
| 43-PepGMV (C. annuum)z | EU155834 | |
| 45-PepGMV C.annuum cv Cubano)z | EU155835 | |
| 120-PepGMV (C. chinense cv Habaneroz | EU155836 | |
| 122-PepGMV (C.chinense cv Habaneroz | EU155838 | |
| 138-PepGMV(C.annuum cv Sweet pepper)z | EU155839 | |
| 144-PepGMV (C.annuum cv Xkaticz | EU155841 | |
| 151-PepGMV (C.annuum cv Sweet pepper)z | EU155843 | |
| 164-PepGMV (C.annuum cv Xkaticz | EU155844 | |
| 240-2-PepGMV (C.annuum cv Maax)z | EU155846 | |
| 250-2-PepGMV (H. crispa)z | EU155847 | |
| R3-PepGMV (C.annuum)z | EU155853 | |
| R6-PepGMV (C. frutescens) z | EU155854 | |
| R10-PepGMV (C.chinense cv Habaneroz | EU155855 | |
| R13-PepGMV C.chinense cv Scotch bonnetz | EU155856 | |
| R15-PepGMV (C.annuum cv Maax)z | EU155858 | |
| R16-PepGMV (C.chinense cv Habaneroz | EU155859 | |
| R12-PepGMV (C. annuum cv Maax)z | ||
| Pepper huasteco yellow vein virus | PHYVV | X70418 |
| Pepper huasteco yellow vein virusz | 40-PHYVV (C. annuum)z | EU155833 |
| 120- PHYVV (C. chinense cv Habanero)z | EU155837 | |
| 183- PHYVV (C. chinense cv Habanero)z | EU155845 | |
| Rhynchosia golden mosaic virus | RhGMV | AF239671 |
| Rhynchosia golden mosaic virus-[Chiapas] | RhGMV-[Chi] | AF408199 |
| Sida golden mosaic Costa Rica virus | SiGMCRV | X99550 |
| Sida golden mosaic virus-[Florida] | SiGMV-[Flo] | AF049336 |
| Sida golden mosaic Honduras virus | SiGMHV | Y11097 |
| Squash leaf curl virus | SLCV | M38183 |
| Tomato golden mosaic virus-Yellow vein | TGMV-YV | K02029 |
| Tomato mild yellow leaf curl Aragua virus | TMYLCAV | AY927277 |
| Tomato mottle virus-[Florida] | ToMoV-[Flo] | L14460 |
| Tomato severe leaf curl virus-[Guatemala] | ToSLCV-[GT] | AF130415 |
| Tomato severe leaf curl virusz | 144- Tomato severe leaf curl virus Guz | EU155842 |
| Tomato yellow leaf curl virus-Mild | TYLCV-Mld | X76319 |
| Tomato yellow leaf curl virus-[Dominican Republic] | TYLCV-[DR] | AF024715 |
| Tomato yellow leaf curl virusz | 138-TYLCV DR (C. annuum cv Sweet pepper)z | EU155840 |
zClonas de begomovirus aislados de plantas recolectadas en los estados de Campeche y Yucatán.
Resultados
Búsqueda de síntomas virales en plantas cultivadas y de campo.
En un estudio de la enfermedad durante temporadas de alta infestación de mosquita blanca, en localidades de YS y CS (Figura 1), se encontraron plantas de chile de diferentes especies y maleza con síntomas similares a los causados por virus. Todas las plantas de chile cultivadas colectadas, como C. chinense, C. a. var annuum, variedad local Dulce, X´kat iik y Ya´ax iik, C. frutescens y la especie silvestre C. annuum var. aviculare Maax iik, mostraron síntomas como mosaicos dorados, deformación en las hojas y retraso en el crecimiento. En C. frutescens los síntomas se observaban claramente, mientras que en el chile Maax iik, a menudo aparecían moderadamente presentes en una o dos ramas y rara vez afectaban a toda la planta (Figura 2A). Las variedades locales X´kat iik (Figura 2B), Chile Dulce (Figura 2C y D) y Chile Habanero (Figura 2E y F) mostraron síntomas foliares notables de diversa gravedad, que incluyen amarillamiento, hojas rizadas hacia abajo, mosaicos dorados y retraso en el crecimiento. Se observaron síntomas comparables en especies de maleza (imágenes no mostradas). En la mayoría de los casos, las plantas de chile mostraron hojas pequeñas y rizadas y tenían un aspecto atrofiado y tupido, debido a la longitud reducida de los entrenudos (Figura 2D).

Figura 2 Síntomas observados en plantas silvestres (A) y cultivadas (B, C, D, E y F) muestreadas en Catmis (A), Muna (B), Mérida (C), Cuzama (D) y Tekax (E y F) infectadas con Begomovirus (PCR+). (A) Capsicum annuum var. aviculare (Maax iik) que muestra ramas con mosaicos dorados, hojas distorsionadas y crecimiento atrofiado; (B) C. annuum var. annuum (X´kat iik) con mosaico dorado severo y hojas distorsionadas; (C y D) C. annuum var. annuum (Chile Dulce) con mosaicos dorados severos, hojas distorsionadas y crecimiento atrofiado; (E y F) C. chinense (tipo Habanero) con mosaicos dorados, hojas rizadas hacia abajo y crecimiento gravemente atrofiado.
Identificación preliminar de Begomovirus
Del total de muestras de plantas colectadas (151), incluyendo 146 chiles y cinco plantas de maleza, el 90.1% fueron PCR positivas con los cebadores que amplifican el core Cp (Cuadro 3). Es decir, el 89.7% de los chiles (131), incluyendo las muestras de plantas de todas las especies de chile y todas las muestras de maleza (5), fueron positivas para begomovirus. La secuenciación de los amplicones, en promedio de dos a tres por muestra de planta (52), reveló la probable incidencia de infecciones simples y mixtas de begomovirus. Las especies de virus detectadas en 120 clonas secuenciadas se identificaron provisionalmente en base a comparaciones de identidad de nucleótidos (> 89% nt) con secuencias del core Cp viral de begomovirus reportados previamente (Cuadro 3). Las infecciones simples con el virus del mosaico dorado del chile (PepGMV) fueron las más frecuentes, representando el 82.5% de las cloneas secuenciadas (99), detectados en hojas de las diferentes especies de Capsicum y en las especies de maleza Callicarpa spp., C. malvaviscifolius, D. sexangularis, H. crispa y N. tabacum (Cuadro 3). Le siguió el virus de la vena amarilla del pimiento huasteco (PHYVV) (5%), que se encontró en dos plantas de C. chinense, una clona aislada de Habanero y el otro de tipo Scotch Bonnet. Además, se encontró el virus del enrollamiento severo de la hoja del tomate (ToSLCV) (4.2%) en plantas de Habanero y Dulce. Curiosamente, cuatro clonas secuenciadas (3.3%) de una sola planta de chile habanero (muestra H6 EuMV-YP[C. chinense H]) mostraron una alta identidad (98-99%) con el virus del mosaico de Euphorbia-[Península de Yucatán] EuMV-YP aislado en Euphorbia heterophylla (Hernandez-Zepeda et al., 2007b). Las posibles infecciones mixtas se confirmaron realizando una búsqueda bidireccional de la secuencia parcial de tres a cinco clonas de cada especie begomoviral aislada en cada planta. Estas incluyeron una mezcla del PepGMV-PHYVV en chile Habanero (muestra 120, C. chinense H), PepGMV-TYLCV en chile Dulce (muestra 138, C. annuum D) y PepGMV-ToSLCV en una planta X´kat iik (muestra 144, C. annuum, Cuadro 3).
Cuadro 3 Identificación provisional de Begomovirus (identidad >89% nt) basado en secuencias parciales, encontrada en muestras de plantas de chile cultivadas*, semicultivadas☼, silvestres† y plantas de maleza (w) recolectadas en el estado de Yucatán y Campeche. Virus amplificados por PCR con cebadores universales para core Cp. Número de clonas positivas +PCR sobre el total de plantas muestreadas por especie y/o tipo de raza local. Entre paréntesis, el número de clonas secuenciadas positivas. Las posibles infecciones virales mixtas aparecen subrayadas.
| Host species | No. +PCR / total samples | Virus species identity >89% nt (No. of sequenced clones to core Cp) |
|---|---|---|
| Capsicum chinense (habanero)z | 53/57 | (35) PepGMV, (6) PHYVV, (2) ToSLCV, (4) EuMV-YP, 1PepGMV:1PHYVV |
| C. annuum var. Annuum (dulce)* | 16/18 | (12) PepGMV, (3) ToSLCV, 1PepGMV:1TYLCV. |
| C. a. var. Annuum (X´kat iik)* | 11/12 | (9) PepGMV, 1PepGMV:1ToSLCV. |
| C. a. var. Annuum (Ya´ax iik ) ☼ | 8/8 | (7) PepGMV |
| C. frutescens (Pico de paloma) ☼ | 6/8 | (5) PepGMV |
| C. a. var. aviculare (Maax iik)† | 37/43 | (18) PepGMV |
| Callicarpa spp (w) | 1/1 | (3) PepGMV |
| Croton malvaviscifolius (w) | 1/1 | (3) PepGMV |
| Nicotiana tabacum (w) | 1/1 | (3) PepGMV |
| Dicliptera sexangularis(w) | 1/1 | (2) PepGMV |
| Herissantia crispa (w) | 1/1 | (2) PepGMV |
Variación genética de aislados del PepGMV basada en análisis de distancia de nucleótidos a partir de secuencias parciales del ADN-A de begomovirus
El análisis de distancia genética basado en el análisis por parejas de 23 secuencias de nucleótidos del core Cp seleccionadas (Cuadro 4), indicó que los aislados del PepGMV son cepas estrechamente relacionadas, que compartían entre 95 y 99% de identidad de secuencia de nucleótidos. El porcentaje de identidad de nt se calculó como [1-distancia genética] x100 (Brown et al., 2005). La distancia promedio de nt del CPR estimada para el número de sustituciones de bases por sitio en los 23 pares de secuencias fue igual a 0.027 (± 0.004 SE), con una distancia de transición a transversión calculada por proporción de sitio (R) igual a 0.641 (± 0.21 SE). En general, la divergencia promedio estimada basada en codones de todos los pares de secuencias mostró un valor bajo de sustituciones no sinónimas (dN) a sustituciones sinónimas (dS) (dN = 0.003 < dS = 0.130) y una proporción dN/dS baja de 0.023 (Cuadro 4).
Análisis filogenético basado en secuencias parciales del ADN-A de begomovirus
El análisis filogenético analizó la relación probable entre los begomovirus encontrados que infectan chiles y aquellos que infectan especies de maleza colectadas en YS y CS, según sus secuencias correspondientes en el GenBank. El análisis incorporó las secuencias de nt de la región central del gen de la proteína de la capside (core Cp) de 61 taxones. La matriz se construyó con secuencias de 23 nt aisladas de Capsicum spp., dos de maleza (Cuadro 3) y 36 de secuencias publicadas en el GenBank (Cuadro 2). La historia evolutiva se infirió utilizando el método de Máxima Parsimonia. La Figura 3 muestra el árbol más parsimonioso, con una longitud =3191. El índice de consistencia es 0.376378 (0.309704), el índice de retención es 0.634911 (0.634911) y el índice compuesto es 0.238966 (0.196634) para todos los sitios y sitios informativos de parsimonia (entre paréntesis). El porcentaje de árboles replicados en los que los taxones asociados se agruparon por Bootstarp (1000 repeticiones) se muestra junto a las ramas. El árbol MP se generó utilizando el algoritmo Subtree-Pruning-Regrafting (SPR) (nivel de búsqueda 1). Los árboles iniciales se obtuvieron mediante la adición aleatoria de secuencias (10 repeticiones). Hubo un total de 566 posiciones en el conjunto de datos final. Los análisis evolutivos se realizaron en MEGA11. El grupo de begomovirus del hemisferio oriental con un valor de Bootstrap del 99% incluye el virus del enrollamiento de la hoja amarilla del tomate (Tomato yellow leaf curl virus), del cual la clona 138-TYLCV-[C. annuum D], aislado de una planta con una probable infección mixta (Cuadro 3, Figura 3), mostró una alta identidad (99%), con el aislado de TYLCV de la República Dominicana. Curiosamente, se observó un clado divergente que contenía tres clones de aislados del PepGMV de diferentes cultivares de C. annuum cerca del MSV-CP, el Mastrevirus, que se seleccionó como la raíz del árbol filogenético, con un 89 % del valor de Bootstrap. El grupo del Hemisferio Occidental se divide en dos subclados (63% soporte del nodo). Un grupo incluye el virus del enrollamiento severo de la hoja del tomate (Tomato severe leaf curl virus) clon 144-ToSLCV-[C. annuum X]), aislado de una planta X´kaat ik con una probable infección mixta (Cuadro 3, Figura 3). Este grupo está asociado con el aislado de referencia ToSLCV-[GT] (82% de Bootstrap), comparte nodos comunes con otros begomovirus y se agrupa con especies del virus del mosaico dorado de Sida (Sida golden mosaic virus -SiGMV), el virus del moteado del tomate (Tomato mottle virus -ToMoV), el virus del Chino del tomate (Chino del tomate virus - CdTV) y el virus del mosaico de Abutilon (Abutilon mosaic virus - AbMV). El segundo grupo, con dos ramas, incluye una clona del virus del mosaico de Euphorbia (Euphorbia mosaic virus), H6 EuMV-YP (C. chinense H), aislado de chile, con su especie hermana, el aislado de referencia EuMV-YP (99% de Bootstrap) y el virus del encrespado amarillo de la hoja del tomate (Tomato mild yellow leaf curl Aragua virus - TMYLCAV), relación sustentada con un valor de Bootstrap del 77% (Figura 3). Los primeros grupos se ensamblan con una especie de begomovirus que no recibió soporte de Bootstrap, incluido el clado SLCV. Las otras relaciones inferidas para las colecciones no tienen soporte de Bootstrap y se subdividen en dos subclados. Un subclado que comprende el cluster del virus huasteco de la vena amarilla del chile (Pepper huasteco yellow vein virus - PHYVV) se agrupa con tres aislados de C. chinense, las clonas 120 y 183 de PHYVV- de Habanero y el aislado 40-PHYVV de la planta Maax iik, con 88 a 99 % de valores Bootstrap. El segundo subclado está formado por secuencias del virus del mosaico dorado del chile (Pepper golden mosaic virus - PepGMV) de la mayoría de los aislamientos de chile y maleza con aislamientos de referencia de begomovirus bien conocidos, formando un clado principal del PepGMV respaldado por Bootstrap (83%). Este clado del PepGMV se dividió visiblemente en tres subclados (Bootstrap 89%) (Figura 3). Dos clonas del PepGMV, R3 y 164, se agruparon en un clado separado con el TYLCV-DR, con un 89% de soporte de Bootstrap. Es necesario un análisis posterior con la secuencia completa del Componente A de ambos clonas para corroborar esta relación filogenética.
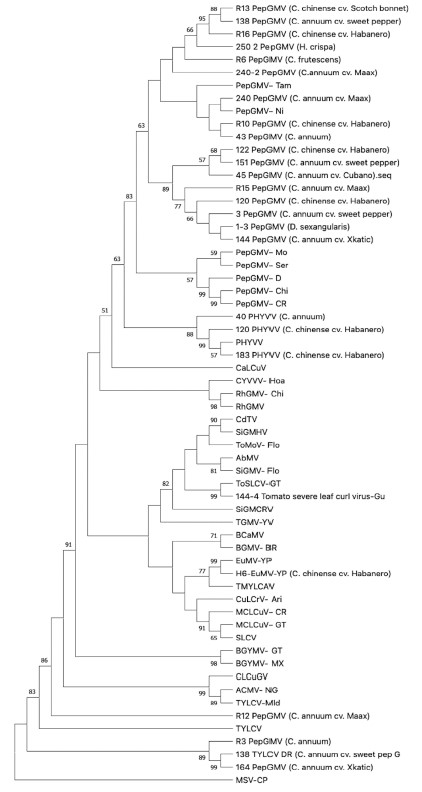
Figura 3 Relaciones filogenéticas inferidas por la secuencia parcial del core Cp de begomovirus identificados en plantas de chile cultivadas y silvestres provenientes de Yucatán y Campeche, México. El análisis también incluye la secuencia del core Cp de begomovirus seleccionados y bien estudiados que están disponibles en el GenBank. La figura muestra el árbol más parsimonioso. Los números colocados en los nodos representan valores de Bootstrap superiores al 50%. Los valores más bajos no se muestran (1000 iteraciones).
Discusión
Los virus de las plantas tienen un enorme potencial para la variación genética y la rápida evolución. La mutación, el reordenamiento y la recombinación son mecanismos conocidos que generan variación genética, pero se sabe poco sobre las presiones de selección que operan e impulsan su evolución (Font et al., 2007; Garcia-Arenal et al., 2003; Seal et al., 2006). Aquí se proporciona evidencia de que las infecciones en la YPM causadas por miembros del género Begomovirus son comunes en chiles cultivados y plantas de maleza. Casi todas las plantas de chile cultivadas en campo estudiadas en el presente trabajo, incluidas las variedades locales Dulce, X´kat ikk, Ya´ax ikk, Pico de Paloma y chile habanero, mostraron síntomas graves de infección por Begomovirus. De hecho, la mayoría de las muestras recolectadas (90%) fueron positivas para begomovirus por PCR (PCR-BG-+). La región central conservada del gen AV1 de la proteína de la capside (core Cp) se utilizó para la identificación provisional de los begomovirus. Esta secuencia parcial permitió la identificación provisional del virus. Para una clasificación definitiva, se requieren secuencias completas de los componentes ADN-A y ADN-B (Brown et al., 2001; Fauquet et al., 2003). Centroamérica, México y Brasil parecen ser importantes centros de diversidad de begomovirus en el hemisferio occidental (Ala-Poikela et al., 2005; Fernandes et al., 2006; Hernandez-Zepeda et al., 2007a; Rodriguez-Negrete et al., 2019). Se han reportado más de 440 especies de begomovirus, incluyendo un gran número de patógenos emergentes que afectan chile, frijol (Phaseolus vulgaris), algodón (Gossypium sp.), cucurbitáceas, okra (Abelmoschus esculentus), papaya (Carica papaya), tomate (Solanum lycopersicum) y maleza (ICTV, 2022). En un estudio anterior, de 119 plantas sintomáticas pertenecientes a 16 especies diferentes, solo el 58% de las muestras de la YPM fueron positivas a begomovirus. En ese estudio, se identificaron 13 especies distintas de begomovirus, incluido el PepGMV aislado de C. annuum (Hernandez-Zepeda et al., 2007c). Por el contrario, casi el 90% de las muestras analizadas en el presente trabajo fueron positivas para PepGMV. En ambos casos, todas las plantas recolectadas presentaron síntomas virales. Como se comentó anteriormente, una posible explicación es que algunas plantas sintomáticas recolectadas en el presente estudio estaban infectadas con otros virus que producen síntomas similares (Hernandez-Zepeda et al., 2007c). Otro reporte mostró que una infección de tomate con síntomas similares a los causados por geminivirus fue inducida por Potyviridae y Tobamoviridae (Polston y Anderson, 1997). Es notable que las plantas de C. anuumm var. s aviculare Maax iik examinadas en el presente estudio mostraron síntomas leves en una rama de la planta y rara vez en dos. Sin embargo, el 86% del material recolectado de Maax iik fue PCR-BG-+. La remisión de los síntomas o la recuperación del huésped se ha asociado con la resistencia específica de las plantas de chile al PepGMV y se han sugerido mecanismos de silenciamiento en el proceso de recuperación (Carrillo-Trip et al., 2007). Esto es importante, ya que la evidencia de la literatura sugiere que las fuentes de resistencia a PHYVV y PepGMV se encuentran entre los parientes silvestres de C. annuum y algunas accesiones de C. chinense (Anaya-Lopez et al., 2003; Hernandez-Espinal et al., 2018; Hernández-Verdugo et al., 2001).
En base a las secuencias del core Cp, las cinco especies de begomovirus parcialmente identificadas asociadas a plantas de chile cultivadas en la YPM concuerdan con las descritas por otros autores (Anaya-Lopez et al., 2003; Godinez-Hernandez et al., 2001; Hernandez-Espinal et al., 2018; Hernández-Zepeda et al., 2007b; Morales-Aguilar et al., 2019). El hallazgo inusual del EuMV-YP infectando una planta de chile habanero, reportado aquí, es notable dado que esta especie viral comúnmente infecta la maleza neotropical Euphorbia heterophylla (Ala-Poikela et al., 2005; Gregorio-Jorge et al., 2010; Hernández-Zepeda et al., 2007a; Hernández-Zepeda et al., 2007c). En México, este virus ha sido reportado en la YPM y en Jalisco (Gregorio-Jorge et al., 2010; Hernandez-Zepeda et al., 2007a). En Cuba se reportó que el EuMV infecta plantas de tabaco recolectadas en el campo (Fiallo-Olive, 2010). Aunque los datos experimentales han confirmado que las plantas P. vulgaris, S. lycopersicum, Nicotiana benthamiana, C. annuum, C. chinense, Cnidoscolus chayamansa, Jatropha curcas, Ricinus communis, Arabidopsis thaliana y N. tabacum son hospederos del EuMV-YP (Hernández- Zepeda et al., 2007b; Villanueva-Alonzo et al., 2013), se requieren estudios de campo adicionales para establecer la extensión del rango de hospederos y la distribución del EuMV-YP en México y otros países de América Central y del Sur.
En el presente estudio, el PepGMV fue la especie de begomovirus más extendida que se encontró infectando a C. chinense cultivada, C. annuum variedad local Dulce y X´kat iik, a C. frutescens cultivada en huertos domésticos y la planta silvestre Maax ikk, así como a la maleza recolectada en la YPM. Se deben analizar mas muestras de maleza para establecer la distribución del PepGMV en estas plantas en la YPM. Esta especie de virus también ha sido reportada en América Central, infectando chiles en Costa Rica y Honduras (Lotrakul et al., 2000; Morales et al., 2005), así como plantas de tomate, chile y cucurbitáceas en Nicaragua (Ala-Poikela et al., 2005). Además, las muestras analizadas de chile habanero presentaron una infección simple con el PHYVV y el ToSLCV, en concordancia con los resultados de otros estudios en otras partes de México y Nicaragua (Ala-Poikela et al., 2005; Hernandez-Espinal et al., 2018; Morales- Aguilar et al., 2019). En el presente estudio, el hallazgo de un aislado de TYLCV en posible combinación con el PepGMV que infecta una planta de C. annuum Dulce, está respaldado por reportes anteriores de la presencia de TYLCV en plantas de tomate en Yucatán desde 1999 (Ascencio-Ibañez et al., 1999). Esta cepa de TYLCV comparte un 99% de identidad con la cepa de República Dominicana (Ascencio-Ibañez et al., 1999), una cepa que se considera un componente del clado del este de América del Norte y el Caribe (ENAC) del TYLCV (Duffy y Holmes, 2007). Las otras posibles infecciones mixtas, de PepGMV-PHYVV, de PepGMV-TYLCV y de PepGMV-ToSLCV, se presentaron frecuentemente en asociación solo con chiles cultivados, como el chile habanero y C. annuum. Var. annuum, variedad local Dulce y X´kat ikk, en áreas de cultivo con registros de infestaciones de mosquita blanca (Escobedo-García Medrano, datos no publicados). Es de destacar que las infecciones mixtas con el PepGMV y el PHYVV ocurran naturalmente en muchos cultivos hortícolas en México en donde la distribución de los dos virus a menudo se superpone (Hernandez-Espinal et al., 2018; Morales-Aguilar et al., 2019). La expresión de síntomas en las interacciones de PepGMV-PHYVV parece depender del hospedero (Mendez-Lozano et al., 2003). Las investigaciones futuras sobre las interacciones PepGMV-PHYVV, PepGMV-TYLCV y PepGMV-ToSLCV deberían abordar la importancia biológica de cada combinación, cómo interactúan sus respectivos genomas con el genoma de la célula hospedera durante cada una de estas infecciones mixtas y cuál es su relación con el biotipo del vector implicado en su transmisión.
Los resultados del presente estudio sobre C. annuum var. aviculare, Maax iik y maleza (p. ej., D. sexangularis y H. crispa) son relevantes porque demuestran que los hospederos silvestres podrían ser reservorios de diversidad genética para los virus asociados con el PepGMV. Esto puede ser importante para la comprensión de la dinámica epidemiológica y la evolución de los virus en las condiciones imperantes en la YPM. Esta conclusión está respaldada por este hecho: aunque las plantas de Maax ikk mostraron síntomas leves del virus cuando se recolectaron, la mayoría de las que dieron positivo a BG por PCR (86.05%) resultaron estar infectadas por el PepGMV. Es interesante notar que la evidencia genética mostrada aquí, de una baja diversidad genética dentro del PepGMV (particularmente las bajas sustituciones de nucleótidos de la proporción dN/dS en la región del core Cp) sugieren fuertemente que una presión selectiva negativa (dN/dS) está operando en la región del gen analizado. Esto es similar a los resultados que se han reportado para el TYLCV (Font et al., 2007).
Conclusiones
En el presente trabajo, casi el 90% de las plantas muestreadas fueron positivas a begomovirus, incluidas las especies parcialmente identificadas del PepGMV, el PHYVV, el ToSLCV, el TYLCV y el EuMV-YP. Predominaron las infecciones simples del PepGMV, que representaron el 82.5% de las clonas secuenciadas. Se observaron infecciones mixtas, incluyendo PepGMV-PHYVV, PepGMV-TYLCV y PepGMV-ToSLCV en diferentes especies de plantas. Las secuencias parciales proporcionan la identificación provisional de virus aislados en la YPM. La clasificación definitiva y el análisis filogenético requieren secuencias completas de los componentes ADN-A y ADN-B.
Cited literature
Ala-Poikela M, Svensson E, Rojas A, Horko T, Paulin L, Valkonen JPT and Kvarnheden A. 2005. Genetic diversity and mixed infections of begomoviruses infecting tomato, pepper and cucurbit crops in Nicaragua. Plant Pathology 54: 448-459. https://doi.org/10.1111/j.1365-3059.2005.01226.x. [ Links ]
Altschul SF, Madden TL, Schaffer AA, Zhang J, Zhang Z, Miller W and Lipman DJ. 1997. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleid Acid Research 25: 3389-3402. https://doi.org/10.1093/nar/25.17.3389. [ Links ]
Anaya-Lopez JL, Torres-Pacheco I, Gonzalez-Chavira M, Garzon-Tiznado JA and Pons-Hernandez JL. 2003. Resistance to geminivirus mixed infections in mexican wild peppers. HortScience 38: 251-255. https://doi.org/10.21273/HORTSCI.38.2.251. [ Links ]
Ascencio-Ibañez JT, Diaz-Plaza R, Mendez-Lozano J, Monsalve-Fonnegra ZI, Argüello-Astorga GR and Rivera-Bustamante RF. 1999. First Report of Tomato yellow leaf curl Geminivirus in Yucatán, México. Plant Disease 83: 1178. https://doi.org/10.1094/PDIS.1999.83.12.1178A. [ Links ]
Bosland PW and Votova EJ. 2000. Peppers: Vegetable and spice Capsicums, Crop Production Science in Horticulture 12, CABI International UK. https://doi.org/10.1079/9781845938253.0000. [ Links ]
Brown JK, Frohlich D and Rosell R. 1995. The sweetpotato or silverleaf whiteflies: biotypes of Bemisia tabaci or a species complex? Annual Review of Entomology 40: 511-534. https://www.annualreviews.org/toc/ento/40/1. [ Links ]
Brown JK, Idris AM, Ostrow KM, Goldberg N, French R and Stenger DC. 2005. Genetic and Phenotypic Variation of the Pepper golden mosaic virus complex. Phytopathology 95: 1217-1224. https://doi.org/10.1094/PHYTO-95-1217. [ Links ]
Brown JK, Idris AM, Torres-Jerez I, Banks GK and Wyatt D. 2001. The core region of the coat protein gene is highly useful for establishing the provisional identification and classification of begomoviruses. Archives of Virology 146: 1581-1598. https://doi.org/10.1007/s007050170080. [ Links ]
Carrillo-Tripp J, Lozoya-Gloria E and Rivera-Bustamante RF. 2007. Symptom remission and specific resistance of pepper plants after infection by Pepper golden mosaic virus. Phytopathology 97: 51-59. https://doi.org/10.1094/PHYTO-97-0051. [ Links ]
Cazares-Sanchez E, Ramirez-Vallejo P, Castillo-Gonzalez F, Soto-Hernandez M, Rodriguez-Gonzalez MT y Chavez-Servia JL. 2005. Capsaicinoides y preferencia de uso en diferentes morfotipos de chile (Capsicum annuum L.) del Centro-Oriente de Yucatán. Agrociencia 39: 627-638. https://www.redalyc.org/pdf/302/30239606.pdf. [ Links ]
Dellaporta SL, Wood J and Hicks JB. 1983. A Plant DNA Minipreparation: Version II. Plant Molecular Biology Reports 1: 19-21. https://link.springer.com/content/pdf/10.1007/BF02712670.pdf. [ Links ]
Duffy S and Holmes EC. 2007. Multiple introductions of the Old World Begomovirus Tomato yellow leaf curl virus into the New World. Applied Environmental Microbiology 73: 7114-7117. https://doi.or/10.1128/AEM.01150-07. [ Links ]
Echevarría-Machado I, Sanchez-Cach LA, Hernandez-Zepeda C, Rivera-Madrid R and Moreno-Valenzuela OA. 2005. A simple and efficient method for isolation of DNA in high mucilaginous plant tissues. Molecular Biotechnology 31: 129-136. https://doi.org/10.1385/MB:31:2:129. [ Links ]
Fauquet CM, Bisaro DM, Briddon RW, Brown JK, Harrison BD, Rybicki EP, Stenger DC and Stanley J. 2003. Revision of taxonomic criteria for species demarcation in the family Geminiviridae, and an updated list of begomovirus species. Archives of Virology 148: 405-421. https://doi.org/10.1007/s00705-002-0957-5. [ Links ]
Fauquet CM and Stanley J. 2005. Revising the way we conceive and name viruses below the species level: A review of geminivirus taxonomy calls for new standardized isolate descriptors. Archives of Virology 150: 2151-2179. https://doi.org/10.1007/s00705-005-0583-0. [ Links ]
Fernandes JJ, Carvalho MG, Andrade EC, Brommonschenkel SH, Fontes EPB and Zerbini FM. 2006. Biological and molecular properties of Tomato rugose mosaic virus (ToRMV), a new tomato-infecting begomovirus from Brazil. Plant Pathology 55: 513-522. https://doi.org/10.1111/j.1365-3059.2006.01395.x. [ Links ]
Fiallo-Olive E, Lett JM, Martin D P, Roumagnac P, Varsani A, Murilo-Zerbini F and Navas-Castillo J. 2021. ICTV Virus Taxonomy Profile: Geminiviridae 2021. Journal of General Virology 102: 1-2. https://doi.org/10.1099/jgv.0.001696. [ Links ]
Fiallo-Olive E, Rivera-Bustamante RF and Martinez-Zubiaur Y. 2010. First report of tobacco as a natural host of Euphorbia mosaic virus in Cuba. Plant Pathology 59: 795. https://doi.org/10.1111/j.1365-3059.2009.02238.x. [ Links ]
Font MI, Rubio L, Martínez-Culebras PV and Jordá C. 2007. Genetic structure and evolution of natural populations of viruses causing the tomato yellow leaf curl disease in Spain. Virus Research 128: 43-51. https://doi.org/10.1016/j.virusres.2007.04.003. [ Links ]
Garcia-Arenal F, Fraile A and Malpica JM. 2003. Variation and evolution of plant virus populations. International Microbiology 6: 225-232. https://doi.org/10.1007/s10123-003-0142-z. [ Links ]
Garrido-Ramirez ER and Gilbertson RL. 1998. First report of tomato mottle geminivirus infecting tomatoes in Yucatan, Mexico. Plant Disease 82: 592. https://doi.org/10.1094/PDIS.1998.82.5.592B. [ Links ]
Garzon-Tiznado JA, Acosta-Garcia G, Torres-Pacheco I, Gonzalez-Chavira M, Rivera-Bustamante RF, Maya-Hernandez V y Guevara-Gonzalez RG. (2002). Presencia de los geminivirus huasteco del chile (PHV), texano del chile variante Tamaulipas (TPV-T) y Chino del tomate (CdTV) en los estados de Guanajuato, Jalisco y San Luis Potosí, México. Revista Mexicana de Fitopatología 20: 45-52. https://www.redalyc.org/pdf/612/61220108.pdf. [ Links ]
Godinez-Hernández Y, Anaya-Lopez JL, Diaz-Plaza R, Gonzalez-Chavira M and Torres-Pacheco I. 2001. Characterization of resistance to Pepper Huasteco Geminivirus in chili peppers from Yucatán, México. HortScience 36: 139-142. https://doi.org/10.21273/HORTSCI.36.1.139. [ Links ]
Gregorio-Jorge J, Bernal-Alcocer A, Bañuelos-Hernandez B, Alpuche-Solís AG, Hernandez-Zepeda C, Moreno-Valenzuela O, Frías-Treviño G and Argüello-Astorga GR. 2010. Analysis of a new strain of Euphorbia mosaic virus with distinct replication specificity unveils a lineage of begomoviruses with short rep sequences in the DNA-B intergenic región. Virology Journal 7: 275. https://doi.org/10.1186/1743-422X-7-275. [ Links ]
Hernandez-Espinal LH, Enríquez-Verdugo I, Melgoza-Villagomez CM, Retes-Manjarrez JE, Velarde-Felix S, Linares-Flores PJ y Garzon-Tiznado JA. 2018. Análisis filogenético y distribución de begomovirus en el cultivo del chile (Capsicum annuum L.) en Sinaloa, México. Revista Fitotecnia Mexicana 41: 149-157. https://doi.org/10.35196/rfm.2018.2.149-157. [ Links ]
Hernandez-Verdugo S, Guevara-Gonzalez RG, Rivera-Bustamante RF and Oyama K. 2001. Screening wild plants of Capsicum annuum for resistance to Pepper huasteco virus (PHV): presence of viral DNA and differentiation among populations. Euphytica 122: 31-36. https://doi.org/10.1023/A:1012624830340. [ Links ]
Hernandez-Zepeda C, Idris AM, Carnevali G, Brown JK and Moreno-Valenzuela O. 2007a. Molecular characterization and experimental host range of Euphorbia mosaic virus-Yucatan Peninsula, a begomovirus species in the Squash leaf curl virus clade. Plant Patholology 56: 763-770. https://doi.org/10.1111/j.1365-3059.2007.01652.x. [ Links ]
Hernandez-Zepeda C, Idris AM, Carnevali G, Brown JK and Moreno-Valenzuela O. 2007b. Molecular characterization and phylogenetic relationships of two new bipartite begomovirus infecting malvaceous plants in Yucatan, Mexico Virus Genes 35: 369-377. https://doi.org/10.1007/s11262-007-0080-5. [ Links ]
Hernandez-Zepeda C, Idris AM, Carnevali G, Brown JK and Moreno-Valenzuela OA. 2007c. Preliminary identification and coat protein gene phylogenetic relationships of begomoviruses associated with native flora and cultivated plants from the Yucatan Peninsula of Mexico. Virus Genes 35: 825-833. https://doi.org/10.1007/s11262-007-0149-1. [ Links ]
Jones DR. 2003. Plant Viruses transmitted by whiteflies. European Journal of Plant Pathology 109: 195-219. https://doi.org/10.1023/A:1022846630513. [ Links ]
Lotrakul P, Valverde R A, De La Torre R, Sim J and Gómez A. 2000. Occurrence of a strain of Texas pepper virus in Tabasco and Habanero pepper in Costa Rica. Plant Disease 84: 168-172. https://doi.org/10.1094/PDIS.2000.84.2.168. [ Links ]
Mendez-Lozano J, Torres-Pacheco I, Fauquet CM, and Rivera-Bustamante RF. 2003. Interactions Between Geminiviruses in a naturally occurring mixture: Pepper huasteco virus and Pepper golden mosaic virus. Phytopathology 93: 270-277. https://doi.org/10.1094/PHYTO.2003.93.3.270. [ Links ]
Morales-Aguilar JJ, Rodriguez-Negrete EA, Camacho-Beltran E, Lopez-Luque CA, Leyva-Lopez NE, Jimenez-Díaz F, Voloudakis A, Santos-Cervantes ME and Mendez-Lozano J. 2019. Identification of Tomato yellow leaf curl virus, Pepper huasteco yellow vein virus and Pepper golden mosaic virus associated with pepper diseases in northern Mexico. Canadian Journal of Plant Pathology 41: 544-550. https://doi.org/10.1080/07060661.2019.1591509. [ Links ]
Morales F, Hilje L, Vallejos J, Sibaja G, Araya C and Araya R. 2005. Whiteflies as vectors of viruses in legume and vegetable mixed cropping systems in the tropical lowlands of Central America, Mexico and the Caribbean: Costa Rica, 217-221. In: Whitefly and Whitefly-borne Viruses in the Tropics: Building a Knowledge Base for Global Action. editors: Anderson P. K. and Morales F. J. CIAT publication No. 34, Cali, CO. http://ciat-library.ciat.cgiar.org/Articulos_CIAT/9586940748.pdf. [ Links ]
Rausch FF, de Albuquerque LC, de Brito GL, Boiteux LS, de Ávila AC and Inoue-Nagata K. 2008. Diversity and prevalence of Brazilian bipartite begomovirus species associated to tomatoes. Virus Genes 36: 251-258. https://doi.org/10.1007/s11262-007-0184-y. [ Links ]
Polston JE and Anderson PK. 1997. The Emergence of Whitefly-Transmitted Geminiviruses in Tomato in the Western Hemisphere. Plant Disease 81: 1358. https://doi.org/10.1094/PDIS.1997.81.12.1358. [ Links ]
Rodelo-Urrego M, García-Arenal F and Pagan I. 2015. The effect of ecosystem biodiversity on virus genetic diversity depends on virus species: A study of chiltepin-infecting begomoviruses in Mexico. Virus Evolution 1:1-13. https://doi.org/10.1093/ve/vev004. [ Links ]
Rodriguez-Negrete EA, Morales-Aguilar JJ, Dominguez-Duran G, Torres-Devora G, Camacho-Beltran E, Leyva-Lopez NE, Voloudakis AE, Bejarano ER and Mendez-Lozano J. 2019. High-throughput sequencing reveals differential begomovirus species diversity in non-cultivated plants in Northern-Pacific Mexico. Viruses 11: 594. https://doi.org/10.3390/v11070594. [ Links ]
Seal SE, vandenBosch F and Jeger MJ. 2006. Factors influencing begomovirus evolution and their increasing global significance: implications for sustainable control. Critical Reviews in Plant Sciences 25: 23-46. https://doi.org/10.1080/07352680500365257. [ Links ]
Tamura K, Stecher G and Kumar S. 2021. MEGA 11: Molecular Evolutionary Genetics Analysis (MEGA) Software version 11. Molecular Biology and Evolution 38: 3022-3027. https://doi.org/10.1093/molbev/msab120. [ Links ]
Torres-Herrera SI, Romero-Osorio A, Moreno-Valenzuela O, Pastor-Palacios G, Cardenas-Conejo Y, Ramirez-Prado JH, Riego-Ruiz L, Minero-Garcia Y, Ambriz-Granados S and Argüello-Astorga GR. 2019. A lineage of begomoviruses encode REP and AC4 proteins of enigmatic ancestry: hints on the evolution of geminiviruses in the New World. Viruses 11: 644. https://doi.org/10.3390/v11070644. [ Links ]
Torres-Pacheco I, Garzon-Tiznado JA, Brown JK, Becerra-Flora A and Rivera-Bustamante RF. 1996. Detection and distribution of geminiviruses in Mexico and the Southern United States. Phytopathology 86: 1186-1192. https://doi.org/10.1094/phyto-86-1186. [ Links ]
Villanueva-Alonzo HJ, Us-Camas RY, Lopez-Ochoa LA, Robertson D, Guerra-Peraza O, Minero-Garcia Y and Moreno-Valenzuela OA. 2013. A new virus-induced gene silencing vector based on Euphorbia mosaic virus-Yucatan peninsula for NPR1 silencing in Nicotiana benthamiana and Capsicum annuum var. Anaheim. Biotechnology Letters 35: 811-823. https://doi.org/10.1007/s10529-013-1146-1. [ Links ]
Recibido: 16 de Junio de 2022; Aprobado: 24 de Agosto de 2022











 texto en
texto en