Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista mexicana de fitopatología
versão On-line ISSN 2007-8080versão impressa ISSN 0185-3309
Rev. mex. fitopatol vol.40 no.3 Texcoco Set. 2022 Epub 14-Nov-2022
https://doi.org/10.18781/r.mex.fit.2203-3
Artículos científicos
Análisis genético de la resistencia a fungicidas en Fusarium oxysporum asociado a Vanilla planifolia
1 Instituto de Biotecnología y Ecología Aplicada, Universidad Veracruzana, Campus para la Cultura, las Artes y el Deporte Av. de las Culturas Veracruzanas No. 101, Col. Emiliano Zapata, CP 91090, Xalapa, Veracruz, México;
2 Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias CIR Golfo Centro, Campo Experimental Ixtacuaco, Km 4.5 carretera federal Martínez de la Torre - Tlapacoyan, Cong. Rojo Gómez, CP 93650, Tlapacoyan, Veracruz, México;
3 Laboratorio de Genética e Interacciones Planta Microorganismos, Facultad de Ciencias Agrícolas, Universidad Veracruzana, Lomas del Estadio s/n, CP 91000, Xalapa, Veracruz, México.
Vanilla planifolia es atacada por F. oxysporum f. sp. vanillae. Dado que no existen genotipos resistentes disponibles para los productores, éstos optan por el uso de fungicidas sintéticos para su control; sin embargo, no hay información sobre la respuesta del patógeno a estos productos comerciales. El objetivo del trabajo fue analizar la respuesta de cepas de F. oxysporum, patógenas (nueve) y no patógenas (tres) de V. planifolia, a tres bencimidazoles (tiofanato metílico, benomilo y carbendazim) y a clorotalonil, así como las bases genéticas de la respuesta al fungicida mediante el estudio de las proteínas blanco de los mismos, la β-tubulina y la GPDH, respectivamente. Se observó que las 12 cepas estudiadas fueron susceptibles a los fungicidas benomilo y carbendazim, mientras que 11 de ellas fueron resistentes al tiofanato metílico y nueve al clorotalonil. No se encontraron diferencias entre el carácter patogénico o la procedencia de las cepas en relación con la susceptibilidad a los fungicidas. Se encontró que la resistencia al tiofanato metílico no es otorgada por una mutación puntual de la secuencia codificante de la β-tubulina, como se ha hipotetizado anteriormente; al parecer, pudiera estar más relacionada con la ausencia de motivos de la proteína. No se observó relación entre variación del gen GPDH con la resistencia o susceptibilidad de las cepas.
Palabras clave: Vainilla; pudrición de raíz; pudrición de tallo; GPDH; β-tubulina
Vanilla planifolia is attacked by the fungus F. oxysporum f. sp. vanillae. Since there are no resistant genotypes available to producers, they opt for the use of synthetic fungicides to control the disease, however, there is no information on a possible resistance of the pathogen to these commercial fungicides. The goal of the work was to analyze the response of F. oxysporum strains, pathogenic (nine) and non-pathogenic (three) of V. planifolia, to three fungicides of the benzimidazole family and to chlorothalonil, as well as the genetic bases by studying the target proteins of these active ingredients, β-tubulin and GPDH. It was observed that the 12 strains studied were susceptible to the fungicides benomyl and carbendazim, while 11 of them were resistant to thiophanate methyl and nine to chlorothalonil. No relationship was found between the pathogenic character or the origin of the strains and the susceptibility to fungicides. It was found that resistance to thiophanate methyl is not conferred by a point mutation of the β-tubulin coding sequence, as previously hypothesized; apparently, it could be more related to the absence of protein motifs. No relationship was observed between variation in the gene GPDH with the resistance or susceptibility of the strains.
Keywords: Vanilla; root rot; stem rot; GPDH; β-tubulin
La vainilla (Vanilla planifolia) es una orquídea de alto valor comercial debido a que produce el compuesto vainillina. La vainillina es buscada por varias industrias por su aroma y sabor. Este cultivo enfrenta una importante amenaza por la enfermedad conocida como “pudrición de raíz y tallo” causada por el ascomicete Fusarium oxysporum f. sp. vanillae (Pinaria et al., 2010; Adame-García et al., 2015). Se ha demostrado que la vainilla está asociada a múltiples genotipos de F. oxysporum, sin necesidad de que todos se comporten como patógenos (Casillas-Isiordia et al., 2017). Lo anterior, sucede también con otras especies vegetales, lo que ha llevado a subdividirlos en patogénicos (formae speciales) y endófitos benéficos, buscando de esta forma evitar perder valiosa información genética que ayude a entender la naturaleza del patógeno (Lecomte et al., 2016).
Al no existir genotipos de vainilla resistentes al alcance de los productores mexicanos, éstos buscan alternativas para el control de la enfermedad, siendo el control químico mediante fungicidas el principal método utilizado, sobre todo en la principal región productora de Papantla, Veracruz (Hernández-Hernández, 2011), zona considerada como el centro de origen, domesticación y distribución de la vainilla (Lubinsky et al., 2008). El control químico de la fusariosis en la vainilla se lleva a cabo principalmente con fungicidas de la familia de los bencimidazoles (Hernández-Hernández, 2019). Estos fungicidas inciden directamente en la degradación de la β-tubulina de los microtúbulos, inhibiendo de esta forma la división celular fúngica (Zhang et al., 2015). Otros sugeridos para el control de F. oxysporum f. sp. vanillae son los fungicidas de amplio espectro, como el clorotalonil, perteneciente a la familia de los carbamatos, los cuales tienen múltiples sitios de acción en las células del hongo, entre ellas la enzima glicerol-3-fosfatodeshidrogenasa (GPDH) involucrada en la glucólisis, que al inactivarse ocasiona la muerte del hongo (Long y Siegel, 1975; Baćmaga et al., 2018).
Sin embargo, en el caso de los bencimidazoles, se han reportado numerosos casos de generación de resistencia en diversas formae speciales de F. oxysporum (Arie, 2019), la cual ha sido previamente asociada a una mutación puntual en la posición 200 de la secuencia codificante de la proteína β-tubulina (Petkar et al., 2017), por tal razón es de suma importancia determinar la susceptibilidad real de aislados de F. oxysporum a estos fungicidas, antes de ser utilizados para el control de estos patógenos. Con base en lo anterior, el objetivo del presente trabajo fue evaluar el efecto de tres fungicidas de la familia de los bencimidazoles y el clorotalonil en cepas de F. oxysporum, patógenas y no patógenas de Vanilla planifolia, además de determinar si la resistencia en cepas de esta forma specialis está relacionada con mutaciones puntuales en las secuencias de las proteínas blanco, β-tubulina y glicerol-3-fosfatodeshidrogenasa.
Materiales y métodos
Cepas de F. oxysporum. Para el presente estudio se utilizaron 12 cepas de F. oxysporum aisladas de vainilla: nueve patógenas de V. planifolia y tres no patógenas (Cuadro 1). Con excepción de las cepas BC1, Chic3 y JF9, la identificación (morfológica y molecular) y capacidad patogénica de las cepas restantes se encuentra publicada en los trabajos de Adame-García et al. (2015), Casillas-Isiordia et al. (2017) y Flores-de la Rosa et al. (2018). La evaluación morfológica, genética y capacidad patogénica en V. planifolia de las cepas BC1, Chic3 y JF9 fueron determinadas siguiendo los protocolos descritos en las referencias antes mencionadas (datos no mostrados). Todas las cepas están resguardadas en el Laboratorio de Genética e Interacciones Planta Microorganismos de la Facultad de Ciencias Agrícolas de la Universidad Veracruzana. Las plantas de vainilla de donde se obtuvieron los hongos fueron colectadas de acahuales, selva o plantaciones de vainilla, sin manejo con fungicidas químicos.
Cuadro 1 Procedencia y susceptibilidad a bencimidazoles y clorotalonil, de cepas de Fusarium oxyspo rum (patógenas y no patógenas) aisladas de Vanilla planifolia.
| ID Cepa | Procedencia | Carácter | Fuente | Benomilo | Carbendazim | Tiofanato metílico | Clorotalonil |
|---|---|---|---|---|---|---|---|
| H5 | Veracruz | Patógeno | 1 | S | S | R | R |
| H9 | Veracruz | Patógeno | 1 | S | S | R | R |
| JF5 | Veracruz | Patógeno | 3 | S | S | R | R |
| A3 | Veracruz | Patógeno | 3 | S | S | R | R |
| V6M2 | Veracruz | Patógeno | 3 | S | S | R | R |
| JF9 | Veracruz | No Patógeno | 4 | S | S | S | S |
| N12 | Nayarit | Patógeno | 2 | S | S | R | R |
| N3 | Nayarit | Patógeno | 2 | S | S | R | R |
| BC1 | Nayarit | No Patógeno | 4 | S | S | R | R |
| Chic 2 | Oaxaca | Patógeno | 3 | S | S | R | R |
| Chic 6 | Oaxaca | No Patógeno | 3 | S | S | R | S |
| Chic 3 | Oaxaca | No Patógeno | 4 | S | S | R | S |
S= susceptible; R= resistente. Fuente: 1) Adame-García et al. 2015; 2) Casillas-Isiordia et al. 2017; 3) Flores de la Rosa et al. 2018; 4) este estudio.
Preparación de suspensión de esporas. Las cepas se activaron y posteriormente se subcultivaron en PDA durante 10 días a 25 °C en incubadora microbiológica (Mca. Felisa). Concluido el periodo de incubación se adicionaron 5 mL de agua destilada estéril con tres gotas de Tween 20 sobre el medio de cultivo para transferir una muestra de suspensión de micelio y conidias a tubos cónicos de 15 mL, para centrifugación a 1000 rpm durante 2 min. A partir del sobrenadante se prepararon suspensiones de conidias de 1x109 UFC por dilución con agua destilada esterilizada. Para el conteo de esporas se utilizó una cámara de Neubauer y observación microscópica.
Pruebas de resistencia a fungicidas. Se evaluó la susceptibilidad de las 12 cepas de F. oxysporum a cuatro fungicidas comerciales: tiofanato metílico (Cercobin M®), benomilo (Promyl®), carbendazim (Prozycar®) y clorotalonil (Talonil 75®). Las pruebas se llevaron a cabo en cajas de Petri mediante la técnica de placa-orificio, para ello, se prepararon cultivos fúngicos con 100 μL de la suspensión de esporas 1x109 UFC y 25 mL de PDA. Para ello, la suspensión de esporas se colocó directamente en la base de la caja de Petri y posteriormente, se vaciaron encima ⁓ 25 mL de medio de cultivo a una temperatura de ⁓ 40 °C. Las conidias fueron distribuidas en el agar con movimientos circulares a favor y en contra de las manecillas del reloj antes de la solidificación del medio de cultivo. Después de la solidificación del agar, con una varilla metálica hueca estéril (sacabocado) se removieron cuatro cilindros de agar distribuidos equidistantemente en la superficie de cada placa, donde se probaron tres concentraciones: una de acuerdo a la recomendación de uso del producto (1% P:V), una más alta (2.5% P:V) y una más diluida (0.5%), por cada tipo de fungicida. Para ello, en cada orificio se colocaron 10 μL de la solución del fungicida a la concentración evaluada. Como testigo, en uno de los orificios se colocaron 10 μL de agua destilada estéril. Esta prueba se repitió cinco veces por cada fungicida y por cada cepa de F. oxysporum en estudio. Los cultivos se incubaron durante cinco días a 25 - 26 ºC en incubadora microbiológica (Mca. Felisa). El diámetro del halo de inhibición de cada tratamiento se midió con un vernier digital. Las cepas resistentes fueron aquellas en las que no se observó un halo de inhibición alrededor del orificio donde se depositó el fungicida.
Determinación de la relación entre el carácter patogénico/no patogénico de la cepa de F. oxysporum, con la respuesta a los fungicidas. Para determinar la posible relación entre el carácter patogénico y no patogénico con la susceptibilidad o resistencia a los fungicidas, se aplicó un Modelo Lineal Generalizado (Dist. Binomial, Función de liga Logit) con base en una matriz de datos binarios (0 resistente, 1 susceptible). Asimismo, se realizó un análisis de conglomerados jerárquico y de ligamiento completo, empleando la distancia euclidiana. Los análisis se desarrollaron en el software para análisis estadísticos R 2.12.2.
Extracción de ADN. Para la extracción de ADN se empleó un protocolo basado en el uso de CTAB propuesto por Adame-García et al. (2016a) a partir de ⁓ 50 mg de micelio de cada aislado de Fusarium oxysporum de 10 a 14 días de crecimiento en medio PDA. El ADN se cuantificó con un espectrofotómetro UV-Visible compacto (Thermo Scientific™ NanoDrop™ One).
Diseño de iniciadores para el gen GPDH. Para la amplificación del gen GPDH fue necesario diseñar iniciadores por no encontrar reportes en la literatura. El diseño de los iniciadores consistió en buscar en NCBI, la secuencia de aminoácidos de la proteína GPDH entre las reportadas para F. oxysporum f. sp. lycopersici, posteriormente, convertir la secuencia en nucleótidos utilizando tBLASTn y posteriormente, se buscó la secuencia resultante en los genomas reportados de F. oxysporum. Las secuencias encontradas se alinearon en el software Mega (Kumar et al., 2018) y mediante los softwares Primer3 Plus (Untergasser et al., 2007) y Primer-BLAST que arrojaron un total 15 parejas de posibles iniciadores (datos no mostrados). Cada par de iniciadores arrojados se probaron mediante la realización de PCR in silico en la plataforma del NCBI. Se eligieron los pares de iniciadores P4f (5’-AAAGTCCACGCACTCCTCTG-3’) y P4r (5´- AGAATGGCGACGGAAACCTT-3’) generados con el software Primer3 Plus, cuya secuencia resultante abarcó desde la base 506 hasta la 1146 del tamaño total del gen, y los iniciadores P13f (5’-CCTGCACAAGTCAACAAGCC-3’) y P13r (5’-GCGCAACTGTTGTCAACCAT-3’) generados con el software Primers BLAST, que abarcaron desde la base 766 hasta la 1710.
Amplificación y secuenciación de los genes de β-tubulina y GPDH. La amplificación del gen β-tubulina se realizó con el juego de iniciadores Tub1 (5’-AACATGCGTGAGATTGTAAGT-3’) y Tub22 (5’-TCTGGATGTTGTTGGGAATCC-3’) (Nosratabadi et al., 2018), con las siguientes condiciones térmicas: desnaturalización inicial a 94 °C durante 5 min; 35 ciclos de desnaturalización por 30 s a 94 °C, alineamiento de 45 s a 60 °C, extensión a 80 s a 72 °C; y extensión final a 72 °C por 7 min. Para la amplificación del gen GPDH se utilizaron los pares de iniciaadores P13f - P13r y P4f - P4r diseñados en este estudio, bajo las siguientes condiciones térmicas: desnaturalización inicial a 94 °C por 2 min; 35 ciclos de desnaturalización a 94 °C por 2 min, alineamiento por 60 °C, extensión a 72 °C por 60 s; extensión final de 72 °C por 7 min. Los productos de amplificación fueron analizados por electroforesis horizontal en gel de agarosa al 1.8 % y tinción con bromuro de etidio, donde se corroboró que los productos de amplificación de los genes β-tubulina y GPDH correspondieran a 950 pb y 450 pb, respectivamente. Después de la electroforesis, los productos de amplificación se purificaron siguiendo el protocolo del kit Wizard® SV Gel and PCR Clean-Up System y se enviaron a secuenciar al Instituto de Biotecnología (UNAM).
Análisis bioinformático. Se editaron las secuencias mediante el software MEGA X y se obtuvieron las secuencias consenso de cada gen a partir de las secuencias generadas con los iniciadores “sentido” y “antisentido”, para cada cepa en estudio. Cada secuencia resultante se analizó mediante el algoritmo BLAST. Con el fin de hacer los diseños en 3D de la molécula de la proteína β-tubulina e identificar los motivos, las secuencias se alinearon y compararon en el software BioEdit. Para la búsqueda de dominios conservados en el gen β-tubulina en cepas susceptibles y resistentes a tiofanato metílico, las secuencias se tradujeron a proteínas y se alinearon con el software MEGA X (Kumar et al., 2018), para tal propósito se incluyeron las secuencias de F. oxysporum f. sp. niveum Fon12105 (acceso KX369333) y Fon53 (acceso KX369332) usadas en el estudio de Petkar et al. (2017). En el alineamiento se identificaron de forma manual las mutaciones y se compararon las secuencias de tres cepas de F. oxysporum resistentes versus la cepa susceptible Fon12105. Para la identificación de motivos proteícos se utilizó el software EzMol, en tanto que, para la modelación 3D de las moléculas se empleó el software Phyre2.
Resultados
Ensayos de resistencia a fungicidas. De acuerdo con la respuesta ante los fungicidas probados en cepas de F. oxysporum aisladas de vainilla, el análisis de conglomerados estableció tres grupos: A) cepas susceptibles a benomilo y carbendazim, pero resistentes a tiofanato metílico y clorotalonil; B) cepas susceptibles a benomilo, carbendazim y clorotalonil, y resistentes a tiofanato metílico; y C) cepas susceptibles a los cuatro fungicidas (Figura 1). En el grupo A se encontraron nueve cepas provenientes de Veracruz, Nayarit y Oaxaca; en el grupo B, dos cepas de Oaxaca; y en el grupo C, una cepa de Veracruz (Cuadro 1). No se encontraron diferencias entre el carácter patogénico o la procedencia de las cepas en relación con la susceptibilidad a los fungicidas (P > 0.05), según las pruebas del modelo lineal generalizado susceptibilidad ~ patogenicidad + procedencia. Es importante destacar que no se observó diferencia en la respuesta de los hongos por las diferentes concentraciones utilizadas del fungicida.

Figura 1 Agrupamiento de las cepas de F. oxysporum según su susceptibilidad a bencimidazoles y clorotalonil. A) susceptibilidad a benomilo y carbendazim, resistentes a tiofanato metílico y clorotalonil; B) susceptibilidad a benomilo, carbendazim y clorotalonil, resistentes a tiofanato metílico; C) susceptible a todos los fungicidas.
Amplificación de los genes β-tubulina y GPDH. En las 12 cepas evaluadas, la amplificación produjo un fragmento de 950 pb para el gen β-tubulina utilizando los iniciadores previamente reportados en la literatura para este gen y de 450 pb para el gen GPDH usando los iniciadores P4F (Figura 3). No se obtuvo amplificación con los iniciadores P13 para el gen GPDH.
Análisis bioinformático. El análisis BLAST confirmó que todas las secuencias obtenidas correspondieron a los genes β-tubulina y GPDH, respectivamente (Cuadro 2). Con el fin de encontrar diferencias en el gen β-tubulina entre las cepas susceptibles y resistentes a los fungicidas tiofanato metílico y clorotalonil se emplearon las secuencias de las cepas patógenas V6M2 y N3 y las cepas no patógenas Chic6 y BC1. El análisis de alineamiento evidenció que la cepa susceptible al tiofanato metílico Fon12105 (Petkar et al., 2017) y la cepa resistente Chic6, compartieron la base timina en la posición 200, mientras que en el resto de las secuencias se encontró adenina (Figura 2). Para el análisis del gen GPDH se consideraron seis secuencias de cepas resistentes a clorotalonil (N3, JF5, A3, V6M2, Chic2 y N12) y una cepa susceptible (JF9), en éstas no se encontraron mutaciones del gen GPDH, lo que lleva a proponer que la respuesta de susceptibilidad o resistencia no está relacionada con variaciones en la secuencia de la región analizada de este gen.
Cuadro 2 Secuencias de cepas de Fusarium oxysporum registradas en la base de datos del GenBank-NCBI, que produjeron alineamientos significativos mediante BLAST, a las secuencias de los genes β-tubulina y GPDH de cepas de Fusarium oxysporum aisladas de Vanilla planifolia.
| Secuencias de F. oxysporum en GenBank-NCBI | Porcentaje de similitud | ID cepa de F. oxysporum |
|---|---|---|
| F. oxysporum f. sp. vasinfectum strain ATCC 16612 beta-tubulin (BTU) gene, complete cds (Clave de acceso KT323825.1) | 98.72 | BC1 |
| F. oxysporum isolate TX85 beta-tubulin (BTU) gene, complete cds (Clave de acceso MH827996.1) | 97.85 | V6M2 |
| F. oxysporum isolate MS15 beta-tubulin (BTU) gene, complete cds (Clave de acceso MH827993.1) | 96.91 | N3 |
| F. solani strain MICMW-30.1 beta-tubulin gene, partial cds (Clave de acceso KX912242.1) | 94.34 | Chic6 |
| F. oxysporum f. sp. lycopersici 4287 glycerol-3-phosphate dehydrogenase Mrna (Clave de acceso XM_018394006.1) | 97.84 | N3 |
| F. oxysporum f. sp. lycopersici 4287 glycerol-3-phosphate dehydrogenase mRNA (Clave de acceso XM_018394006.1) | 95.60 | JF5 |
| F. oxysporum f. sp. lycopersici 4287 glycerol-3-phosphate dehydrogenase mRNA (Clave de acceso XM_018394006.1) | 95.32 | JF9 |
| F. oxysporum f. sp. lycopersici 4287 glycerol-3-phosphate dehydrogenase mRNA (Clave de acceso XM_018394006.1) | 99.29 | A3 |
| F. oxysporum f. sp. lycopersici 4287 glycerol-3-phosphate dehydrogenase mRNA (Clave de acceso XM_018394006.1) | 99.64 | V6M2 |
| F. oxysporum f. sp. lycopersici 4287 glycerol-3-phosphate dehydrogenase mRNA (Clave de acceso XM_018394006.1) | 100 | Chic2 |
| F. oxysporum f. sp. lycopersici 4287 glycerol-3-phosphate dehydrogenase mRNA (Clave de acceso XM_018394006.1) | 99.82 | N12 |
Identificación de motivos. Se encontró que las secuencias de la proteína β-tubulina de las cepas N3, BC1 y Fon53 resistentes a tiofanato metílico y la cepa susceptible Fon 12105, comparten los motivos 237537 y 335235. La cepa N3 se diferencia por presentar los motivos 236152 y 279881, en tanto que la cepa Fon 12105 fue la única en presentar el motivo 237844 (Figura 3).
Estructuras 3D de las proteínas blanco. La estructura tridimensional de la proteína β-tubulina mostró diferencias entre la cepa susceptible y las resistentes al tiofanato metílico, específicamente se encontró que las cepas resistentes V6M2, BC1 y Fon53 tienen el arreglo de aminoácidos histidina-glicina-serina, en tanto que, en la misma posición, la cepa Fon12105 tiene cisteína-cisteína-prolina (Figura 4a). Las proteínas GPDH de las cepas JF5 (resistente a clorotalonil) y JF9 (susceptible a clorotalonil) mostraron una estructura diferente a las proteínas de las cepas resistentes A3, V6M2, Chic2 y N12 (Figura 4b).
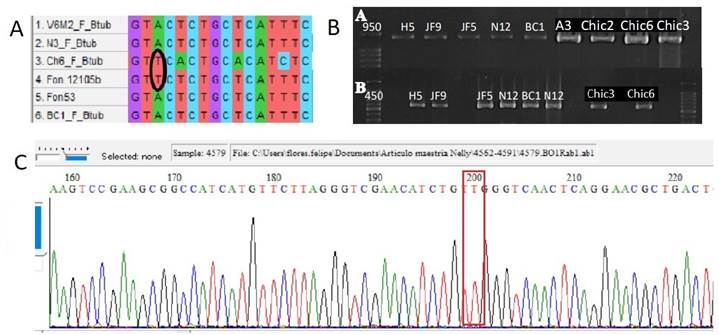
Figura 2 A) Fragmento del alineamiento de las secuencias del gen β-tubulina donde se observa en la posición 200 (enmarcada en el rectángulo) que las cepas Ch6 (Chic6, este estudio) y Fon12105b (F. oxysporum f. sp. niveum, Petkar et al., 2017) comparten el nucleótido timina, siendo Chic6 resistente al tiofanato metílico, mientras que Fon12105b es susceptible. B) Amplificación de productos de PCR en gel de agarosa del gen β-tubulina (A) y gen GPDH (B) de algunas cepas de Fusarium en estudio. C) Electroferograma de la secuencia del gen β-tubulina de la cepa Chic6, resistente al tiofanato metílico, donde se muestra la timina en la posición 200.

Figura 3 Búsqueda de dominios conservados en el gen β-tubulina en cepas de F. oxysporum, susceptibles y resistentes a tiofanato metílico, aisladas de V. planifolia. Se encontró que el motivo 237844 en la cepa Fon12105 susceptible (Petkar et al., 2017) no está presente en las cepas resistentes Fon53 (Petkar et al., 2017), N3 y BC1.
Discusión
La limitada variabilidad genética que tiene la vainilla (V. planifolia) la hace altamente vulnerable ante diversos patógenos (Koyyappurath et al., 2015). El hongo F. oxysporum f. sp. vanillae, agente causal de la enfermedad de pudrición de raíz y tallo, representa un importante problema fitosanitario para los productores de vainilla a nivel mundial (Pinaria et al., 2010; Adame-García et al., 2016b; Casillas-Isiordia et al., 2017).

Figura 4 Modelos 3D de las proteínas blanco de: A) tiofanato metílico (1, cepa V6M2; 2, cepa Fon 53; 3, cepa BC1; 4, cepa Fon12105); B) clorotalonil (1, cepa JF5; 2, cepa JF9; 3, cepa A3; 4, cepa V6M2; 5, cepa Chic2; 6, cepa N12). Se observan diferencias estructurales en ambos casos asociadas a la resistencia a los fungicidas.
Para combatir esta enfermedad se tienen avances en la investigación o la aplicación de varios tipos de control: genético, cultural, biológico y químico. Este último es el más utilizado por los productores, ya que su costo es bajo, efectivo y de fácil acceso; no obstante, el uso excesivo de los fungicidas puede provocar que los hongos generen resistencia a los principios activos de estos, como ha sucedido en patógenos de otros cultivos (Hobbelen et al., 2011; Steinberg y Gurr, 2020). Algunas especies del género Fusarium, como F. fujikuroi (Chen et al., 2014), F. graminearum (Duan et al., 2014; Zhou et al., 2016; Zhu et al., 2018), F. asiaticum (Suga et al., 2011), entre otras, han sido reportadas como resistentes a algunos fungicidas como el benomilo, el tiofanato metílico y el carbendazim. En el caso de F. oxysporum f. sp. vanillae no existen reportes previos acerca de su resistencia o suceptibilidad a fungicidas.
En el cultivo de la vainilla, los fungicidas más utilizados son de la familia de los bencimidazoles (Hernández-Hernández, 2019), como el benomilo, carbendazim y el tiofanato metílico, que tienen actividad sistémica; así mismo, fungicidas de contacto multisitio de amplio espectro, como el clorotalonil, son ampliamente recomendados. Los bencimidazoles en contacto con el hongo inhiben el ensamblaje de los microtúbulos mediante la degradación de la β-tubulina, impidiendo la generación del huso mitótico, interrumpiéndose así la división celular (Chung et al., 2009). En tanto que, el clorotalonil inhibe la función de la enzima glicerol-3-fosfato-deshidrogenasa (GPDH) que forma parte de la ruta de la glucólisis del hongo (Long y Siegel, 1975).
El uso de los bencimidazoles está ampliamente extendido en la producción agrícola, aunque, en la lista 2019 del FRAC se describe la resistencia a los bencimidazoles como una característica común entre las especies fúngicas, entre ellas algunas formas especiales de F. oxysporum, por ejemplo, lilii, gladioli (Chung et al., 2009), lycopersici (Amini y Sidovich, 2010) y niveum (Petkar et al., 2017). En este estudio, todas las cepas de F. oxysporum, patógenas y no patógenas de vainilla, presentaron susceptibilidad a benomilo y a carbendazim, en tanto que, nueve cepas fueron resistentes a tiofanato metílico, sin distinción de su carácter patogénico.
Petkar et al. (2017) plantearon que el cambio de timina (T) por adenina (A) en la posición 200 del gen β-tubulina codifica dos aminoácidos distintos, fenilalanina (TTC) y tirosina (TAC), lo que daría la cualidad de susceptibilidad o resistencia al tiofanato metílico, respectivamente. Partiendo del hecho de que la comparación de las secuencias de las cepas de F. oxysporum, patógenas y no patógenas de vainilla, resistentes al tiofanato metílico, contra la secuencia de la cepa susceptible (Fon12105) y la resistente (Fon53) de F. oxysporum f. sp. niveum empleadas en el estudio de Petkar et al. (2017) mostró que las cepas Fon12105 (cepa susceptible) y Chic6 (cepa resistente de F. oxysporum f. sp. vanillae) tienen en la posición 200 una timina (T), y que el resto de las cepas resistentes poseen una adenina (A), se descarta la hipótesis de que la resistencia a este fungicida está explicada por un cambio en esta posición específica de la secuencia codificante de este gen. Con anterioridad se ha propuesto que en Fusarium fujikuroi (Chen et al., 2014), así como en otras formae speciales de F. oxysporum (Chung et al., 2009; Amini y Sidovich, 2010), se encuentra la fenilalanina en la posición 200 del gen, independiente a la resistencia o susceptibilidad hacia el tiofanato metílico. Otros estudios confieren la resistencia a los bencimidazoles en Sclerotinia homoeocarpa a la mutación de fenilalanina a tirosina (Dowling et al., 2017), así como, que la mutación inversa (tirosina-fenilalanina) es la responsable de la resistencia en Colletotrichum musae (dos Santos-Vieira et al., 2017).
Debido a la contraposición encontrada, respecto a la mutación en el codón 200 como la responsable de la resistencia o susceptibilidad del hongo al fungicida, el estudio se condujo hacía la búsqueda de motivos conservados en las secuencias del gen, encontrándose que el motivo 237844 (Pfam) únicamente se presentó en la cepa susceptible, lo que sugiere que en la resistencia al tiofanato metílico, el sitio de unión y acción del fungicida podría no incluir al residuo del codón, como lo propuso Vela-Corsía et al. (2018) al estudiar el acoplamiento entre el sitio de acción de los bencimidazoles con la proteína β-tubulina. No se encontró mayor información biológica para el motivo 237844 (Pfam).
Los bencimidazoles interfieren en ciertos sitios de la polimerización de los monómeros de α1-tubulina, α2-tubulina y β2-tubulina, sin embargo, una vez realizada la polimerización de los microtúbulos, ésta ya no se revierte (Zhou et al., 2016). La inhibición de la polimerización depende del principio activo, así, el benomilo y carbendazim actúa en las subunidades α1/β2 y α2/β2, mientras que en el caso del tiabendazol se ha observado una menor capacidad de inhibir α2/β2 (Zhou et al., 2016), lo que estaría relacionado con una mayor probabilidad de resistencia. Para el tiofanato metílico no se tienen datos sobre cómo afecta estos procesos de polimerización, siendo posible que se comporte de manera similar al tiabendazol. Son necesarios más estudios acerca del modo de acción del tiofanato metílico para determinar si es éste el mecanismo de resistencia en F. oxysporum f. sp. vanillae.
El clorotalonil inhibe la actividad enzimática fúngica de deshidrogenasas, catalasas y fosfatasas ácidas (Baćmaga et al., 2018); de manera específica, inhibe la actividad de la enzima GPDH (Long y Siegel, 1975). Los altos niveles de resistencia al clorotalonil encontrados en cepas patógenas y no patógenas de F. oxysporum asociadas a la raíz de V. planifolia contradice a las anotaciones del FRAC (2019), que considera al clorotalonil como parte de un grupo con bajo nivel de riesgo en la generación de resistencia por su naturaleza multisitio de acción. No obstante, este fungicida ha mostrado alta efectividad contra otras formae speciales de F. oxysporum como F. oxysporum f. sp. lentis (Dahal y Shrestha, 2018).
Si bien, las cepas de F. oxysporum asociado a vainilla no presentaron variaciones en el gen GPDH que expliquen la diferencia en la respuesta al clorotalonil, hay indicios dentro de los motivos conservados de la enzima que podrían estar asociados a la susceptibilidad. Para determinar el mecanismo de resistencia es necesario enfocar investigaciones que involucren de manera integral otros sistemas enzimáticos blanco reportados para este y otros fungicidas, toda vez que las cepas de Fusarium asociadas a vainilla han revelado alta variación en la actividad enzimática (Adame-García et al., 2011) y alta variabilidad genética (Flores-de la Rosa et al., 2018), que explica su respuesta variable ante los fungicidas probados. Para otros fungicidas multisitio, como el mancozeb, se ha documentado una reducción sustancial de la sensibilidad en muchas especies de patógenos, la cual ha sido asociada con los genes que involucran la formación de la maquinaria transcripcional, la regulación del pH celular y los transportadores de múltiples fármacos (Yang et al. 2019).
Conclusiones
Las cepas de F. oxysporum, patógenas y no patógenas de V. planifolia, fueron altamente susceptibles a los fungicidas benomilo y carbendazim, y altamente resistentes a tiofanato metílico y clorotalonil. La mutación puntual de adenina a timina en el codón 200 del gen β-tubulina, que codifican fenilalanina/tirosina, respectivamente, no es la responsable de la resistencia al tiofanato metílico, como ha sido propuesto por otros autores, no obstante, se encontraron indicios de que la presencia de motivos conservados pueda estar relacionada con la susceptibilidad del hongo a este fungicida. No se encontró evidencia de que la variabilidad en la secuencia analizada del gen GPDH esté relacionada con la respuesta de susceptibilidad o resistencia al clorotalonil.
Agradecimientos
N. A. González-Oviedo agradece al CONACYT por la beca número 629405 para estudios de maestría.
REFERENCIAS
Adame-García J, Trigos-Landa A, Iglesias-Andreu LG, Flores-Estevez N and Luna-Rodríguez M. 2011. Isozymic and pathogenic variations of Fusarium spp. associated with vanilla stem and root rotting. Tropical and Subtropical Agroecosystems 13:299-306. https://www.revista.ccba.uady.mx/ojs/index.php/TSA/article/view/1330/663 [ Links ]
Adame-García J, Rodríguez-Guerra R, Iglesias-Andreu LG, Ramos-Prado JM and Luna-Rodríguez M. 2015. Molecular identification and pathogenic variation of Fusarium species isolated from Vanilla planifolia in Papantla Mexico. Botanical Sciences 93:669-678. https://doi.org/10.17129/botsci.142 [ Links ]
Adame-García J, Flores-de la Rosa FR, Ricaño-Rodríguez J and Luna-Rodríguez M. 2016a. Adequacy of a protocol for amplification of EF-1α gene of Fusarium oxysporum f. sp. vanillae. ARPN Journal of Agricultural and Biological Science 11:236-241. http://www.arpnjournals.org/jabs/research_papers/rp_2016/jabs_0616_804.pdf [ Links ]
Adame-García J, Luna-Rodríguez M and Iglesias-Andreu LG. 2016b. Vanilla rhizobacteria as antagonists against Fusarium oxysporum f. sp. vanillae. International Journal of Agriculture and Biology 18:23-30. https://doi.org/10.17957/IJAB/15.0053 [ Links ]
Amini J and Sidovich D. 2010. The effects of fungicides on Fusarium oxysporum f. sp. lycopersici associated with Fusarium wilt of tomato. Journal of Plant Protection Research 50:172-178. http://www.plantprotection.pl/pdf-91523-25801?filename=The%20effects%20of%20fungicides.pdf [ Links ]
Arie T. 2019. Fusarium diseases of cultivated plants, control, diagnosis, and molecular genetic studies. Journal of Pesticide Sciences 44(4): 275 - 281. https://doi.org/10.1584/jpestics.J19-03 [ Links ]
Baćmaga M, Wyszkowska J and Kucharski J. 2018. The influence of chlorothalonil on the activity of soil microorganisms and enzymes. Ecotoxicology 27. https://doi.org/10.1007/s10646-018-1968-7 [ Links ]
Casillas-Isiordia R, Flores-de la Rosa FR, Can-Chulim Á, Luna Esquivel G, Rodríguez-Guerra R, Ramírez-Guerrero LG, Luna-Rodríguez M and Aguirre Beltrán G. 2017. Fusarium sp. associated with Vanilla sp. rot in Nayarit, Mexico. ARPN Journal of Agricultural and Biological Science. 12:43-50. http://www.arpnjournals.org/jabs/research_papers/rp_2017/jabs_0217_844.pdf [ Links ]
Chen Z, Gao T, Liang S, Liu K, Zhou M and Chen C. 2014. Molecular mechanism of resistance of Fusarium fujikuroi to benzimidazole fungicides. FEMS Microbiology Letters 357:77-84. https://doi.org/10.1111/1574-6968.12504 [ Links ]
Chung WH, Chung WC, Ting PF, Ru CC, Huang HC and Huang JW. 2009. Nature of resistance to methyl benzimidazole carbamate fungicides in Fusarium oxysporum f. sp. lilii and F. oxysporum f. sp. gladioli in Taiwan. Journal of Phytopathology 157:742-747. https://doi.org/10.1111/j.1439-0434.2009.01545.x [ Links ]
Dahal N and Shrestha R. 2018. Evaluation of efficacy of fungicides against Fusarium oxysporum f. sp. lentis in vitro at Lamjung, Nepal. Journal of the Institute of Agriculture and Animal Science 35:105-112. https://doi.org/10.3126/jiaas.v35i1.22520 [ Links ]
dos Santos Vieira WA, Guerreiro Lima W, Souza Nascimento E, Michereff S, Doyle VA and Saraiva Camara M. 2017. Thiophanate-methyl resistance and fitness components of Colletotrichum musae isolates from banana in Brazil. Plant Disease 101:1659-1665. https://doi.org/10.1094/PDIS-11-16-1594-RE [ Links ]
Dowling ME, Hu MJ and Schnabel G. 2017. Fungicide resistance in Botrytis fragariae and species prevalence in the Mid-Atlantic United States. Plant Disease 102:964-969. https://doi.org/10.1094/PDIS-10-17-1615-RE [ Links ]
Duan Y, Zhang X, Ge C, Wang Y, Cao J, Jia X, Wang J and Zhou M. 2014. Development and application of loop-mediated isothermal amplification for detection of the F167Y mutation of carbendazim-resistant isolates in Fusarium graminearum. Scientific Reports 4:7094 https://doi.org/10.1038/srep07094 [ Links ]
Flores-de la Rosa FR, De Luna E, Adame-García J, Iglesias-Andreu LG and Luna-Rodríguez M. 2018. Phylogenetic position and nucleotide diversity of Fusarium oxysporum f. sp. vanillae worldwide based on translation elongation factor 1α sequences. Plant Pathology 67:1278-1285. https://doi.org/10.1111/ppa.12847 [ Links ]
Hernández-Hernández J. 2019. Mexican vanilla production. Pp: 3-38. In: Havkin-Frenkel D and Belanger FC (eds.). Handbook of Vanilla Science and Technology. Second edition. Hoboken, Wiley Blackwell, New Jersey, USA. 500p https://www.wiley.com/en-us/Handbook+of+Vanilla+Science+and+Technology%2C+2nd+Edition-p-9781119377276 [ Links ]
Hobbelen P, Paveley N and Bosch F. 2011. Delaying selection for fungicide insensitivity by mixing fungicides at a low and high risk of resistance development: a modeling analysis. Phytopathology 101:1224-1233. https://doi.org/10.1094/PHYTO-10-10-0290 [ Links ]
Koyyappurath S, Conejero G, Dijoux JB, Lapeyre-Montes F, Jade K, Chiroleu F, Gatineau F, Verdeil JL, Besse P and Grisoni M. 2015. Differential responses of vanilla accessions to root rot and colonization by Fusarium oxysporum f. sp. radicis-vanillae. Frontiers in Plant Science 6: 1125. https://doi.org/10.3389/fpls.2015.01125 [ Links ]
Kumar S, Stecher G, Li M, Knyaz C and Tamura K. 2018. MEGA X: Molecular evolutionary genetics analysis across computing platforms. Molecular biology and evolution 35(6): 1547-1549. https://doi.org/10.1093/molbev/msy096 [ Links ]
Lecomte C, Edel-Hermann V, Cannesan MA, Gautheron N, Langlois A, Alabouvette C, Robert F and Steinberg C. 2016. Fusarium oxysporum f. sp. cyclaminis: underestimated genetic diversity. European Journal of Plant Pathology 145(2): 421-431. https://doi.org/10.1007/s10658-016-0856-3 [ Links ]
Long JW and Siegel MR. 1975. Mechanism of action and fate of the fungicide chlorothalonil (2,4,5,6-Tetrachloroisophthalonitrile) in biological systems. Chemico-biological Interactions 10(6): 383-394. https://doi.org/10.1016/0009-2797(75)90069-1 [ Links ]
Lubinsky P, Bory S, Hernández-Hernández J, Kim SC and Gómez-Pompa A. 2008. Origins and dispersal of cultivated vanilla (Vanilla planifolia Jacks. [Orchidaceae]). Economic Botany 62(2): 127-138. https://doi.org/10.1007/s12231-008-9014-y [ Links ]
Nosratabadi M, Kachuei R, Rezaie S and Harchegani AB. 2018. Beta-tubulin gene in the differentiation of Fusarium species by PCR-RFLP analysis. Le Infezioni in Medicina, 26(1): 52-60. https://www.infezmed.it/media/journal/Vol_26_1_2018_7.pdf [ Links ]
Petkar A, Langston DB, Buck JW, Stevenson KL and Ji P. 2017. Sensitivity of Fusarium oxysporum f. sp. niveum to prothioconazole and thiophanate-methyl and gene mutation conferring resistance to thiophanate-methyl. Plant Disease 101:366-371. https://doi.org/10.1094/PDIS-09-16-1236-RE [ Links ]
Pinaria A, Liew ECY and Burgess L. 2010. Fusarium species associated with vanilla stem rot in Indonesia. Australasian Plant Pathology 39:176-183. https://doi.org/10.1071/AP09079 [ Links ]
Steinberg G and Gurr SJ. 2020. Fungi, fungicide discovery and global food security. Fungal Genetics and Biology 144: 103476. https://doi.org/10.1016/j.fgb.2020.103476 [ Links ]
Suga H, Nakajima T, Kageyama K and Hyakumachi M. 2011. The genetic profile and molecular diagnosis of thiophanate-methyl resistant strains of Fusarium asiaticum in Japan. Fungal Biology 115:1244-1250. https://doi.org/10.1016/j.funbio.2011.08.009 [ Links ]
Untergasser A, Nijveen H, Rao X, Bisseling T, Geurts R and Leunissen JA. 2007. Primer3Plus, an enhanced web interface to Primer3. Nucleid Acid Research 35. https://doi.org/10.1093/nar/gkm306 [ Links ]
Vela-Corcía D, Romero D, de Vicente A and Pérez-García A. 2018. Analysis of β-tubulin-carbendazim interaction reveals that binding site for MBC fungicides does not include residues involved in fungicide resistance. Scientific Reports 8:7161. https://doi.org/10.1038/s41598-018-25336-5 [ Links ]
Yang LN, He MH, Ouyang HB, Zhu W, Pan ZC, Sui QJ, Shang LP and Zhan J. 2019. Cross-resistance of the pathogenic fungus Alternaria alternata to fungicides with different modes of action. BMC Microbiology 19:205. https://doi.org/10.1186/s12866-019-1574-8 [ Links ]
Zhang X, Chen X, Jiang J, Yu M, Yin Y and Ma Z. 2015. The tubulin cofactor A is involved in hyphal growth, conidiation and cold sensitivity in Fusarium asiaticum. BMC Microbiology 15:35. https://doi.org/10.1186/s12866-015-0374-z [ Links ]
Zhou Y, Xu J, Zhu Y, Duan Y and Zhou M. 2016. Mechanism of action of the benzimidazole fungicide on Fusarium graminearum: Interfering with polymerization of monomeric tubulin but not polymerized microtubule. Phytopathology 106(8):807-813. https://doi.org/10.1094/PHYTO-08-15-0186-R [ Links ]
Zhu Y, Liang X, Li Y, Duan Y, Zheng Z, Wang J and Zhou M. 2018. F240 of β 2- Tubulin explains why Fusarium graminearum is less sensitive to carbendazim than Botrytis cinerea. Phytopathology 108:352-361. https://doi.org/10.1094/PHYTO-09-17-0295-R [ Links ]
Recibido: 15 de Marzo de 2022; Aprobado: 26 de Junio de 2022











 texto em
texto em 


