Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de fitopatología
versión On-line ISSN 2007-8080versión impresa ISSN 0185-3309
Rev. mex. fitopatol vol.39 no.spe Texcoco 2021 Epub 30-Nov-2022
https://doi.org/10.18781/r.mex.fit.2021-23
Fitosanidad y COVID-19
Los intervalos serial y de generación de la dinámica de transmisión de SARS-CoV-2 y su aplicación potencial en la epidemiología de dos enfermedades de los cítricos
1 Department of Plant Sciences and Plant Pathology, Montana State University, Bozeman, MT 59717, USA;
2 Facultad de Agronomía, Universidad de Buenos Aires, Buenos Aires, Argentina;
3 Campo Experimental Ixtacuaco, Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias, Tlapacoyan, Veracruz, CP 93600, México;
4 Department of Botany and Plant Pathology, Purdue University, West Lafayette, IN, 47907, USA;
Desde el inicio del síndrome respiratorio agudo grave del Coronavirus 2 (SARS-CoV-2) responsable de la pandemia COVID-19, los conceptos de intervalo serial e intervalo de generación se han usado como medidas epidemiológicas clave para entender la dinámica de transmisión de la enfermedad. En esta revisión, estos conceptos se han examinado y reorientado al entendimiento de la cadena de transmisión y dinámica de dos enfermedades importantes de los cítricos: la tristeza de los cítricos (causada por Citrus tristeza virus, CTV) y el Huanglongbing (causado por Candidatus Liberibaster asiaticus). Basados en la definición fundamental de los conceptos, la revisión define la cadena de transmisión del SARS-CoV-2 y del CTV y CLas, señalando sus mayores diferencias y similitudes. Posteriormente, la revisión discute el cálculo de los intervalos y sus distribuciones para ambas enfermedades. La identificación de pares de árboles infector-infectado en una cadena de transmisión dentro de huertos se propone a través del uso de datos de incidencia de enfermedad de mapeos intensivos, análisis de patrón espacial, probabilidad condicional y métodos de simulación. Al igual que en la dinámica del SARS-CoV-2, la transmisión pre-sintomática en estos dos patosistemas es de significancia epidemiológica. De ahí que el cálculo del intervalo serial y de generación puede ayudar a sentar las bases para el entendimiento de la dinámica de transmisión temprana, y con ello, la implementación de medidas de control del vector o la erradicación de árboles enfermos. Se espera que esta revisión motive discusiones sobre el cálculo y uso de estos conceptos para mejorar el entendimiento de la epidemiología de las enfermedades examinadas en este trabajo.
Palabras clave: COVID-19; patrón espacial; VTC; HLB; simulación Monte Carlo.
Since the start of the severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) causing the coronavirus disease 2019 (COVID-19) pandemic, the concepts of serial and generation intervals have been used as key epidemiological measures to understand the transmission dynamics of the disease. We carefully examined and repurposed these concepts to the understanding of the transmission chain and dynamics of two major citrus diseases: tristeza virus (caused by Citrus tristeza virus, CTV) and Huanglongbing (caused by Candidatus Liberibacter asiaticus). Following the fundamental definition of the concepts, the review delineates the transmission chain in the SARS-CoV-2 and that of CTV and CLas, pointing out their major similarities and differences. Then, it discusses estimation of the serial and generation intervals and their distributions for both plant diseases. Identification of infector-infectee tree pairs in a transmission chain within orchards is proposed through use of disease incidence data from intensive mapping, spatial pattern analysis, conditional probability, and simulation approaches. Like in SARS-CoV-2 dynamics, pre-symptomatic transmission in these two plant pathosystems is of epidemiological significance. Hence, estimation of the serial and generation interval can lay the foundations to understanding of early disease transmission dynamics, thus the implementation of vector control measures or eradication of infected trees. We hope this review motivates discussions on estimation and usage of these concepts to enhance understanding of the epidemiology of both of the herein examined citrus diseases.
Key words: COVID-19; spatial pattern; CTV; HLB; Monte Carlo simulation
Análisis del contagio SARS-CoV-2
La rápida propagación global del síndrome respiratorio agudo severo coronavirus 2 (SARS-CoV-2) causante de la enfermedad del coronavirus COVID-19 desató una enorme cantidad de investigación para el desarrollo de medidas de control y mitigación de la enfermedad en regiones afectadas. Con ello, varios conceptos epidemiológicos han sido clave para entender la transmisión y la propagación del virus y, con ello, planificar para su intervención (Li et al., 2021). Entre esos conceptos se encuentran el intervalo serial, IS: el período entre el inicio de los síntomas de un infector y un infectado, y el intervalo de generación, IG: el tiempo entre eventos de infección en un par de infector-infectado (Lehtinen et al., 2021). El IS ha demostrado ser un indicador para la vigilancia efectiva de epidemia y es un factor necesario para el cálculo del IG. El IG es esencial para estimar el número reproductivo básico, es decir, el promedio de individuos infectados por un solo infector en una población susceptible (Rothman et al., 2013). Tanto el IS como el IG son las bases de modelos epidemiológicos que buscan guiar las estrategias de intervención de enfermedades infecciosas (Ganyani et al., 2020).
En esta revisión examinamos la relevancia del IS y del IG para comprender la dinámica de dos enfermedades importantes en cítricos: el virus de la tristeza de los cítricos (VTC) y Huanglongbing (HLB). La tristeza de los cítricos es una enfermedad causada por un virus limitado al floema de la familia Closteroviridae, género Closterovirus, que infecta a cítricos y a especies relacionadas (Dawson et al., 2013). HLB, antes conocida por su nombre en inglés, Citrus greening disease, es una de las enfermedades más destructivas de los cítricos en el mundo, causada por al menos tres especies de la bacteria Candidatus Liberibacter (Bové, 2006), limitada al floema. Estas enfermedades son las más destacadas para la producción de cítricos en México y otras regiones citrícolas del mundo, en donde han matado a millones de árboles de cítricos (Moreno et al., 2008; Gottwald et al., 2007; Spreen et al., 2014; Dawson et al., 2015; Dala-Paula et al., 2019). Ambas enfermedades son transmitidas por vectores: el VTC transmitido principalmente por el pulgón café Toxopera citricida y CLas, por Diaphorina citri.
Al igual que el SARS-CoV-2, VTC y HLB presentan un período de transmisión en el que los individuos enfermos no presentan síntomas, aunque ya son contagiosos (Lee et al., 2015; Moreno et al., 2008). Sin embargo, la propagación de VTC y HLB durante esta fase asintomática es un tema del cual todavía se tiene poco conocimiento mediante el uso de parámetros epidemiológicos tradicionales, tales como tasas de infección, el área bajo la curva de progreso de la enfermedad y distancias de dispersión. Por el contrario, las dinámicas tempranas de transmisión de COVID-19 han sido caracterizadas con precisión con el uso del IS y el IG como medidas epidemiológicas (Ganyani et al., 2020; Ferretti et al., 2020; Lehtinen et al., 2021; Li et al. 2021; Mettler et al., 2020; Ng et al., 2021). El objetivo de esta revisión es discutir el significado de IS e IG para poner en perspectiva su utilidad potencial, o ausencia de la misma, para comprender más a fondo la epidemiología del VTC y HLB. Aquí tratamos el tema de los enfoques para la identificación de pares de árboles infector-infectado, que sería un paso importante en la determinación del IS e IG; también tratamos la estimación de los intervalos y su distribución para ambas enfermedades. Con esta revisión buscamos generar discusiones sobre la estimación y el uso de estos conceptos para promover la comprensión de la epidemiología de las dos enfermedades de cítricos aquí tratadas.
Aplicaciones de los intervalos serial y de generación
Definiciones. El intervalo serial (IS) y el intervalo de generación (IG, también conocido como el tiempo de generación) son parámetros epidemiológicos clave de uso en el sector de salud pública para caracterizar las dinámicas de transmisión de enfermedades como el COVID-19 (Ganyani et al., 2020; Ferretti et al., 2020; Li et al. 2021; Mettler et al., 2020; Ng et al., 2021; Lethtinen et al., 2021). El IS implica la diferencia de tiempo entre el inicio de síntomas en un infector y un infectado, mientras que el IG implica la diferencia de tiempo entre infectarse e infectar a otros, o el tiempo entre eventos de infección en un par infector-infectado (Figura 1) (Lehtinen et al., 2021). Otros conceptos usados en el estudio de enfermedades infecciosas son el número reproductivo básico (R0) y el período de incubación. El número reproductivo básico, una medida del potencial de transmisión de una enfermedad, cuantifica el promedio de infecciones secundarias producidas por un caso típico de infección en una población de sólo individuos susceptibles (Rothman et al., 2013). El período de incubación (PI) es el tiempo transcurrido desde la infección hasta el inicio de los síntomas. En fitopatología, el PI se define como el tiempo entre la infección por un patógeno y el inicio de la expresión de síntomas en la planta hospedante. La latencia o el período de latencia se define, para las enfermedades virales o enfermedades causadas por bacterias fastidiosas similares a los virus, como el tiempo entre una infección arbórea y el inicio de la infectividad. Para las enfermedades de cultivos, los períodos de incubación y latencia empiezan con la infección y son procesos temporales relacionados (Agrios, 2005). Tanto en humanos como en plantas, los individuos asintomáticos recién infectados permanecen, inicialmente, en su período latente y no transmiten el patógeno (Leung et al., 2018). Sin embargo, cuando el IG se completa, los individuos asintomáticos pueden volverse infecciosos y transmitir el agente causal de la enfermedad (Gottwald et al., 2002; Rimbaud et al., 2015; Qian et al., 2020; Johansson et al., 2021). Por definición, la latencia termina cuando la infectividad empieza. Por lo general, el período de incubación es más largo que el período de latencia. A diferencia del proceso de infección causado por la cepa viral de SARS-CoV-2 directamente entre humanos, las infecciones virales y bacterianas en árboles suelen transmitirse entre árboles frutales por un insecto vector. Por ello, en el último caso, la transmisión no es directa, sino que depende de variables que afectan la biología y ecología del vector (Gottwald et al., 1999; Canale et al., 2017). La evaluación o determinación de los períodos de incubación y latencia para enfermedades transmitidas por vectores continúa siendo todo un desafío (Rimbaud et al., 2015).
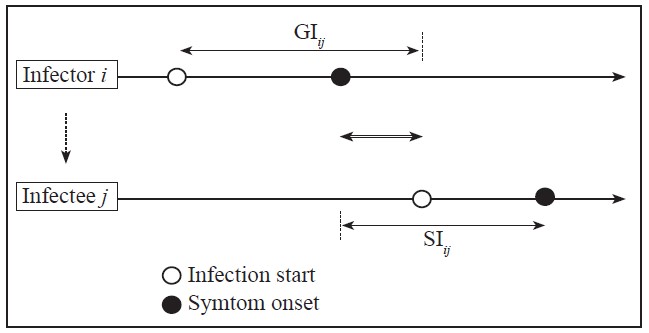
Figura 1 Representación del intervalo serial (SIij) y el intervalo de generación o el tiempo de generación (GIij) en una cadena de transmisión de infectante i-infectado j de COVID-19. La flecha hueca en el medio representa el tiempo desde el inicio de los síntomas de i hasta la infección de j. El período de incubación (no indicado) es el tiempo desde el inicio de la infección (círculo blanco) hasta el inicio de los síntomas (círculo negro) en cada individuo. Basado en Lehtinen et al. (2021).
Cadena de transmisión de las enfermedades estudiadas
COVID-19. El COVID-19 es una enfermedad infecciosa causada por un Coronavirus (Coronaviridae: Beta-coronavirus) identificado recientemente; un virus de ARN monocatenario positivo, envuelto con los característicos picos tipo “corona” en su superficie. El primer caso fue identificado en Wuhan, China, en diciembre de 2019 (Asselah et al., 2021). Los síntomas de esta enfermedad pueden ser descritos como leves en la mayoría de las personas, aunque también puede causar malestares severos e incluso la muerte en otros (Mavrodiev et al., 2020; Wu et al., 2020). La cadena de infección del COVID-19, como la define el sector de salud pública, se refiere a la serie de etapas interconectadas, desde que un agente infeccioso sale de su hospedante y es transportado por algún mecanismo de transmisión hasta que ingresa a un nuevo hospedante susceptible y lo infecta. La cadena de infección de COVID-19 tiene una fase exponencial cuando hospedantes infectados y sanos se acercan a una distancia muy próxima entre ellos, lo cual favorece la transmisión directa o indirecta (Wu et al., 2020). La transmisión directa puede ocurrir por medio de besos o por compartir cubiertos al comer, mientras que la transmisión indirecta ocurre al tocar una superficie contaminada con secreciones que contienen el virus y después tocar un portal de entrada al cuerpo, tal como los ojos, nariz o boca.Otra forma de transmisión del COVID-19 ocurre cuando gotículas y aerosoles que contienen el virus son expulsados de una persona infectada al hablar, respirar, toser o estornudar (Prather et al., 2020). La cadena de transmisión comienza cuando un infector sintomático o asintomático, o una fuente primaria infecta a uno o varios individuos (infectados) que se encuentran en proximidad física (Qian et al., 2020; Wei et al., 2020). Este patrón se ve afectado por la movilidad de infectores sintomáticos o asintomáticos y la exposición a los mismos. La transmisión presintomática ocurre cuando el IG es más corto que el PI del infector. Según Nishiura et al. (2020), la mediana del IS es más breve que la del PI, lo cual sugiere una proporción importante de transmisión presintomática. En otro estudio se estimó que al menos 50% de las nuevas infecciones de SARS-CoV-2 se originaron de la exposición a individuos con infección asintomática (Johansson et al., 2021). Por este motivo, las medidas de mitigación tales como la cuarentena, el distanciamiento social, el uso de cubrebocas, la higiene de manos y la realización estratégica de pruebas qPCR ayudan a reducir el riesgo de transmisión, y por ello a interrumpir cadenas de infección lo más temprano posible.
La duración del IS y el IG de COVID-19 se calcula del rastreo de datos de contacto en una cadena de transmisión de casos confirmados (Ganyani et al., 2020; Ferretti et al., 2020; Li et al. 2021; Mettler et al., 2020; Ng et al., 2021). Las estimaciones sugieren una media de IS de 7.5 días (95% CI: 5.3-19); para IG, una media de 3.95 días (95% CI: 3.01-4.91), y una media para PI de 5.2 días (95% CI: 4.1-7.0) (Ganyani et al., 2020; Li et al. 2020). En la cadena de transmisión de COVID-19, la longitud del IS se ve afectada por la interacción infector-infectado, ya que este par puede incluir a personas de diferentes edades y con diferentes respuestas a la infección por COVID-19. Por ejemplo, se ha observado que, mientras algunas personas solo presentan síntomas muy leves, otros pueden expresar un síndrome extremadamente severo o morir.
Virus tristeza de los cítricos (VTC). En la epidemiología de plantas, el término “cadena de transmisión” no es de uso común. En cambio, la cadena de infección de una enfermedad es mejor conocida como el ciclo de la enfermedad, lo cual se refiere a los diferentes eventos que conducen a la enfermedad en una porción o en la totalidad de una población de plantas (Agrios, 2005). El VTC no se transmite por semilla. Normalmente, la propagación a larga distancia es por el transporte de material vegetal infectado asintomático por humanos. Así, en huertos de cítricos recién establecidos, la fuente principal de inóculo de VTC suele ser el material vegetal infectado. Una vez introducido en un huerto de cítricos, la propagación local o de corto alcance dentro de huertos o plantaciones se lleva a cabo por insectos vectores (pulgones). A partir de árboles infectados, el VTC es adquirido por poblaciones de pulgones responsables de la propagación secundaria. Varias especies de pulgones transmiten VTC (Lee et al. 1994; Yokomi et al. 1994), de las cuales Aphis citricidus (Syn. Toxoptera citricida) es el vector más eficiente de VTC y transmite el virus de manera semipersistente (Gottwald et al., 1998; Dawson et al., 2013). La adquisición require de un período de prueba de 4-6 horas y los pulgones permanecen virulíferos por 24-48 horas sin atravesar un período de latencia (Rocha-Peña et al., 1994). La cadena de transmisión de VTC puede ser descrita, simplemente, de la siguiente manera: fuente primaria/dispersión del inóculo primario/primer árbol infectado en un huerto → adquisición del virus por un pulgón avirulífero → transmisión del virus por el pulgón a un árbol sano → transmisión a otro árbol sano y → fin de la cadena de transmisión en 30 días con el fin del ciclo de vida del vector. Aumento en el número de vectores infecciosos en una población (aumento de inóculo secundario/transmisión) → aumento en el número de árboles infectados. En un estudio de transmisión realizado en la Península de Yucatán, en México, se estimó un rango de transmisión para A. citricidus de 0 - 14.8%, dependiendo del origen de los aislamientos de VTC (Hernández, 2013). La implicación epidemiológica de estas características biológicas se refleja en las altas tasas de dispersión temporal del VTC en condiciones de campo (Rivas-Valencia et al., 2017). Por ejemplo, en México, la incidencia de árboles con VTC aumentó 40% en cuatro años. La dispersión de aislamientos severos aumenta gracias a A. citricidus, incluso en patrones considerados tolerantes a VTC (Rivas-Valencia et al., 2020).
Normalmente, VTC tiene un período de incubación de varios años, a medida que los árboles de cítricos se deterioran de repente, años después de infectarse (Fishman et al., 1983). Esto se llama comúnmente una infección crónica. La enfermedad, en regiones endémicas, está presente en más de 90% de los árboles de cítricos en condiciones asintomáticos (Mora-Aguilera, 2008; Mora-Aguilera et al., 2005; Rivas-Valencia et al., 2017). Para detector árboles infectados con VTC (asintomáticos o sintomáticos), se toman muestras de tejido vegetal y se aplican técnicas de diagnóstico serológicas (ELISA) o moleculares (RT-PCR) (Hughes y Gottwald, 1998; Huang et al., 2004; Rosa et al., 2007; Ruiz-Ruiz et al., 2007; Korkmaz et al., 2008; Saponari et al., 2008). Existen pocos estudios sobre los períodos de incubación de VTC. Balaraman y Ramakrishnan (1979) determinaron experimentalmente que una transmisión del virus 100% efectiva solo se logró con al menos 15 T. citricida virulíferos que se alimentaron de una planta, y que los pulgones requirieron 24 h para cada período de adquisición y transmisión. El período de incubación se redujo 15-20 días cuando más de 100 pulgones se alimentaron de cada planta. En otro experimento, un aislamiento inoculado en plántulas de naranjo agrio permaneció por 51 días en las partes basales de las plantas, pero se distribuyó sistemáticamente por toda la planta a los te 58 días después de la inoculación (Rocha-Peña et al., 1994). Gottwald et al. (2002) determinaron que el VTC inoculado en la primavera o en el otoño en árboles de 2-3 años de edad permanece serológicamente indetectable hasta la siguiente primavera o más tarde. Incluso durante la siguiente primavera, solo algunos de los árboles podrían tener infecciones detectables. Los mismos autores encontraron que el movimiento sistémico de VTC ocurre en las semanas siguientes a la inoculación, pero que el umbral de detección (la cantidad de carga viral) permaneció bastante bajo. Por lo tanto, existe una variación considerable en el período de latencia entre la infección inicial y la sistémica.
Huanglongbing (HLB). HLB es uno de los problemas más importantes de los cítricos a nivel mundial. HLB es causado por Candidatus Liberibacter spp., una bacteria fastidiosa limitada al floema transmitida por chicharritas, y se caracteriza por tener un periodo de incubación que dura de 3 meses a varios años antes de que los síntomas sean visibles (Manjunath et al., 2008; Flores-Sánchez et al., 2017; Canale et al., 2020). El psílido asiático Diaphorina citri es el vector más eficiente y transmite el patógeno en un ciclo de transmisión circulativo-propagativo (persistente) (Manjunath et al., 2008; Hall et al., 2013; Torres-Pacheco et al., 2013; Keremane et al., 2015; Galdeano et al., 2020). De acuerdo con Canale et al. (2017), los psílidos pudieron transmitir el patógeno hasta por 5 semanas después de un periodo de acceso de adquisición de 14 días como ninfas. El psílido puede adquirir el patógeno después de alimentarse hasta por 15 a 30 minutos y permanece infectivo por el resto de su vida (3 a 4 meses) (Hall et al., 2013; Galdeano et al., 2020); la transmisión transovárica sólo ha sido reportada en el psílido africano Trioza erytreae (Manjunath et al., 2008). Gottwald et al. (2008) analizaron el efecto del contagio por psílidos a cortas distancias sobre el patrón de dispersión global de la enfermedad en el tiempo, y encontraron que la infección de árboles ocurre principalmente por psílidos provenientes de bloques de árboles contaminados, no cercanos a árboles no infectados y no por psílidos de huertos cercanos. Estudios realizados en huertos de cítricos en Florida sobre la distribución espacial de HLB evidenciaron la existencia de dos procesos de infección (Gottwald et al. 2008): un proceso de dispersión primaria, donde el contagio ocurre principalmente por la transmisión de vectores a largas distancias, y otro, por dispersión local, que ocurre dentro del huerto. Los autores concluyeron que el contagio más devastador está relacionado con los vectores provenientes de largas distancias, independientemente del control local mediante insecticidas; dado que los psílidos se alimentan en árboles distantes (detectados como infectados con CLas) continuarán infectando huertos locales antes de morir debido a las aplicaciones de insecticidas. La cadena de transmisión de CLas se puede describir simplemente como: fuente primaria o primer árbol infectado → adquisición de la bacteria por una Diaphorina no infectiva → transmisión a un árbol sano, y así sucesivamente.
Varios investigadores han reportado períodos variables de incubación para CLas, desde pocos meses hasta uno o varios años (Gottwald et al., 1989; Gottwald, 2010; Canale et al., 2020). También se ha observado que en los huertos más jóvenes el patógeno tiene períodos de incubación de entre 6 y 12 meses. En un estudio realizado por Irey et al. (2006), la detección de RT-PCR reveló que 50% de los árboles infectados eran sintomáticos y 50% asintomáticos. En la práctica se asume que puede haber dos o muchas veces más infecciones asintomáticas ya establecidas en comparación con aquellas que son visualmente sintomáticas en un huerto dado. Por ello, la detección visual de HLB es inadecuada porque conduce a subestimar la cantidad de enfermedad (falsos negativos). Además, la distribución sistémica dentro de los árboles es incompleta o difusa, lo que dificulta aún más la detección temprana mediante técnicas moleculares (Gottwald et al., 2008). Esta es la explicación del por qué CLas es altamente invasivo cuando se introduce en áreas nuevas. Es decir, la enfermedad es normalmente muy dispersa espacialmente en la región antes de la expresión de síntomas (debido al largo período de incubación).
El prolongado período de incubación y una dispersión regional pueden hacer no viables los esfuerzos de erradicación (Gottwald, 2010). Recientemente se calculó la mediana del período de latencia en 16.8 y 17.8 días para las ninfas y los adultos de los psílidos que adquirieron el patógeno, respectivamente (Canale et al., 2017).
Estimación de los intervalos serial y de generación en VTC y HLB y sus distribuciones
En el cálculo del IS y el IG para COVID-19, los pares infector-infectado normalmente son creados ex post facto a partir de datos de rastreo der contacto de casos infectados de registros públicos oficiales. Luego se determina una cantidad de cadenas de transmisión a partir de esos datos y las mediciones obtenidas para el IS se usan para estimar la distribución del IG por medio de modelos matemáticos o estadísticos con enfoques frecuentistas o bayesianos (Li et al., 2021; te Beest et al., 2013). El cálculo de los intervalos para VTC y HLB puede ser más dificultoso porque debe de considerar el modo indirecto de adquisición y transmisión de patógenos por sus vectores móviles y la naturaleza sésil de los árboles de cítricos. Sin embargo, esta última condición puede ser ventajosa para identificar pares de árboles infectores-infectados dentro de los huertos, siempre y cuando la enfermedad y la población de vectores sean monitoreadas tanto espacial como temporalmente. Una forma de asignar pares puede ser examinando mapas de incidencia de la tristeza o HLB desde evaluaciones periódicas, seguido de análisis de autocorrelación espacial de datos de incidencia para determinar la presencia o ausencia de agregación de árboles enfermos y, de encontrarse agregación, determinar también su direccionalidad e intensidad. El grado de dependencia espacial indicado por la cantidad de ubicaciones espaciales con correlaciones significativas (p<0.05) puede ser usado para guiar la identificación de los árboles en el mapa de incidencia como los posibles pares de infectores-infectados. Las correlaciones significativas de primer, segundo o tercer orden se podrían considerar como equivalentes a la distancia de dispersión de vectores (Kobori et al., 2011; Loxdale et al., 1993) y las hileras de cítricos y el espaciamiento entre árboles comúnmente usado en huertos comerciales. Los métodos de simulación Monte Carlo (un ejemplo del cual se da en el Cuadro 1) y de cadena Markov Monte Carlo pueden ser usados como alternativa a la autocorrelación espacial para estimar la aleatoriedad o ausencia de ésta en los árboles contagiados dentro de un huerto. En el ejemplo que se da en el Cuadro 1, la distancia observada entre 12 árboles contagiados y su vecino más cercano en un huerto de limón Persa fue de 7.13, mientras que la distancia estimada con la prueba de aleatorización (de 1000 simulaciones) fue de 7.1552. La diferencia fue de 0.0252. Esto significa que la probabilidad de que un valor diste tanto o más que 7.13 de 7.1552 se estima como la proporción de los 1000 valores que son de 7.13 ó menos ó 7.1804 (de 7.1552 + 0.252) o más. Existen 496 valores tan extremos como esto, por lo que la significancia de los resultados se estima en 0.496, lo cual sugiere que hay evidencia de que el arreglo de los árboles contagiados en el huerto fue aleatorio. Los métodos de cadena de Markov Monte Carlo también pueden ser usados para identificar pares de árboles infector-infectado. Además, siempre y cuando se cumpla la suposición de independencia de eventos, se pueden usar la probabilidad condicional y el teorema de Bayes para estimar la probabilidad de que un árbol específico haya sido el infector de su primero, segundo o tercer vecino más cercano. De manera explícita, la probabilidad de que un árbol sintomático A sea el infector del árbol B, dado que B es su vecino más cercano [P(A|B)], se estimaría con la probabilidad de que el árbol B sea el infectado, dado que el árbol sintomático A es la fuente primaria de infección a su vecino más cercano [P(B|A)], multiplicado por la probabilidad de B y dividido por la probabilidad de B. La forma de la ecuación es la siguiente: P(A|B) = [P(B|A) P(B)] / P(B), donde P(A) es la probabilidad de que el árbol cítrico A sintomático sea una fuente primaria o infector de su vecino más cercano y P(B) es la probabilidad de que un árbol sintomático B sea infectado por el árbol A, asumiendo que es su vecino más cercano y que fue asintomático antes que A.
Cuadro 1 Distancias observadas (m) entre 12 árboles de limón Persa enfermos deHLB y sus cuatro vecinos más cercanos en un huerto de 180 árboles, las distancias medias estimadas con 1000 datos generados de manera aleatoria usando la simulación Monte Carlo, y los niveles de significación estimados de los valores observados.
| Distance (m) | 1st nearest neighbor | 2nd nearest neighbor | 3rd nearest neighbor | 4th nearest neighbor |
|---|---|---|---|---|
| Observed | 7.13 | 9.78 | 14.52 | 17.19 |
| Estimated Mean | 7.1552 | 9.7746 | 14.5525 | 17.2107 |
| Significancez | 0.4961 | 0.5040 | 0.5001 | 0.4986 |
z El nivel de significancia para cada ier árbol vecino es la probabilidad estimada de obtener un valor tan alto como el observado, que es interpretado como la probabilidad de un valor tan lejos o más lejos que la media de la distribución simulada.
Los datos fueron obtenidos de un estudio de HLB realizado en la región norte-centro de Veracruz, México del 2019 al 2020 (Dominguez et al., no publicado) y los datos se simularon en R.
La distribución de los intervalos serial y generacional de VTC y HLB puede estimarse con los datos de IS observados de los pasos anteriores usando metodologías similares a las usadas para el COVID-19 (Lehntine et al., 2021). Debido a que el IS puede tomar un continuo de valores y ser negativo (más corto que el periodo de incubación), entonces requiere un modelo de distribución probabilística para una variable aleatoria continua y que acepte valores negativos. Se sugieren las distribuciones lognormal, Gamma y Weibull como opciones de distribución (Ganyani et al., 2020). Después de las pruebas de bondad de ajuste, el modelo puede ser usado para obtener los intervalos de confianza (IC) relacionados a la media de IS y IG. Metodologías como los de Monte Carlo y cadena de Markov Monte Carlo con un número deseado de simulaciones se pueden usar para aproximar una distribución y obtener los parámetros del modelo propuesto. Finalmente, el método de tiempo aun evento puede ser una alternativa para determinar el tiempo hasta que un árbol asintomático en particular comienza a mostrar síntomas en un nuevo brote foliar durante un período predeterminado.
Discusión
La epidemiología agrícola ha tomado muchos términos útiles, principios y metodologías de la epidemiología humana. En esta revisión se discuten los conceptos de intervalo serial (IS) e intervalo de generación (IG), que han sido instrumentales en la caracterización de la dinámica de la transmisión (sintomática y asintomática) de una enfermedad humana de interés global: COVID-19. Hasta donde se tiene conocimiento, los conceptos de IS e IG no han sido usados, al menos de manera explícita, en la epidemiología agrícola.
Con el entendimiento de lo que implican estos conceptos, las preguntas que permanecen son las siguientes: i) ¿son útiles estas medidas epidemiológicas para comprender epidemias fitopatológicas? - específicamente de enfermedades causadas por virus y patógenos limitados al floema, tales como VTC y HLB. Y si la respuesta a esta pregunta es afirmativa, entonces ii) ¿Cuáles serían algunas metodologías factibles y confiables para la observación en campo, experimentación y estimación de la distribución de estos parámetros? Tal como el SARS-CoV-2, el VTC y HLB presentan una transmisión presintomática durante la cual los árboles ya pueden ser contagiosos (Gottwald et al., 1999; Manjunath et al., 2008; Canale et al., 2020). El IS, y derivado de él, el IG puede ayudar a elucidar este proceso, ya que está basado en el inicio de los síntomas (que son fácilmente observables), en lugar de en tiempos de infección (que por lo general son desconocidos). Tanto el VTC como el HLB tienen un largo periodo de incubación que incluso puede tardar años en completarse. Además, la distribución del patógeno en un árbol no es perfectamente sistémica al inicio de la infección, y el periodo de incubación podría variar con la edad de las plantas y con la región. Todas estas características dificultan aún más la detección temprana de individuos infectados en un huerto. En la reducción de la transmisión y propagación de estas enfermedades, la detección temprana de individuos infectados es esencial para implementar medidas de mitigación tales como el control de poblaciones de vectores o la erradicación de árboles infectados. La detección confiable se debe llevar a cabo por medio de técnicas serológicas tales como ELISA (para VTC) o técnicas moleculares tales como RT-PCR (para CLas y VTC). Recientemente, la detección por medio del olfato canino demostró ser una herramienta de detección más rápida, confiable y económica que los métodos moleculares. Por ejemplo, la detección de individuos asintomáticos infectados por medio de perros eficazmente entrenados fue demostrada, tanto para COVID-19 (Grandjean et al., 2020, 2021; Jendrny et al., 2020) como para HLB (Gottwald et al., 2020) como una alternativa eficiente para este problema. Los métodos de detección temprana podrían ayudar a estimar los IS e IG. Al estimar los intervalos SI y GI para CTV y HLB, se deben hacer varias consideraciones y suposiciones importantes con respecto al cultivo hospedante, el vector, el ambiente, así como las prácticas de manejo que podrían afectar la transmisión del patógeno.
De ello se deduce que tanto el IS como el IG podrían ser útiles para conocer más sobre las epidemias de estas enfermedades. Nuestra revisión sugiere que el uso y aplicación de estos parámetros ofrece tanto retos como oportunidades de investigación en la epidemiología de VTC y HLB. El reto principal es la elucidación del papel del vector en la transmisión y propagación del patógeno en lo que se relaciona con la duración del tiempo de infección, el periodo de incubación, IS e IG. Por otro lado, es extremadamente difícil determinar cuándo se infecta un árbol de cítricos. Determinar la infectividad y transmisión requeriría de experimentos de liberación artificial de insectos, enjaulamiento de árboles o protección de árboles por medio de la aplicación de insecticidas, tal como se ha hecho para otras enfermedades diseminadas por vectores. Esto podría hacer juego con la detección de vectores y árboles infectados usando métodos serológicos, moleculares o incluso métodos de olfato animal. En contraste con epidemias de COVID-19 en las que la movilidad del hospedante afecta la transmisión, en las epidemias de VTC y HLB, el hospedante principal no presenta dispersión ni movilidad. Por lo tanto, existe menos incertidumbre y subjetividad en la asignación de pares de infección que en los datos de rastreo de contactos usados para la estimación del IS en humanos. Otra pregunta es si es de relevancia epidemiológica definir los IS e IG en el vector mismo.
Conclusiones
Esta revisión ha presentado a fitopatólogos los intervalos serial y de generación, dos medidas epidemiológicas clave que han sido usados en la epidemiología humana para caracterizar las dinámicas de transmisión (especialmente la transmisión presintomática o la propagación asintomática) de una enfermedad humana de interés global. Se presentaron la definición y los enfoques para estimar la distribución de estos parámetros, con especial énfasis en las metodologías para identificar los pares de árboles infector-infectado en una cadena de transmisión dentro de un huerto, así como las metodologías usadas para estimar medias e intervalos de confianza para COVID-19. Al igual que en COVID-19, la transmisión asintomática de VTC y HLB ha sido un problema importante para la implementación efectiva de medidas de control de enfermedades como el control y la erradicación de vectores. Se espera que esta revisión despierte el interés en la realización de más análisis de las medidas del IS y el IG o la búsqueda de su uso o adopción. Estos parámetros epidemiológicos podrían ayudar a entender más sobre la epidemiología de enfermedades de importancia de cuarentenaria en las que la detección debe ser seguida de la erradicación de los individuos contagiados.
Literature cited
Agrios GN. 2005. Plant Pathology. Fifth ed. Academic Press. Burlington, Massachusetts, United States of America. https://doi.org/10.1016/C2009-0-02037-6 [ Links ]
Asselah T, Durantel D, Pasmant E, Lau G and Schinazi RF. 2021. COVID-19: Discovery, diagnostics and drug development. Journal of Hepatology 74(1):168-184. http://dx.doi.org/10.1016/j.jhep.2020.09.031 [ Links ]
Balaraman K and Ramakrishnan K. 1979. Transmission studies with strains of citrus tristeza virus on acid lime (Citrus aurantifolia). Journal of Plant Diseases and Protection 86: 653-661. Link: https://www.jstor.org/stable/43214591?seq=1 [ Links ]
Bové JM. 2006. Huanglongbing: a destructive, newly emerging, century-old disease of citrus. Journal of Plant Pathology 88:7-37. http://dx.doi.org/10.4454/jpp.v88i1.828 [ Links ]
Canale MC, Tomaseto AF, Haddad ML, Della Coletta-Filho H and Lopes JR. 2017. Latency and Persistence of ‘Candidatus Liberibacter asiaticus’ in Its Psyllid Vector, Diaphorina citri (Hemiptera: Liviidae). Phytopathology 107: 264-272. https://doi.org/10.1094/PHYTO-02-16-0088-R [ Links ]
Canale MC, Komada KMA and Lopes JRS. 2020. Latency and incubation of ‘Candidatus Liberibacter asiaticus’ in citrus after vector inoculation. Tropical plant pathology 45: 320-326. https://doi.org/10.1007/s40858-019-00311-1 [ Links ]
Dala-Paula BM, Plotto A, Bai J, Manthey JA, Baldwin EA, Ferrarezi RS and Gloria MBA. 2019. Effect of Huanglongbing or Greening Disease on Orange Juice Quality, a Review. Frontiers in Plant Science 9:1976. https://doi.org/10.3389/fpls.2018.01976 [ Links ]
Dawson WO, Garnsey SM, Tatineni S, Folimonova SY, Harper SJ and Gowda S. 2013. Citrus tristeza virus-host interactions. Frontiers in Microbiology 4:88. https://doi.org/10.3389/fmicb.2013.00088 [ Links ]
Dawson WO, Bar-Joseph M, Garnsey SM and Moreno P. 2015. Citrus tristeza virus: making an ally from an enemy. Annual Review of Phytopathology 53: 137-55. https://doi.org/10.1146/annurev-phyto-080614-120012 [ Links ]
Ferretti L, Wymant C, Kendall M, Zhao L, Nurtay A, Abeler-Dörner L, Parker M, Bonsall D and Fraser C. 2020. Quantifying SARS-CoV-2 transmission suggests epidemic control with digital contact tracing Science 368(6491). https://doi.org/10.1126/science.abb6936 [ Links ]
Fishman S, Marcus R, Talpaz H, Bar-Joseph M, Oren Y, Salomon R and Zohar M. 1983. Epidemiological and economic models for spread and control of citrus tristeza virus disease. Phytoparasitica 11: 1. https://doi.org/10.1007/BF02980710 [ Links ]
Flores-Sánchez JL, Mora-Aguilera G, Loeza-Kuk E, López-Arroyo JI, Gutiérrez-Espinosa MA, Domínguez-Monge S, Bassanezi RB, Acevedo-Sánchez G and Robles-García P. 2017. Diffusion model for describing the regional spread of Huanglongbing from first-reported outbreaks and basing an area wide disease management strategy. Plant Disease 101 (7): 1119-1127. https://doi.org/10.1094/PDIS-04-16-0418-RE [ Links ]
Galdeano DM, de Souza Pacheco I, Alves GR, Moreira GL, Rashidi M, Turner D, Levy A and Machado MA. 2020. Friend or foe? Relationship between ‘Candidatus Liberibacter asiaticus’ and Diaphorina citri. Tropical plant pathology 45: 559-571. https://doi.org/10.1007/s40858-020-00375-4 [ Links ]
Ganyani T, Kremer C, Chen D, Torneri A, Faes C, Wallinga J and Hens N. 2020. Estimating the generation interval for COVID-19 based on symptom onset data. MedRxiv 2020.03.05.20031815. https://doi.org/10.1101/2020.03.05.20031815 [ Links ]
Gottwald TR, Aubert B and Zhao X-Y. 1989. Preliminary analysis of citrus greening (Huanglongbing) epidemics in the People’s Republic of China and French Reunion Island. Phytopathology 79: 687-93. http://dx.doi.org/10.1094/Phyto-79-687 [ Links ]
Gottwald TR, da Graça JV and Bassanezi RB. 2007. Citrus Huanglongbing: The pathogen and its impact. Online. Plant Health Progress. https://doi.org/10.1094/PHP-2007-0906-01-RV [ Links ]
Gottwald TR, Garnsey SM and Borbón J. 1998. Increase and patterns of spread of citrus tristeza virus infections in Costa Rica and the Dominican Republic in the presence of the brown citrus aphid, Toxoptera citricida. Phytopathology 88:621-636. https://doi.org/10.1094/PHYTO.1998.88.7.621 [ Links ]
Gottwald TR, Garnsey SM and Riley TD. 2002. Latency of Systemic Infection in Young Field-Grown Sweet Orange Trees Following Graft-Inoculation with Citrus tristeza virus. Fifteenth International Organization of Citrus Virologists Conference Proceedings (1957-2010) 15(15): 48-53. Link: https://escholarship.org/uc/item/9rb677sf [ Links ]
Gottwald TR, Gibson GJ, Garnsey SM and Irey M. 1999. Examination of the effect of aphid vector population composition on the spatial dynamics of citrus tristeza virus spread by stochastic modeling. Phytopathology 89(7): 603-8. https://doi.org/10.1094/PHYTO.1999.89.7.603 [ Links ]
Gottwald T, Parnell S, Taylor E, Poole K, Hodge J, Ford A, Therrien L, Mayo S and Irey M. 2008. Within-tree spatial distribution of Candidatus Liberibacter asiaticus. Proceeding of the International Research Conference Huanglongbing, pp. 270-73. http://www.plantmanagementnetwork.org/proceedings/irchlb/2008/ [ Links ]
Gottwald TR. 2010. Current Epidemiological Understanding of Citrus Huanglongbing. Annual Review of Phytopathology 48: 119-39. https://doi.org/10.1146/annurev-phyto-073009-114418 [ Links ]
Gottwald T, Poole G, McCollum T, Hall D, Hartung J, Bai J, Luo W, Posny D, Duan YP, Taylor E, da Graça J, Polek M, Louws F and Schneider W. 2020. Canine olfactory detection of a vectored phytobacterial pathogen, Liberibacter asiaticus, and integration with disease control. Proceedings of the National Academy of Sciences of the United States of America 117(7): 3492‐3501. https://doi.org/10.1073/pnas.1914296117 [ Links ]
Grandjean D, Sarkis R, Lecoq-Julien C, Benard A, Roger V, Levesque E, et al. 2020. Can the detection dog alert on COVID-19 positive persons by sniffing axillary sweat samples? A proof-of-concept study. PLoS ONE 15(12): e0243122. https://doi.org/10.1371/journal.pone.0243122 [ Links ]
Grandjean D, Marzooqi DHA, Lecoq-Julien C, et al. 2021. Use Of Canine Olfactory Detection For COVID-19 Testing Study On U.A.E. Trained Detection Dog Sensitivity. bioRxiv 2021.01.20.427105. https://doi.org/10.1101/2021.01.20.427105 [ Links ]
Hall DG, Richardson ML, Ammar E and Halbert SE. 2013. Asian citrus psyllid, Diaphorina citri, vector of citrus huanglongbing disease. Entomolgia Experimentalis et Applicata 146: 207-223. https://doi.org/10.1111/eea.12025 [ Links ]
Hernandez GA. 2013. Prevalencia de Toxoptera citricida y tasa de adquisición del Citrus tristeza virus en la Península de Yucatán. Tesis de maestría. Colegio de Postgraduados, México. Link: https://www.repositorionacionalcti.mx/recurso/oai:colposdigital.colpos.mx:10521/2066 [ Links ]
Huang Z, Rundell PA, Guan X and Powell CA. 2004. Detection and Isolate Differentiation of Citrus tristeza virus in Infected Field Trees Based on Reverse Transcription-Polymerase Chain Reaction. Plant Disease 88(6): 625-629. https://doi.org/10.1094/PDIS.2004.88.6.625 [ Links ]
Hughes G, Gottwald TR. 1998. Survey methods for assessment of citrus tristeza virus incidence. Phytopathology 88: 715-723. https://doi.org/10.1094/PHYTO.1998.88.7.715 [ Links ]
Irey MS, Gast T and Gottwald TR. 2006. Comparison of visual assessment and polymerase chain reaction assay testing to estimate the incidence of the Huanglongbing pathogen in commercial Florida citrus. Proceedings of Florida State Horticultural Society 119:89-93 [ Links ]
Jendrny, P., Schulz, C., Twele, F. et al. 2020. Scent dog identification of samples from COVID-19 patients - a pilot study. BMC Infectious Diseases 20: 536. https://doi.org/10.1186/s12879-020-05281-3 [ Links ]
Johansson MA, Quandelacy TM, Kada S, et al. 2021. SARS-CoV-2 Transmission from People Without COVID-19 Symptoms. JAMA Netw Open 4(1): e2035057. https://doi.org/10.1001/jamanetworkopen.2020.35057 [ Links ]
Keremane ML, Ramadugu C, Rodriguez E, Kubota R, Shibata S, Hall DG, Roose ML, Jenkins D and Lee RF. 2015. A rapid field detection system for citrus huanglongbing associated ‘Candidatus Liberibacter asiaticus’ from the psyllid vector, Diaphorina citri Kuwayama and its implications in disease management. Crop Protection 68: 41-48. https://doi.org/10.1016/j.cropro.2014.10.026 [ Links ]
Kobori Y, Nakata T, Ohto Y and Takasu F. 2010. Dispersal of adult Asian citrus psyllid Diaphorina citri Kuwayama (Homoptera: Psyllidae), the vector of citrus greening disease, in artificial release experiments. Applied Entomology and Zoology. https://doi.org/10.1007/s13355-010-0004-z [ Links ]
Korkmaz S, Cevik B, Onder S, Koc K and Bozan O. 2008. Detection of Citrus tristeza virus (CTV) from Satsuma Owari mandarins (Citris unshiu) by direct tissue blot immunoassay (DTBIA), DAS‐ELISA, and biological indexing. New Zealand Journal of Crop and Horticultural Science 36: 239-246. https://doi.org/10.1080/01140670809510240 [ Links ]
Lee JA, Halbert SE, Dawson WO, Roberton CJ, Keesling JE and Singer BH. 2015. Asymptomatic spread of huanglongbing and implications for disease control. Proceedings of the National Academy of Sciences. 112: 7605-7610. https://doi.org/10.1073/pnas.1508253112 [ Links ]
Lee RF, Baker PS and Rocha-Peña MA. 1994. The Citrus Tristeza Virus (CTV). Intl. Inst. Biological Control, CAB International, Silwood Park, UK. 197 p. [ Links ]
Lehtinen S, Ashcroft P and Bonhoeffer S. 2021. On the relationship between serial interval, infectiousness profile and generation time. Journal of the Royal Society Interface 18:20200756. https://doi.org/10.1098/rsif.2020.0756 [ Links ]
Leung KY, Trapman P and Britton T. 2018. Who is the infector? Epidemic models with symptomatic and asymptomatic cases. Mathematical Biosciences 301: 190-198. https://doi.org/10.1016/j.mbs.2018.04.002 [ Links ]
Li Q, Guan X, Wu P, Wang X, Zhou L, Tong Y, et al. 2020. Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus-Infected Pneumonia. The New England Journal of Medicine 382(13): 1199-207. https://doi.org/10.1056/NEJMoa2001316 [ Links ]
Li M, Liu K, Song Y, Wang M and Wu J. 2021. Serial Interval and Generation Interval for Imported and Local Infectors, Respectively, Estimated Using Reported Contact-Tracing Data of COVID-19 in China. Frontiers in Public Health 8: 577431. https://doi.org/10.3389/fpubh.2020.577431 [ Links ]
Manjunath KL, Halbert SE, Ramadugu C, Webb S and Lee RF. 2008. Detection of ‘Candidatus Liberibacter asiaticus’ in Diaphorina citri and its importance in the management of citrus huanglongbing in Florida. Phytopathology 98(4): 387-396. https://doi.org/10.1094/PHYTO-98-4-0387 [ Links ]
Mavrodiev EV, Turskly ML, Mavrodiev NE, Ebach MC and Williams DM. 2020. On classification and taxonomy of Coronaviruses (Riboviria, Nidovirales, Coronaviridae) with the special focus on severe acute respiratory syndrome related coronavirus 2 (SARS-Cov-2). https://doi.org/10.1101/2020.10.17.343749 [ Links ]
Mettler SK, Kim J and Maathuis MH. 2020. Diagnostic serial interval as a novel indicator for contact tracing effectiveness exemplified with the SARS-CoV-2/COVID-19 outbreak in South Korea. International Journal of Infectious Diseases 99: 346-351. https://doi.org/10.1016/j.ijid.2020.07.068 [ Links ]
Mora-Aguilera G, Ochoa-Martínez DL, Gutiérrez-Espinosa MA, Villegas MA, Álvarez-Ramos R, Jasso J, Góngora-Canul CC, Ruiz-García N, Rivas-Valencia P, Loeza-Kuk E, Palacios TC, Pérez Molphe-Bach E, Febres V, Moore G, Moreno P, Mendoza A and Quiroz J. 2005. Citrus tristeza closterovirus: consideraciones biológicas y epidemiológicas. pp. 1-25. In: Memorias del IX Simposio Internacional de citricultura. 19-23 abril. Cd. Victoria, Tamaulipas. México. [ Links ]
Mora-Aguilera G. 2008. Bases de estudios epidemiológicos para el manejo de patosistemas agrícolas. Taller internacional de vigilancia epidemiológica fitosanitaria para el pronóstico de plaga. México. Link: http://langif.uaslp.mx/documentos/presentaciones_internacionl/01/drmora.pdf [ Links ]
Moreno P, Ambros S, Albiach-Marti MR, Guerri J and Pena L. 2008. Citrus tristeza virus: a pathogen that changed the course of the citrus industry. Molecular Plant Pathology 9: 251-268. https://doi.org/10.1111/j.1364-3703.2007.00455.x [ Links ]
Ng SH, Kaur P, Kremer C, Tan WS, Tan AL, Hens N, Toh MP, Teow KL and Kannapiran P. 2021. Estimating transmission parameters for COVID-19 clusters by using symptom onset data, Singapore, January-April 2020. 27: 582-585. https://doi.org/10.3201/eid2702.203018 [ Links ]
Nishiura H, Linton NM and Akhmetzhanov AR. 2020. Serial interval of novel coronavirus (COVID-19) infections. International Journal of Infectious Diseases 93: 284-286. https://doi.org/10.1016/j.ijid.2020.02.060 [ Links ]
Prather KA, Wang CC and Schooley RT. 2020. Reducing transmission of SARS-CoV-2. Science 368(6498): 1422-1424. https://doi.org/10.1126/science.abc6197 [ Links ]
Qian G, Yang N, Ma AHY, Wang L, Li G, Chen X, et al. 2020. A COVID-19 Transmission within a family cluster by presymptomatic infectors in China. Clinical Infectious Diseases 71:861-2. https://doi.org/10.1093/cid/ciaa316 [ Links ]
Rimbaud L, Dallot S, Delaunay A, Borron S, Soubeyrand S, Thébaud G and Jacquot E. 2015. Assessing the Mismatch Between Incubation and Latent Periods for Vector-Borne Diseases: The Case of Sharka. Phytopathology 105(11): 1408-16. https://doi.org/10.1094/PHYTO-01-15-0014-R [ Links ]
Rivas-Valencia P, Loeza-Kuk E, Domínguez-Monge S and Lomas-Barrié CT. 2017. Infección crónica del virus de la tristeza de los cítricos en árboles de Citrus sinensis/C. aurantium en un régimen térmico restrictivo en Yucatán. Revista Chapingo. Serie horticultura 23(3): 188-202. https://doi.org/10.5154/r.rchsh.2016.11.028 [ Links ]
Rivas-Valencia P, Domínguez-Monge S, Santillán-Mendoza R, Loeza-Kuk E, Pérez-Hernández O, Rodríguez-Quibrera C and Lomas-Barrié C. 2020. Severe Citrus tristeza virus Isolates from Eastern Mexico Are Related to the T36 Genotype Group. American Journal of Plant Sciences 11: 1521-1532. https://doi.org/10.4236/ajps.2020.1110110. [ Links ]
Rocha-Peña MA, Lee RF, Lastra R, Niblett CL, Ochoa-Corona FM, Garnsey SM and Yokomi RK 1994. Citrus Tristeza Virus and Its Aphid Vector Toxoptera citricida: Threats to Citrus Production in the Caribbean and Central and North America. Plant Disease 79(5): 437-444. https://doi.org/10.1094/PD-79-0437 [ Links ]
Rosa C, Polek M, Falk BW and Rowhani A. 2007. Improved Efficiency for Quantitative and Qualitative Indexing for Citrus tristeza virus and Citrus psorosis virus. Plant Disease 91(9): 1089-1095. https://doi.org/10.1094/PDIS-91-9-1089 [ Links ]
Rothman KJ, Lash T and Greenland S. 2012. Modern Epidemiology, third ed. Lippincott Williams & Wilkins. ISBN: 9781451190052. 758 p. [ Links ]
Ruiz-Ruiz S, Moreno P, Guerri J and Ambrós S. 2007. A real-time RT-PCR assay for detection and absolute quantitation of Citrus tristeza virus in different plant tissues. Journal of Virological Methods 145: 96-105. https://doi.org/10.1016/j.jviromet.2007.05.011 [ Links ]
Saponari M, Manjunath K and Yokomi RK. 2008. Quantitative detection of Citrus tristeza virus in citrus and aphids by real-time reverse transcription-PCR (TaqMan). Journal of Virological Methods 147(1): 43-53. https://doi.org/10.1016/j.jviromet.2007.07.026 [ Links ]
Spreen TH, Baldwin J and Futch SH. 2014. An Economic Assessment of the Impact of Huanglongbing on Citrus Tree Plantings in Florida, HortScience 49(8): 1052-1055. Retrieved Feb 15, 2021, from https://journals.ashs.org/hortsci/view/journals/hortsci/49/8/article-p1052.xml [ Links ]
te Beest DE, Wallinga J, Donker T and van Boven M. 2013. Estimating the generation interval of influenza A (H1N1) in a range of social settings. Epidemiology 24(2): 244-50. https://doi.org/10.1097/EDE.0b013e31827f50e8 [ Links ]
Torres-Pacheco I, Lopez-Arroyo JI, Aguirre-Gómez JA, Guevara-González RG, Yanez-López R, Hernández-Zul MI and Quijano-Carranza JA. 2013. Potential distribution of Diaphorina citri (Hemiptera: Psyllidae) vector of huanglongbing in Mexico. Florida Entomologist 96: 36-47 https://doi.org/10.1653/024.096.0105 [ Links ]
Wei W, Li Z, Chiew C, Yong S, Toh M and Lee V. 2020. Presymptomatic transmission of SARS-CoV-2-Singapore, January 23 March 16, 2020. MMWR Morb Mortal Wkly Rep. 69:411-5. https://doi.org/10.15585/mmwr.mm6914el [ Links ]
Wu Y, Chen C and Chan Y. 2020. The outbreak of COVID-19: An overview Journal of Chinese Medical Association 83: 217-220. https://doi.org/10.1097/JCMA.0000000000000270 [ Links ]
Yokomi R, Lastra R, Stoetzel MB, Damsteegt VD, Lee RF, Garnsey SM, Gottwald TR, Rocha-Peña MA and Niblett CL. 1994. Establishment of the brown citrus aphid (Homoptera: Aphididae) in Central America and the Caribbean basin and Transmission of citrus tristeza virus. Journal of Economic Entomology 87: 1078-1085. https://doi.org/10.1093/jee/87.4.1078 [ Links ]
Recibido: 03 de Abril de 2021; Aprobado: 29 de Abril de 2021











 texto en
texto en 


