En 2018, México fue el cuarto productor de naranja (Citrus sinensis) en el mundo con 4.7 millones de toneladas, de las cuales el 98% se destinó al consumo interno (SIAP, 2019; Rivera-López et al., 2020). Sin embargo, la producción histórica no exhibe el potencial productivo nacional y representa una citricultura altamente heterogénea determinada por la diversidad tecnológica, régimen hídrico, factores edáficos y problemas fitosanitarios. Diversos programas federales y/o estatales se han implementado para impulsar la productividad e incluso promover la reconversión citrícola. En particular, en las últimas dos décadas se han establecido campañas fitosanitarias nacionales para el control del Citrus tristeza virus (CTV) y Candidatus Liberibacter asiaticus (CLas) (Flores-Sánchez et al., 2017; Mora-Aguilera et al., 2014b). Estos patógenos poseen alto potencial epidémico por la capacidad de dispersión y adaptación agroecológica de sus vectores, condición infectiva sistémica y su variabilidad genética (Flores-Sánchez et al., 2017; Domínguez-Monge et al., 2014; Rivas-Valencia et al., 2010). El CTV y recientemente CLas, han causado fuertes pérdidas productivas en Brasil, Argentina, EUA, Venezuela y otros países, y han modificado los modelos productivos citrícolas mundiales (Flores-Sánchez et al., 2015; Dowson et al., 2015). En México, CLas, causante de la enfermedad denominada huaglongbing o HLB, es una bacteria restringida al floema transmitida eficientemente por Diaphorina citri (Hemyptera: Liviidae). En este país, la expresión de síntomas en C. sinensis está influenciada por el portainjerto y la variante de secuencia de la bacteria pudiendo incluso ser asintomática en campo (Martínez-Bustamante et al., 2015). Por el contrario, la expresión de síntomas en cítricos agrios es de mayor intensidad pero también dependiente de la genética del portainjerto y patógeno. Así, en C. aurantifolia/C. macrophylla se manifiesta clorosis foliar asimétrica, aborto de semillas y deformación de frutos, típicamente reportadas en naranjos de Brasil y Florida, pero además exhibe amarillamiento generalizado de follaje (Esquivel-Chávez et al., 2015; Robles-González et al., 2013; Esquivel-Chávez et al., 2012).
CTV, causal de la enfermedad ‘tristeza de los cítricos’, es un closterovirus asociado al floema y transmitido en forma semipersistente por un complejo de áfidos vectores (Hemyptera: Aphididae) entre los que destacan Aphis gossypii y Toxoptera citricida en México (Dowson et al., 2015; Loeza-Kuk et al., 2011). Contrario al escenario epidémico de los 30´s en Sudamérica, caracterizado por marchitez (tristeza) y muerte rápida de aproximadamente 15 millones árboles C. sinensis/C. aurantium, en México la condición predominante ha sido asintomática y subendémica a partir de la detección del patógeno en los 80’s (G. Mora-Aguilera, 2018. Comunicación personal; Rivas-Valencia et al., 2010). Modelos simulativos demuestran que la selección de razas moderadas ha operado en este virus en detrimento de razas severas manteniendo una estructura poblacional relativamente estable con limitada variabilidad genotípica (G. Mora-Aguilera, 2018. Comunicación personal). Sin embargo, este aparente equilibrio puede ser temporalmente alterado con introducción de nuevas razas, desbalances inducidos por factores climáticos y microbiológicos, como es el caso del reciente brote reportado en el centro de Veracruz (Martínez-Bustamante et al., 2020). La dificultad de controlar estos patógenos con estrategias convencionales (Mora-Aguilera et al., 2014a; Loeza-Kuk et al., 2011), y la necesidad de producir materiales genéticos resistentes en menor tiempo posible (CTV) o ante la ausencia de fuentes de resistencia viables para estos patógenos (CLas), propició en los 90’s la obtención exitosa de cítricos transgénicos, primero en CTV y posteriormente en CLas (Soares et al., 2020; Loeza-Kuk et al., 2011; Gutiérrez-Espinosa et al., 1997). Sin embargo, regulaciones de bioseguridad ha limitado su implementación comercial. Nuevas tecnologías genómicas han favorecido el estudio de genes endógenos del hospedero, en oposición a un gen exógeno implícito en la tecnología transgénica, explotando la expresión contrastante de síntomas en función de genética del patógeno implicado en la infección y la composición injerto/portainjerto. Este fenómeno natural, conocido como resistencia sistémica adquirida puede ser potenciado a través de estudios transcriptómicos mediante análisis de expresión diferencial a nivel de ARNm de genes que responden a un proceso patogénico respecto a genes de referencia del hospedero o ‘housekeeping’ (Rawat et al., 2017; Fu et al., 2016; Mafra et al., 2013). Estos últimos genes son esenciales para la fisiología de la planta por lo que su expresión no es dependiente, teóricamente, de un proceso infeccioso (Yan et al., 2012; Mafra et al., 2012; Xu et al., 2008; He et al., 2006). En un trabajo previo se logró clonar tres genes endógenos que podrían eventualmente conferir resistencia a patógenos en cítricos, incluyendo a CLas y CTV, a través de su sostenida expresión en altas concentraciones en las plantas cisgénicas induciendo resistencia con sus propios genes (Ventura-Medina et al., 2019). Una etapa fundamental en este enfoque cisgénico es cuantificar la expresión de un gen endógeno potencial en respuesta a la interacción planta-patógeno, lo cual es dependiente de la correcta selección de un gen de referencia (Yan et al., 2012; Mafra et al., 2012). En este contexto, este trabajo tuvo como objetivo desarrollar un protocolo que permitiera evaluar la expresión de cinco genes de referencia con base a criterios de eficiencia y estabilidad en el proceso de amplificación por RT-qPCR respecto al gen endógeno CDR13 transcrito en respuesta a la infección de CLas y CTV en plantas C. sinensis/C. aurantium para futuros estudios de resistencia sistémica adquirida en las condiciones citrícolas y fitosanitarias mexicanas.
Material vegetal. En mayo 2019 se seleccionaron nueve plantas de C. sinensis injertadas en C. aurantium provenientes de la colección COLPOS-INIFAP establecida en el Instituto Nacional de Investigaciones Forestales Agrícolas y Pecuarias, Campo Experimental Tecomán (INIFAP-CE Tecomán). Este material se mantuvo durante la investigación en condiciones de invernadero en COLPOS Montecillo. El experimento incluyó tres plantas por cada condición sanitaria: a) CTV positivas, b) CLas positivas y c) sanas. La condición enferma y sana se confirmó por qPCR en el laboratorio de Fruticultura, COLPOS. Las plantas enfermas se inocularon en 2014 con inóculo procedente de Jalisco y Puebla, respectivamente. Las plantas sanas fueron de 18 meses de edad. La muestra vegetal empleada para la extracción de ácidos nucleicos consistió de ocho hojas maduras por planta provenientes de diferente orientación y estrato.
Extracción, cuantificación y calidad de ARN. Para la extracción de ARN total se empleó 0.1 g de nervadura central de hojas de naranjo (C. sinensis) mediante el protocolo de CTAB al 2%. Se cuantificó el rendimiento y calidad de ácidos nucleicos totales empleando el NanoDrop 2000 (Thermo Fisher Scientific). Se verificó que la tasa de densidad óptica a 260/280 y 260/230 nm de longitud de onda se mantuviera en el rango 1.8-2.0 para garantizar ausencia de contaminación por proteínas o fenoles. La extracción de ácidos nucleicos totales permitió obtener concentraciones de 200-1000 ng µL-1 a partir de nervadura central foliar de C. sinensis (Cuadro 1). En todas las muestras las cantidades obtenidas fueron suficientes para homogenizar una submuestra de 50 ng µL1. Esta etapa fue indispensable para la correcta comparación de la expresión diferencial entre los genes evaluados evitando sobre o subestimarlos. La tasa de densidad óptica (260/280) estuvo en el rango 1.93-2.01 (Cuadro 1), lo cual implicó que la calidad y pureza de los ácidos nucleicos estuvo dentro de rango óptimo. Valores inferiores a 1.8 indican presencia de proteínas, fenoles u otros contaminantes (Taylor et al., 2015).
Cuadro 1 Cantidad y calidad de ARN total extraído a partir de nervadura central de una muestra foliar compuesta por planta de Citrus sinensis/C. aurantium.
| Material VegetalX | Conc. Ácido Nucleico (ng µL-1) | Tasa 260/280 | Tasa 260/230Z |
|---|---|---|---|
| Planta sana/R1 | 976.2 | 2.02 | 1.96 |
| Planta sana/R2 | 779.3 | 1.93 | 1.92 |
| Planta sana/R3 | 628.2 | 1.94 | 1.94 |
| +CLas (Jalisco)/R1 | 1186.7 | 2.01 | 1.97 |
| +CLas (Jalisco)/R2 | 1070.5 | 1.94 | 1.8 |
| +CLas (Jalisco)/R3 | 236.2 | 1.94 | 1.9 |
| +CTV (Puebla)/R1 | 750.3 | 1.94 | 1.86 |
| +CTV (Puebla)/R2 | 983.2 | 1.95 | 1.93 |
| +CTV (Puebla)/R3 | 756.7 | 1.97 | 1.81 |
x CLas: Candidatus Liberibacter asiaticus; CTV: Citrus tristeza virus; R1-3: repetición.
z Tasa de densidad óptica obtenida con dos longitudes de onda luz UV.
Retrotranscripción. Para síntesis de ADNc se empleó 50 ng µL-1 de ARN previa homogenización con agua libre de nucleasas. La retrotranscripción con oligos dT se realizó en dos etapas mediante un protocolo no comercial. Se realizaron cinco ensayos con el fin de optimizar la concentración de ADNc (Cuadro 2). La mezcla de reacción de la primera etapa se incubó a 85 °C por 3 min en un termociclador Bio-Rad T100™Thermal Cycler. Transcurrido este tiempo las muestras fueron colocadas en hielo durante 5 min.
Cuadro 2 Reactivos y volumen empleado en cinco ensayos para generar una mezcla óptima de reacción (1X) aplicable a la primera y segunda etapa de retrotranscripción (RT) a par tir de ARN total obtenido de tejido foliar de C. sinensis con tres condiciones sanitarias.
| Primera etapa de retrotranscripción | |||||
|---|---|---|---|---|---|
| Reactivos | Ensayo 1 | Ensayo 2 | Ensayo 3 | Ensayo 4 | Ensayo 5 |
| RNA (50 ng µL-1) | 3.00y | 3.0 | 3.50 | 3.00 | 3.00 |
| zOligo (dT)15 Iniciador (50 mM) | 0.25 | 0.3 | 0.50 | 0.50 | 0.50 |
| H2O libre de nucleasas | 10.50 | 9.2 | 13.25 | 14.35 | 8.75 |
| Volumen final (µL) | 13.75 | 12.5 | 17.25 | 16.85 | 12.25 |
| Segunda etapa de retrotranscripción | |||||
| zBuffer-RT (5X) | 4.00 | 4.0 | 4.50 | 4.20 | 4.50 |
| zdNTP Mix (10 mM) | 1.00 | 1.2 | 1.50 | 1.20 | 1.50 |
| zM-MLV (2000 U µL-1) | 1.00 | 2.0 | 1.00 | 1.50 | 1.50 |
| zRNasin(40 U µL-1) | 0.25 | 0.3 | 0.25 | 0.25 | 0.25 |
| Volumen final (µL) | 20.00 | 20.0 | 25.00 | 25.00 | 20.00 |
y Todas las cantidades por ensayo en μL.
z Proveedor: Promega Corp. USA.
Posteriormente, a cada tubo se adicionó el volumen correspondiente a la mezcla de reacción de la segunda etapa. El ensayo 1 y 2 se incubaron a 44 °C por 60 min, mientras que los ensayos 3-5 se incubaron a 44 °C por 100 min. En todos los ensayos se desactivó la retrotranscriptasa a 92 °C por 10 min. El ADNc obtenido se mantuvo a -20 °C y se cuantificó por espectrofotometría empleando el NanoDrop 2000 para evaluar el rendimiento obtenido.
La síntesis de ADNc se realizó exitosamente incubando a 44 °C por 100 min mediante la configuración del ensayo 5. Este ensayo prolongó el tiempo de síntesis enzimática lo cual permitió obtener concentraciones promedio de 1069 ng µL-1. Los ensayos restantes generaron rendimientos promedio menores con 666-680 ng µL-1 (ensayo 1 y 4) y 865-880 ng µL-1 (ensayos 2 y 3). Un óptimo rendimiento garantiza la cantidad suficiente de ADNc para implementar RT-qPCR simultáneos con varios iniciadores asociados a múltiples genes. También reduce reactivos y consumibles empleados por muestra. Por otra parte, el Oligo-dT es empleado en 40% de ensayos RT-qPCR debido a que se hibrida a las colas de poly A de ARNm en comparación con un iniciador aleatorio alternativo. La combinación de ambos iniciadores se considera la mejor opción de optimización (Taylor et al., 2015).
Genes de referencia. Cinco genes de referencia o constitutivos se seleccionaron bibliográficamente debido a su condición altamente conservada en organismos eucariotas. Estos genes tienen expresión continua e independiente de una condición fitosanitaria (Cuadro 3). Posteriormente, se evaluó su viabilidad para normalizar la expresión de genes potencialmente asociados con resistencia adquirida bajo las condiciones experimentales de este trabajo. Para este fin se empleó el gen endógeno CDR13, el cual se ha asociado con este tipo de resistencia en cítricos (Rawat et al., 2017). Los iniciadores o cebadores empleados para detectar y cuantificar el producto transcrito de este gen fueron F: CAAGCTGATATAATACCCAATATCGGAG, y R: GAGGCTCGCACTGCGT (Rawat et al., 2017; Fu et al., 2016).
Cuadro 3 Iniciadores empleados para amplificar el transcrito primario de cinco genes constitutivos de eucariotas selec cionados para evaluar su implementación como genes de referencia en un protocolo RT-qPCR para analizar la expresión génica inducida por la infección de CTV y CLas en C. sinensis/C. aurantium.
| Gen | zIniciador 5´- 3´ | Función del gen | Referencia |
|---|---|---|---|
| ACTINA | F:CTGAGCGTGGCTA | Regula la interacción entre filamentos de actina y otras fibras de proteínas que actúan como agentes de unión | Fu et al., 2016 |
| TTCCTTC | |||
| R:GACTTCAGGACAG | |||
| CGGAAAC | |||
| Gliceraldehído- 3-fosfato deshidrogenasa | F:GGAAGGTCAAGAT | Produce la enzima que interfiere en óxido-reducción del proceso de glicólisis | Quiñones et al., 1995 He et al., 2006 Mafra et al., 2013 |
| CGGAATCAA | |||
| (GAPDH) | R:CGTCCCTCTGCAA | ||
| GATGACTCT | |||
| F:TTGGAAACTCTTT | Producen la subunidad 18S del RNA ribosomal involucrado en la síntesis de proteínas | Quiñones et al., 1995 He et al., 2006 Yan et al., 2012 | |
| CGCCACT | |||
| 18S rRNA genes | R:AGCAGACCTGGCT | ||
| ATTATACGACTG | |||
| Citocromo C Oxidasa | F:GTATGCCACGTCG | Sintetiza la enzima que cataliza la cadena de trasporte de electrones de la unidad citocromo c- oxidasa a la molécula de oxígeno y es clave en la respiración celular | Quiñones et al., 1995 He et al., 2006 Li et al., 2006 |
| CATTCCAGA | |||
| (COX) | R:GAATGCCCTTAGC | ||
| AGTTTTGGCT | |||
| F:TTGGAAACTCTTT | Genera la proteína relacionada con el reconocimiento de sustrato de la ubiquitina ligasa | Xu et al., 2008 Mafra et al., 2012 Chen et al., 2018 | |
| CGCCACT | |||
| F-BOX | R:AGCAGACCTGGCT | ||
| ATTATACGACTG | |||
| zF: ´forward’; R:’reverse’ | |||
zF: ´forward’; R:’reverse’
Optimización de RT-qPCR. La técnica RT-qPCR se empleó para amplificar y cuantificar el transcrito primario (en su forma complementaria ADNc) de los cinco genes de referencia y del gen de endógeno CDR13, para lo cual se empleó el equipo C1000 Touch™ (Bio-Rad). Con fines de optimizar la técnica, por cada gen se probaron 20 y 10 µL de volumen final de reacción. Ambas mezclas contenían 1X de SsoAdvanced Universal SYBR® Green Supermix (2X). Así mismo, se evaluaron 200, 250 y 500 nM de cada iniciador y 50 ng de ADNc (Cuadro 3). La configuración del experimento consistió en tres replicas por cada muestra obtenida por planta y condición fitosanitaria. Cada replica se usó para amplificar el transcrito de cada gen de referencia y el gen endógeno. Se incluyó el mismo número de testigos negativos por cada iniciador. En total, el experimento consistió de 135 pruebas RT-qPCR. Se evaluó la eficiencia de amplificación de cada iniciador mediante un rango dinámico de 1x103 - 1x10-1 ADNc con factor de dilución 1:10. Se evaluaron dos programas de termociclaje: el primero consistió de desnaturalización a 95 °C por 30 s, seguido de 40 ciclos 95 °C por 5 s y un gradiente de temperatura de anillamiento, para lo cual se evaluó el rango 57-60 °C por 30 s. El segundo programa incluyó desnaturalización a 95 °C por 3 min, 30 ciclos de 94 °C por 30 s y anillamiento a 57 °C por 40 s. Para ambos programas de amplificación se evaluaron tres rangos de temperatura para generar curvas de disociación con incrementos de 0.5 °C/ciclo: A) 70-85 °C; B) 60-95 °C, y C) 65-95 °C.
La forma de curva y valores bajos del umbral de amplificación (Ct) para cada muestra, así como la identificación de la temperatura óptima de disociación Td mediante la conformación de un solo pico de amplificación fueron considerados para optimizar el protocolo RT-qPCR. Además de Td, se verificó la amplificación específica de un simple fragmento esperado, sin la formación de dímeros, mediante electroforesis con gel de agarosa al 1.5% preparado en solución buffer TBE 1X, el cual fue sometido a 110 voltios durante 40 min. Finalmente se visualizó en el fotodocumentador UVP Bioimaging Systems.
El protocolo óptimo monoplex RT-qPCR incluyó un volumen final de reacción de 10 uL con 500 nM por iniciador y 1X de SsoAdvanced Universal SYBR® Green. Esto permite reducir la cantidad de reactivos sin afectar el Ct o Td. Sin embargo, debido a la precisión requerida en la cuantificación el empleo de sondas puede ser otra opción al SYBR® Green. El perfil térmico elegido incluyó desnaturalización inicial a 95 °C durante 30 s, seguido de 40 ciclos de amplificación a 95 °C durante 5 s y 60 °C durante 30 s.
La Td se estimó con la temperatura asociada al pico único de la curva de disociación para cada iniciador de los transcritos de GAPDH (81 °C), F-BOX (83.5) y ACTINA (84) (Figura 1A, B). El iniciador asociado al gen 18S rRNA conformó un pico moderado a diferentes temperatura para las muestras vegetales sanas e infectadas con CTV y CLas contrario a lo que se esperaría de un gen de referencia (Figura 1B). En el caso de COX no se conformó una curva de disociación por lo que ambos se eliminaron para su posible uso como genes de referencia. El gen putativo de resistencia CDR13 tuvo una Td de 81 °C (Figura 1C). La curva de disociación con pico único indicó que no existen problemas de hibridación específica, concentración del iniciador o condiciones ineficientes de reacción del iniciador (Taylor et al., 2015).
Las curvas de amplificación con los iniciadores asociados a ACTINA y GADPH mostraron Ct constantes entre 22.5-26 con 3500-4200 RFU (Unidades relativas de fluorescencia) para las muestras vegetales sanas e infectadas con CLas y CTV. (Figura 1D), mientras que el gen F-BOX presentó Ct entre 25-27 con similar RFU (Figura 1E). El iniciador relativo al gen 18S rRNA tuvo Ct tardíos de 35-37 con 600 RFU (Figura 1E), congruente con la curva de disociación inadecuada, lo que confirmó su eliminación. Contrario a lo esperado, en todos los genes’ muestras de cítricos infectados con CTV exhibió los Ct´s más tardíos (25-27) seguido por las muestras sanas (24-25), indicando posible efecto de la infección en la expresión de estos genes. La curva de amplificación con el iniciador relativo al gen CDR13 tuvo un Ct de 23 para las tres repeticiones (Figura 1F). La visualización en gel de agarosa de fragmentos amplificados por cada repetición se muestra con productos obtenidos con los iniciadores asociados a los genes ACTINA y GADPH. Una banda simple, sin productos secundarios o dímeros, confirmaron la especificidad de los iniciadores empleados (Figura 1D).
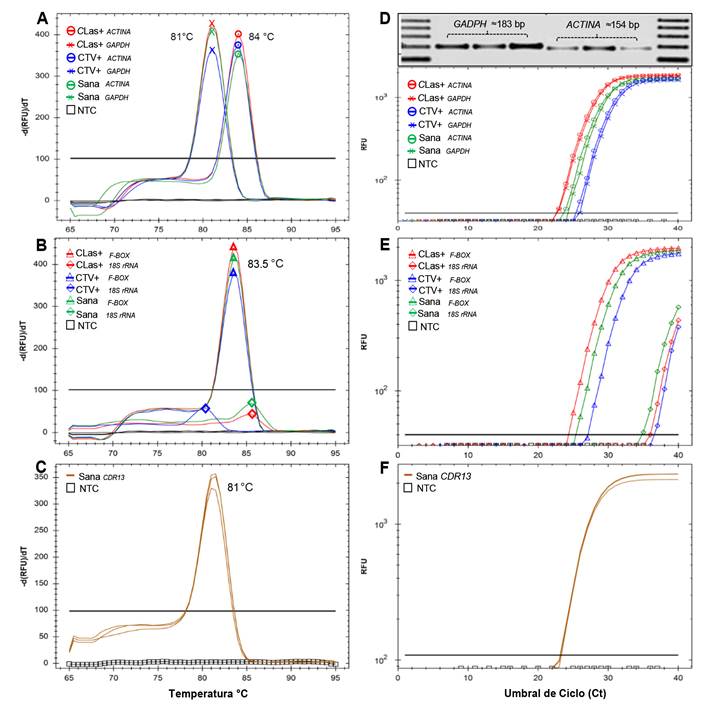
Figura 1 Curvas de disociación (A, B, C) y amplificación (D, E, F) por RT-qPCR empleando SYBR® Green para iniciadores de productos transcritos de los genes GAPDH y ACTINA (A, D), F-BOX y 18S rRNA (B, E), y CDR13 (C, F), en muestras foliares compuestas de C. sinensis/C. aurantium con tres condiciones fitosanitarias: sana (verde y café), positiva a CLas (rojo) y positiva a CTV (azul). Gel de agarosa al 1.5 % con fragmentos amplificados con iniciadores relativos a los genes GAPDH y ACTINA (D, recuadro superior).
RT-qPCR curva estándar. El rango dinámico se determinó con cuatro factores de dilución base 10 de ADNc (1x103-1x100 ng µL-1), cada uno con tres repeticiones, para establecer la cantidad óptima de ADNc requerida para la reacción RT-qPCR y para estimar la eficiencia (E) (Taylor et al., 2015). Este proceso se ilustra con los iniciadores relativos a los genes ACTINA y CDR13 (Figura 2A-D).
Para estos genes, la concentración de ADNc tuvo una relación inversa con valores Ct (Figura 2 A, C), permitiendo ajuste a un modelo de regresión lineal simple con pendientes negativas de -3.33 (r2 = 0.99) y -3.49 (r2 = 0.99), y eficiencias (E) de 99.5 y 93.3 %, respectivamente. Con la línea ajustada, se determinó 1 ng µL-1 como umbral mínimo de concentración ADNc en la muestra vegetal para detectar la transcripción del gen ACTINA con Ct = 32-33 y de CDR13 con Ct = 33.5-34 (Figura D, E). Las eficiencias de reacción estimadas estuvieron dentro del rango 90-110 % considerado aceptable (Taylor et al., 2015). Estos resultados son ilustrativos del procedimiento por lo que el rango dinámico debe ser estimado por cada gen de interés ya que la expresión génica no es homogénea (Mafra et al., 2012; Yan et al., 2012).
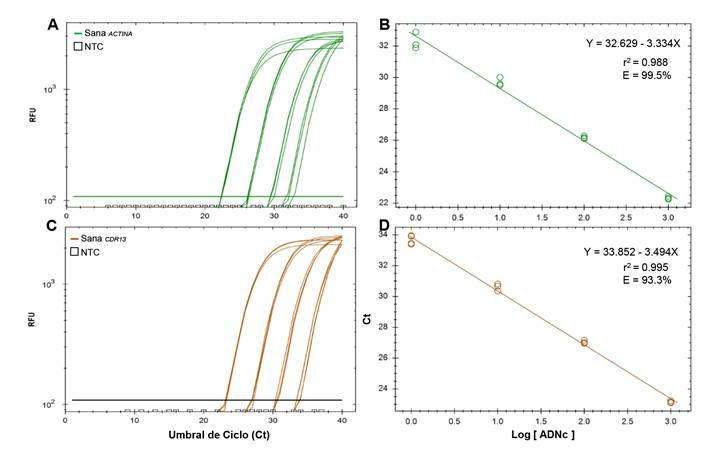
Figura 2 Curvas de estandarización y ajuste a un modelo de regresión lineal simple de las reacciones RT-qPCR obtenidas de cuatro factores de dilución (1x103-1x100) de ADNc empleado como templete para iniciadores asociados a la expresión de ACTINA (A, B) y CDR13 (C, D). E= eficiencia de la reacción.
Selección de genes de referencia. Con base en la cuantificación relativa obtenida con el protocolo RT-qPCR óptimo se procedió a analizar la estabilidad de la expresión de GAPDH, ACTINA y F-BOX mediante el programa CFX Manager™ ver 3.1 de Bio-Rad. GAPDH y ACTINA tuvieron el menor valor de M (0.059) por lo que tuvieron la mayor estabilidad con 2.84 (ln 1/M). Esto indica que la expresión de estos genes tuvieron una variabilidad atribuible el proceso experimental pero no a la infección por CLas o CTV lo cual era esperado para un gen de referencia (Figura 3). Sin embargo, M < 0.5 se considera aceptable efectos de normalización. Por la sincronía de GAPDH y ACTINA (Figura 3), el empleo de ambos genes para fines de normalización son recomendables. Mafra y colaboradores (2012) evaluaron un conjunto de genes, entre los que se incluían ACTINA, GAPDH, F-BOX y 18S rRNA en diferentes especies citrícolas y porta injertos. Conclusivamente, identificaron únicamente a F-BOX como el más estable. También se han reportado a los genes 18S rRNA y ACTB (Yan et al., 2012). Estos resultados indican que la selección de genes de referencia, previo a un estudio específico de resistencia genética adquirida, requiere evaluarse en las condiciones experimentales y con el material biológico de interés. Adicionalmente, es fundamental optimizar el protocolo RT-qPCR con el iniciador(es) diseñado o elegido en función al tipo de muestra vegetal y objetivo experimental (Taylor et al., 2015). En este trabajo también se identificó la importancia de estandarizar al máximo las unidades experimentales ya la expresión de genes constitutivos pueden ser influenciados por factores abióticos como estrés hídrico y temperatura.
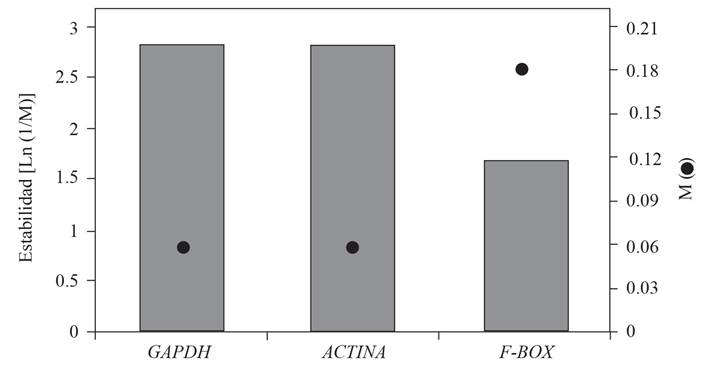
Figura 3 Estabilidad y promedio de variación de expresión entre pares de genes de referencia (M) obtenidos a partir de cuantificación por RT-qPCR de muestras de nervadura central de hojas de naranjo dulce (C. sinensis/C. auran tium) sanas e infectadas con CLas y CTV.
En conclusión, se desarrolló exitosamente un protocolo de laboratorio, no comercial, RT-qPCR para normalizar el gen endógeno CDR13, asociado con resistencia sistémica adquirida en cítricos. Los genes de referencia GAPDH, ACTINA y F-BOX exhibieron curvas de disociación sin expresión de dímeros, Ct ≤ 28 y eficiencias de reacción en el rango de 90-110%. Sin embargo, GAPDH y ACTINA tuvieron la expresión génica más estable (2.83) por lo que estos genes se proponen para normalizar la expresión de genes endógenos potenciales en estudios de resistencia de C. sinensis/C. aurantium en respuesta a la infección de CTV y CLas en condiciones experimentales análogas a las de este estudio.











 texto en
texto en 


