Los bananos y plátanos (Musa spp.) están entre los cultivos más importantes en los países del trópico y el subtrópico. Constituyen una fuente importante de ingresos en casi 135 países productores (FAO, 2017). Lo anterior indica que los bananos y plátanos, representan uno de los productos más importantes para la seguridad alimentaria y la generación de ingresos. Sin embargo, la producción de estos cultivos se encuentra amenazada por el ataque de enfermedades como el Mal de Panamá o marchitez. Esta enfermedad causada por Fusarium oxysporum f. sp. cubense (FOC) representa una de las enfermedades más destructivas y de importancia económica en el género Musa (Ploetz, 2015). Durante los primeros 50 años del siglo XX esta enfermedad causó el reemplazo en las plantaciones del cultivar Gros Michel susceptible a la raza 1, por cultivares Cavendish con importantes transformaciones en la exportación de la industria bananera (Bubici et al., 2019). La introducción de la raza tropical 4 de FOC (RT4) en las plantaciones de cultivares Cavendish representa un gran impacto económico y social en la industria bananera, en América Latina y el Caribe (Ploetz, 2015). En la actualidad no existe control químico sostenible para esta enfermedad. Por ello, en los últimos años existe un consenso general de que la única forma de control efectivo y segura para esta enfermedad lo constituye el mejoramiento genético para la resistencia (Dita et al., 2011). Se han desarrollado estrategias basadas en la biotecnología vegetal para el mejoramiento genético del banano en la obtención de individuos resistentes o tolerantes a la marchitez por Fusarium (Saraswathi et al., 2016). Pero, la búsqueda de nuevos cultivares Cavendish resistentes o tolerantes a la enfermedad continúa siendo una prioridad. Por otra parte, la mayoría de los estudios de campo carecen de resultados a largo plazo que contribuyan a evaluar la eficacia de los genes in situ (Ploetz et al., 2015). En este sentido, el desarrollo de métodos para la selección temprana de individuos resistentes a las enfermedades, con el empleo de la biotecnología constituye un valioso utilitario para los programas de mejoramiento genético de los cultivos. Lo cual se ha convertido en las últimas décadas en un tema necesario y atractivo para los fitomejoradores (Saraswathi et al., 2016). El presente trabajo se realizó con el objetivo de ofrecer una revisión de literatura científica relacionada con la utilización de técnicas biotecnológicas en apoyo al mejoramiento genético para la resistencia a la marchitez por Fusarium en banano, con énfasis en la selección in vitro, ex vitro y en campo para la resistencia al patógeno.
Fusarium oxysporum f. sp. cubense: mecanismo de infección y síntomas de la enfermedad
El género Fusarium constituye el más amplio e importante de la familia Tuberculareacea; y desde el punto de vista taxonómico es uno de los más difíciles de todos los grupos de hongos. Este hongo comprende numerosas especies y diversas formas especiales (f. sp.) dentro de cada especie. Por otra parte, presenta una gran variabilidad y se hace imprecisa su identificación en las distintas especies (Dean et al., 2012). La fase perfecta de F. oxysporum (FOX) no se conoce aún. Pero, la supervivencia y proliferación de esta especie y sus formas especiales existentes dependen de sus esporas asexuales. FOC produce tres tipos de esporas asexuales que incluyen macroconidios, microconidios y clamidosporas en su ciclo de vida (Figura 1 A-C), lo que le permite dispersarse y sobrevivir; además, comparte un ciclo de infección similar a Fusarium oxysporum f. sp. lycopercisi (FOL), agente causal de la enfermedad del marchitamiento del jitomate (Solanum lycopersicum) (Guo et al., 2014). Las clamidosporas son de paredes gruesas, formadas dentro de la hifa o del macroconidio y pueden sobrevivir en ausencia del hospedante por varios años en estado latente o consiguen germinar y crecer como saprófito en restos de plantas no hospedantes para producir nuevas clamidosporas (Nelson et al., 1983). Los conidios de FOC germinan y forman hifas fúngicas bajo diversas condiciones nutricionales y ambientales; las hifas se desarrollan alrededor de las raíces y colonizan su superficie. Posteriormente, penetran la epidermis e invaden; y colonizan los vasos del xilema de la raíz (Figura 1 D-F). Después del proceso de infección en las raíces de las plantas de banano, el hongo crece hacia el rizoma y pseudotallo y causa la muerte del tejido o de toda la planta. Entre la infección inicial por el hongo y los síntomas externos transcurre un tiempo en que el limbo de las hojas se torna amarillo brillante, y se marchita o colapsa alrededor del pseudotallo (Figura 1 G). Mientras que los haces vasculares del pseudotallo y del rizoma de la planta enferma se tornan de color rojizo a marrón (Figura 1 H-I) (Guo et al., 2014).
El hongo puede sobrevivir en ausencia del hospedante por largos períodos de tiempo; en estado latente o pueden germinar o crecer como saprófito en restos de plantas no hospedantes para producir nuevas clamidosporas. Estas a su vez le permiten al patógeno adaptarse a condiciones extremas (Ploetz et al., 2015). Como consecuencia, los genotipos susceptibles no pueden cultivarse en un campo infectado por un período de hasta 30 años (Buddenhagen, 2009). Sin embargo, el proceso de infección puede terminar y no progresar cuando la planta no es susceptible o no está predispuesta por un estrés ambiental. Dong et al. (2012) encontraron que el hospedante resistente responde a una señal en los vasos infestados por la formación de calosa, geles vasculares y tilosas que inmovilizan las esporas alrededor del sitio de almacenamiento; el desarrollo de estas estructuras fue examinado en detalle por microscopía electrónica, finalmente determinaron que la síntesis y liberación de las sustancias fenólicas que producen y lignifican estas estructuras son las que detienen la invasión del patógeno durante la interacción hospedante-patógeno.
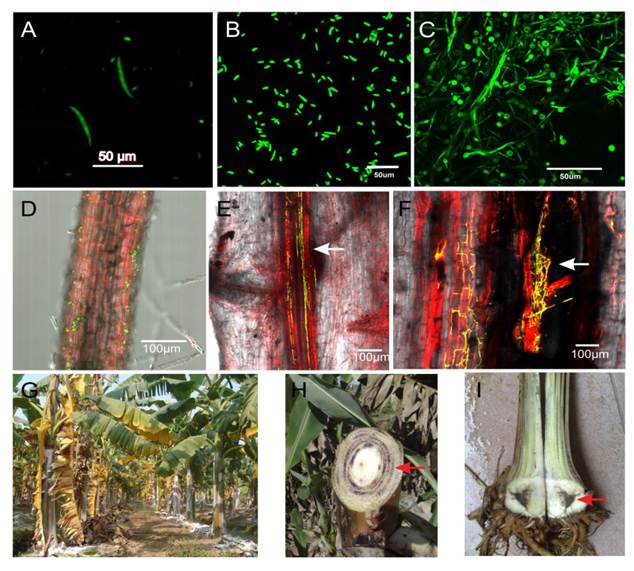
Figura 1 Ciclo de infección de la marchitez vascular en banano causada por Fusarium oxysporum f. sp. cubense. A) macroconidios; B) microconidios; C) clamidospora producida por el aislado FOC marcado con proteína fluorescentes verde; D) acoplamiento de la hifa de FOC a las raíces de banano; E) colonización de los haces vasculares de las raíces de banano, por la hifa de FOC (indicado por la flecha); F) una sección longitudinal de las raíces de banano muestra las hifas del hongo creciendo en los haces vasculares; G) las plantas de banano enfermas con los síntomas dominantes de hojas amarillentas; H-I) los haces vasculares del pseudotallo, y el rizoma de la planta de banano enfermos que se tornan de color rojizo a marrón (indicado por la flecha). Fuente: Guo et al. (2014).
Complejidad patogénica de FOC
La variabilidad de las poblaciones de FOC ha sido diferenciada en base a su patogenicidad donde se han identificado tres razas que afectan a Musa spp.: la raza 1 responsable de la epifitia en las plantaciones de los cultivares Manzano (AAB) y Gros Michel (AAA); la raza 2 patogénica al cultivar Bluggoe (ABB), y a algunos tetraploides (AAAA); y la raza 4 que ataca a los cultivares del subgrupo Cavendish (AAA) y a todos los susceptibles a las razas 1 y 2 (Ploetz, 2006). Esta última fue descubierta por primera vez en 1990 en Taiwán; y fue posteriormente dividida en las cepas tropical y subtropical (Ploetz, 2006). En la actualidad, la raza 4 de FOC se encuentra presente en veinte países de 135 productores de bananos (Martínez et al., 2020). Sin embargo, la designación de razas de FOC era engorrosa y por ello se desarrollaron otros métodos que revelaron su diversidad genética. El análisis de compatibilidad vegetativa constituye una técnica rápida y confiable que la prueba de patogenicidad. Su utilización ha permitido determinar la diversidad de razas de FOC dentro de una región. Los análisis de los grupos de compatibilidad vegetativa (GCV) dividieron los aislados de FOC en 24 GCV (GCV0120 a GCV0126, y GCV0128 a GCV01224) (Ploetz, 2006). Después, marcadores de ácido desoxirribonucleico (ADN) revelaron el origen polifilético de FOC ya que algunos GCV están taxonómicamente más cerca de otras formas especiales de FOX que a otros GCVs de FOC (Fourie et al., 2009). Además, las cepas pertenecientes a diversos GCVs infestan cultivares particulares de banano y, por lo tanto, se agruparon en la misma raza. Lo que sugiere que la patogenicidad hacia un cultivar específico ha evolucionado convergentemente o ha resultado de la transferencia horizontal de genes entre los miembros del complejo de FOX (Ploetz, 2006). En general, los linajes de FOC muestran una notable dicotomía referida como tipos o clados (Groenewald et al., 2006). La genotipificación de alta resolución por análisis de secuenciación usando DArTseq genera lecturas de secuencias cortas después de reducir la complejidad de un genoma por digestión con enzimas de restricción. En este sentido, marcadores de genomas enteros por DArTseq dividieron las 24 cepas de FOC (que representan todos los GCV hasta ahora conocidos) en dos grupos (Cruz et al., 2013). Por otro lado, el análisis del genoma reveló que las estructuras genómicas de los aislados de la raza 1 y la raza 4 eran altamente sinténicas (que ocurren en el mismo cromosoma) con las de FOL cepa Fol4287. Guo et al. (2014) lograron la identificación de genes ortólogos SIX primariamente descritos para el genoma de aislados de FOL mediante el análisis del genoma de aislados de la raza 1 y la RT4 de FOC. En este aspecto, se han diseñado varios protocolos para la detección molecular de aislados de FOC, los cuales han sido revisados de forma exhaustiva por Lin y Lin (2016) y Ying y Yi (2016). De este modo FOC se compone de tres razas, ocho linajes y 24 GCVs. La mayor parte del daño es causado por la RT4 de FOC que se encontraba sólo en Asia. Sin embargo, García-Bastidas et al. (2019) informaron el primer reporte de la RT4 de FOC asociado con la enfermedad fuera del sudeste asiático, en la zona de la Guajira colombiana clasificado como Fusarium odoratissimum (Figura 2). Lo cual representa un alto riesgo para el resto de los países del continente americano. Por lo que se requiere el desarrollo de estrategias para el combate de esta enfermedad; y reducir los riesgos de las afectaciones que puede causar la entrada de esta nueva raza del patógeno en el resto de los países exportadores de América Latina y el Caribe.
Control de la marchitez por Fusarium en banano
La producción perenne del cultivo de banano y la naturaleza policíclica de la enfermedad marchitez por Fusarium impiden el desarrollo de estrategias de manejo eficientes. A pesar de ello, con el conocimiento y las tecnologías disponibles, hoy es más fácil y efectivo implementar políticas cuarentenarias e impedir así la diseminación de patógenos. En relación con ello, en México la SADER (Secretaría de Agricultura y Desarrollo Rural) a través de SENASICA (Servicio Nacional de Sanidad, Inocuidad y Calidad Agroalimentaria) realizan un conjunto de medidas importantes para prevenir la entrada y diseminación de la RT4 de FOC en el país. Dentro de estas medidas encontramos acciones de vigilancia epidemiológica fitosanitaria para la detección oportuna de la RT4; la capacitación de técnicos y productores, con el interés de lograr conciencia en los productores del riesgo de trasladar material vegetativo; y por último, la exploración y monitoreo de las regiones bananeras (Manzo et al., 2014). Otras de las medidas de manejo utilizadas tenemos las siembras anuales escalonadas; la utilización de plantas de cultivo de tejidos; la rotación de cultivo; contar en el terreno de siembra con un buen sistema de drenaje; la incorporación de materia orgánica; y la aplicación del biocontrol mediante Trichoderma harzianum. No obstante, estos métodos de control sólo permiten sembrar estos cultivares por períodos cortos, dado a que el hongo vuelve a devastar las plantaciones (Fu et al., 2017). Por ello, se ha considerado que el único método eficaz de control es sembrar o plantar hospedantes resistentes a la enfermedad (Dita et al., 2011). En este sentido, desde la aparición de la marchitez por Fusarium en los diferentes países productores de banano, se desarrolla un amplio programa de mejoramiento genético para la resistencia a las tres razas de FOC, mediante técnicas convencionales y biotecnológicas con énfasis en los últimos años en la RT4 de FOC.
Técnicas biotecnológicas en apoyo al mejoramiento genético de Musa spp. para la resistencia a FOC
Las mayores posibilidades que brindan las técnicas biotecnológicas están dadas en la mejora de la resistencia a patógenos. Las cuales ofrecen varias ventajas sobre los métodos convencionales por la posibilidad de seleccionar individuos a partir de grandes poblaciones de plantas en un espacio reducido de tiempo. Lo cual aumenta de esta forma la posibilidad de encontrar los caracteres deseados. Por otra parte, permiten un control de la concentración de inóculo, y de las condiciones ambientales que pudieran interferir en los resultados obtenidos. A su vez, permiten el acceso a las plantas libres de la enfermedad; y por último mejoran la eficiencia de los programas de mejoramiento genético de los cultivos (Ortiz y Swennen, 2014). Por estas razones, los resultados de la mejora de bananos y plátanos mediante la biotecnología vegetal se han introducido con mayor rapidez en comparación con los métodos del mejoramiento genético convencional (Li et al., 2015); en donde la micropropagación ha jugado un papel importante, en particular ha mejorado el manejo del germoplasma sano a escala mundial. La variación somaclonal, la mutagénesis in vitro, la transformación genética y la selección temprana en el género Musa se han empleado con éxito en varios programas de mejoramiento genético.
Utilización de la variación somaclonal
La variación somaclonal que comprende los cambios genéticos o epigenéticos inducidos durante la fase de callo de las células vegetales cultivadas in vitro ha sido usada para el mejoramiento genético de varios caracteres agronómicos. Molina et al. (2011) determinaron la respuesta a la susceptibilidad o resistencia a la RT4 de FOC en dos variantes somaclonales de Cavendish (AAA) de Taiwán, dos cultivares comerciales del subgrupo Cavendish, tres cultivares locales, y un híbrido mejorado de Honduras, en un campo previamente infestado con el patógeno en Filipinas. En dicha investigación observaron los mayores valores de incidencia de la enfermedad (100%) en los dos cultivares comerciales Cavendish (Gran Enano y Williams) y en el cultivar local ‘Lakatan’. Sin embargo, no encontraron plantas infectadas a la RT4 de FOC en las variantes somaclonales de Taiwán (‘GCTCV119’ y ‘Formosana’). En otros estudios Molina et al. (2016) compararon cuatro selecciones de variantes somaclonales (‘GCTCV-105’, ‘GCTCV-119’, ‘GCTCV-218’ y ‘GCTCV-219’) de Cavendish (AAA) de Taiwán; y tres cultivares locales importantes de Filipinas: ‘Latundan’ (AAB, subgrupo de Seda), ‘Lakatan’ (AAA, subgrupo Lakatan) y ‘Saba’ (ABB, subgrupo Saba) con el Cavendish comercial Gran Enano (AAA, subgrupo Cavendish) en un suelo severamente infestado con la RT4 de FOC en el sur de Filipinas. Los resultados mostraron susceptibilidad con una incidencia de la enfermedad de 64 y 76% respectivamente en los cultivares comerciales de Gran Enano y ‘Lakatan’ en el primer ciclo de evaluación de la resistencia; y de 79 y 92% respectivamente, en el segundo ciclo. Sin embargo, las variantes somaclonales ‘GCTCV’ manifestaron una incidencia de la enfermedad de 0 a 6% en el primer ciclo; y de 0 a 8% en el segundo ciclo de las pruebas de evaluación de los síntomas de la enfermedad. El cultivar ‘Saba’ mostró una incidencia de la enfermedad del 0% tanto en el primero como en el segundo ciclo de evaluación de la resistencia al hongo. Estos resultados confirmaron la estabilidad de la resistencia a la RT4 de FOC en las variantes somaclonales ‘GCTCV’. Por otra parte, muestran que la selección de variantes somaclonales constituye una herramienta factible en el mejoramiento genético para la búsqueda de resistencia a FOC en banano.
Utilización de la mutagénesis in vitro inducida
Las técnicas mutagénicas que inducen cambios heredables en la constitución genética de una célula mediante la alteración de su ácido desoxirribonucleico (ADN) han sido utilizadas como una herramienta muy eficiente en el mejoramiento genético de plantas. En este sentido, Chen et al. (2013) emplearon la mutagénesis in vitro inducida por el metanosulfonato de etilo para evaluar la obtención de líneas de banano brasileño (Musa spp., AAA) resistentes a la RT4 de FOC. Las plantas regeneradas a partir de las cinco líneas de banano brasileño resistentes a la RT4 del patógeno fueron sometidas a un método de selección in vitro posterior; donde mostraron una reducción en la incidencia de la enfermedad con relación a las plantas utilizadas como control. Mientras que Saraswathi et al. (2016) obtuvieron mutantes de banano del cultivar Rasthali (Silk, AAB) resistentes a la raza 1 de FOC, grupo de compatibilidad vegetativa (GCV) [0124/5]. Estos resultados evidencian que es posible adoptar diferentes técnicas biotecnológicas para el mejoramiento genético del banano y obtener resistencia a FOC.
Utilización de la transformación genética
Los métodos de transformación genética que se utilizan para introducir al interior de las células vegetales, el ADN exógeno que se quiere expresar en la planta constituye otra estrategia biotecnológica para el desarrollo de la resistencia a hongos en cultivos económicamente importantes como el banano. Varios genes han sido utilizados para combatir este patógeno. En este sentido Paul et al. (2011) ensayaron los genes Bcl-xl, Ced-9 y Bcl-2 3’ UTR en el cultivar de banano ‘Lady Finger’ (AA). Los resultados demostraron la sobreexpresión del gen Bcl-2 3’ UTR en las líneas que manifestaron resistencia a FOC. Por otra parte, observaron como característico la apoptosis en plantas hospederas contra patógenos necrotróficos, donde el patógeno provocó la muerte del tejido e incrementó su potencial de crecimiento más rápido. En otro estudio, Ghag et al. (2012) insertaron genes de petunia (Petunia hybrida) que codifican las defensinas PhDef1 y PhDef2 en plantas de banano usando como explantes suspensiones celulares embriogénicas; y como sistema de transformación el empleo de Agrobacterium tumefaciens, se encontraron altos niveles de expresión constitutiva de estas defensinas en plantas élites de banano en el cultivar Rasthali (Musa AAB); que a su vez mostraron resistencia contra la infección de la raza 1 de FOC en estudios tanto in vitro como in vivo.
Por otro lado, Mahdavi et al. (2012) demostraron la expresión de genes de taumatina de arroz (Oriza sativa) en plantas transgénicas de banano con resistencia a la TR4 de FOC. Posteriormente, genes obtenidos de cebolla (Allium cepa) codificantes para una proteína antimicrobiana (Ace-AMP1) fueron introducidos y expresados en bananos transgénicos (Mohandas et al., 2013). Por otra parte, Ghag et al. (2014) desarrollaron un procedimiento de transformación en banano mediada por Agrobacterium para la expresión de siRNAs dirigidos contra genes vitales de FOC. Después de ocho meses de evaluación obtuvieron cinco líneas transgénicas con niveles de resistencia a FOC. Mientras que Zhuang et al. (2016) demostraron el efecto antagónico de la proteína multifuncional B4 del virus Banana bunchy top virus(BBTV) contra F. oxysporum en banano. Lo cual ofrece nuevos conocimientos para el mejoramiento de la resistencia transgénica a Fusarium teniendo en cuenta la interacción virus-hongo. Por otro lado, Dale et al. (2017) obtuvieron dos líneas de Cavendish transgénico: la primera línea fue transformada con el gen RGA2 aislado de un banano diploide resistente a la RT4 de FOC; y la segunda fue transformada con el gen Ced9 derivado de nemátodos. Después de tres años de pruebas en campo demostraron resistencia a la RT4 de FOC.
Como se puede apreciar, en los últimos años la transformación genética constituye la técnica biotecnológica de mayor expectativa para el mejoramiento genético en el cultivo. Sin embargo, los resultados de campo a largo plazo que contribuyan a evaluar la eficacia de los genes in situ no están presentes en la mayoría de los estudios (Ploetz, 2015). Por ello, la comprensión de la interacción hospedero-patógeno en términos de defensa; y las rutas relacionadas con los mecanismos de virulencia permite identificar pasos críticos para desarrollar cultivares resistentes empleando enfoques genéticos.
Selección temprana de resistencia
La selección temprana de la resistencia en plantas frente a diferentes formas especiales (f. sp.) de F. oxysporum ha sido un objetivo primordial en el mejoramiento genético convencional y biotecnológico. La misma se ha incrementado por el avance en los estudios tanto de los procesos de la planta, y la biología de los patógenos como del entendimiento de la interacción planta-patógeno. El empleo de filtrados del cultivo de hongos (FCH) y toxinas e incluso cultivos duales para este proceso son prometedores. El primero y más crucial de todos los pasos al evaluar un nuevo clon de banano es la selección de la resistencia a las diferentes razas de marchitez por FOC (Buddenhagen, 2009). Por lo tanto, constituye una prioridad el desarrollo de métodos eficientes que permitan seleccionar cultivares susceptibles y resistentes a este patógeno, en el mejoramiento genético en el cultivo. Hasta el momento, diferentes grupos de investigadores han desarrollado dos sistemas de selección para la resistencia del banano a FOC: 1) selección ex vitro e in vivo mediante plantación de propágulos de banano en macetas, y en campo con inóculo del hongo natural o artificial; 2) selección in vitro utilizando como agente de selección propágulos o FCH y toxinas. A continuación, se recogen algunas experiencias de la selección de la resistencia del banano a FOC con el uso de los sistemas de selección mencionados.
Evalución ex vitro e in vivo de la resistencia a la RT4 de FOC en propágulos de banano
La selección ex vitro e in vivo para evaluar la resistencia a enfermedades constituyen los principales métodos de selección desde hace muchos años. Consisten en la infección natural con esporas provenientes del hongo y en la actualidad continúan siendo utilizados. Seguidamente, se muestran resultados en la evaluación para resistencia a la RT4 de FOC de cultivares de bananos, plátanos y variantes somaclonales de Cavendish mediante estos sistemas de selección proporcionados por el Instituto de Investigaciones del Plátano de Taiwán (Cuadro 1) (Huang et al., 2005).
Cuadro 1. Resultados en la evaluación para resistencia a la RT4 de FOC de cultivares de bananos, plátanos y variantes somaclonales de Cavendish.
| Cultivar | Lugar | Respuesta a RT4 de FOC | Referencia |
|---|---|---|---|
| ‘FHIA 01’ | FAO, Malasia | Resistente | Huang et al. (2005) |
| ‘FHIA 02’ | International Musa Testing Programme, Phase III (IMTP III) China | Resistente | Huang et al. (2005) |
| ‘FHIA 03’ | Journal of Tropical Crops China | Resistente | Zisi et al. (2009) |
| ‘FHIA 17’ | Papua New Guinea | Resistente | Molina et al. (2011) |
| ‘FHIA 18’ | Journal of Tropical Crops China | Altamente Resistente | Zisi et al. (2009) |
| ‘FHIA 21’ | International Musa Testing Programme, Phase III (IMTP III), China | Resistente | Molina et al. (2011) |
| Huang et al. (2005) | |||
| ‘FHIA 23’ | FHIA: Bananas Database | Susceptible | Houbin et al. (2004) |
| ‘FHIA 25’ | Journal of Tropical Crops China | Altamente Resistente | Zisi et al. (2009) |
| Papua New Guinea | Resistente | Molina et al. (2011) | |
| ‘FORMOSANA’ | Taiwan | Resistente | Hwang y Ko (2007) |
Dita et al. (2011) obtuvieron un bioensayo rápido y confiable para la evaluación de la resistencia a la RT4 de FOC en condiciones de invernadero. Para ello utilizaron un sistema de doble maceta según Mohamed et al. (2000). Las plántulas provenientes del cultivo in vitro del cultivar Gran Enano (grupo AAA) les realizaron cortes a las raíces a una longitud aproximadamente de 40 cm. Después, realizaron el proceso de inoculación de forma individual con tres aislados de la RT4 del hongo (NRRL36114, FOC-115 y BPS3.4) del GCV [01213]; y un aislado de la raza 1, CNPMFO8-R1. La inoculación se realizó mediante la inmersión de la raíz durante 30 minutos en una suspensión de 105 conidios mL-1 del hongo. Las plántulas inoculadas fueron sembradas en macetas de 8 L que contenían como sustrato arena de río estéril; y suplementada con 20 g de granos de maíz pre-colonizados con FOC. La evaluación de la resistencia se realizó a los siete hasta los 40 días después de la inoculación de las plántulas, mediante la evaluación de los síntomas internos y la decoloración del rizoma. Los resultados mostraron que el bioensayo descrito en condiciones controladas de invernadero fue confiable para obtener una respuesta de resistencia en la reacción del huésped a FOC. Además, el bioensayo mostró una herramienta útil para realizar estudios precisos en la interacción planta-patógeno. En este mismo sentido, Li et al. (2015) estudiaron la resistencia a la RT4 de FOC en ocho genotipos silvestres de banano (Musa acuminata subsp. burmannica, M. balbisiana, M. basjoo, M. itinerans, M. nagensium, M. ruiliensis, M. velutina y M. yunnanensis) en condiciones de invernadero y en campo. Para las pruebas en invernadero procedieron a la inoculación de las plántulas de banano provenientes del cultivo in vitro mediante la punción de orificios en la base del pseudotallo; con una suspensión de 5 x 106 esporas mL-1 del hongo. Después de este proceso, realizaron la selección de la resistencia cada dos días hasta los 65 días mediante la evaluación de la expresión de los síntomas internos y externos de la enfermedad en las plántulas. Para los síntomas externos, fue mediante una escala de calificación de cuatro clases: 0 = sin síntomas; 1 = amarillamiento inicial, principalmente en las hojas inferiores; 2 = amarillamiento de todas las hojas inferiores con cierta decoloración de las hojas más jóvenes; 3 = todas las hojas con intenso amarilleamiento o planta muerta. Mientras que los síntomas internos fueron evaluados por medio de una escala basada en la decoloración del rizoma: 0 = sin síntomas, 1 = 1 - 20%, 2 = 21 - 40% y 3 = > 40% de rizoma descolorido. La prueba de selección en campo se realizó en una parcela experimental infestada de forma natural con la RT4 de FOC. La evaluación de expresión de los síntomas externos de la enfermedad se realizó cada dos semanas hasta los 12 meses después de la siembra; y, los síntomas internos se evaluaron al final del experimento (12 meses) de acuerdo a la escala. Los resultados mostraron que existen diferentes fuentes de resistencia a la RT4 de FOC, lo cual constituyen un importante recurso genético para los programas de mejoramiento genético del banano con el interés de obtener cultivares resistentes al marchitamiento para la RT4 de FOC. Sin embargo, se continúan los estudios relacionados a la resistencia a RT4 de FOC como se indican solo algunos.
Selección in vitro con uso de propágulos o filtrados del cultivo del hongo y toxinas
Wu et al. (2010) desarrollaron una metodología de bioensayo in vitro para la temprana detección de la resistencia o susceptibilidad a la RT4 de FOC en seis cultivares de Musa (Brazil Xiangjiao subgrupo AAA, Guangfen No.1 subgrupo ABB, Formosana subgrupo AAA, Nongke No.1 subgrupo AAA, GCTCV-119 subgrupo AAA y Dongguan Dajiao subgrupo AAA). Las plántulas enraizadas in vitro de banano fueron inoculadas en su medio de cultivo mediante una suspensión de 106 conidios mL-1 de FOC. Después de 24 días de la inoculación se realizó la selección de la resistencia al patógeno sobre las plántulas de banano mediante una escala de 0 a 6 grados de severidad, propuesta por los mismos autores. Los resultados mostraron que los dos cultivares susceptibles (Brazil Xiangjiao y Guangfen No.1) presentaron los mayores grados de severidad de la enfermedad, en comparación con los cuatro cultivares resistentes probados (Formosana, Nongke No.1, GCTCV-119 y Dongguan Dajiao). Lo cual evidencia que los prometedores clones resistentes obtenidos mediante técnicas de mejoramiento genético tradicionales pueden ser analizados de forma directa mediante el bioensayo in vitro descrito; este método eficientiza los tiempos dado a que no necesita una etapa de aclimatización para las plántulas. Mientras, Saraswathi et al. (2016) realizaron la selección de los mutantes de banano del cultivar Rasthali (Silk, AAB) con resistencia a la raza 1 del GCV [0124/5] de FOC mediante la selección in vitro. Para ello, utilizaron como agentes de selección el FCH y la toxina (ácido fusárico). Los brotes individuales de los mutantes de banano obtenidos después del tercer o cuarto subcultivo fueron transferidos al medio de cultivo de multiplicación, suplementado con diferentes concentraciones de la toxina (ácido fusárico comercial) (Sigma Aldrich, EE. UU.) (0.0125; 0.025; 0.0375; 0.05 y 0.0625 mM); y a las concentraciones de 3, 4, 5, 6, 7 y 8% del FCH. Además, añadieron el regulador de crecimiento. Después de tres semanas observaron el 50% de supervivencia de los explantes en la concentración de 0.050 mM de la toxina y 7% con el filtrado del cultivo del hongo. En concentraciones superiores observaron una rápida disminución en el crecimiento de los brotes de los mutantes de banano. A los tres meses siguientes, los mutantes seleccionados fueron transferidos para realizar las pruebas en macetas, en condiciones controladas mediante la inoculación al sustrato con una suspensión de esporas de 12 × 109 conidios mL-1 del hongo. Posterior a los seis meses del proceso de evaluación de la resistencia obtuvieron tres mutantes putativos resistentes a la raza 1 de FOC. Los cuales fueron multiplicados in vitro de forma masiva para continuar otros estudios en la interacción.
Es importante señalar que, para el establecimiento de sistemas de selección de la resistencia es necesaria la determinación de una concentración correcta del filtrado del cultivo patógeno o toxina, para la expresión de la actividad fitotóxica diferencial entre variedades. Lo cual incrementa las posibilidades de obtener líneas estables de plantas con resistencia a las enfermedades (Švábová y Lebeda, 2005). Como se puede mostrar, aunque se realicen selecciones in vitro las plantas supuestamente resistentes deben someterse a estudios en campo. La enfermedad causada por FOC tiene períodos relativamente largos de incubación bajo estas condiciones. En otras palabras, desde la infección de las raíces hasta el amarillamiento y colapso del limbo de las hojas (síntomas externos) pueden transcurrir varios meses. Por lo tanto, se debe realizar un seguimiento a un gran número de plantas durante un prolongado tiempo.
Los planteamientos anteriores indican la necesidad del desarrollo de métodos alternativos, rápidos y no destructivos para la selección de la resistencia del banano a FOC en las pruebas de selección de resistencia en condiciones de campo. En este aspecto, Companioni et al. (2003) desarrollaron un procedimiento para la diferenciación foliar de la resistencia y susceptibilidad de cultivares de banano a la raza 1 de FOC, GCV [01210] mediante el bioensayo de punteadura en hojas (bioensayo en hojas de banano desprendidas); y la aplicación de FCH. El bioensayo de evaluación consistió en la colecta en campo de hojas de mediana edad de dos cultivares de banano: Gros Michel (grupo AAA, susceptible) y FHIA-01 (grupo AAAB, resistente); a los que se les aplicó el FCH. La aplicación se realizó en tres posiciones diferentes en el lado adaxial del limbo de la hoja: distal, media y proximal. Transcurridas 48 horas se evaluó la actividad fitotóxica de los filtrados del cultivo del hongo, a través de la expresión sintomatológica de necrosis formada alrededor del punto de aplicación del FCH, expresada como área de la lesión elíptica (mm2) (Figura 3). A las 48 horas de la aplicación del FCH en las hojas de banano se observaron las mayores diferencias estadísticas entre los cultivares.
En otros estudios (Companioni et al., 2005; 2012) incluyeron evaluaciones de otros indicadores, tales como componentes bioquímicos (Figura 4). El análisis de la discriminante para la diferenciación de la resistencia y susceptibilidad de cultivares de banano, utilizado en el programa de mejoramiento genético constituye un aspecto novedoso en esta investigación. Tal estimación se realizó a partir de una matriz de datos que incluyó el efecto del FCH (área de la lesión y niveles de fenoles libres y ligados a las paredes celulares, aldehídos, excepto malondialdehído, y proteínas) en hojas de dieciocho cultivares. Tras el análisis discriminante, se obtuvieron dos funciones, una para los cultivares resistentes, y otra para los susceptibles. Se evaluó cada planta de la matriz, según las funciones discriminantes; y estas se clasificaron en resistentes o susceptibles al 94.4% de los casos (68 de 72 plantas). El método descrito constituye una herramienta útil para incrementar la eficiencia de la selección en condiciones ex vitro e in vivo, sin tener en cuenta las condiciones ambientales, y la época del año favorable para el desarrollo de la enfermedad. También permite evaluar un número importante de muestras en condiciones de laboratorio, y acelerar los programas de mejoramiento genético a esta enfermedad. Lo cual evidencia el potencial de la biotecnología en el campo del mejoramiento genético en el cultivo. Por tal motivo, García et al. (2020) demostraron que el método para la diferenciación de la susceptibilidad o resistencia a marchitez por Fusarium descrito con anterioridad puede realizarse de forma rápida, y no destructiva con el uso del FCH tratados en las hojas para diferentes poblaciones del patógeno. Lo cual podría ser aplicable para los programas de mejoramiento genético de musáceas tanto convencional como biotecnológicas. Por otro lado, propician las bases para futuros proyectos relacionados con la identificación de los metabolitos fitotóxicos, y de la especificidad de presuntos genes de avirulencia del agente causal. En este aspecto, Portal et al. (2018) estudiaron mediante el bioensayo descrito por Companioni et al. (2003) los componentes tóxicos de los FCH de la raza 1 de FOC GCV [01210]; y aislaron los metabolitos microbianos extracelulares implicados en la respuesta de la planta. Donde obtuvieron un filtrado de cultivo del hospedero específico, y caracterizaron los compuestos fitotóxicos como ácido fusárico, bovericina y fumonisina B1. Estos resultados constituyen la base para el aislamiento directo de genes de avirulencia del patógeno, y genes de resistencia en banano mediante la genética avanzada para su uso en programas de mejoramiento genético en el cultivo.

Figura 3 Expresión sintomatológica de necrosis formada alrededor del punto de aplicación del filtrado del cultivo del hongo, expresada como área de la lesión elíptica (mm2).
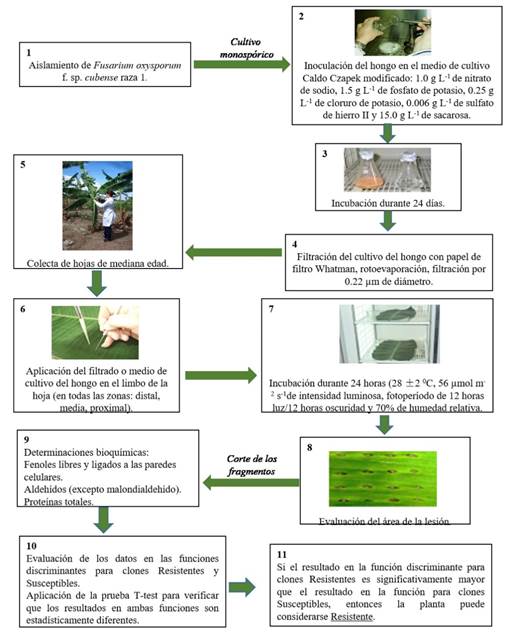
Figura 4 Método para la diferenciación a nivel foliar de la resistencia a Fusarium oxysporum f. sp. cubense raza 1 en banano según Companioni et al. (2012).
Los resultados presentados en esta revisión evidencian el potencial de la biotecnología vegetal y otras herramientas en el campo del mejoramiento genético en el cultivo. Donde las técnicas biotecnológicas constituyen herramientas factibles en la búsqueda de la resistencia a FOC. En este sentido, la transformación genética representa la técnica biotecnológica de mayor expectativa en los últimos años. Sin embargo, la mayoría de los estudios de campo carecen de resultados a largo plazo que contribuyan a evaluar la eficacia de los genes in situ. Por ello, la comprensión de la interacción hospedero-patógeno en términos de defensa; y las rutas relacionadas con los mecanismos de virulencia permite identificar pasos críticos para desarrollar cultivares resistentes empleando enfoques genéticos. Pero, los sistemas de selección para la resistencia a esta enfermedad en condiciones de campo resultan trabajosos, destructivos y consumen mucho tiempo. Por estas razones, el desarrollo de métodos eficientes para la selección temprana de la resistencia a FOC constituye una herramienta útil para realizar estudios precisos en la interacción planta-patógeno; y aumentar la eficiencia de los programas de mejoramiento genético en el cultivo.
Perspectivas
Estudios científicos reportados en todo el mundo relacionados a la interacción hospedante-patógeno del binomio Musa spp. y Fusarium oxysporum f. sp. cubense indican que la tendencia en el presente y futuro es la búsqueda de materiales resistentes al patógeno mediante el uso de las diversas técnicas de la biotecnología como lo son la variación somaclonal, mutagenisis in vitro inducida y la transformación genética mismas que son apoyadas con metodologías para la detección temprana de la resistencia; con resultados parcialmente exitosos hasta ahora. Grupos diversos de trabajo se han formado para afrontar este problema fitopatológico, destacan los del continente asiático donde se detectó por primera vez e FOC RT4 en 1990. En junio de 2019 se observaron plantas con síntomas de FOC en Colombia, tan solo 19 años después. Esto alertó a los países miembros del OIRSA del cual México forma parte; para el 25 de julio de 2019, a través de la SADER-SENASICA (2019) señaló estar preparado para prevenir la entrada al país del hongo Fusarium oxysporum f. sp. cubense RT4. Reforzar las medidas de vigilancia y prevención sanitarias para mitigar el riesgo de entrada; así como la capacitación de técnicos y productores son prioridad, donde el programa de vigilancia epidemiológica forma parte fundamental. En relación a investigación científica para enfrentar este problema que se avecina poco se ha avanzado en México. Por lo que, es una necesidad inminente que instituciones enfocadas a la investigación aborden este problema.
CONCLUSIONES
Los resultados presentados en esta revisión muestran el potencial de la biotecnología vegetal y otras herramientas en el campo del mejoramiento genético en el cultivo. Los cuales permiten acelerar los programas de mejoramiento genético de la resistencia a esta enfermedad. La transformación genética representa la técnica biotecnológica de mayor expectativa en los últimos años. Sin embargo, la mayoría de los estudios de campo carecen de resultados a largo plazo que contribuyan a evaluar la eficacia de los genes in situ. Por ello, el desarrollo de métodos eficientes para la selección temprana de la resistencia a FOC constituye una herramienta útil para realizar estudios precisos en la interacción planta-patógeno; y aumentar la eficiencia de los programas de mejoramiento genético para la resistencia a FOC en el cultivo.











 texto en
texto en 



