El hongo fitopatógeno Guignardia bidwellii (anamorfo: Phyllosticta ampelicida) es el agente causal de la pudrición negra de la vid, una de las enfermedades más importantes de la viticultura en Europa (Ullrich et al., 2009). Es nativo de Norteamérica y fue introducido a Europa a partir de la movilización de híbridos infectados a finales del siglo XIX (Wicht et al., 2012). El patógeno se encuentra distribuido en importantes zonas vitícolas como Francia, Alemania y norte de Italia, lo que representa un potencial riesgo fitosanitario en aquellas regiones donde la humedad relativa es alta durante el verano, que se traduce en pérdidas económicas considerables si no se realiza un manejo adecuado de la enfermedad (Wicht et al., 2014).
La pudrición negra afecta principalmente a la vid (Vitis spp.), pero también se presenta en otros hospedantes de la familia Vitaceae: Parthenocissus spp., Ampelopsis spp., Cissus spp. (Van der Aa, 1973; Wicht et al., 2012). Se han observado diferencias de susceptibilidad entre diversos cultivares, sin embargo, todas las variedades de importancia comercial son susceptibles a la enfermedad (Ullrich et al., 2009). Luttrell (1946) propuso que el hongo presenta tres variantes o formas (f.) en función del hospedante (f. euvitis, f. parthenocissi y f. muscadinii), sin embargo, los estudios moleculares sólo han demostrado la diferenciación en dos formas distintas: las provenientes de los hospedante Vitis y Parthenocissus (Wicht et al., 2014). La identificación tradicional de G. bidwellii se basa en la descripción de los caracteres morfológicos y la expresión de síntomas (Wicht et al., 2012); sin embargo, el estado anamorfo posee características similares con varias especies relacionadas, por ejemplo: picnidios con conidios hialinos sin septos, cubiertos por una capa mucoide y un apéndice apical (Wikee et al., 2013a).
El hongo endófito Guignardia endophyllicola (Phyllosticta capitalensis) es una especie cosmopolita y un parasito débil en un amplio rango de hospedantes (Okane et al., 2003) que incluye a miembros de la familia Vitaceae (Wikee et al., 2013b). El estado teleomorfo y anamorfo pueden confundirse morfológicamente con G. bidwellii y P. ampelicida, conduciendo a una incorrecta identificación, por lo que es necesario la corroboración mediante el uso de técnicas moleculares (Wicht et al., 2014).
Existen pocos estudios moleculares sobre G. bidwellii. En una primera aproximación, Wicht et al., (2012) analizaron filogenéticamente la región ITS1 e ITS2 de muestras provenientes de diversos orígenes geográficos, concluyendo que existe evidencia de dos variantes asociadas a los hospedantes de los géneros Vitis y Parthenocissus. Zhang et al. (2013) llegaron a una conclusión similar mediante un análisis multilocus de cuatro genes, propusieron recategorizar a esta forma a nivel de especie como P. parthenocissi. Mientras, el uso de microsatélites ha permitido determinar que existe una gran variación alélica en las poblaciones presentes en Europa y entre ambos hospedantes (Wicht et al., 2014), sin embargo, no existe evidencia suficiente para asociar un genotipo de G. bidwellii a una reacción especifica en el hospedante Vitis (Rinaldi et al., 2017).
En la actualidad la pudrición negra de la vid es una enfermedad de importancia económica en Europa y cuarentenaria para México (SENASICA, 2019), como parte del sustento científico en los programas de vigilancia epidemiológica, es importante contar con un protocolo basado en técnicas moleculares que permita la correcta identificación de la especie, por lo cual el objetivo del presente estudio fue diseñar, desarrollar y validar un marcador molecular basado en la técnica de PCR con oligos específicos para el diagnóstico de G. bidwellii.
Materiales y metodos
Obtención de aislamientos. Se utilizaron las cepas de referencia Guignardia bidwellii f. euvitis ATCC 9560 (Georgia, Estados Unidos), Guignardia endophyllicola CNRF-MICO-C1/001 (Michoacán) y Guignardia citricarpa ATCC 26254 (Sudáfrica), además de aislamientos pertenecientes a la colección de hongos fitopatógenos del Centro Nacional de Referencia Fitosanitaria (CNRF) de la Dirección General de Sanidad Vegetal (DGSV) en México, de las especies fitopatógenas Verticillium dahliae, Fusarium oxysporum, Alternaria alternata, Colletotrichum gloeosporioides, así como hojas de vid infectadas con Plasmopara viticola.
Los aislamientos de la colección del CNRF-SENASICA se mantienen conservados a 4 °C en aceite mineral de acuerdo a la metodología empleada por Humber (1997), se reactivaron en medio de cultivo Papa Dextrosa Agar (PDA) y se incubaron a 25 °C ± 3 °C durante diez días. A continuación, los cultivos monospóricos, monopicnidiales para el caso del género Phyllosticta, se realizaron con base en Crous et al. (2005), en PDA y se incubaron a la temperatura mencionada anteriormente bajo 12/12 horas luz-oscuridad durante 15 días. Para P. viticola se mantuvo preservado en forma de herbario en prensa botánica.
Extracción de ADN. La extracción de ADN genómico total se realizó mediante el método CTAB (Doyle y Doyle, 1987) para las especies del género Guignardia y para el tejido de vid sano e infectado con P. viticola, mientras que para las cuatro especies restantes se utilizó la metodología propuesta por Cenis (1992) a partir de placas de PDA, omitiéndose el primer centrifugado. La cantidad y calidad del ADN se verificó por la relación de absorbancias A260/280 y A260/230 (Manchester, 1995) en un espectrofotómetro Nanodrop 2000C.
Diseño de oligos. In silico se utilizaron secuencias de la región ITS del ADNr de 46 especies del género Guignardia reportadas por Wikee et al., (2013a), más las secuencias de referencia AB095505, AB095509 a AB095511, AB454268, AB454276, AB454313, HM008727 a HM008728, FJ824766, EU683672, KC193586, KF015253 a KF015268 y KF851288 a KF851317 correspondientes a la f. euvitis y f. parthenocissus (Okane et al., 2003, Motohashi et al., 2009, Wicht et al., 2012; Zhang et al., 2013; Rinaldi et al., 2017). Las secuencias se alinearon con el algoritmo ClustalW en BioEdit 7.2.5 (Hall, 1999). Se identificaron los polimorfismos de un solo nucleótido (SNP´s) característicos de la secuencia consenso de referencia de G. bidwellii f. euvitis respecto a las otras 46 especies, la región ITS se utilizó para diseñar el oligonucleótido forward Bidwell.
El oligonucleótido reverse Ampel2 se diseñó utilizando el software Primer3 (Untergasser et al., 2012) a partir de la misma secuencia consenso. El par de oligonucleótidos se estructuró para amplificar un fragmento único de 173 pb. Los parámetros termodinámicos, la formación de estructuras internas, heterodímeros y autodímeros se examinaron in silico con mFold (Zuker, 2003). La especificidad de ambos oligonucleótidos fue ensayada in silico mediante Primer BLAST (Ye et al., 2012).
Amplificación por PCR. Después de la extracción se realizó un ensayo de PCR de gen endógeno con los oligonucleótidos ITS1 (5´ TCCGTAGGTGAACCTGCGG 3´) e ITS4 (5´ TCCTCCGCTTATTGATATGC 3´) (White et al., 1990) para verificar la calidad del material genético. La amplificación se realizó con el Kit Taq DNA Polymerase de Invitrogen®, la mezcla de reacción se hizo a una concentración final de Buffer 1X, 1.5 mM de MgCl2, 0.2 mM de dNTP´s, 0.5 µM de cada iniciador, 2.5 U Taq Polymerase y 2 ng µL-1 de ADN en un volumen de 25 µL. El programa térmico utilizado fue una desnaturalización inicial a 95 °C por 3 min, seguida de 35 ciclos a 95 °C por 45 s, hibridación a 58 °C por 45 s, extensión de 72 °C por 45 s y una extensión final de 72 °C por 10 min. Se utilizó un T100™ Thermal Cycler de BioRad.
La amplificación del fragmento de 173 pb con los oligonucleótidos específicos se realizó usando la misma mezcla de reacción con un programa térmico de desnaturalización inicial a 95 °C por 5 min, seguido de 35 ciclos a 95 °C por 45 s, hibridación a 62 °C por 30 s, extensión de 72 °C por 30 s y una extensión final de 72 °C por 5 min. Los productos de PCR se analizaron en geles de agarosa ultra pura al 2% en buffer TAE 1X marcado con 1X de GelRed® Biotium. El tamaño de los fragmentos amplificados se estimó utilizando un marcador TrackIt 100 bp DNA Ladder de Invitrogen.
Control positivo. Se generó a partir del fragmento específico del gen amplificado con los oligonucleótidos Bidwell y Ampel2. El producto de PCR se clonó utilizando pGEM® T Easy Vector de Promega. El plásmido portador del fragmento se utilizó para transformar las células competentes One Shot® Mach1™ T1R E. coli de Invitrogen. Se seleccionaron las colonias bacterianas que lograron introducir el plásmido y se realizó extracción de ADN con el método de lisis alcalina (Sambrook y Russell, 2001). A continuación, el inserto clonado se amplificó por PCR y se secuenció en ambos sentidos con el equipo ABI PRISM 3130 de Applied Biosystems® mediante la química BigDye™ Terminator v3.1.
Las secuencias obtenidas se compararon en la base de datos del Centro Nacional para la Información Biotecnológica (NCBI) mediante el algoritmo denominado Herramienta Básica de Búsqueda de Alineación Local (nucleotide BLAST) (Altschul et al., 1990) y con las secuencias reportadas por Wicht et al., (2012) y Rinaldi et al., (2017).
Ensayo de especificidad. Se evaluó el par de oligonucleótidos contra los hongos G. bidwellii f. euvitis, G. citricarpa, V. dahliae, F. oxysporum, A. alternata, C. gloeosporioides, P. viticola y G. endophyllicola, este último reportado como hongo cosmopolita endófito (Okane et al., 2003) relacionado morfológica y filogenéticamente a G. bidwellii. Como control negativo se utilizó ADN de tejido de vid sano y un control blanco (sin ningún tipo de ADN). El ensayo se realizó por duplicado incluyendo cada vez dos repeticiones por espécimen. El resultado se consideró como positivo cuando se obtuvo la amplificación del fragmento de 173 pb.
Ensayo de sensibilidad. A partir del control clonado de G. bidwellii f. euvitis a una concentración inicial de 3000 ng µL-1 se realizaron diluciones seriales para obtener ocho concentraciones de trabajo (3000, 300, 30, 3, 0.3, 0.15, 0.06 y 0.03 ng µL-1). Cada dilución se amplificó por triplicado usando los oligos Bidwell-1 y Ampel-2 siguiendo las condiciones de reacción y programa térmico mencionados con anterioridad. A continuación, se realizó un segundo ensayo con siete diluciones del control clonado (1000, 500, 300, 30, 3, 0.3 y 0.03 ng µL-1), dos repeticiones por dilución y se agregó 1 µL de ADN extraído de vid sana (50 ng µL-1) a los 25 µL de la reacción final.
Resultados y discusión
Diseño de oligos. El análisis bioinformático de los oligonucleótidos diseñados para G. bidwellii (Cuadro 1), señala que el valor de ΔG fue menor a 2 kcal mol-1, lo que indica que no hay formación de estructuras secundarias importantes que afecten negativamente la reacción (Matveeva et al., 2003); En consecuencia los valores de ΔG para el extremo 3´, autodimeros y heterodimeros estuvieron dentro de los rangos tolerados de acuerdo a Untergasser et al. (2012). Otros criterios como la temperatura de fusión (Tm), longitud, % de GC, presencia de C o G en el extremo 3´, dinucleótidos en tándem y bases repetidas, estuvieron dentro de los parámetros aceptables de acuerdo a Rychlik (1995).
Respecto a la especificidad, el análisis in silico mediante BLAST del oligonucleótido forward Bidwell mostró un 100% de cobertura e identidad sólo con secuencias de G. bidwellii, ningún otro hongo presentó una región homologa, los organismos más cercanos fueron Streptomyces sp. y Bacillus subtilis con un 85 y 76% de cobertura. Para el oligo reverse Ampel2 también hubo 100% de cobertura e identidad con G. bidwellii, sin embargo, en este caso hubo homología completa con muchas especies dentro del género Guignardia, no así con los géneros Streptomyces y Bacillus.
Cuadro 1 Oligonucleótidos específicos para la detección de Guignardia bidwellii f. euvitis (Phyllosticta ampelicida) mediante PCR punto final.
| Oligo | Secuencia 5´a 3´ | pb | Tm (°C)X | %GCX | ΔGy | Valor de Ez | % Coberturaz | % Identidadz |
|---|---|---|---|---|---|---|---|---|
| Bidwell | GAAAAGCCGTCCGAAAGAGCC | 173 | 66.47 | 57.14 | 1.98 | 0.083 | 100 | 100 |
| Ampel2 | CAGGACTTCACGAAATAATCG | 57.38 | 42.86 | 0.84 | 0.083 | 100 | 100 | |
x Valores calculados mediante Primer3 (Untergasser et al., 2012).
y Valor de energía libre de Gibbs calculado con mFOLD (Zuker, 2003).
z Valor de E, cobertura e identidad determinado con nBLAST del NCBI para las accesiones KF851289 a KF851315.
El alineamiento múltiple con las secuencias de referencia de Wikee et al. (2013a) confirmó que Bidwell es específico, dado que los 21 nucleótidos del oligonucleótido tienen homología completa con las secuencias reportadas en el hospedante Vitis (Figura 1a), adicionalmente, los primeros once nucleótidos del extremo 3´ de este oligo incluyen a la variante reportada en el hospedante Parthenocissus (Wicht et al., 2012; Zhang et al., 2013; Rinaldi et al., 2017). Por otra parte, Ampel2 se ubica en una región conservada, puesto que los últimos ocho nucleótidos hacia el extremo 3´ son constantes para todas las especies del género Guignardia (Figura 1b). Ambos resultados fueron acordes a lo observado en BLAST.
Amplificación y especificidad. Se obtuvo ADN genómico de buena calidad tanto para los cultivos monospóricos como para las muestras provenientes de tejido vegetal. El ensayo de gen endógeno de la región ITS del ADNr tuvo una amplificación de PCR eficiente que varió entre 550 a 750 pb de acuerdo a cada especie (Figura 2a), lo que indica que el ADN obtenido es apto para ser amplificado, esta íntegro y no existe inhibición proteica de la reacción.
La reacción de PCR con el par de oligonucleótidos específicos Bidwell y Ampel2 generó el fragmento esperado de 173 pb sólo para el ADN de G. bidwellii (Figura 2b). Los oligonucleótidos no mostraron reacción cruzada con ninguno otro hongo, incluido G. endophyllicola, el cual es la especie molecularmente más relacionada. Tampoco se observó reacción en el tejido de vid, por lo tanto el genoma de la planta no produce falsos positivos. En adición, se confirmó la especificidad de los oligonucleótidos mediante la secuenciación de los productos de PCR que resultaron positivos en el ensayo, obteniendo un 100% de cobertura e identidad con G. bidwellii accesiones KF015253 a KF015255 de Rinaldi et al. (2017).

Figura 1 Filogenia de la región ITS del ADNr para las secuencias representativas de Guignardia bidwellii f. euvitis y f. parthenocissi. A) Alineamiento de las secuencias para diseñar el forward Bidwell-1. B) Diseño en sentido 5´- 3’ del reverse Ampel-2. Se utilizó a C. gloeosporioides como nodo raíz externo al grupo.

Figura 2 Amplificación de los productos de PCR. A) Ensayo de gen endógeno con los oligos ITS-1 e ITS-4 (550-750 pb). B) Ensayo con los oligos específicos Bidwell-1 y Ampel-2 (173 pb). Para ambos ensayos carril 1-2: G. bidwellii, 3-4: ADN de vid con Plasmopara viticola, 5-6: G. endophyllicola, 7-8: G. citricarpa, 9-10: Verticillium dahliae, 11-12: F. oxysporum, 13-14: A. alternata, 15-16: C. gloeosporioides, 17: Blanco con H2O. MM: TrackIt 100 bp DNA Ladder de Invitrogen.
Ensayo de sensibilidad. La amplificación del fragmento clonado se inhibió al usar una concentración de 3000 ng µL-1, por lo que en un segundo ensayo se incluyó una concentración de 1000 ng µL-1 donde la banda fue tenue o imperceptible. Todas las demás diluciones mostraron una óptima amplificación, lo que indica que el método es sensible ya que permite detectar a G. bidwellii en concentraciones menores a 500 ng µL-1 con un límite de detección inferior evaluado en 0.03 ng µL-1. El ensayo con el extracto de ADN de vid mostró resultados similares, por lo que la sensibilidad de la técnica no se vio afectada por la presencia del genoma de la planta (Figura 3).
Se diseñaron los oligonucleótidos específicos para G. bidwellii tomando como referencia todas las secuencias disponibles de G. bidwellii f. euvitis para la región ITS del ADNr, la cual se eligió debido a que es polimórfica para diferenciar a las especies del género Guignardia (Wikee et al., 2013a). Además, los espaciadores transcritos internos de esta región han sido propuestos como DNA Barcoding universal para el reino Fungi debido a su alta variabilidad inter especifica en un amplio rango de Eumycetos (Schoch et al., 2012). Asimismo, Bonants et al. (2003), Everett y Rees (2006) y Peres et al. (2007) han desarrollado con éxito oligonucleótidos específicos basados en la misma región ITS del ADNr para otras especies del género Guignardia.
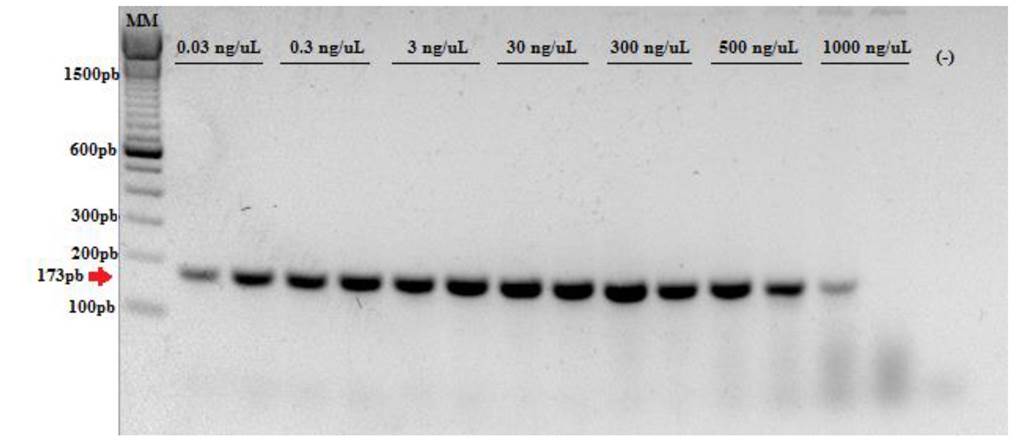
Figura 3 Ensayo de sensibilidad de los oligonucleótidos Bidwell-1 y Ampel-2 utilizando gradiente de diluciones del control clonado en mezcla con 50ng μL-1 de ADN de Vid. (-): Blanco con H2O. MM: TrackIt 100 bp DNA Ladder de Invitrogen.
En el diseño de los oligos se incluyeron secuencias de los aislamientos representativos de las principales regiones vitícolas donde se presenta la enfermedad: Estados Unidos (Zhang et al., 2013) y Europa (Rinaldi et al., 2017). Las secuencias del control de referencia ATCC 9560 y el control clonado fueron homologas. Asimismo, para este gen en particular no se detectaron polimorfismos de un solo nucleótido para los distintos orígenes geográficos de la misma especie, tal como lo refiere Wicht et al. (2012), por lo tanto, los oligos aquí diseñados tienen robustez interespecífica, sin embargo, la variación intraespecifica, de subpoblaciones o haplotipos requerirá de marcadores más robustos (Wicht et al., 2014; Rinaldi et al., 2017).
Respecto al rango de hospedantes y la posible especialización o variantes citadas por Luttrell (1946) y Wicht et al. (2012), los oligos diseñados Bidwell y Ampel2 son complementarios en un 100% con las secuencias reportadas para los aislamientos obtenidos del hospedante del género Vitis, los ensayos in silico e in vivo generaron el fragmento esperado de 173 pb. No obstante, el alineamiento con las secuencias de la f. parthenocissi mostró un SNP de guanina por timina en la posición 12 del forward Bidwell y dos SNP´s más en las posiciones ocho y diez del reverse Ampel2. En los ensayos in silico un solo SNP es suficiente para determinar especificidad entre dos organismos (Stadhouders et al., 2010); sin embargo, in vivo la especificidad puede estar dada sólo por los primeros nucleótidos del extremo 3´ (Ayyadevara et al., 2000; Stadhouders et al., 2010). En futuras investigaciones se sugiere evaluar los oligos para los aislamientos obtenidos de los hospedantes Parthenocissus, Ampelopsis y Cissus.
Considerando que de acuerdo con Wich et al. (2012) la tasa de crecimiento del hongo en medios de cultivo es lenta, pudiendo requerir más de dos semanas para la formación de cuerpos fructíferos, el protocolo aquí descrito representa una herramienta rápida y confiable para la detección e identificación del patógeno, al poder realizarse a partir de material vegetal infectado o de micelio cultivado in vitro con menos de una semana de crecimiento. Protocolos similares se han implementado para la detección de otros hongos de difícil aislamiento como P. citricarpa (Bonants et al., 2003; Peres et al., 2007) y Elsinoë fawcettii (Hyun et al., 2007). Los oligos permiten diferenciar rápidamente a G. bidwellii del resto de especies, sin embargo, no permiten diferenciar teleomorfo de anamorfo, por lo que la identificación biológica es necesaria. Por otra parte, este ensayo de PCR puede ser complementado por el propuesto por Everett y Rees (2006) para la detección especifica del endófito G. endophyllicola, a fin de obtener resultados más robustos en el diagnóstico de G. bidwellii f. euvitis (P. ampelicida).
Finalmente, debido a que la principal fuente de dispersión de la pudrición negra de la vid es la movilización de material propagativo (Wicht et al., 2012), su introducción a regiones vitícolas representa un gran riesgo para la producción, tal como se ha reportado en Europa (Rinaldi et al., 2017). Para México, su introducción pondría repercutir en más de 33 000 ha de vid (SIAP, 2018), debido a que no se cuenta con cultivares resistentes (Ullrich et al., 2009), por lo que la detección oportuna del hongo a partir de material vegetal propagativo y la aplicación de medidas adecuadas de confinamiento y delimitación demandan como una prioridad el desarrollo de métodos de diagnóstico rápidos y precisos como lo es la PCR.
Conclusiones
Esta investigación reporta el desarrollo de un ensayo de PCR punto final con base en la región ITS del ADNr para la detección e identificación específica, rápida y sencilla de Guignardia bidwellii f. euvitis (Phyllosticta ampelicida), agente causal de la pudrición negra de la vid, lo cual permite contar con un diagnóstico confiable del hongo siendo punto de partida para la planeación y aplicación de medidas de prevención de la introducción y manejo oportuno de la enfermedad, contribuyendo así con los programas de vigilancia epidemiológica y manejo fitosanitario.











 texto en
texto en 


