El maíz (Zea mays) es actualmente el segundo cultivo alimenticio más importante en términos de fuentes de energía y un contenido de proteína de aproximadamente 9.2% en la nutrición humana (FAO, 2016). México ocupa el séptimo lugar en producción del cereal con aproximadamente 25 millones de toneladas al año (FIRA, 2016). La productividad de esta gramínea se limita por factores bióticos por varias plagas y enfermedades y abióticos como sequía, deficiencia de nutrientes, salinidad y altas temperaturas (Grover et al., 2007). La sequía es el término meteorológico para la escasez de agua y es uno de los factores ambientales de estrés más importantes que afectan la germinación, vigor de la planta y productividad de los cultivos agrícolas (Kamara et al., 2003; Wilkinson y Davies, 2010) y una posible alternativa para hacer frente a este problema es la generación de conocimiento sobre los microorganismos de las plantas que desempeñan una función importante en la expresión de resistencia al estrés por factores abióticos (Gond et al., 2015).
Los microorganismos endófitos son bacterias, hongos o virus que habitan, parte o todo su ciclo de vida, en el tejido interno de las plantas sin causar daño a su hospedante y que establecen una interacción simbiótica que modulan la sanidad de la planta y su habilidad para adaptarse a diferentes factores de estrés del ambiente (Hardoim et al., 2015). Las bacterias endófitas representan un subgrupo dentro de la comunidad de bacterias de la rizosfera y rizoplano que colonizan el tejido interno de las raíces de la planta hospedante y que le confiere ventajas ecológicas sobre otras poblaciones que colonizan en forma epífita. Diversas especies de bacterias endófitas Gram positivas y Gram negativas se han aislado de diferentes tipos de tejido en numerosas especies de plantas (Rosenblueth y Martínez-Romero, 2006).
Existen evidencias del efecto positivo que inducen los microorganismos endófitos mediado por la expresión de mecanismos como el antagonismo, resistencia sistémica inducida (RSI) y adquirida (RSA), promotores del crecimiento de la planta, y la inducción de una respuesta de adaptación al estrés ambiental (Liu et al., 2013). La relación entre la planta hospedante y la comunidad bacteriana endofítica refleja la coevolución en el proceso de colonización influenciado por el genotipo, etapa de crecimiento, estado fisiológico y tejido de la planta, así como por las características del suelo, prácticas agronómicas y condiciones ambientales como la temperatura, agua y el suministro de nutrientes (Higgins et al., 2007).
En Maíz, la inoculación de estos microorganismos se relaciona con un incremento en la germinación, altura de la planta, biomasa radical y aérea que mejora el rendimiento (Morales et al., 2011). Así mismo, se ha demostrado la eficiencia con la inoculación de estas bacterias en maíz promoviendo tolerancia al stress por sequía (Fan et al., 2015), mediado por el incremento de la longitud y biomasa de la raíz mejorando la absorción de agua y nutrientes (Naseem y Bano, 2014; Yasmin et al., 2013). Las bacterias endófitas pueden inducir tolerancia contra enfermedades abióticas como salinidad y sequía, mientras que algunas poblaciones confieren tolerancia a factores de estrés específico y son responsables de la supervivencia de las plantas bajo esas condiciones particulares del ambiente (Gond et al., 2015; Montañez et al., 2011). Así, los microorganismos que establecen una interacción positiva con las raíces de las plantas desempeñan un papel clave en sistemas agrícolas con un promisorio potencial biotecnológico para su uso en una agricultura sostenible.
En México no existen investigaciones sobre las comunidades bacterianas endófitas en raíces de líneas de maíz tolerantes y susceptibles a sequía. Tales bacterias endófitas, potencialmente podrían utilizarse para el desarrollo de futuras estrategias biotecnológicas como inductores de tolerancia a sequía y a otros factores bióticos y abióticos limitantes en la productividad del cultivo. Por lo anterior, el objetivo de este estudio fue estimar la densidad y diversidad de las poblaciones bacterianas cultivables endófitas en la raíz en líneas de maíz homocigóticas (S6) tolerantes y susceptibles a sequía previamente identificadas con este carácter en condiciones de campo y cultivadas en tres localidades de México durante tres ciclos del cultivo.
Materiales y métodos
Sitio de muestreo. Se sembraron siete líneas homocigóticas de maíz tolerantes (CLQRQ108, CML384, CML445, CML544, DTMA90, DTMA224, DTMA256) y siete susceptibles a sequía (CML181, DTMA34, DTMA41, DTMA43, DTMA109, DTMA144, DTMA182), las cuales se seleccionaron por ser líneas endocriadas debido a su uniformidad en la respuesta a sequía. Cada línea se sembró con tres repeticiones en el Campo Experimental del Centro Internacional de Mejoramiento de Maíz y Trigo (CIMMyT) en las localidades de Tlaltizapán, Morelos (18.68 N; 99.11 O), El Batán, Texcoco, México (19.53 N; 98.85 O), y Agua Fría, Puebla (20.5 N; 97.6 O) durante los ciclos de cultivo verano 2012 (V-2012), invierno 2012 (I-2012) y verano 2013 (V-2013). Se realizaron muestreos del tejido interno formado en la zona apical de la raíz en tres plantas de cada línea por localidad y ciclo de siembra a los 25, 52 y 75 días después de la siembra (dds) para su análisis en el Laboratorio de Patología de Maíz del CIMMyT, El Batán, Edo. México. El tamaño de muestra se determinó por el método propuesto por Cochran (1982).
Aislamiento de bacterias endófitas de raíces. Se definieron como cepas endófitas aquellos aislamientos bacterianos obtenidos del tejido interno de las raíces del maíz superficialmente esterilizadas. Las raíces de maíz sin síntomas se lavaron con agua destilada estéril y se cortaron en trozos de 2 cm. Se esterilizaron superficialmente 10 g de raíces en frascos de vidrio estériles con lavados en serie con etanol 70% durante 5 min; hipoclorito de sodio 0.53% durante 10 min y tres lavados con agua destilada estéril. Del último lavado con agua destilada estéril, se sembraron 100 µL de cada frasco en placas Petri con medio de cultivo NB (1L): Agar Bacteriológico 15 g (BIOXON® México), Peptona 5 g (BD DIFCOTM EE. UU), Extracto de levadura 3 g (DIBICO® México, NaCl JT BAKER® México) y se incubaron a 28±1 ºC durante 2-5 días. La ausencia de crecimiento bacteriano en el medio de cultivo confirmó la eficiente esterilización superficial de las raíces. Estas raíces se molieron en morteros fríos con 20 mL de una solución amortiguadora (50 mM KH2PO4, 150 mM NaCl, pH 7.6) estéril. Las suspensiones obtenidas se diluyeron en serie (1:9) desde 100 hasta 10-3 y se sembraron 100 µL de cada dilución con tres repeticiones en medio NB incubados a 28 °C durante 24 h con luz continua. Se cuantificaron y clasificaron los diferentes morfotipos bacterianos con base al color, forma, textura y tipo de crecimiento. El crecimiento bacteriano se consideró representativo de bacterias cultivables endófitas en la raíz de maíz. Las cepas bacterianas se preservaron por congelación en medio NB y glicerol 20% para estudios posteriores.
Densidad poblacional de bacterias endófitas. La densidad poblacional de bacterias por tejido de raíz se estimó por conteo directo de colonias en placa. El número de colonias bacterianas aisladas de las 14 líneas de maíz se cuantificó con un contador de colonias (Quebec®, Darkfield Colony Counter). La población bacteriana de las muestras de tejido de raíz se expresó como unidades formadoras de colonias (UFC g-1 de raíz). Los datos obtenidos se transformaron a Log10 UFC g-1 de raíz y se realizó un análisis de varianza (ANOVA) siguiendo un diseño de análisis factorial de donde se obtuvo la separación de medias por DMS (α=0,05) con el programa estadístico SAS (Statistical Analysis System, version 9.1.3 SAS Institute, Cary, NC, EE.UU) en función del ciclo de cultivo, localidad, días después de la siembra (dds) y líneas de maíz tolerantes/susceptibles a sequía estudiadas.
Amplificación del gen 16S rADN e identificación de bacterias endófitas. La extracción de ADN de las bacterias se realizó por el protocolo descrito por Mahuku (2004), con las siguientes modificaciones: las células bacterianas se obtuvieron a partir del crecimiento en medio NB a 28 °C durante 48 h. Después de suspender el precipitado en 100 µL de 1X TE, se agregaron 2 µL de RNAsa (1 mg mL-1), y se incubó en baño María a 37 °C durante 1 h. La calidad del ADN se verificó por electroforesis en gel de agarosa. Las bacterias se identificaron mediante la amplificación parcial del gen 16S rADN con los iniciadores universales 27F (5´AGAGTTTGATCMTGGCTCAG-3´) y 1492R (5´TACGGHTACCTTGTTACGACTT-3´) con las condiciones de PCR descritas por Galkiewicz y Kellogg (2008). La amplificación y secuenciación se hizo en Macrogen (DNA Sequency Service. Korean Biotechnology Company); las secuencias obtenidas se alinearon con secuencias depositadas en el GenBank del Centro Nacional de Biotecnología de los Estados Unidos (NCBI) utilizando el programa BLASTn (http://www.ncbi.nlm.nih.gov/BLAST/).
Resultados y discusión
Identificación taxonómica de bacterias endófitas. La secuenciación parcial del gen 16S rADN permitió identificar 22 especies de bacterias endófitas en la raíz en ambos grupos de líneas de maíz. La mayor abundancia de bacterias se asoció a los phyla Proteobacteria, seguido de Firmicutes y Bacteroidetes. Dentro de Firmicutes, el 100% de las cepas se identificaron como Bacillus sp. y dentro de la clase Gammaproteobacteria, 33% como Pseudomonas sp.
La diversidad y riqueza de bacterias naturalmente asociadas a las raíces de maíz es amplia y la estimación de esta depende del método de análisis. En este estudio se utilizó un enfoque de cultivo dependiente y los resultados coinciden con investigaciones previas que reportaron el phylum Proteobacteria como el más dominante entre las poblaciones bacterianas endófitas aisladas en raíces, tallos y hojas de maíz (Rosenblueth y Martínez-Romero, 2006). Así mismo, otros estudios sobre la diversidad de las comunidades endófitas de la raíz en maíz utilizando métodos de cultivo dependiente e independiente también coinciden en que las bacterias más frecuentemente asociadas a este cultivo son Firmicutes (Bacillus), Gammaproteobacteria (Pseudomonas) (Pereira et al., 2011) y Burkholderia spp. (Ikeda et al., 2013). El análisis de estas poblaciones por cromatografía de gases y perfiles de ácidos grasos identificaron a Bacillus pumilus, B. subtilis, Pseudomonas aeruginosa y P. fluorescens como las especies relativamente más predominantes en el tallo de maíz (Rai et al., 2007), mientras que mediante pyrosecuenciación se identificó como los más abundantes en la rizosfera de maíz a los phyla Proteobacteria, Bacteroidetes y Actinobacteria (Li et al., 2014).
Experimentaciones en maíz han mostrado el potencial uso de estas poblaciones como inoculantes bacterianos para el control de estrés por sequía, sugiriendo que la inducción de tolerancia a sequía se debe a la producción de fitohormonas como el ácido abscísico, giberélico, indol-3-acético, citocininas, enzimas como la ACC deaminasa, producción de exopolisacáridos bacterianos y la inducción de tolerancia sistémica (Dimkpa et al., 2009; Kim et al., 2012; Timmusk et al., 2014; Yang et al., 2009). En otros cultivos se han señalado como inductores de tolerancia en trigo (Triticum sativum) y leguminosas (Vigna radiata) al ser inoculados en semilla y promoviendo el incremento en los niveles de regulación de genes relacionados con la tolerancia a sequía y la actividad de diversas enzimas (Kasim et al., 2013; Saravanakumar et al., 2011).
Por la frecuencia de los aislamientos, en el presente estudio se destacan tres grupos de bacterias endófitas en la raíz de maíces tolerantes y susceptibles a sequía: 1) altamente frecuentes (Bacillus subtilis, Bacillus megaterium y Pseudomonas geniculata), aislados durante los tres ciclos de cultivo en las tres localidades, 2) frecuentes (Bacillus firmus, Pseudomonas hibiscola y Sinorhizobium meliloti), aislados durante los tres ciclos de cultivo en dos localidades y 3) baja frecuencia (Acinetobacter soli, Stenotrophomonas maltophila y Burkholderia gladioli, aisladas solo en dos localidades y no en todos los ciclos del cultivo (Cuadro 1).
Cuadro 1 Especies de bacterias endófitas identificadas en la raíz de 14 líneas de maíz en tres localidades y durante tres ciclos del cultivo.
| ID | Endófito | Línea | Identidad de Nucleótidos | Clase | Localidad/ciclo de cultivo | ||
| T/S | TL | AF | EB | ||||
| AF101 | Acinetobacter soli Z | T, S | 96% (KU551890) | γ Proteobacteria | 1 | 1 | |
| AF105 | Agrobacterium tumefaciens | T, S | 97% (KX518841) | α Proteobacteria | 1 | ||
| TL007 | Bacillus asahii | T, S | 95% (KU551893) | Firmicutes | 1, 2, 3 | 1 | |
| TL008 | Bacillus firmus Y | T, S | 98% (KU551896) | Firmicutes | 1, 2, 3 | 1, 2, 3 | |
| AF103 | Bacillus megaterium X | T, S | 96% (KC414697) | Firmicutes | 1, 2, 3 | 1, 2, 3 | 1, 2, 3 |
| AF102 | Bacillus subtilis X | T, S | 97% (KU551891) | Firmicutes | 1, 2, 3 | 1, 2, 3 | 1, 2, 3 |
| AF111 | Burkholderia cenocepacia | T, S | 96% (GU433447) | β Proteobacteria | 1, 2, 3 | ||
| AF129 | Burkholderia gladioli Z | S | 97% (EU1611873) | β Proteobacteria | 1, 2 | 2 | |
| AF109 | Chryseobacterium indologenes | T, S | 98% (KU551895) | Flavobacteria | 1, 2, 3 | 2 | |
| TL032 | Enterobacter aerogenes | T, S | 97% (AM184247) | γ Proteobacteria | 1 | 1, 2, 3 | |
| BT011 | Enterobacter spp. | T, S | 97% (KX518848) | γ Proteobacteria | 1 | ||
| TL012 | Flavobacterium johnsoniae | T, S | 96% (KU551897) | Flavobacteria | 1 | ||
| AF116 | Klebsiella oxytoca | T, S | 97% (KU551901) | γ Proteobacteria | 2 | ||
| AF128 | Klebsiella pneumoniae | T, S | 96% (KX518847) | γ Proteobacteria | 1 | ||
| TL015 | Pseduomonas hibiscola Y | T, S | 97% (KX518846) | γ Proteobacteria | 1, 2, 3 | 1, 2, 3 | |
| AF112 | Pseudomonas chlororaphis | T, S | 96% (KX518844) | γ Proteobacteria | 1, 2, 3 | ||
| AF115 | Pseudomonas geniculata X | T, S | 96% (KU551900) | γ Proteobacteria | 1, 2, 3 | 1, 2, 3 | 1, 2, 3 |
| TL011 | Pseudomonas lini | T, S | 99% (KX518842) | γ Proteobacteria | 1 | ||
| AF107 | Salmonella bongori | T, S | 96% (KU551899) | γ Proteobacteria | 1 | ||
| AF106 | Serratia marcescens | T, S | 96% (KX518843) | γ Proteobacteria | 1, 2, 3 | 1 | |
| TL010 | Sinorhizobium meliloti Y | T, S | 98% (KU551892) | α Proteobacteria | 1, 2, 3 | 1, 2, 3 | |
| TL009 | Stenotrophomas maltophilia Z | T, S | 98% (KU551894) | γ Proteobacteria | 1, 2, 3 | 1 | |
T= Línea de maíz tolerante a sequía, S= Línea de maíz susceptible a sequía.
TL= Tlaltizapán, Mor., AF= Agua Fría, Pue., EB= El Batán, Méx.
1=Verano 2012; 2=Invierno 2012; 3=Verano 2013.
X Endófitos altamente frecuentes.
Y Endófitos frecuentes.
Z Endófitos de baja frecuencia.
La estructura taxonómica y funcional de las comunidades bacterianas en el suelo está influenciada por factores bióticos y abióticos como las características físicoquímicas del suelo, condiciones climáticas, genotipo de la planta y la interacción con otros procariontes y eucariontes en el suelo, lo cual indica que son interacciones complejas. En este estudio, la estructura y abundancia de las bacterias aisladas puede asociarse con la interacción de varios factores, incluyendo el genotipo de la planta, las características genéticas de la bacteria, el suelo, la temperatura, ciclo de cultivo y fenología de la planta de maíz (Li et al., 2014; Oliveira et al., 2009) que influyen en la colonización y dinámica de las comunidades bacterianas endófitas (Bodenhausen et al., 2013). Se ha mostrado la relación específica entre planta-endófito mediante una quimiotaxia bacteriana hacia exudados de la planta hospedante como fuentes de carbono que actúan como moléculas de señalización (Albareda et al., 2006) y que las diferencias en la composición y patrones de exudación son dependientes del cultivar, etapa de desarrollo, exposición a estrés de la planta y tipo de suelo, los cuales influencian la colonización por comunidades bacterianas (Haichar et al., 2008). Estudios en maíz indicaron que los exudados de la raíz están compuestos de 65% de azúcares, 33% de ácidos orgánicos y 2% de aminoácidos y que los cambios en cantidad y calidad en estos patrones de exudación en las diferentes etapas del crecimiento y fisiología de la raíz, influyó en la biomasa y estructura de las comunidades bacterianas incrementando la actividad y deposición de nutrientes por estas comunidades microbianas beneficiando el crecimiento de la planta (Baudoin et al., 2003). Algunas investigaciones en maíz mediante un enfoque de cultivo dependiente han demostrado cambios dinámicos en la comunidad bacteriana de la rizosfera en diferentes etapas de crecimiento del cultivo (Cavaglieri et al., 2009; Nacamulli et al., 1997).
En este estudio se identificaron bacterias específicas como Burkholderia gladioli, la cual se aisló con baja frecuencia únicamente en las líneas susceptibles en dos localidades y ciclos de cultivo (Cuadro 1).
B. gladioli se ha identificado previamente en raíces del ancestro silvestre (Zea mays ssp. parviglumis) y en genotipos modernos de maíz como un endófito con propiedades antifúngicas (Shehata et al., 2016); así mismo, la inoculación de Burkholderia sp. en maíz mostró efectos positivos en la tolerancia a sequía (Fan et al., 2015; Naveed et al., 2014).
Densidad de poblaciones endófitas en líneas de maíz. La densidad de bacterias endófitas en las 14 líneas de maíz tuvo rangos entre 1, 6 Log10 UFC g-1 de raíz. El ANOVA mostró diferencias altamente significativas (**= p≤0,01) en la densidad de población endófita de B. subtilis, B. megaterium, P. hibiscola y S. meliloti en función de la localidad, ciclo de cultivo, días después del trasplante (dds) y grupo (tolerancia/susceptibilidad a sequía) de línea de maíz (Cuadro 2).
Cuadro 2 Análisis de varianza y comparación de medias de la densidad de población (Log10 UFC g-1 de raíz) en las raíces de 14 líneas de maíces tolerantes y susceptibles a sequía.
| B. subtilis | B. megaterium | P. hibiscola | S. meliloti | |
| Localidad | ** | ** | ** | ** |
| Ciclo | NS | ** | ** | NS |
| Días | NS | ** | NS | ** |
| Tolerancia | ** | ** | ** | ** |
| Tolerante | 2.2684 a | 3.7374 a | 2.9388 a | 4.8880 a |
| Susceptible | 1.7410 b | 3.3976 b | 2.4501 b | 4.6389 b |
Medias con letras iguales no son estadísticamente diferentes (DMS, 0.05)
NS= No existen diferencias significativas
**= Diferencias altamente significativas
Por localidad, en El Batán, las mayores densidades de poblaciones se registraron con B. subtilis y P. hibiscola, mientras que en Tlaltizapán fueron con B. megaterium y S. melilloti. Por ciclo de cultivo y fecha de muestreo, B. megaterium y P. hibiscola fueron significativamente diferentes (p≤0,05) durante el ciclo V-2012 a los 52 dds. En el presente estudio se identificaron un total de 22 bacterias endófitas en raíces de líneas de maíces tolerantes y susceptibles a sequía. Sin embargo, la densidad de población de B. subtilis, B. megaterium, P hibiscola y S. meliloti fueron significativamente mayores en las líneas de maíz tolerantes que en las susceptibles a sequía (Cuadro 2).
La densidad de poblaciones en bacterias es un elemento clave y se considera como el mayor mecanismo de regulación del metabolismo en la interacción con el medioambiente biótico y abiótico, a través del cual coordina la expresión de genes especializados dependiente de una densidad celular. Este comportamiento conocido como Quorum sensing (QS), en bacterias asociadas a plantas se ha demostrado que regula la expresión de genes en la rizosfera para la síntesis de metabolitos secundarios, compuestos antifúngicos, antibióticos y enzimas extracelulares que están involucrados en el biocontrol. (Somers et al., 2004; Whitehead et al., 2001).
En este estudio, la densidad total de población de B. subtilis en las líneas de maíz en la localidad de El Batán fue de 3, 4 Log10 UFC g-1 de raíz, no hubo diferencia significativa por ciclo y días después de la siembra, pero fue significativamente mayor en las líneas de maíz tolerantes a sequía (Cuadro 2, Figura 1).
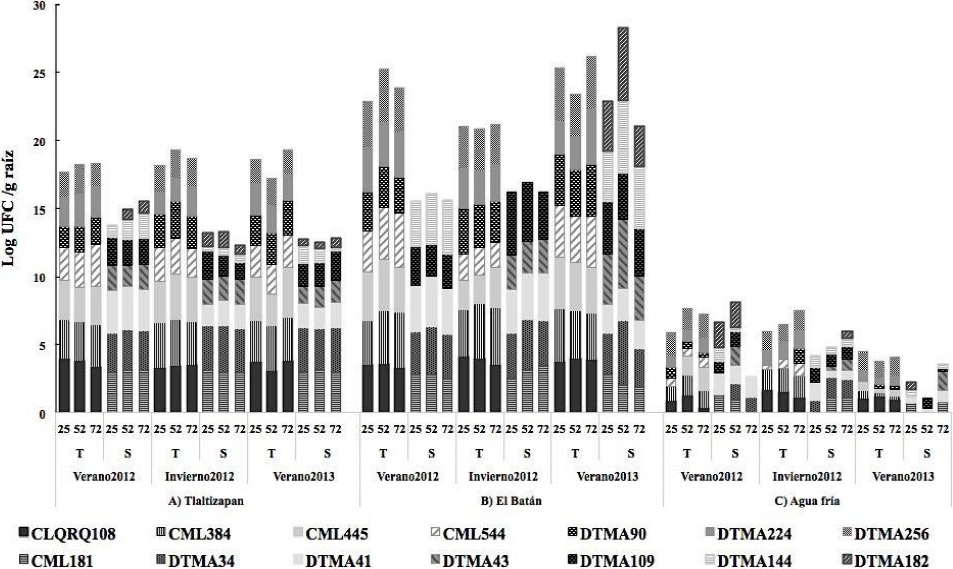
Figura 1 Población de B. subtilis en 7 líneas de maíz tolerantes y 7 susceptibles a sequía a los 25, 52 y 75 dds en los ciclos de cultivo Verano 2012, Invierno 2012 y Verano 2013, en tres localidades de México.
Como endófito, B. subtilis es un microorganismo de gran interés en aplicaciones biotecnológicas como agente de biocontrol de enfermedades bióticas y abióticas, mediada por la eficiente colonización de las raíces de las plantas que desencadenan diferentes mecanismos de biocontrol y adaptación a diferentes ambientes (Marulanda et al., 2006); además, demostrando su alta capacidad de producir compuestos orgánicos volátiles (VOCs) que actúan como moléculas de señalización para disparar una respuesta de defensa mediante la resistencia sistémica inducida (ISR) específica (Farag et al., 2013). Estos VOCs activan las vías de producción de hormonas incluyendo auxinas, giberelinas, citocininas y ácido salicílico, que promueven el desarrollo de la planta hospedante en condiciones de estrés, principalmente por el incremento de biomasa radicular resultando en una mejor absorción de agua (Zhang et al., 2007). Otro estudio demostró que la inoculación en semilla de maíz con cepas seleccionadas de B. subtilis aportó importantes beneficios como promotor de crecimiento y mayor capacidad de absorción de nutrientes (Canbolat et al., 2006).
En Tlaltizapán, la densidad de población de B. megaterium en las líneas de maíz estuvo entre 3, 5Log10 UFC g-1 de raíz en los tres ciclos y fue significativamente mayor (5 Log10 UFC g-1 de raíz) en el ciclo V-2012 a los 52 dds, fue significativamente mayor en las líneas de maíz tolerantes a sequía (Cuadro 2, Figura 2).
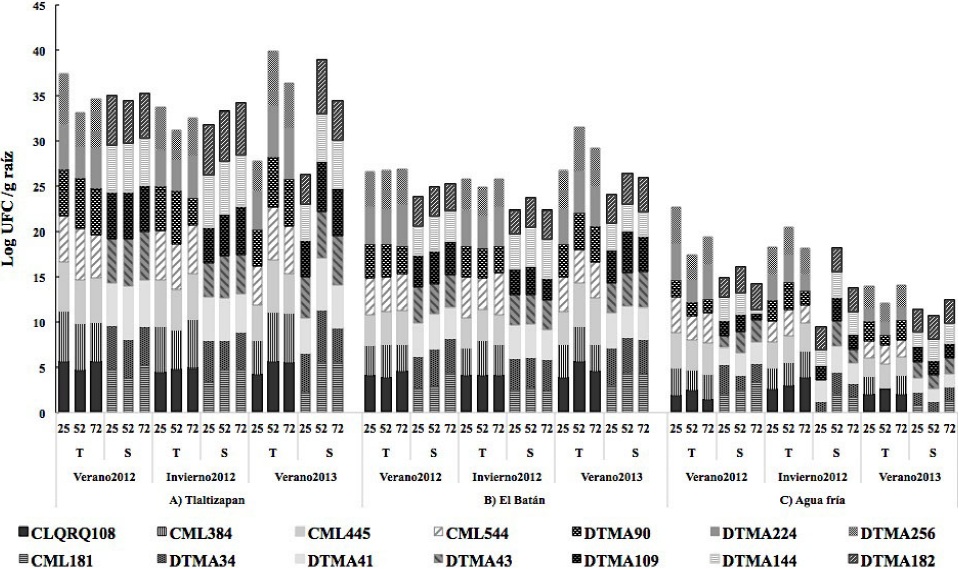
Figura 2 Población de B. megaterium en 7 líneas de maíz tolerantes y 7 susceptibles a sequía a los 25, 52 y 75 dds en los ciclos de cultivo Verano 2012, Invierno 2012 y Verano 2013, en tres localidades de México.
Investigaciones recientes demostraron que B. megaterium modifica la respuesta de la planta del maíz ante varios estreses abióticos, incluyendo la importancia de la densidad poblacional de algunas especies de Bacillus en la respuesta de tolerancia a sequía y transporte de agua en Retama sphaerocarpa, al estimular el crecimiento de la raíz y la capacidad de absorción de agua y los niveles de prolina y ácido indol acético (Marulanda et al., 2006). Particularmente, destacan que la presencia de B. megaterium en su planta hospedante aumenta la biomasa y contenido de agua de la raíz (Marulanda et al., 2009), que sugiere un uso potencial de estas poblaciones endófitas en plantas de maíz cultivadas en zonas áridas y semiáridas. La función biológica de otras especies de Bacillus endofíticos en maíz se relaciona con una eficiente respuesta de defensa de la planta contra patógenos relacionada con la producción de lipopéptidos antifúngicos que induce la expresión de genes de defensa (Gond et al., 2015).
En El Batán, la densidad de población de P. hibiscola en líneas de maíz estuvo entre 3, 5 Log10 UFC g-1 de raíz en los tres ciclos y fue significativamente mayor (5 Log10 UFC g-1 de raíz) en el ciclo V-2012. No se aisló en la localidad de Agua Fría. No hubo diferencias significativas entre los días después de la siembra (dds), fue significativamente mayor en las líneas de maíz tolerantes a sequía (Cuadro 2, Figura 3).
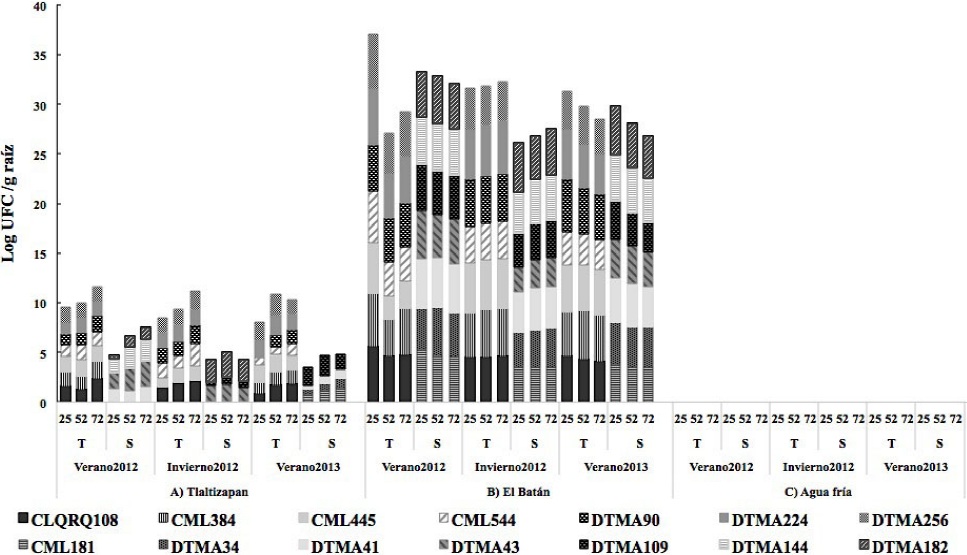
Figura 3 Población de P. hibiscola en 7 líneas de maíz tolerantes y 7 susceptibles a sequía a los 25, 52 y 75 dds en los ciclos de cultivo Verano 2012, Invierno 2012 y Verano 2013, en tres localidades de México.
P. hibiscola no se ha citado como endófito en maíz; sin embargo, la afiliación filogenética de la cepa tipo ATCC 19867 de P. hibiscola, basados en el análisis comparativo de secuencias del gen 16S rADN y perfiles quimio-taxonómicos, lo reclasifican como Stenotrophomonas sp. (Anzai et al., 2000). S. maltophilia se ha citado como endófito en maíz (McInroy y Kloepper, 1995) y se reporta su función como agente de control biológico contra patógenos con origen en el suelo como Pythium spp., Fusarium spp. y Rhizoctonia solani además de la capacidad para producir ácido indol acético que en plantas regula el desarrollo de brotes y raíces laterales (Mehnaz et al., 2010).
La densidad de población de S. meliloti en líneas tolerantes a sequía en Tlaltizapán estuvo entre 4, 6 Log10 UFC g-1 de raíz en los tres ciclos y fue significativamente mayor (6 Log10 UFC g -1 de raíz) a los 52 dds. No se aisló en la localidad de Agua Fría. No hubo diferencias significativas por ciclo de cultivo, pero fue significativamente mayor en las líneas de maíz tolerantes a sequía (Cuadro 2, Figura 4).
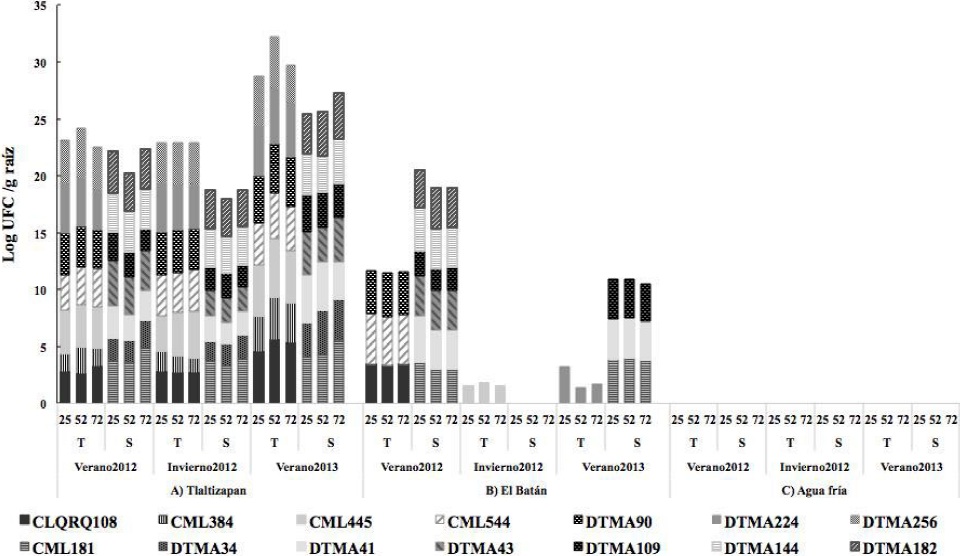
Figura 4 Población de S. meliloti en 7 líneas de maíz tolerantes y 7 susceptibles a sequía a los 25, 52 y 75 dds en los ciclos de cultivo Verano 2012, Invierno 2012 y Verano 2013, en tres localidades de México.
S. meliloti se conoce como una bacteria noduladora fijadora de nitrógeno en plantas del género Medicago, con implicaciones en la respuesta de tolerancia a sequía y salinidad (Roumiansteva y Muntyan, 2015). En México se determinó la existencia de una gran diversidad genética de poblaciones nativas de S. meliloti en alfalfa (Medicago spp.) y actualmente se han identificado nuevas especies en leguminosas leñosas en el género Leucaena (Wang et al., 2002) y Acacia (Toledo et al., 2003). El género Sinorhizobium se encontró en la rizosfera de maíz (Rosenblueth y Martínez-Romero, 2006) y a Rhizobium etli bv. phaseoli como endófito en raíces de maíz en México (Gutiérrez-Zamora y Martínez-Romero, 2001); sin embargo, no existe información sobre S. meliloti como endófito en la raíz de maíz, por lo que el presente estudio aporta información de referencia de la colonización natural endófita de S. meliloti en esta gramínea en México. En las localidades estudiadas es común el cultivo intercalado de alfalfa con maíz, que podría explicar la colonización endófita de raíces de maíz por S. meliloti. Estudios en arroz (Oryza sativa) sugieren que S. meliloti podría producir la molécula señal lumicromo en la rizosfera de estas plantas promoviendo el crecimiento por inducción de una mejor respiración de las raíces, conductancia estomática, transpiración de la hoja y eficiencia fotosintética (Chi et al., 2010). Su aplicación en concentraciones nanomoleculares promovió el crecimiento en leguminosas y gramíneas e incrementó la biomasa de las raíces de la leguminosa Lotus japonicus y jitomate (Solanum lycopersicum) (Gouws et al., 2012).
La frecuencia de ciertos géneros de bacterias aisladas de la raíz en este estudio podría tener relación con la filogenia de estas líneas de maíz. Por ejemplo, entre las bacterias con mayor frecuencia y densidad en líneas tolerantes de maíz destacan dos especies de Bacillus; Bacillus se considera una bacteria endófita importante que se ha aislado tanto de Teozintle (ancestro del maíz) como de genotipos de maíces modernos y se demostró que la composición de la comunidad bacteriana endófita en semilla de maíz, es variable y conservada a través de la evolución, etnografía y ecología de la planta de maíz como hospedante (Johnston-Monje y Raizada, 2011).
En esta investigación se determinó que algunos de los factores: tolerancia/susceptibilidad a sequía de las líneas de maíz estudiadas, localidad, ciclo de cultivo, tiempo (días después del trasplante) afectaron significativamente la densidad de bacterias cultivables endófitas en la raíz de estos materiales genéticos de maíz. Las rizobacterias como promotores del crecimiento (PGPR), nutrición y manejo de enfermedades en plantas ha sido ampliamente estudiado; sin embargo, su función en el manejo de enfermedades abióticas como el estrés a sequía es de gran interés en los últimos años (Dimpka et al., 2009; Grover et al., 2010; Kavamura et al., 2013; Yang et al., 2009). El uso de microorganismos endófitos puede ser una alternativa viable contra estreses bióticos y abióticos; las cepas aisladas en este estudio constituyen un recurso microbiano que está íntimamente asociados al maíz con potencial de uso biotecnológico en la agricultura que merecen ser evaluados en el futuro como inoculantes microbianos e inductores de tolerancia al estrés hídrico y otros factores bióticos y abióticos en regiones con condiciones limitantes para la producción de este cultivo en México.
Conclusiones
Las bacterias cultivables endófitas de la raíz que tuvieron mayor presencia en las 14 líneas de maíz probadas pertenecen a los phyla Proteobacteria y Firmicutes. La mayor densidad de población de bacterias endófitas se estimó en líneas de maíz tolerantes a sequía. Las bacterias de los géneros Bacillus, Pseudomonas y Sinorhizobium fueron las que presentaron mayor frecuencia y densidad de población en las líneas de maíz tolerantes a sequía en las tres localidades y ciclos de cultivo estudiados. Sinorhizobium meliloti es capaz de colonizar endofíticamente la raíz de maíz.











 texto en
texto en 


