Introducción
Las enfermedades causadas por fitopatógenos constituyen un factor importante para la producción de caña de azúcar. En México, aproximadamente el 80% de las enfermedades que afectan este cultivo son de origen fúngico, el 20% restante es causado por bacterias, virus y fitoplasmas (CONADESUCA, 2015). Entre las primeras epidemiologías sobre el cultivo de la caña de azúcar reportadas en el mundo a comienzos del siglo XX están las ocasionadas por virus, causando grandes pérdidas económicas (Grisham, 2000). El Virus del mosaico de la caña de azúcar (SCMV), agente causal de la enfermedad del mosaico, es uno de los patógenos virales de mayor importancia económica a nivel mundial (Gonçalves et al., 2012). La enfermedad fue responsable de drásticas epidemias en Argentina, Brasil, Cuba, Puerto Rico y Estados Unidos (Koike y Guillaspie, 1989; Yang y Mirkov, 1997). Lo anterior condujo a la introducción de híbridos interespecíficos del género Saccharum (tolerantes al mosaico) importados de Java, con el fin de controlar la rápida propagación de la enfermedad en las cañas nobles (S. officinarum) obtenidas en aquel entonces en esos países (Koike y Gillaspie, 1989). En variedades susceptibles infectadas con SCMV las pérdidas en rendimiento se estiman de 11-50% (Singh et al., 2003; Singh et al., 2005). En México, el primer reporte (basado en sintomatología) de este virus afectando a caña de azúcar fue realizado en 1929, en El Potrero, Veracruz. En 1947 más del 80% de las áreas plantadas con variedades de caña de azúcar criollas, del Ingenio Independencia en el municipio de Martínez de la Torre, presentaron la enfermedad (CONADESUCA, 2015). En ese mismo país el primer reporte (basado en métodos moleculares) del SCMV afectando plantas de maíz fue realizado en el año 2006 (Espejel et al., 2006), sin embargo, no hay información cuantitativa sobre los daños que ocasiona esta enfermedad en los cultivos de caña de México.
El síntoma general causado por el SCMV en caña de azúcar se caracteriza principalmente por presentar decoloraciones en la lámina foliar, en la cual se observan zonas de color verde normal alternado con áreas verde pálido o amarillentas; estas decoloraciones son resultado de los niveles de variación en la concentración de la clorofila en la hoja (Grisham, 2000; CONADESUCA, 2015). Los síntomas de esta enfermedad también pueden ser causados por otros virus y pueden confundirse con el mosaico estriado (SCSMV) o el mosaico del sorgo (SrMV), los cuales también infectan a caña de azúcar (Viswanathan et al., 2008; Xie et al., 2009). Además, las plantas asintomáticas también pueden resultar positivas al SCMV (Xu et al., 2008). En México, son escasos los reportes sobre la detección del SCMV por métodos moleculares, y mayoritariamente se han basado en síntomas (CONADESUCA 2015), por lo que no hay certeza de que el daño visual de mosaico corresponda realmente al SCMV o algún otro virus que manifiesta síntomas similares. Existen varios reportes que han estandarizado protocolos para la detección del SCMV mediante RT-PCR (Smith y Van de Velde, 1994; Xie et al., 2009; Filippone et al., 2010). Smith y Van de Velde (1994) desarrollaron los iniciadores S400-551 y S400-910, los cuales amplifican un fragmento de 359 pb, correspondiente a una región parcial que codifica para la proteína de la cápside (CP) del SCMV. En otro estudio, Yang y Mirkov (1997) desarrollaron los cebadores SCMV-F3/SCMV-R3, los cuales amplifican una banda de aproximadamente 900 pb, correspondiente a una región que codifica para la proteína CP del SCMV. Xie et al. (2009), diseñaron el juego de oligonucleótidos SCMV-F1/ SCMV-R1 basados en secuencias depositadas en la base de datos de NCBI; dichos iniciadores amplifican un fragmento de 720 pb, correspondiente a una región parcial de la proteína HC-Pro del SCMV. Recientemente, Filippone et al. (2010) optimizaron un protocolo para detectar al SCMV utilizando los oligonucleótidos descritos previamente por Yang y Mirkov (1997) y Smith y Van de Velde (1994), respectivamente.
Por otra parte, resulta importante conocer el origen filogenético del SCMV para conocer probables ingresos de esta enfermedad a México. En el año 2012 se reportó un análisis filogenético de 185 secuencias CP del SCMV provenientes de varios países, sugiriendo diferentes orígenes filogeográficos de dos aislados mexicanos que contemplo ese estudio (Chaves y Ortiz, 2012). Por otra parte, los resultados de Xie et al. (2016) del análisis filogenético de 24 aislados de SCMV provenientes de China y diversos países del mundo revelaron que podrían dividirse en dos grupos, los cuales fueron asociados con la especie vegetal hospedera del SCMV. Más recientemente, los resultados de Moradi et al. (2017) a partir de secuencias CP del SCMV de varios países sugieren cinco linajes evolutivos divergentes, donde se relacionan parcialmente el origen geográfico y/o plantas hospederas del SCMV. Por lo anterior, el objetivo del presente estudio fue detectar mediante RT-PCR la presencia y distribución del SCMV en las zonas cañeras del Pacífico de México y determinar el origen filogenético de un aislado del estado de Jalisco. La información generada en este trabajo podrá ser utilizada por el Centro de Investigación y Desarrollo de la Caña de Azúcar (CIDCA), tanto en su estación cuarentenaria para el intercambio de germoplasma, como dentro del programa de mejoramiento genético, en el que la selección y eliminación de clones susceptibles al SCMV son procedimientos rutinarios.
Materiales y métodos
Material vegetal
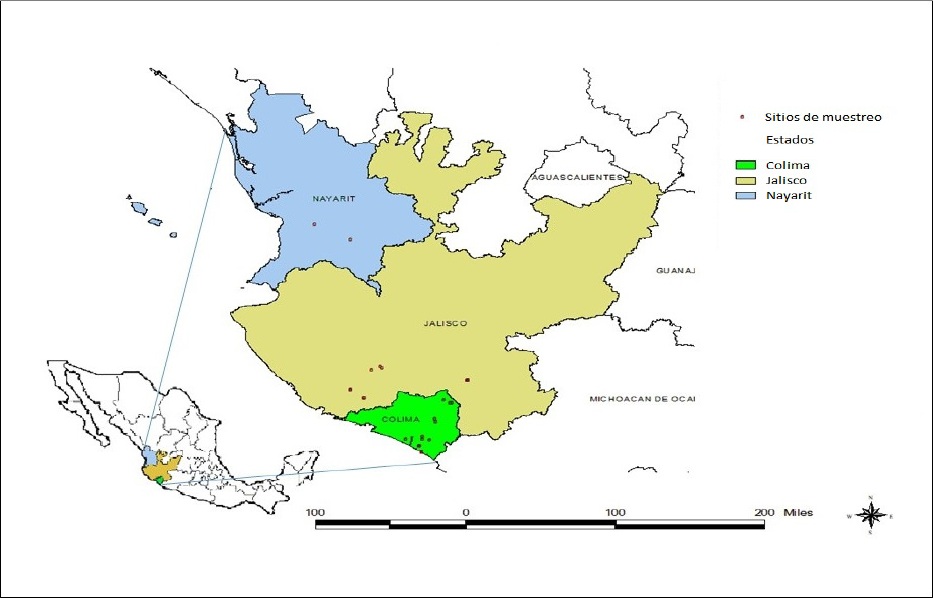
Durante el año 2014 se colectaron muestras foliares de diversas variedades de caña de azúcar sembradas comercialmente en los estados de Colima, Jalisco y Nayarit (Figura 1). Las plantas presentaron sintomatología típica de mosaico o fueron asintomáticas. Las muestras se envolvieron en bolsas plásticas, fueron colocadas en hieleras con refrigerantes térmicos y se trasladaron al laboratorio de biotecnología de plantas del Campo Experimental Tecomán del INIFAP ubicado en el km 35 de la carretera Colima-Manzanillo en Colima.
Extracción y cuantificación del RNA total
Se tomaron aproximadamente 200 mg de varias partes del tejido foliar de una misma planta. Estas hojas se pulverizaron con nitrógeno líquido hasta obtener un polvo fino, el cual fue depositado en un tubo de 1.5 mL y se homogeneizó con 500 μL del reactivo Tripure (Roche). Se siguieron las recomendaciones del fabricante para la extracción del RNA total. Finalmente, el RNA obtenido fue resuspendido en agua libre de RNasa (0.01% DEPC) y almacenado a -70°C. La cuantificación se realizó con un espectrofotómetro NanoDrop (Thermo Scientific) utilizando 1 μL del RNA total extraído y se midieron las relaciones de A260/280 y A260/230 para determinar su pureza.
Clonación de un fragmento del SCMV en pGEM-T Easy
Con el objetivo de contar con un control positivo para la detección del virus, el producto de PCR de 720 pb correspondiente a una muestra de follaje de caña de azúcar, obtenida de una localidad del estado de Jalisco fue purificada a partir del gel de agarosa con el kit “QIAquick Gel Extraction” (QIAGEN), según las indicaciones del fabricante. Posteriormente, el fragmento se clonó con el sistema de “pGEM-T Easy” (Promega) y se transformó en células de E. coli JM109 (Promega), las cuales se hicieron competentes previamente con CaCl2 de acuerdo a Riley et al., 2008. La comprobación del inserto clonado en el vector se realizó mediante la extracción de DNA plasmídico (Engebrecht et al., 2001) a las colonias recombinantes y posterior reacción de digestión con las enzimas EcoR I y Not I.
Análisis de secuencias
El DNA plasmídico del fragmento clonado en E. coli fue secuenciado en un secuenciador ABI PRISM 310 Genetic Analyzer en ambas direcciones por el método de terminación con el Big Dye de Applied Biosystems. La edición y el ensamblado de las secuencias “forward” y “reverse” se realizó con el programa CLC Main Workbench 7. Finalmente se comparó la similitud de las secuencias obtenidas contra las reportadas para SCMV en la base de datos del “National Center for Biotechnology information” (NCBI) empleando la herramienta “Basic Local Alignment Searh Tool” (BLAST).
Detección del SCMV por RT-PCR
La reacción de transcripción inversa (RT) del RNA total se realizó utilizando el kit “Reverse Transcription System” (Promega). El RNA fue desnaturalizado a 70 °C durante 10 min; la mezcla de reacción RT se llevó a cabo en un volumen de 20 µL conteniendo 5 mM de MgCl2, amortiguador RT 1 X, 1 mM de cada dNTP, 1 u/µL de inhibidor de ribonucleasa RNasin® recombinante, 15 u/µg de transcriptasa inversa AMV, 0.5 µg de mezcla de oligonucleótidos por µg de RNA y 5 µL de RNA total (1.5 µg aproximadamente). La mezcla anterior fue incubada a temperatura ambiente por 10 min y luego a 42°C durante 45 min. Inmediatamente después se incubó a 95°C durante 5 min y finalmente 0-5°C por 5 min. Los cDNA resultantes fueron utilizados como molde para la amplificación por PCR con los oligonucleótidos descritos por Xie et al. (2009). El volumen final de la mezcla de reacción fue de 25 µL, conteniendo 12.5 µL de REDTaq® ReadyMixTM (SIGMA-ALDRICH), 1 μM de cada oligonucleótido, 3 µL de cDNA y H2O grado molecular. La mezcla de reacción se incubó en un termociclador (Labnet) con el siguiente programa: un ciclo de 50°C por 30 min y 94°C por 2 min; 35 ciclos de 94°C por 30 seg, 56°C por 30 seg y 72°C por 1 min seguido de una extensión final a 72°C por 5 min. La electroforesis se realizó en gel de agarosa al 1% con buffer TAE 1 X y fueron cargados 12.5 µL de los productos de PCR; las muestras se corrieron con un voltaje de 120 V. Finalmente los geles se tiñeron con BrEt y se visualizaron en un transiluminador con luz UV (UVP) para el análisis de los resultados.
Análisis filogenético
Todos los análisis bioinformáticos se realizaron con el software CLC Workbench versión 7.0.3. Se seleccionaron y descargaron secuencias del análisis BLAST en NCBI con los mayores porcentajes de identidad con la secuencia de SCMV denominada JalMex-126 (KT334297) y se emplearon otras más de la misma base de datos (Cuadro 2). Para todas las muestras se realizaron PCR in silico, con los primers para SCMV descritos por Xie et al. (2009). En todas las secuencias en las que se generaron fragmentos de 720 pb, estos fueron extraídos y almacenados en una base de datos local. Las secuencias que no generaron el fragmento esperado fueron alineadas con las generadas en la base de datos. Se seleccionaron y guardaron las regiones alineadas de estas secuencias. Se utilizó como grupo externo la región HC-Pro del PRSV (NC_001785). Se realizaron alineamientos múltiples de secuencias con el algoritmo MUSCLE y se empleó el método de máxima verosimilitud y Neighbor-joining para la construcción del árbol filogenético. Se utilizó “General Time Reversible” (GTR) como modelo de sustitución de nucleótidos. El análisis bootstrap se realizó con 1,000 réplicas y finalmente el árbol filogenético fue editado con el software TreeGraph 2.
Cuadro 1 Relación de muestras de follaje de caña de azúcar positivas al SCMV mediante RT-PCR en la región cañera del Pacífico de México.
| No. | Muestra | Estado | Municipio | Georeferencia | Variedad |
| 1 | 87 | Colima | Colima | N 19°11’33.0’’ W 103°43’15.4’’ | CP 72-2086 |
| 2 | 88 | Colima | Colima | N 19°09’43.6’’ W 103°43’14.0’’ | CP 72-2086 |
| 3 | 35 | Colima | Cuauhtémoc | N 19°24’34.14’’ W 103°38’19.79’’ | Mex 69-290 |
| 4 | 82 | Colima | Cuauhtémoc | N 19°22’36.61’’ W 103°33’37.49’’ | CP 72-2086 |
| 5 | 90 | Colima | Cuauhtémoc | N 19°22’09.12’’ W 103°34’49.81’’ | CP 72-2086 |
| 6 | 148 | Colima | Tecomán | N 18°52’36.24” W 103°52’1.49” | CP 72-2086 |
| 7 | 336 | Colima | Tecomán | N 18°52’40.12” W 103°51’50.62” | CP 72-2086 |
| 8 | 23 | Colima | Tecomán | N 18°56’37.4’’ W 103°46’26.03’’ | Mex 69-290 |
| 9 | 33 | Colima | Tecomán | N 18°48’33.23’’ W 103°50’37.89’’ | Mex 69-290 |
| 10 | 146 | Colima | Tecomán | N 18°57’57.26’’ W 103°50’22.01’’ | Saccharum spp. |
| 11 | 2 | Colima | Tecomán | N 18°56’06.52’’ W 104°00’03.54’’ | Saccharum spp. |
| 12 | 37 | Colima | Tecomán | N 18°48’32.91’’ W 103°50’33.48’’ | Mex 69-290 |
| 13 | 133 | Colima | Tecomán | N 18°52’37.61” W 103°51’57.66” | Saccharum spp. |
| 14 | 387 | Colima | Tecomán | N 18°57’56.03’’ W 103°50’22.02’’ | Saccharum spp. |
| 15 | 350 | Jalisco | Autlán | N 19°45’07.23’’ W 104°19’36.69’’ | Mex 69-290 |
| 16 | 342 | Jalisco | Cuautitlán | N 19°26’49.58’’ W 104°24’10.45’’ | Saccharum spp. |
| 17 | 344 | Jalisco | Cuautitlán | N 19°26’54.74’’ W 104°23’40.70’’ | Mex 79-431 |
| 18 | 355 | Jalisco | El Grullo | N 19°48’49.81’’ W 104°14’35.95’’ | Mex 69-290 |
| 19 | 349 | Jalisco | El Grullo | N 19°47’44.20” W 104°13’38.63” | Atemex 96-40 |
| 20 | 413 | Jalisco | La Huerta | N 19°31’07.39’’ W 104°32’11.61’’ | Saccharum spp. |
| 21 | 341 | Jalisco | La Huerta | N 19°31’08.14’’ W 104°32’11.61’’ | Saccharum spp. |
| 22 | 369 | Jalisco | La Huerta | N 19°31’11.23’’ W 104°32’10.97’’ | Saccharum spp. |
| 23 | 340 | Jalisco | La Huerta | N 19°31’05.15’’ W 104°32’06.51’’ | Atemex 96-40 |
| 24 | 114 | Jalisco | Zapotiltic | N 19°38’43.00’’ W 103°24’31.01’’ | Atemex 96-40 |
| 25 | 120 | Jalisco | Zapotiltic | N 19°38’42.90’’ W 103°24’29.40’’ | Atemex 96-40 |
| 26 | 125 | Jalisco | Zapotiltic | N 19°38’24.12’’ W 103°25’03.64’’ | CP 72-2086 |
| 27 | 110 | Jalisco | Zapotiltic | N 19°38’27.25’’ W 103°25’04.85’’ | CP 72-2086 |
| 28 | 113 | Jalisco | Zapotiltic | N 19°38’42.9’’ W 103°24’29.7’’ | Atemex 96-40 |
| 29 | 126 | Jalisco | Zapotiltic | N 19°38’42.68’’ W 103°24’30.04’’ | Atemex 96-40 |
| 30 | 123 | Jalisco | Zapotiltic | N 19°38’42.31’’ W 103°24’31.21’’ | Atemex 96-40 |
| 31 | 138 | Jalisco | Zapotiltic | N 19°38’42.58’’ W 103°24’31’’ | Atemex 96-40 |
| 32 | 186 | Nayarit | Santa María del Oro | N 21°17’56.3’’ W 104°32’14.9’’ | Mex 69-290 |
| 33 | 170 | Nayarit | Xalisco | N 21°27’50.1’’ W 104°53’11.4’’ | Saccharum spp. |
Cuadro 2 Secuencias parciales del fragmento HC-Pro del SCMV obtenidas de la base de datos de NCBI y utilizadas en este estudio para el análisis filogenético.
| No. | Nombre del aislado | Origen | Hospedero | Año | No. Acceso |
| 1 | JalMex-126 | México | Saccharum spp. (Var. Atemex 96-40) | 2015 | KT334297 |
| 2 | Brisbane | Australia | Saccharum spp. | 2005 | AJ278405 |
| 3 | FZ-C1 | China | Saccharum spp. (Var. Badila) | 2014 | KR108212 |
| 4 | CBTCT-Seng | India | Saccharum sinense | 2014 | KX266877 |
| 5 | ARG-915 | Argentina | Saccharum spp. | 2007 | JX237863 |
| 6 | FZ-C2 | China | Saccharum spp. (Var. Badila) | 2014 | KR108213 |
| 7 | CBMungo | India | Saccharum barberi | 2014 | KX266876 |
| 8 | JAL-1 | México | Zea mays | 2010 | GU474635 |
| 9 | SCMV-VER1 | México | Zea mays | 2011 | EU091075 |
| 10 | Seehausen | Alemania | - | 2012 | JX185303 |
| 11 | SCMVgp1 | China | Zea mays | 2015 | NC_003398 |
Resultados
Síntomas
Los síntomas relacionados con la presencia del SCMV se encontraron en las muestras foliares colectadas de caña de azúcar de diferentes variedades en los estados de Colima, Jalisco y Nayarit, México; así mismo, se observaron variaciones en los patrones de mosaico observados (Figura 2 A). Otras muestras con mosaico también fueron detectadas con algunos síntomas como rayas blancas en nervaduras, lesiones por pústulas, clorosis, necrosis y hoja corrugada (Figura 2 B).
Clonación de un fragmento del SCMV
El control positivo utilizado para detectar la presencia y distribución del SCMV en tres estados del Pacífico de México se obtuvo de una muestra con síntomas severos de mosaico procedente del estado de Jalisco. Se amplificó y clonó un fragmento de 720 pb; posteriormente, fueron obtenidas siete colonias recombinantes y se realizó el análisis restrictivo con EcoR I y Not I para comprobar la presencia del fragmento clonado (Figura 3), el cual finalmente fue secuenciado. El análisis BLAST de la secuencia presentó hasta un 95% de identidad con otras accesiones de SCMV y fue depositada en la base de datos de NCBI con el registro KT334297. Se determinó que la secuencia corresponde a una región parcial del gen HC-Pro del SCMV.
Identificación y distribución del SCMV en tres estados del Pacífico de México
Todos los productos de RT-PCR sometidos a electroforesis en gel de agarosa que mostraron la amplificación de fragmentos de 720 pb del SCMV con el juego de oligonucleótidos SCMV-F1/SCMV-R1 descritos por Xie et al. (2009) fueron considerados como positivos al virus (Figura 4). En total se analizaron 242 muestras foliares de diversas variedades de caña de azúcar sembradas comercialmente, sintomáticas y asintomáticas paramosaico; además de la presencia de otros síntomas descritos anteriormente.

Figura 4 Detección del Sugarcane mosaic virus (SCMV) mediante RT-PCR en muestras de caña de azúcar procedentes de tres estados del Pacifico mexicano. C-: control negativo (H2O). C+: Control positivo (DNA plasmídico). M: Marcador de peso molecular 100 pb (Invitrogen). Los números corresponden a diferentes muestras analizadas.
En el Cuadro 1 se observa la amplia distribución que presenta el SCMV en las regiones tropicales y subtropicales del Pacífico de México que abarcó el presente estudio. En el estado de Colima se detectó en las variedades Mex 69-290 y CP 72-2086, entre otras más en etapa experimental. De igual manera, en Jalisco se presentó afectando a las mismas dos variedades, además de la Atemex 96-40. Finalmente, en Nayarit también la variedad Mex 69-290 fue la más afectada. En el Cuadro 1 se muestra la relación de 33 plantas que resultaron positivas al SCMV y su origen geográfico, lo que representa un 13.63% del total analizado. Las variedades con síntomas más severos de virosis y que resultaron positivas al SCMV fueron Mex 69-290, CP 72-2086 y Atemex 96-40. Algunas muestras presentaron síntomas claros de mosaico, sin embargo, resultaron negativas a la presencia del virus mediante RT-PCR.
Origen filogenético
El análisis filogenético con la secuencia parcial HC-Pro del SCMV de Jalisco y otras secuencias similares de otros países se muestra en la Figura 5. Se observa la formación de dos grupos, el primero incluye dos secuencias de México, EU091075 y GU474635 cuyos porcentajes de identidad con Jal-Mex-126 (aislado de este estudio) fueron de 80.39 y 79.28%, respectivamente; en este grupo también se incluyeron otras dos secuencias procedentes de China y Alemania. En el segundo grupo se encuentra la secuencia JalMex-126, junto con dos secuencias de la India, una de Australia, dos de China y una de Argentina, con las que comparte porcentajes de identidad de entre 92.5-95.14%, por lo que los virus de este grupo podrían tener un origen genético común. Para ambos grupos, los orígenes geográficos de los aislados fueron muy diversos; la principal diferencia entre estos dos grupos que se formaron radica en el tipo de hospedero del virus. Las secuencias virales del primer grupo tuvieron como hospederos a plantas de maíz, exceptuando al aislado de Alemania cuyo hospedero se desconoce. El resto de secuencias de HC-Pro del SCMV que formaron el segundo grupo son provenientes de diversos híbridos y especies de caña de azúcar (Cuadro 2).
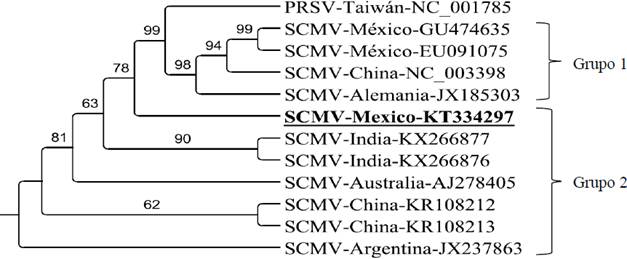
Figura 5 Árbol filogenético basado en el método de Neighbor-joining y máxima verosimilitud utilizando secuencias parciales del fragmento HC-Pro del SCMV. Las ramas con valores de bootstrap menores a 50% fueron colapsadas. Se utilizó como grupo externo al Papaya ringspot virus (PRSV). En negritas y subrayado se resalta el aislado de este estudio.
Discusión
La diversidad de síntomas presentes en las muestras analizadas combinados con patrones de mosaico, posiblemente se deban a infecciones mixtas con hongos, ya que algunos de los síntomas observados corresponden a enfermedades como mancha café, roya café y pokkah boeng, cuyos agentes causales son Cercospora longipes, Puccinia melanocephala y Gibberella fujikuroi, respectivamente (Raid y Comstock, 2000; Saumtally y Sullivan, 2000; Whittle e Irawan, 2000); sin embargo, se deben realizar las pruebas moleculares para confirmar la identidad de estos agentes causales. Por otra parte, según Xie et al. (2009) la enfermedad del mosaico es ocasionada por un complejo de tres virus: SCMV, Sorghum mosaic virus (SrMV) y Sugarcane streak mosaic virus (SCSMV), de los cuales los últimos dos no se han reportado afectando a caña de azúcar en México. Probablemente las muestras con características de mosaico que resultaron negativas a la prueba de RT-PCR para detectar SCMV en el presente estudio se deba a que sus síntomas estén asociados con la presencia de alguno de los virus antes mencionados (Grisham, 1994; Xie et al., 2009), o incluso aquellas muestras con mosaicos severos y positivas al SCMV contengan infecciones virales mixtas. Recientemente Balarabe et al. (2014) mencionan que también el Johnsongrass mosaic virus (JGMV) y el Maize dwarf mosaic virus (MDMV) son responsables de la enfermedad del mosaico en caña de azúcar en Nigeria. Hasta el momento se ha reportado la presencia del SrMV en Estados Unidos, China y Vietnam (Yang y Mirkov, 1997; Grisham y Pan, 2007; Ha et al., 2008; Zhang et al., 2016), mientras que el SCSMV está limitado para las zonas cañeras del continente asiático (Hall et al., 1998; Hema et al., 1999; Rao et al., 2006). Sin embargo, debido a la cercanía de Estados Unidos con México sería muy probable que el SrMV se encuentre infectando a las plantas de caña de azúcar en nuestro país.
La detección del SCMV por RT-PCR en este estudio confirma su amplia distribución en las zonas cañeras de la región del Pacífico de México. El virus incluso está presente en el continente europeo, donde no se cultiva caña de azúcar, debido a la cantidad de especies vegetales que pueden servir como reservorios del mismo (Dallwitz, 1980; Dallwitz et al., 1993; Brunt et al., 1996). En México, las variedades Mex 69-290 y CP-72-2086, además de representar más del 50% del área cultivada con caña de azúcar, tienen más de 30 años de uso, lo que ha conllevado al deterioro genético de los materiales que con el tiempo han presentado susceptibilidad a diversas enfermedades, incluidas las virales. Además, sus características de multiplicación vegetativa (semilla-estaca), así como la presencia de los áfidos vectores Melanaphis sacchari y Rophalosiphum maidis (Figueredo et al., 2004) han favorecido la dispersión del virus.
Hay varios reportes que han identificado la presencia del SCMV en varios países (Smith y Van de Velde, 1994; Xie et al, 2009; Filippone et al., 2010; Sawazaki et al., 2013). El presente estudio se basó en el trabajo descrito por Xie et al. (2009) para la estandarización del método de detección mediante RT-PCR; sin embargo, a pesar de que las técnicas de diagnóstico molecular tienen mayor sensibilidad que los métodos serológicos, estos últimos son más económicos y factibles de utilizar cuando se deben procesar un elevado número de muestras (Gonçalves et al., 2007; Balarabe et al., 2014). En México, se atribuye al SCMV como único agente causal de la enfermedad del mosaico; sin embargo, no existen reportes sólidos o con validez científica para sostener estos hechos debido a que la información que se tiene sobre la presencia del virus en el país datan desde los años 1930-1950, además de que se han basado en la sintomatología típica de la enfermedad (CONADESUCA, 2015), lo cual no es concluyente. En México, los estudios relacionados con el SCMV se han centrado en el cultivo del maíz (Espejel et al., 2006; Chaves et al., 2011; Chaves y Ortiz, 2012). Chaves et al. (2011) obtuvieron un aislado del SCMV de maíz en el estado de Veracruz y utilizaron las variedades de caña de azúcar CP 72-2086 y MY 44-12 como genotipos de referencia para inocular el virus en la lámina foliar tanto de maíz como caña para determinar el movimiento del SCMV-Ver1. Por lo anterior, el presente trabajo representa el primer estudio centrado en caña de azúcar para detectar la presencia y distribución del SCMV en la zona cañera del Pacífico mexicano. Finalmente, cabe destacar que, en México, hacen falta estudios que cuantifiquen el daño ocasionado por la enfermedad del mosaico en caña de azúcar ya que la mayor parte de los productores y la agroindustria desconocen el impacto económico que podrían tener en el cultivo.
Con respecto al origen filogenético del aislado JalMex-126, los resultados indican que podría tener un origen genético común con aislados de India, Australia, China y Argentina, lo que sugiere deficiencias en el control fitosanitario durante el intercambio de germoplasma, lo cual es muy común por el tipo de reproducción de semilla-estaca en caña de azúcar. Sin embargo, debido a que el número de secuencias HC-Pro del SCMV es muy reducido en la base de datos de NCBI a comparación del número de secuencias CP, no fue posible relacionar el origen del aislado mexicano con países cañeros importantes como Brasil, Estados Unidos, Colombia, entre otros. En este estudio se obtuvo la formación de dos grupos basados en secuencias parciales HC-Pro de SCMV, esta misma tendencia fue reportada previamente por Chaves y Ortiz (2012) y Xie et al. (2016), utilizando secuencias CP de SCMV. De forma similar, estos autores coinciden que la formación de estos grupos está asociada con los hospederos que tiene el SCMV: caña de azúcar y maíz, principalmente.
Según Chaves y Ortiz (2012), el aislado mexicano JAL-1 con número de acceso GU474635 es filogenéticamente más cercano a aislados de SCMV de Brasil y Estados Unidos, mientras que secuencias de la CP del SCMV reportadas en China y Alemania son filogenéticamente más cercanas al aislado mexicano SCMV-VER1 con número de acceso EU091075. En este estudio, los aislados mexicanos antes mencionados estuvieron estrechamente relacionados y se agruparon con secuencias de China y Alemania, conformando el grupo uno. Por otra parte, Moradi et al. (2017), también contemplaron en su análisis las mismas dos secuencias mexicanas de SCMV y las colocaron en dos grupos diferentes.
Por lo anterior, es probable que haga falta incluir más secuencias HC-Pro del SCMV al análisis filogenético para observar una tendencia similar a la reportada por estos autores.
Conclusiones
Las pruebas de RT-PCR realizadas a material vegetativo sintomático y asintomático de caña de azúcar permitieron identificar la presencia del SCMV en los estados de Colima, Jalisco y Nayarit. Este virus se encuentra ampliamente distribuido en las zonas cañeras del Occidente de México, probablemente por la presencia de áfidos vectores y la propagación por semilla estaca potencialmente infectada. El análisis filogenético utilizando secuencias parciales HC-Pro del aislado de SCMV JalMex-126 permitieron identificar dos grupos, el primero que tuvo como hospederos a plantas de maíz y el segundo con plantas hospederas de diferentes especies e híbridos de caña de azúcar. El aislado JalMex-126 de este estudio es probable que comparta un origen genético común con aislados de India, Australia, China y Argentina.











 text in
text in