Ralstonia solanacearum (Smith, 1896) es uno de los fitopatógenos bacterianos más destructivos en la agricultura. Existe una alta variabilidad dentro de la especie, por lo que R. solanacearum es considerada una “especie complejo”, abreviado como RSSC por sus siglas en inglés “Ralstonia solanacearum species complex” (Safni et al., 2014; Prior et al., 2016). Una especie complejo es definida como un grupo de aislados relacionados cercanamente, cuyos miembros individuales pueden representar más de una especie (Fegan y Prior, 2005). R. solanacearum tiene una amplia distribución mundial y un alto número de plantas hospedantes, que incluye cientos de plantas de al menos 54 familias botánicas, tanto dicotiledóneas como monocotiledóneas (Prior et al., 2016). Muchas de las especies vegetales afectadas son cultivos agrícolas estratégicos o importantes alimentos de subsistencia. Entre estos figuran la papa (Solanum tuberosum), tomate (S. lycopersicum), berenjena (S. melongena), maní (Arachis hypogea) y los plátanos (Musa spp.) (Cardozo et al., 2010).
El plátano es la fruta tropical de mayor producción a nivel mundial. En el 2013 se reportó una producción de alrededor de 17 millones de toneladas (FAO, 2014). India es el principal productor, con el 26% del total mundial. El plátano es un producto básico para la alimentación y su exportación genera divisas y es fuente de empleo en numerosos países en desarrollo.
México está entre los primeros quince países en la producción de plátano (FAO, 2017; Workman, 2017), con un volumen de producción de 2.2 millones de toneladas y una derrama económica de M$ 6,209,716.85 (SIAP, 2017). Se cultiva en 17 estados de México, siendo Chiapas y Tabasco los principales estados productores, contribuyendo con el 60% de la producción (SIAP, 2017).
El Moko bacteriano es uno de los problemas fitosanitarios más importantes en la agroindustria platanera en países tropicales, siendo la principal enfermedad bacteriana en este cultivo. En la actualidad la enfermedad se encuentra presente en varios países productores de plátano en América, Asia, África y Oceanía (Cardozo et al., 2010).
El patógeno sobrevive o permanece latente en residuos de cosecha infectados y rizósfera de malezas hospedantes, siendo este el inóculo primario e infecta a plantas sanas por las raíces y heridas, colonizando los vasos del xilema. Los síntomas incluyen hojas amarillas y flácidas que finalmente colapsan y quedan adheridas a la planta; los frutos son chiclosos, de aspecto oscuro y tendiendo a degradarse. Se observa una necrosis vascular interna y la presencia de puntos oscuros debido a una infección sistémica. Esta enfermedad es común en el genotipo ABB (plátano de cocción), pero todos los genotipos de plátanos son afectados (Albuquerque et al., 2014). En México esta enfermedad se presenta en algunas áreas y está sujeta a control oficial; la campaña contra Moko del plátano opera en los estados de Chiapas, Tabasco y Nayarit, con el objetivo de reducir el riesgo de dispersión a zonas sin presencia (SENASICA, 2016).
Se tienen reportes que R. solanacearum causante de Moko se introdujo a México en 1960 en la zona platanera de Tapachula, Chiapas. Ocho años más tarde la enfermedad se extendió a otros 12 municipios de dicho estado. En 1991 se presentaron los primeros reportes en Teapa, Tabasco, y en el 2009 la enfermedad se extendió a los municipios de Cunduacán, Huimanguillo, Centro, Cárdenas y Jalapa. El Moko del plátano se encuentra presente en las zonas plataneras de Pichucalco, Huehuetán, Mazatán, Suchiate y Tapachula en el estado de Chiapas; en los municipios de Centro, Cunduacán, Huimanguillo, Jalapa, Teapa y Cárdenas en el estado de Tabasco, y en el municipio de Santiago Ixcuintla en el estado de Nayarit. Su incidencia ha aumentado en el estado de Tabasco en los últimos años (SENASICA, 2015). Actualmente está sujeta a control oficial, mediante la campaña fitosanitaria contra Moko del plátano, que opera en los estados de Chiapas, Tabasco y Nayarit, con el objetivo de reducir el riesgo de dispersión a zonas sin presencia, reduciendo los niveles de infestación en las zonas bajo control fitosanitario (SENASICA 2017).
R. solanacearum se subdivide en cinco biovares, con base en su metabolismo de carbohidratos, y cinco razas, en base al rango de hospedantes que infecta (Denny y Hayward, 2001; Fegan y Prior, 2005; Champoiseau et al., 2009). Las razas pueden ser hospedante-específico como las 4 y 5 que infectan jengibre y mora respectivamente, o pueden presentar un amplio rango como la raza 1 (tomate, papa, berenjena, entre otros), la raza 3 (papa, tomate y geranio) y la raza 2 (heliconias y plátanos) (Denny y Hayward, 2001; Champoiseau et al., 2009).
Debido a que R. solanacearum comprende un grupo heterogéneo de cepas con alta diversidad genética y fenotípica, su clasificación ha estado en continuos replanteamientos desde hace 50 años (Hayward, 1964; Fegan y Prior, 2005; Safni et al., 2014; Prior et al., 2016). La clasificación en biovares no refleja el fondo genético de las cepas, por lo que actualmente se utiliza un esquema de clasificación genética organizada jerárquicamente en las categorías de filotipos y secuevares (Fegan y Prior, 2005; Prior et al., 2016). Los filotipos correlacionan las cepas de R. solanacearum con sus orígenes geográficos: el filotipo I comprende cepas principalmente de Asia; el Filotipo II los de América; el Filotipo III de África y el Océano Índico; y el filotipo IV de Indonesia, Japón y Australia (Fegan y Prior, 2005). Los filotipos se subdividen en secuevares, los cuales están basados en la secuencia nucleotídica del gen Egl, que codifica para una endoglucanasa (Fegan y Prior, 2005). Hasta el momento, se han identificado 54 secuevares en el complejo de R. solanacearum (Li et al., 2016); los secuevares causantes de la enfermedad Moko en plátano son el 3, 4, 6, 24, 25, 41 y 53. En el continente americano la enfermedad Moko está presente desde Brasil hasta Estado Unidos (Sanchez-Perez et al., 2008; Norman et al., 2009; Hong et al., 2012; Albuquerque et al., 2014).
Para estudios genéticos, de patogénesis u otras caracterizaciones, R. solanacearum es usualmente aislada en medio semiselectivo SMSA (“selective media from south africa”), (Álvarez-Restrepo et al., 2008; Cardozo et al., 2010; Elphinstone et al., 1996). Sin embargo, aún sobre este medio el aislamiento de esta bacteria es difícil debido al crecimiento de otros microorganismos (Champoiseau et al., 2009; Kalpage y De Costa, 2015), particularmente bacterias de los géneros Klebsiella, Erwinia y Pseudomonas. Estos tres géneros de bacterias gram-negativas, así como otras especies de Ralstonia como por ejemplo Ralstonia mannitolilytica, son endófitos mayoritarios en las plantas de plátano (Thomas et al., 2008; Ganen et al., 2009; Souza et al., 2013). Estas bacterias son contaminantes comunes en los cultivos de R. solanacearum debido a que presentan mayor capacidad de crecimiento y muestran morfologías microscópicas y coloniales similares (French et al., 1995; Nasim, 2011).
El aislamiento de cepas puras de R. solanacearum puede llevar bastante tiempo (French et al., 1995; Thera, 2007; Döölotkeldieva y Bobuşeva, 2014; Döölotkeldieva y Bobusheva, 2016). En este reporte se describe una estrategia que facilita el aislamiento y caracterización genética de R. solanacearum a partir de plátano enfermo de Moko.
MATERIALES Y MÉTODOS
Muestreo de tejidos vegetales
Se realizaron muestreos en Tabasco (20-24 de octubre, 2014) con apoyo del personal del Comité Local de Sanidad Vegetal de Tabasco (CESVETAB) para la ubicación, diagnóstico visual y colecta del material, de acuerdo a instrucciones del SENASICA. Las muestras se depositaron en bolsas tipo Ziploc y se etiquetaron con la información del tejido colectado, fecha y sitio de muestreo. Las muestras fueron colocadas en hieleras con geles refrigerantes y enviadas al laboratorio de biotecnología molecular de plátano del Centro de Investigación Científica de Yucatán, donde fueron procesadas.
Detección de R. solanacearum por pruebas inmunológicas
Para la detección de R. solanacearum se utilizó el ensayo ELISA (“Enzyme-Linked Immunosorbent Assay”) (Agdia®, N.C. SRP 33900), en placa de 96 pozos, de acuerdo a las instrucciones del fabricante. Los controles positivos y muestras de plantas infectadas con R. solanacearum generan una coloración azul; los controles negativos y muestras de plantas sanas dan resultado incoloro. Se examinó visualmente la placa para evitar falsos positivos por posibles contaminaciones o suciedad en la placa y se midió la absorbancia espectrofotométrica a 655 nm (Bio-Rad Modelo 680XR). La densidad óptica igual o menor de 0.09 se consideró negativo. Para el escrutinio de las muestras en el laboratorio también se utilizaron inmunotiras (Agdia®, N.C. STX33900), siguiendo las instrucciones del fabricante.
Extracción de ADN
Para enriquecer la población de R. solanacearum en el medio SMSA, un fragmento (1 cm2) del tejido vegetal infectado se depositó en el medio líquido y se incubó durante 24 h en agitación a 180 rpm a 28 °C (Álvarez-Restrepo et al., 2008). Una alícuota de este cultivo (1.5 mL) se centrifugó a 5,200 X g durante 5 min. La pastilla bacteriana se resuspendió con 1 mL de buffer CTAB (2% de bromuro de cetil trimetil-amonio, 1% polivinil pirrolidona, 100 mM Tris-HCl, 1.4 M NaCl, 20 mM EDTA), agitando con vórtex durante 30s. Posteriormente se congeló a -20 °C por 10 min y se incubó a 65 °C a baño María por 20 min. Se temperizó 1 min a temperatura ambiente y centrifugó a 20,800 X g. El sobrenadante se recuperó en tubo de 1.5 mL para microcentrífuga, se adicionaron 500 µL de fenol:cloroformo:isoamílico (25:24:1) y se agitó por 10 min a temperatura ambiente (CNRF, 2012). Posteriormente, se centrifugó 5 min a 20,800 X g y se colectó la fase acuosa. Se continuó con el procedimiento estándar de recuperación de ADN con etanol absoluto frío, lavados con etanol al 70%, secado de la pastilla y resuspensión en agua destilada ultrapura (Sambrook y Russell, 2001). La pureza del ADN se determinó por espectrofotometría a 260nm/280nm y la integridad se determinó mediante electroforesis en gel de agarosa.
Detección de R. solanacearum por PCR
La detección se realizó mediante la ampliación de regiones genómicas con los iniciadores de 759: 5’- GTC GCC GTC AAC TCA CTT TCC - 3’ y 760: 5’- GTC GCC GTC AGC AAT GCG GAA TCG- 3 (Diagnóstico universal de R. solanacearum, 280 pb), (Opina et al., 1997); pehA#3-F 5’-CAG CAG AAC CCG CGC CTG ATC CAG- 3’ y pehA#6-R 5’- ATC GGA CTT GAT GCG CAG GCC GTT- 3’ (gen pga, 532pb), (Gillings et al., 1993); ISRso19-F 5’- TGG GAG AGG ATG GCG GCT TT - 3’ y ISRso19-R 5’ - TGA CCC GCC TTT CGG TGT TT - 3’ (iniciadores específicos a la Raza 2; 1884pb), (Lee y Chin, 2003). La mezcla de reacción de PCR se preparó con 20 ng de ADN, 0.1 µM de cada iniciador, 1 U de Taq ADN polimerasa recombinante (Invitrogen®), IX del Buffer de la enzima Taq, 200 µM de cada dNTP y 1.5 mM MgCl2, en 25µL de volumen final. El programa en el termociclador (Bio-Rad modelo T100™) para el diagnóstico universal de R. solanacearum (iniciadores 759/760) tuvo un ciclo de desnaturalización de 2 min a 96 °C, seguido de 35 ciclos de 94 °C por 45 s, alineamiento a 64 °C por 20 s y un ciclo de 72 °C por 1 min y al final, un ciclo de extensión de 72 °C por 5 min. El programa de amplificación para los otros genes fue idéntico, excepto que el paso de alineamiento fue de 70°C por 45 s para el gen pga, y para la raza 2 el alineamiento fue de 55 °C por 30 s y la extensión de 72 °C por 1.5 min en cada ciclo. Los productos (10 µL de cada reacción PCR) se analizaron por electroforesis, en gel de agarosa de 1.5% (p/v) usando buffer TAE (Tris-acetato-EDTA) 1X suplementado con 3 µL de bromuro de etidio (10mg/mL). La visualización de las bandas se realizó sobre un transiluminador con luz UV (Gel Doc™ XR, Bio-Rad).
Aislamiento de R. solanacearum
Se utilizaron tejidos (pseudotallo y fruto) que fueron positivos en los ensayos inmunológicos de ELISA e inmunotiras. Las muestras se cortaron en pequeños trozos con una cuchilla estéril y posteriormente se desinfestaron mediante dos lavados con agua estéril, hipoclorito de sodio al 10% (partiendo de una solución comercial al 6%) durante 1 min, agua estéril, etanol por 30 s, agua estéril y posteriormente se colocó en buffer TE y se dejó incubar por una hora, con agitación suave. Se sembró una asada sobre medio semiselectivo SMSA (formulación por cada litro: 1g de casaminoácidos, 10g de peptona, 5g de glucosa, 5mg de cristal violeta, 100mg de β-sulfato de polimyxina, 25mg de bacitracina, 5mg de cloranfenicol, 0.5mg de penicilina, 17g de agar bacteriológico, 50mg de TZC (2,3,5-trifenil cloruro de tetrazolio) y se incubó a 28 °C durante 72 h (Kelman, 1954; Cardozo et al., 2010). Las colonias resultantes fueron sembradas por estría cruzada sobre medio B-King sólido y cultivadas a 28 °C por 48 h. El medio B-King permite distinguir a las bacterias Pseudomonas spp. debido a que éstas generan fluorescencia al ser expuestas a una longitud UV de 360 nm, mientras que R. solanacearum no fluoresce (King et al., 1954; Dulla y Lindow, 2009; Lamichhane y Varvaro, 2013).
Caracterización del secuevar
Para la caracterización de los secuevares se utilizaron los iniciadores de la serie Mus de Fegan y Prior (2005): 5´-CGGGTCGCTGAGACGAATATC-3´ y 5´-GCCTTGTCCAGAATCCGAATG-3´ (Secuevar 4 , 351 pb); 5´-GCAGTAAAGAAACCCGGTGTT-3´ y 5´-TCTGGCGAAAGACGGGATGG-3´ (Secuevar 3, 400 pb); 5´-GCTGGCATTGCTCCCGCTCAC-3´ y 5´-TCGCTTCCGCCAAGACGC-3´ (Secuevar 4 SFR, 167 pb); 5´-CGTTCTCCTTGTCAGCGATGG-3´ y 5´-CCCGTGTGACCCCGATAGC-3´ (Secuevar 6, 221 pb). Se realizó PCR en Multiplex utilizando 20 ng de ADN, 2 U de Taq ADN polimerasa recombinante (Invitrogen®), 1X del Buffer de la enzima Taq, 200 µM de cada dNTP, 1.5 mM MgCl2 y 6 pmoles de cada iniciador, en un volumen final de 25µL. Las condiciones de PCR fueron similares a las descritas previamente, usando temperatura de alineamiento de 59 °C. Las PCR sencillas para cada secuevar se realizaron en las mismas condiciones, pero usando en cada caso una sola pareja de iniciadores. En cada caso se analizó mediante electroforesis en gel de agarosa 15 µL del producto de la reacción.
RESULTADOS Y DISCUSIÓN
Se realizó el diagnóstico de Moko bacteriano en muestras de pseudotallo y fruto de plátano de plantas sospechosas. El diagnóstico del material vegetal colectado se realizó mediante 2 pruebas serológicas y 2 ensayos de PCR. Un total de 40 muestras fueron analizadas, de las cuales 35 fueron positivas en el diagnóstico con el ELISA y 28 con el diagnóstico con las inmunotiras. Las 40 muestras fueron analizadas por PCR con los dos pares de iniciadores, y ambas reacciones de PCR amplificaron 28 muestras. El diagnóstico con las inmunotiras proporcionó 100% de congruencia con los diagnósticos por PCR, tanto en número de positivas como en las muestras identificadas, mientras que el ensayo ELISA presentó un 20% de error (7 falsas positivas de las 35). Ninguno de los dos métodos inmunológicos presentó falsos negativos (Cuadro 1). Las Figuras 1A y 1B muestran resultados típicos obtenidos en los diagnósticos de ELISA y con las inmunotiras. Algunas inmunotiras eran más oscuras que otras, dependiendo de la fenolización del extracto, pero la coloración no interfirió en la reacción de la línea de control y la del resultado negativo o positivo.
Cuadro 1 Diagnóstico de la enfermedad Moko bacteriano en plantas de plátano con sintomatología sospechosa.

x Se analizaron en total 40 muestras: 14 de pseudotallo y 26 de fruto
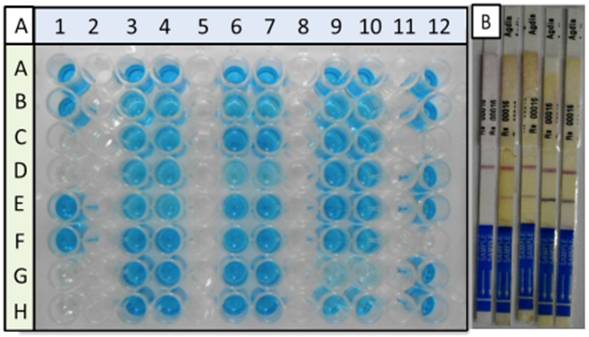
Figura 1 Diagnóstico serológico de Moko bacteriano en muestras de pseudotallo y fruto de plátano. Panel A, diagnóstico por ELISA. Los controles y muestras fueron analizados por duplicado. Columna 1, controles negativos (Filas C, D, G y H) y positivos (Filas A, B, E y F). Las muestras se colocaron en las columnas 3, 4, 6, 7, 9, 10 y 12. Panel B, diagnóstico mediante inmunotiras; se incluye el resultado de una muestra negativa (una sola línea que corresponde al control); muestras positivas, dos líneas. En ambos diagnósticos se observan diferencias en la intensidad de color de la señal en las muestras positiva, lo cual se relaciona con las diferencias en la cantidad de inóculo, de acuerdo al fabricante.
La técnica de ELISA ha sido ampliamente usada en los protocolos de identificación de R. solanacearum entre colonias con fenotipo similares (Álvarez-Restrepo et al., 2008; Döölotkeldieva y Bobusheva, 2016). El diagnóstico se basa en el uso de anticuerpos monoclonales contra exopolisacáridos (EPS) producidos por la bacteria. Naranjo-Feliciano y Martínez-Zubiaur (2013) diagnosticaron tejidos vegetales enfermos comparando los ensayos de ELISA, la aglutinación con partículas látex y la detección de R. solanacearum por PCR. La aglutinación con partículas látex fue la técnica más rápida y más sencilla entre las 3, pero fue menos específica, ya que reaccionó con otros microorganismos, y un orden de magnitud menos sensible (límite 106 ufc/mL) que la otra prueba serológica. De acuerdo al fabricante, el límite de detección de R. solanacearum en la técnica de ELISA es de 105 ufc/mL. Estos autores observaron diferencias en la confiabilidad de la detección de la bacteria patógena por los 3 métodos, cuando compararon los resultados del diagnóstico con el desarrollo de la enfermedad en la planta posteriormente. La confiabilidad del diagnóstico por PCR fue hasta 20% superior en comparación con la prueba de ELISA. En el presente trabajo la prueba de PCR confirmó 80% de los casos positivos diagnosticados con ELISA, indicando 20% de falsos positivos en esta prueba serológica, lo que coincide con el reporte de Naranjo-Feliciano y Martínez-Zubiaur (2013). Otros autores han discutido las limitaciones de la prueba de ELISA y las ventajas sobre ella de los métodos moleculares, que pueden detectar hasta 102 ufc/mL (Álvarez-Restrepo et al., 2008). Eriksson y Aspan (2007) compararon la PCR y el ELISA en el diagnóstico de bacterias del género Salmonella en muestras fecales de cerdo, y encontraron discrepancia entre los resultados de ambas técnicas debido a la baja especificidad de los anticuerpos que usaron, lo que muestra que la problemática del inmunodiagnóstico por ELISA no es exclusivo del diagnóstico de R. solanacearum.
El diagnóstico de R. solanacearum en las muestras de plátano con las inmunotiras fue 100% congruente con la detección posterior por PCR. La inmunotira es un diagnóstico de bolsillo, comercializado también por Agdia®, Inc. y ofrece el mismo límite de detección que el ensayo de ELISA (105 ufc/mL). Estas inmunotiras han sido usadas en otros trabajos como método confirmativo de identidad de cepas aisladas de R. solanacearum (Sánchez-Pérez et al., 2008; Hong et al., 2012). Thera (2007) utilizó inmunotiras para diagnosticar R. solanacearum en tejidos de tomate, de papa, de tabaco y de chile verde. Las inmunotiras tuvieron un falso negativo en una muestra congelada de chile verde y confirmaron 24 de las 25 muestras infectadas (96%), lo que apoya que esta prueba tiene alta certidumbre.
Korus (2011) diagnosticó Clavibacter michiganensis comparando ELISA e inmunotiras y obtuvo resultados similares de 100% de eficiencia de detección usando inmunotiras, en comparación con eficiencias de detección variables (entre 33 y 100%) en las pruebas con ELISA para diferentes subespecies del patógeno. Ambos ensayos pueden usar los mismos anticuerpos, como es el caso de los productos Agdia® para detectar R. solanacearum. La detección con las inmunotiras es sencilla, i.e. la muestra es macerada y aplicada sobre la inmunotira, mientras los ensayos ELISA incluyen múltiples pasos como preparación de buffers, tiempos prolongados de incubación y múltiples lavados, lo cual incrementa la posibilidad de error. También puede ocurrir un efecto de dilución de la muestra en el ensayo ELISA, afectando la detección en algunas muestras (Ruiz-García et al., 2009).
La robustez del diagnóstico de R. solanacearum con las inmunotiras en los tejidos de plátano, en el presente trabajo y otros (Thera, 2007), así como su rapidez, sencillez y portabilidad, hacen que su uso sea aconsejable para monitorear R. solanacearum en campo, y para la colecta de muestras para el aislamiento y estudio de la bacteria. La confirmación del diagnóstico deberá continuar siendo con los métodos moleculares ya que los límites de detección serológicos no permiten detectar casos de infección temprana (Thera, 2007), y tampoco distinguen entre las diferentes razas de R. solanacearum (Rajeshwari et al., 1998).
Aislamiento de R. solanacearum
Mediante el uso secuencial de 2 medios de cultivo, el SMSA y el B-King, se logró aislar 25 cepas de R. solanacearum de las 28 muestras positivas. Las tres muestras donde no se pudo recuperar R. solanacearum fueron las que presentaron las líneas diagnósticas más pálidas en las inmunotiras, lo que indica que esas muestras contenían una baja cantidad de bacteria.
El medio SMSA es frecuentemente utilizado para el aislamiento de R. solanacearum, porque evita el crecimiento de muchos otros microorganismos (Kelman, 1954; Álvarez-Restrepo et al., 2008; Cardozo et al., 2010). Sin embargo, aun así R. solanacearum es difícil de aislar debido a que in vitro presenta crecimiento lento, mientras otras bacterias, e.g. Pseudomonas, crecen rápido y contaminan los aislamientos (Ramesh et al., 2009; Champoiseau et al., 2009; Nasim, 2011; Kalpage y De Costa, 2015). La combinación del medio SMSA y el medio B-King permite distinguir R. solanacearum de Pseudomonas spp. Ambos tipos de bacterias crecen sobre el medio SMSA como colonias mucoides, de bordes blancos irregulares y centros rosas a rojos. Sobre el medio B-King las colonias de Pseudomonas spp. fluorescen mientras que las de R. solanacearum no lo hacen (Figura 2), lo que permite distinguirlas entre sí. Ramesh et al., (2009) señalan que las bacterias del género Pseudomonas son oportunistas durante la colonización del hospedero por R. solanacearum. Además de las Pseudomonas, las bacterias Klebsiella, Erwinia y otras especies del género Ralstonia, como Ralstonia mannitolilytica, son endófitas mayoritarias del plátano (Thomas et al., 2008; Ganen et al., 2009; Souza et al., 2013). Las bacterias recuperadas de las 3 muestras que dieron las líneas tenues en las inmunotiras presentaron fluorescencia sobre el medio B-King y no amplificaron en ninguna de las reacciones de PCR de diagnóstico para R. solanacearum. Algunas de esas bacterias fueron identificadas mediante secuenciación del 16S, y correspondieron a Pseudomonas fulva, Pseudomonas aeruginosa y Pseudomonas citronellolis, consistente con lo reportado en la bibliografía.
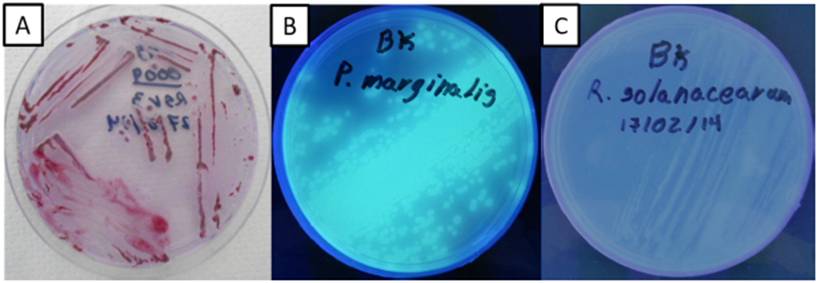
Figura 2 Pre-selección de bacterias con fenotipo similar a R. solanacearum. Aislamiento sobre medio SMSA (panel A). Las colonias con características mucoides, bordes blancos irregulares y centros rojos fueron transferidas sobre medio B-King. Las bacterias del género Pseudomonas fluorescen sobre este medio (Panel B). Se seleccionaron las bacterias no fluorescentes (panel C) para continuar el aislamiento de R. solanacearum.
Confirmación de la identidad R. solanacearum
La identidad de las cepas R. solanacearum fue confirmada mediante secuenciación del fragmento amplificado con los iniciadores pehA#3-F, pehA#6-R (fragmento del gen pga). Las 25 cepas que amplificaron la banda diagnóstica de 532pb fueron confirmadas como R. solanacearum mediante el análisis BlastN con las secuencias obtenidas, y comparando con la base de datos de GenBank (hit 99-100% de identidad con R. solanacearum). Con base en los resultados positivos de la PCR cuando se usaron los iniciadores ISRso19, las 25 cepas aisladas corresponden a la Raza 2.
La identificación de los secuevares se determinó primero mediante PCR Multiplex, usando los iniciadores de la serie Mus (Fegan y Prior, 2005). De acuerdo a estos autores, cada muestra debe de dar una sola banda diagnóstica: de ~167 pb (secuevar 4 ecotipo SFR), ~350 pb (secuevar 4), ~220 pb (secuevar 6), ~400 pb (secuevar 3). Estas bandas se visualizaron en los productos de la Multiplex (Figura 3A), pero no fueron bandas únicas por muestra. De acuerdo a la Multiplex (Figura 3A), las cepas de R. solanacearum Raza 2 genotipifican en el secuevar 3 (carriles 11 y 13), secuevar 4 (carriles 3 y 5), secuevar 4 SFR (carril 1) y secuevar 6 (carriles 2, 4, 6, 8). Otras cepas generaron perfiles que no pudieron asignarse a ninguno de los secuevares (e.g. carriles 7, 10). Debido a la inconsistencia en los resultados de la PCR en Multiplex, cada cepa fue posteriormente genotipificada mediante PCR independiente para cada secuevar. Ninguna cepa de Tabasco amplificó con las parejas de iniciadores para los secuevares 3, 4 y 4 SFR (datos nos mostrados). Las 25 cepas amplificaron la banda diagnóstica de ~220 pb con la pareja de iniciadores SI28, que corresponde al secuevar 6 (Figura 3B). El secuevar 6 se originó en las islas del Caribe (Trinidad) y llegó al continente en Venezuela; en 1950 se introdujo accidentalmente a Costa Rica en cormos de plátano infectados. En 1960 un embarque comercial lo introdujo accidentalmente en Honduras y de allí se dispersó rápidamente por el norte a México y por el sur a Panamá (Sequeira, 1998). En México el primer reporte fue en 1960 en Chiapas. Actualmente el secuevar 6 se ha reportado en Norte América en Hawai (Fegan y Prior, 2005) y Florida (Hong et al., 2012); en Centroamérica en Guatemala (Sánchez-Pérez et al., 2008) y en Sudamérica en Brasil (Alburquerque et al., 2014). El Centro y Sudamérica es considerado el centro de origen de las cepas relacionadas a Moko del plátano (Sequerira, 1998; Fegan y Prior, 2005, 2006). Otros países en América tienen, además del secuevar 6, alguno(s) de los secuevares 3, 4, 24, 25, 41 y 53 (Raymundo et al., 1998; Prior y Fegan. 2005; Alburquerque et al., 2014). El estudio en México debe ampliarse para determinar si existen otros secuevares relacionadas al Moko bacteriano del plátano.
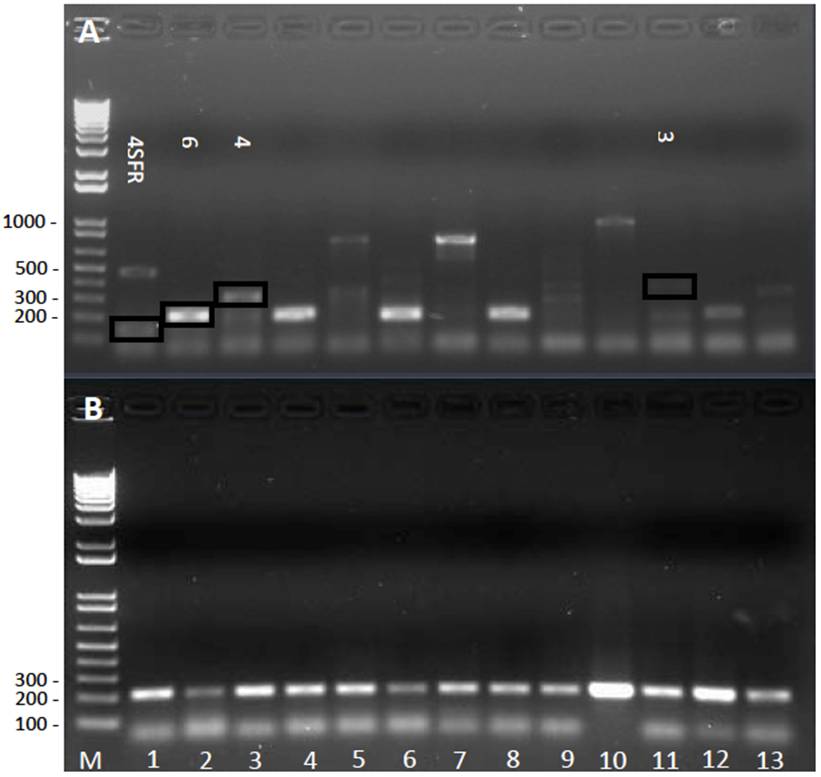
Figura 3 Caracterización de bacterias R. solanacearum aisladas a partir de plantas de plátano; tipificación de los secuevares. Panel A, PCR en Multiplex (Iniciadores de la serie Mus y SI28 juntos). El carril 1 presentó una banda de ~167 pb que corresponde a lo esperado para el secuevar 4 SFR y otra de ~450 pb no descrita para ningún secuevar; el carril 2 muestra una banda de aproximadamente ~220 pb que corresponde a lo descrito para el secuevar 6; en el carril 3 se observó una banda de ~350 pb, lo que corresponde al secuevar 4. En los carriles siguientes se observaron bandas inesperados, como por ejemplo ~850 pb (carriles 5 y 7) y ~1000pb (carril 10). Panel B, PCR con iniciadores específicos para el secuevar 6 (iniciadores SI28, banda diagnóstica de ~220 pb). En ambos paneles los carriles corresponden a: M) Marcador 1 Kb Plus DNA Ladder (Invitrogen® Carlsbad, CA, USA); carriles 1-13, cepas de R. solanacearum, 1) MT01, 2) MT02, 3) MT03, 4) MT04, 5) MT05, 6) MT06, 7) MT07, 8) MT08, 9) MT09, 10) MT10, 11) MT11, 12) MT12, 13) MT13.
La clasificación taxonómica de las cepas del complejo de especies de R. solanacearum (RSSC) ha estado en constante revisión y corrección desde hace 50 años y más frecuentemente en los últimos 10 (Peeters et al., 2013; Prior et al., 2016). A medida que las colecciones mundiales van aumentando y junto con ella la diversidad genética global conocida del RSSC, se han encontrado discrepancias en los resultados de clasificación de algunas cepas mediante diferentes herramientas de diagnóstico (Hong et al., 2012), e incluso en clasificación de poblaciones, como son los casos de las R. solanacaerun aisladas de Solanáceas y Cucurbitáceas en la Guayana Francesa y de tomate en Guadalupe (Álvarez-Restrepo et al., 2008; Deberdt et al., 2014). Aunque las razones de las inconsistencias no son del todo claras, algunas de las diferencias se han convertido en herramientas de diagnóstico. Por ejemplo, muchas cepas del secuevar 4 producen la banda de 351 pb con los iniciadores Mus06, pero adicionalmente amplifican una banda de 167pb (Álvarez-Restrepo et al., 2008), mientras que algunas cepas del secuevar 4 aisladas en Florida en 2008 no producen la banda diagnóstica de 351pb sino únicamente la banda inespecífica de 167pb, con lo que los autores proponen que las cepas de Florida que amplifican únicamente la banda de 167pb son cepas emergentes (Hong et al., 2012).
El Multiplex para identificar entre los secuevares 3, 4 y 6 dentro de la Raza 2 (i.e. cepas causantes de Moko en plátano) fue generado por Fegan y Prior (2005) y ha sido usado para clasificar cepas en Martinica (Wicker et al., 2007, 2009), Colombia (Álvarez-Restrepo et al., 2008), Estados Unidos (Hong et al., 2012), La Guayana francesa (Deberdt et al., 2014), Brasil (Albuquerque et al., 2014), entre otros. En los reportes que muestran los análisis electroforéticos de los productos de PCR se pueden observar en algunos carriles bandas inespecíficas adicionales a la banda diagnóstica (Álvarez-Restrepo et al., 2008; Hong et al., 2012; Albuquerque et al., 2014), como lo observado en el presente trabajo de México. Incluso el análisis de un mismo secuevar muestra discrepancias entre los diferentes grupos de investigación. El secuevar 4 amplifica 2 bandas en el Multiplex de Moko, pero los tamaños de las bandas son diferentes entre el reporte de Álvarez-Restrepo et al., (2008) y el de Albuquerque et al., (2014); a pesar de que en ambos trabajos se utilizaron geles de agarosa de la misma concentración (1.5% p/v).
En el presente trabajo el diagnóstico por Multiplex fue ambiguo. En el caso de las cepas que amplificaron la banda de 221pb (secuevar 6), esta fue una banda única (Figura 3A, carriles 2, 4, 6, 8,12), contrario al resultado de Albuquerque et al., (2014) que obtuvieron 2 bandas para la cepa IBSBF2661 (secuevar 6), pero similar a lo obtenido por Hong et al., (2012), quienes también obtuvieron una sola banda para la cepa 527 (secuevar 6) en la Multiplex de Moko. Con algunas cepas mexicanas se observó amplificación de la banda de diagnóstico esperada para los secuevares 3 (400pb, carril 11) y 4 (351pb, 167pb, carriles 1 y 3), pero otras bandas observadas también en esos mismos carriles no coinciden con ninguno de los trabajos reportados. También se observaron bandas de PCR que no se han reportado previamente en los resultados de Multiplex de cepas de Moko (Figura 3A, carriles 5, 7, 10) es decir, son bandas nuevas. La banda nueva de ~850 pb amplifica en diferentes cepas (Figura 3A, carriles 5 y 7), lo que sugiere que tienen una base de origen genético y refleja la diversidad en las poblaciones de R. solanacearum. Es difícil interpretar con más detalle estos resultados porque los iniciadores de diagnóstico amplifican bandas marcadoras, pero la función en el microorganismo de esas regiones genómicas es desconocida (Deberdt et al., 2014).
La presencia de productos de PCR nuevos y reproducibles en diferentes cepas (e.g. banda de ~850 pb en cepas MT05 y MT07), pudiera interpretarse como emergencia de nuevas cepas de R. solanacearum en Tabasco, como fue propuesto para las cepas de Florida que presentaron resultados atípicos en este diagnóstico Multiplex (Hong et al., 2012). Debido a la ambigüedad de resultados se realizó el diagnóstico individual de cada secuevar. No se obtuvo amplificación de ninguna cepa en los diagnósticos de los secuevares 3 y 4 (datos no mostrados). Todas las cepas aisladas en Tabasco en el presente trabajo pertenecen al secuevar 6 (Figura 3B). La recomendación que surge en el presente reporte es no basarse en resultados de Multiplex de Moko para proponer emergencia de nuevas cepas de R. solanacearum. Se sugiere que los productos amplificados sean secuenciados.
La Figura 4B presenta un resumen del protocolo propuesto a partir del presente trabajo, con el objetivo de simplificar el aislamiento de cepas de R. solanacearum Raza 2. Se compara con el procedimiento seguido en otros trabajos (Figura 4A). El procedimiento 4A es más costoso debido a que el diagnóstico por ELISA o inmunotiras debe realizarse sobre cada colonia aislada. En este trabajo esa estrategia tuvo poco éxito. Cuando no se diagnosticaron las muestras previamente al aislamiento, una gran parte de las bacterias aisladas sobre el SMSA no fueron R. solanacearum. El uso secuencial de medio B-King después del aislamiento sobre SMSA es una opción rápida y económica que permite la preselección fenotípica de R. solanacearum. De acuerdo a la literatura los diagnósticos de identidad más confiables de R. solanacearum son la PCR y secuenciación, ya sea de ITS o de genes específicos (Fegan y Prior, 2005). Una vez confirmada la identidad de R. solanacearum la colección de bacterias aisladas puede ser caracterizada genéticamente, fenotípicamente, filogenéticamente, o usarse para otros estudios. Se recomienda realizar PCR individual con los iniciadores específicos de cada secuevar de Moko, en vez del Multiplex de Moko, cada vez que los resultados no sean claros.
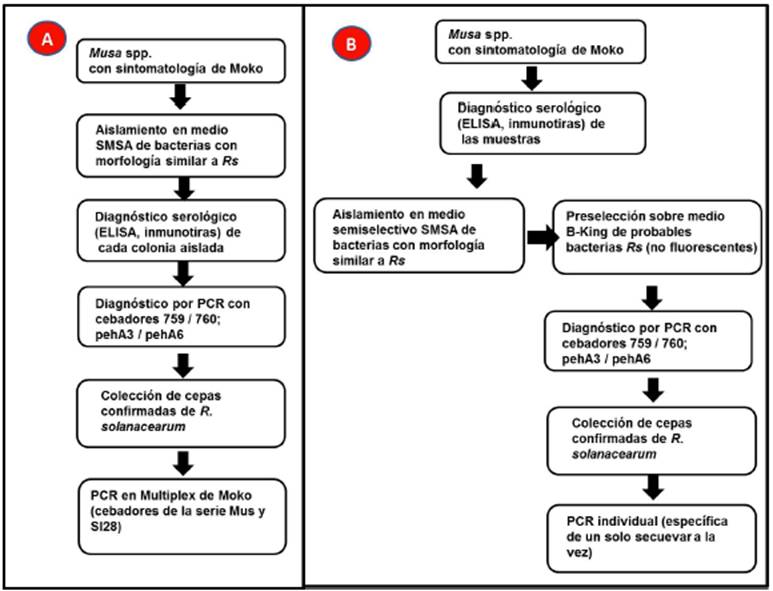
Figura 4 Comparación esquemática del protocolo generalmente usado para el aislamiento de R. solanacearum relacionadas a la enfermedad Moko (Panel A), y el protocolo recomendado en el presente estudio (panel B). El panel A se basa en Álvarez-Restrepo et al. (2008); Dööolotkeldieva y Bobusheva (2016); Hong et al. (2012); Sánchez-Pérez et al. (2008).
Moko es una enfermedad bajo regulaciones cuarentenarias en México y se tiene establecida la campaña fitosanitaria, y por lo tanto se mantiene bajo continua vigilancia y medidas de erradicación de los brotes identificados. Es importante enfatizar que por normatividad y por seguridad alimentaria, es indispensable prevenir la dispersión del patógeno por colecta del material por personal inexperto. Por ello, todos los estudios que se realicen sobre este patógeno en México deberán ser llevados a cabo con el permiso y en colaboración con la Dirección General de Sanidad Vegetal (DGSV), el Servicio Nacional de Sanidad, Inocuidad y Calidad Agroalimentaria (SENASICA) y los Comités Estatales de Sanidad Vegetal.
CONCLUSIONES
Se recomienda incluir el uso sistemático de inmunotiras para fortalecer el diagnóstico de R. solanacearum en campo.
El aislamiento e identificación de R. solanacearum es difícil, como han mencionado otros autores. La modificación del procedimiento citado en la literatura para el aislamiento de R. solanacearum facilitó aislar la bacteria a partir de plátanos enfermos: el diagnóstico serológico de las muestras sospechosas, la inclusión sistemática del medio B-King para descartar bacterias del género Pseudomonas, y el diagnóstico confirmativo por PCR con los iniciadores pehA#3-F -pehA#6-R, e ISRso19F-ISRso19R.
La detección de resultados atípicos en la PCR Multiplex para Moko debe interpretarse con cuidado. Los resultados deben confirmarse empleando PCRs individuales (específicas) para cada secuevar.











 texto en
texto en 


