INTRODUCCIÓN
El café (Coffea spp.) es el principal producto de agro-exportación del Perú, con un área cultivada de 361 671 ha y una producción de 222 047 t producidas principalmente en las regiones de Cajamarca, Amazonas, Junín, Cusco y San Martin (MINAGRI, 2015). El 95% del café peruano proviene de cultivares de Coffea arabica (JNC, 2014) que son muy susceptibles a la roya amarilla, enfermedad cuyo agente causal es el hongo biotrófico Hemileia vastatrix (Berkeley, 1869).
La roya amarilla del café fue reportada por primera vez en el Perú el año 1979, en el departamento de Junín (Scheiber y Zentmyer, 1984). Sin embargo, tuvo su mayor incidencia en el año 2013 ocasionando pérdidas que alcanzaron el 27% de la producción (INEI, 2014), afectando principalmente a los productores orgánicos. Desde entonces esta enfermedad se ha extendido a gran parte de las principales zonas cafetaleras del Perú, entre ellos a Villa Rica (Región Pasco) y a Quillabamba (Región Cusco).
La roya amarilla es considerada, a nivel mundial como la principal enfermedad del café, puede ocasionar pérdidas hasta del 40% en la producción (Rivillas et al., 2011). En tal sentido se han planteado dos enfoques para evitar las pérdidas ocasionadas por la enfermedad, la primera consiste en el uso de fungicidas, que es una alternativa muy costosa y potencialmente perjudicial para el ambiente, y la segunda a través, del desarrollo de variedades mejoradas. Sin embargo, esta última requiere del conocimiento de las fuentes de resistencia y de la diversidad del patógeno.
Los mecanismos que promueven el surgimiento de nuevas razas virulentas de H. vastatrix aún son desconocidos, y se ha planteado que la principal causa para el incremento de la diversidad del hongo son las mutaciones (Varzea y Marques, 2005); aunque, existe evidencia de un nuevo tipo de reproducción sexual oculto dentro de esporas asexuales (criptosexualidad) que podría explicar el origen de las nuevas variantes (Carvalho et al., 2011). Además, se sabe que la elevada tasa de generación de nuevas razas, que ha despertado la atención de los programas de mejoramiento genético, tiene un efecto sobre la forma de generación de nuevas variedades de café, porque el hongo va adquiriendo capacidad de infectar plantas que originalmente fueron resistentes (Alvarado y Moreno, 2005).
Debido a esta capacidad de rápida adaptación a nuevos cultivares de café se ha llegado a considerar a H. vastatrix como un patógeno de alto potencial evolutivo cuyas evidencias de variación vienen siendo estudiadas desde 1930 (Rodrigues et al., 1975), en los cuales se ha reportado la presencia de la raza II en casi todos los casos, por lo tanto, se ha sugerido que este podría ser el posible inóculo inicial a partir del cual se han generado naturalmente todas las nuevas razas del hongo encontradas (Zambolim et al., 2005). Estos estudios iniciales fueron realizados por medio de las lecturas de las expresiones fenotípicas de una serie de clones de cafetos diferenciales del Centro de Invesgação das Ferrugens do Cafeeiro (CIFC) en Portugal, identificando hasta antes de 2005 a 45 razas de H. vastatrix (Várzea y Marques, 2005). Sin embargo, estos conjuntos de cafetos diferenciales fueron insuficientes para la caracterización de nuevas razas virulentas del hongo, por lo que, se plantearon nuevas técnicas para determinar su variabilidad, como el uso de marcadores moleculares (Nandris et al., 1998) y la secuenciación de los ITS del ADN ribosomal (Cristancho et al., 2007). Si bien es cierto con estas técnicas moleculares no fue posible la identificación de razas, sin embargo, permitieron determinar la diversidad genética del patógeno. Además, las secuencias ITS han sido muy utilizadas en el análisis molecular de la diversidad de hongos fitopatógenos, así como en la caracterización de nuevas especies de hongos, y en la separación de especies muy cercanas o de razas de una misma especie (Grube y Kroken, 2000). Su aplicación a H. vastatrix viene ofreciendo nuevos panoramas sobre su diversidad que puede ser utilizada en programas de mejoramiento genético. En el Perú, donde pocos estudios fueron realizados con esta finalidad, la generación de este conocimiento es fundamental para el manejo del cultivo y en los programas de mejoramiento genético del café peruano. Por ello en el presente trabajo se analizó la diversidad genética de H. vastatrix de dos zonas productoras importantes de café en el Perú mediante secuenciación de la región ITS, las que fueron comparadas con las secuencias reportadas en el GenBank para Colombia.
MATERIALES Y MÉTODOS
Material biológico
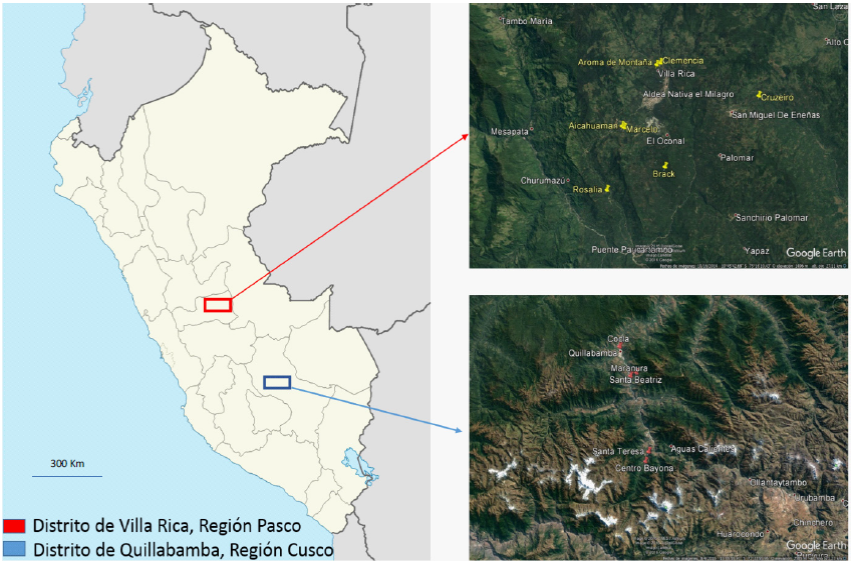
Esporas de Hemileia vastatrix fueron colectados en 5 predios de la zona cafetalera de Quillabamba, (Región Cusco) y en 7 predios de la zona cafetalera de Villa Rica (Región Pasco) (Cuadro 1). Estas zonas se encuentran distanciadas geográficamente y están consideradas dentro de las regiones cafetaleras con mayor producción de café en el Perú (Figura 1).
En cada predio se colectaron 25 hojas de cafetos de las variedades “Typica” y “Caturra” infectados con H. vastatrix, a las que se les extrajeron las esporas. Con la finalidad de obtener suficiente concentración de ADN, las muestras de uno o dos predios se agruparon en una muestra compuesta (Cuadro 1).
Cuadro 1 Información georreferenciada de los predios en los que se muestrearon las pústulas de un total de 25 hojas de Hemileia vastatrix de las dos zonas cafetaleras del Perú y se indican las claves de las muestras compuestas generadas.
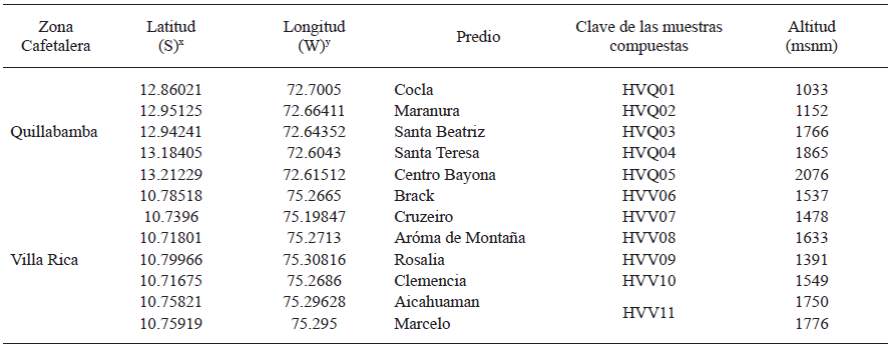
x S= Sur.
y W= Oeste.
Análisis de la diversidad genética de Quillabamba y Villa Rica
Extracción de ADN
El ADN se extrajo utilizando el protocolo descrito por Cristancho et al. (2007) con modificaciones: las uredosporas se depositaron en tubos de 1.5 ml, luego se adicionó 150 μL de buffer de lisis (50 mM de tris HCl pH 7,5; 50 mM de EDTA pH 8,0; 3% de SDS; 1% de b Mercaptoetanol), posteriormente se trituraron las uredosporas con ayuda de un pistilo de polipropileno durante 30 a 45 minutos. La calidad se determinó mediante electroforesis en geles de agarosa al 1% teñidos con GelRed (Biotium®). Finalmente se determinó la concentración del ADN mediante espectrofotometría.
PCR y Purificación
Se realizó la amplificación de la región ITS mediante PCR empleando los iniciadores universales ITS1L e ITS4R (White et al., 1990), como referencia se utilizó el protocolo descrito por Cristancho et al. (2007). La reacción de amplificación se realizó en un volumen de 10 μL conteniendo 2 mM de MgCl2, 0.2 mM de dNTPs, 0.4 μM de cada iniciador, Buffer de PCR 1X, 1.5 Unidades de Taq ADN polimerasa y 40 ng de ADN. El programa de amplificación consistió de una etapa inicial a 95 °C por 5 min; seguido de 30 ciclos de desnaturalización a 94 °C por 1.5 min, alineamiento a 60 °C por 1 min y extensión a 72 °C por 2 min; y un paso final de extensión a 72 °C por 5 min. Los productos de amplificación fueron separados mediante electroforesis en geles de agarosa al 1%, la purificación del fragmento de ADN de la Región ITS se realizó utilizando el QIAquick gel extracción Kit®.
Clonación y Secuenciación
Los productos de amplificación purificados fueron clonados en Escherichia coli quimio competente (JM109) utilizando el Pgem T-easy vector system II Kit® según el protocolo recomendado por el fabricante. Posteriormente, se seleccionaron 10 colonias blancas por cada muestra compuesta para ser sembradas cada una en 5 ml de medio LB con 60 μg/mL de ampicilina, e incubadas a 37 °C por 24 h. La extracción del plásmido de E. coli conteniendo el fragmento de ADN de interés, se realizó empleando el protocolo Wizard®Plus SV Minipreps DNA purification system de PROMEGA. La confirmación de la presencia de la secuencia del ADN de la región ITS se realizó mediante digestión con la enzima EcoRI. Los fragmentos fueron visualizados en gel de agarosa al 1.5%. La secuenciación de los fragmentos clonados fue realizada por la empresa GenBioteck (Argentina).
Análisis de datos
Las secuencias obtenidas fueron alineadas y editadas con el programa Codon Code Aligner v 2.0.6 (Codon Code Corporation). Como referencia se utilizaron las secuencias de ADN de la región ITS de H. vastatrix almacenadas en el GenBank (Cristancho et al., 2007).
Las secuencias obtenidas fueron alineadas utilizando el programa MAFFT implementado en la plataforma UGENE (Golosova, et al., 2014). La diversidad nucleotídica y la haplotípica (Nei y Li, 1979) se calcularon con el programa DNAsp v 5 (Librado y Rozas, 2009), y para la observación de las semejanzas entre los haplotipos se construyó una red utilizando el algoritmo median Joining (MJ) implementado en el programa PopArt (Bandelt et al., 1999) para el cual se excluyeron los sitios gap.
Adicionalmente se analizaron secuencias de las regiones ITS de Hemileia vastatrix almacenadas en el GenBank, que corresponden a H. vastatrix de Colombia, y de las razas II y XXII del CIFC (Cristancho et al., 2007).
RESULTADOS Y DISCUSIONES
Análisis de la diversidad genética de Quillabamba y Villa Rica
A partir de las muestras de H. vastatrix colectadas en los predios de Quillababa (5 predios) y de Villa Rica (7 predios) se obtuvieron 106 secuencias de ADN de la región ITS de Hemileia vastatrix. Las longitudes de las secuencias se encontraron entre el rango de 910 pb y 918 pb.
Del alineamiento de estas secuencias se obtuvieron 921 sitios alineados, de los cuales 21 resultaron ser gaps (inserciones o deleciones). Asimismo, se encontró 101 sitios polimórficos, con 11 posiciones parsimoniosas informativas (Cuadro 2).
Cuadro 2 Valores de los parámetros obtenidos sobre las medidas descriptivas de la variabilidad de Hemileia vastatrix de las secuencias de la región ITS asociadas a muestras de Quillabamba, Villa Rica, Colombia y CIFC.
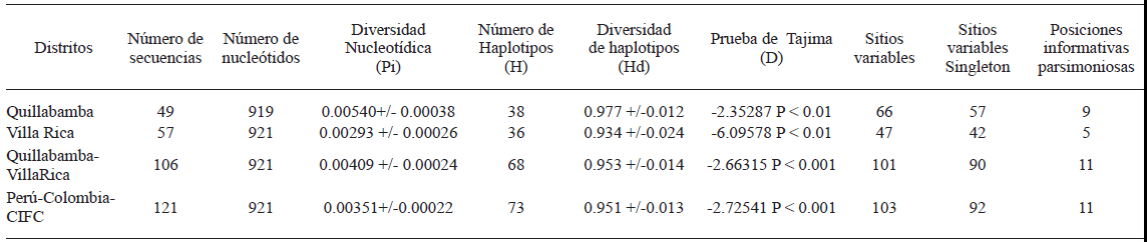
CIFC: Centro de Invesgação das Ferrugens do Cafeeiro.
Entre las dos zonas cafetaleras se halló un total de 68 haplotipos con una diversidad haplotípica (Hd = 0.953 +/- 0.014) y nucleotídica (Pi = de 0.00409 +/- 0.00024); con 62 haplotipos con secuencias únicas y 6 haplotipos conformados por más de dos secuencias (Cuadro 3). Los haplotipos Hap_4, Hap_8, Hap_36 y Hap_21, que se encontraron en mayor frecuencia, se podrían considerar como los ancestrales (Figura 2); además, éstos se encuentran distribuidos en ambas zonas cafetaleras, a donde debieron haber llegado en tiempos diferentes adaptándose a las condiciones ambientales de cada zona. Los haplotipos Hap_4 y Hap_36 son más frecuentes en Villa Rica; mientras que, Hap_8 y Hap_21 son más frecuentes en Quillabamba. Esta distribución de los ancestrales en las dos zonas obedece a la teoría genética de coalescencia observada también por Santana et al. (2007) en poblaciones de este hongo en Brasil.
Cuadro 3 Distribución regional de frecuencias de haplotipos de H. vastatrix generados a partir de las secuencias ITS.
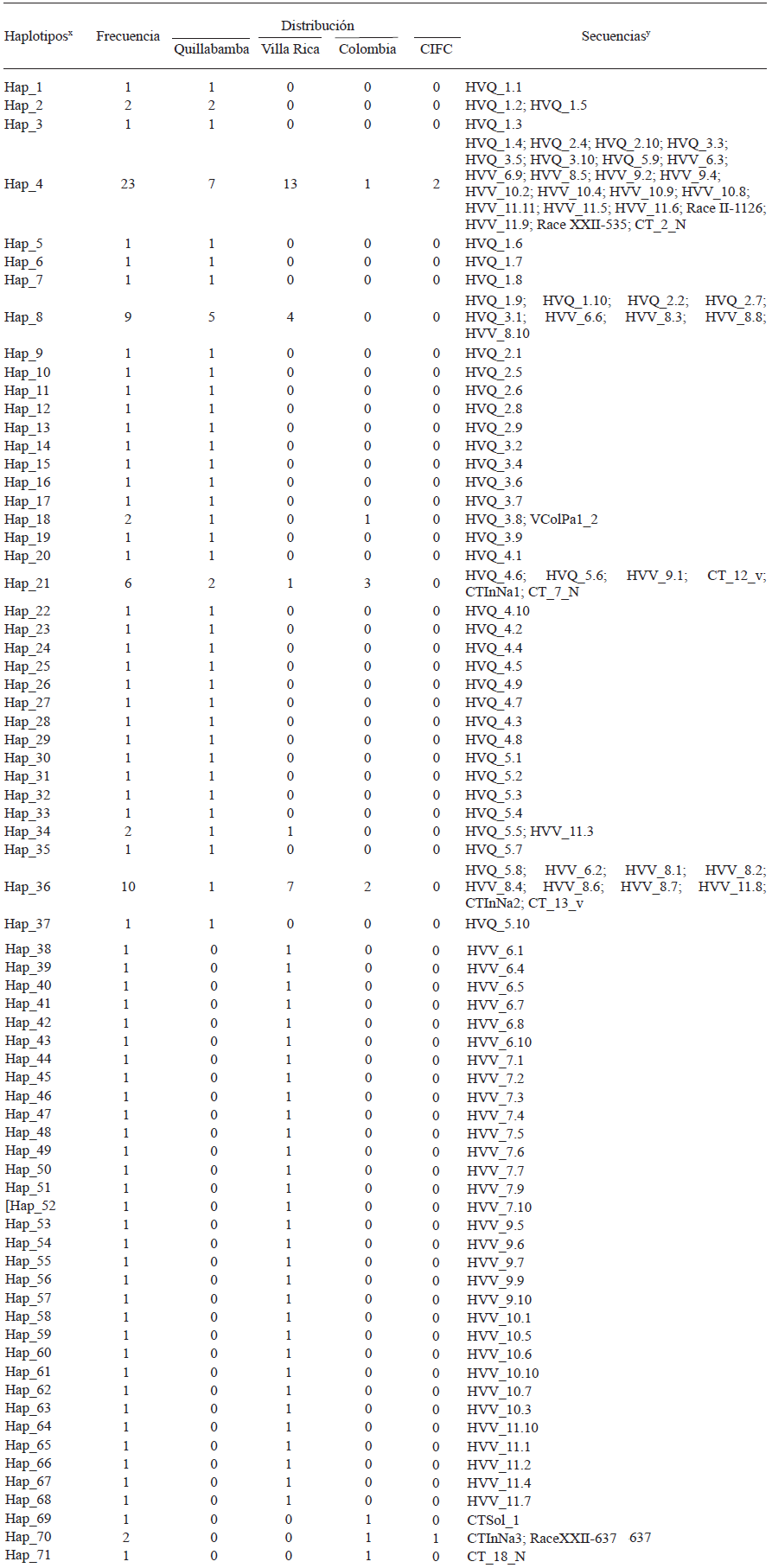
x Hap: Haplotipo
y HVQ: Hemileia vastatrix de Quillabamba; HVV: Hemileia vastatrix de Villa Rica; CT: Muestras de roya amarilla de Colombia; Race XXII y Race II: Muestras que corresponden al CIFC
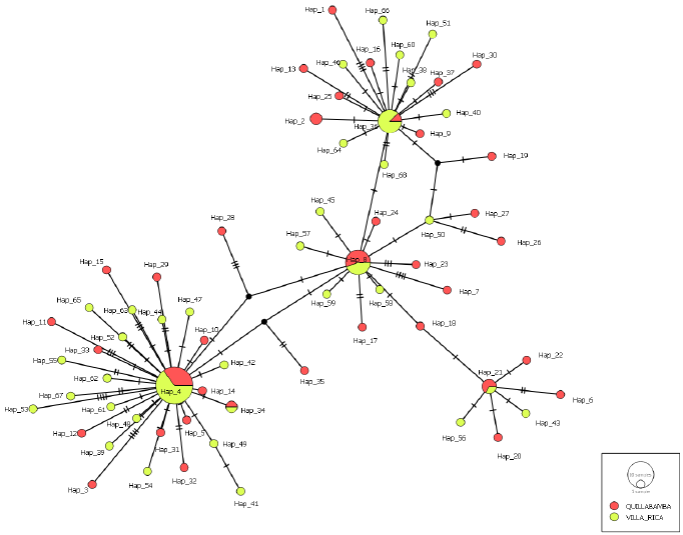
Figura 2 Red haplotípica con el método de unión por la mediana (Median-Joinig) de 68 haplotipos detectados para la región ITS de Hemileia vastatrix de las zonas cafetaleras de Quillabamba y Villa Rica. Los círculos representan un único haplotipo, el tamaño de los círculos es proporcional al número de secuencias que posee un haplotipo, eventos de mutaciones simples están indicados por líneas transversales y los haplotipos perdidos o no muestreados están representados por nodos de color negro.
Entre las dos zonas cafetaleras se halló un total de 68 haplotipos con una diversidad haplotípica (Hd = 0.953 +/- 0.014) y nucleotídica (Pi = de 0.00409 +/- 0.00024); con 62 haplotipos con secuencias únicas y 6 haplotipos conformados por más de dos secuencias (Cuadro 3). Los haplotipos Hap_4, Hap_8, Hap_36 y Hap_21, que se encontraron en mayor frecuencia, se podrían considerar como los ancestrales (Figura 2); además, éstos se encuentran distribuidos en ambas zonas cafetaleras, a donde debieron haber llegado en tiempos diferentes adaptándose a las condiciones ambientales de cada zona. Los haplotipos Hap_4 y Hap_36 son más frecuentes en Villa Rica; mientras que, Hap_8 y Hap_21 son más frecuentes en Quillabamba. Esta distribución de los ancestrales en las dos zonas obedece a la teoría genética de coalescencia observada también por Santana et al. (2007) en poblaciones de este hongo en Brasil.
Del análisis de las poblaciones de H. vastatrix de Quillabamba y Villa Rica, se ha determinado que la mayor diversidad genética se encuentra en la zona cafetalera de Quillabamba, porque presenta los valores de diversidad nucleotídica y haplotípica más altos (Cuadro 2).
Al no observarse en la red haplotípica de la Figura 2, nodos exclusivos por región geográfica, se estaría evidenciando una población de H. vastatrix grande e indiferenciada, que pudo haberse generado por el flujo de las esporas entre ambas zonas, favorecido por el movimiento de personas, semillas y/o plantas infectadas. Estos resultados son similares a los obtenidos por Cabral et al. (2016) en Brasil, quienes no observan una estructuración de la población de H. vastatrix respecto a su origen, hospedero ni razas fisiológicas; sin embargo, la diversidad genética fue relativamente menor a la hallada en el presente trabajo. No obstante, Maia et al. (2013) también en Brasil, obtuvieron resultados similares al presente trabajo, a través del estudio de 91 aislados del hongo, proponiendo que H. vastatrix se comporta como una gran población indiferenciada con una alta diversidad genotípica, y de igual forma, sin estructuración con respecto a su origen geográfico y de hospedero. Por lo tanto, la falta de estructuración de las dos poblaciones de H. vastarix por regiones podría explicarse por la dispersión del hongo a grandes distancias.
La prueba de neutralidad de Tajima se realizó para las poblaciones de H. vastatrix de Villa Rica (D=-2.50181; P<0.001), Quillabamba (D=-2.35287; P<0.001), así como, de ambas zonas juntas (D=-2.66315; P<0.001). Estos valores menores a 0 indican que las poblaciones del hongo se encuentran en proceso de expansión. Además, éstas se habrían originado a partir de pocos genotipos que fueron introducidos recientemente, o serían los descendientes de la raza II, la cual, según Scheiber y Zentmyer (1984), existe desde el año 1979 en el Perú. También, estos valores negativos de D de Tajima explicarían la presencia en la red haplotípica de un elevado número de haplotipos únicos que vienen incrementando la variabilidad, y que es el resultado de un crecimiento acelerado de la población. Por lo tanto, se refleja el estado de la población de H. vastatrix y se estaría demostrando que el origen de la epidemia de roya amarilla del café en el Perú que se inició en el año 2013 fue a partir de pocos individuos. Sin embargo, no se ha podido establecer un efecto de la altitud sobre el nivel del mar en la población, puesto que el valor negativo de la prueba de Tajima indicaría que aún no es evidente la existencia de un proceso selección.
Los mecanismos que conducen al incremento de la variabilidad y la creación de nuevas razas de H. vastatrix no se conocen. Por lo tanto, se podría considerar a las mutaciones espontáneas como el principal mecanismo responsable de la creación de variabilidad en el hongo, principalmente en las épocas de mayor incidencia de la enfermedad. Además, se debe sumar la presión de selección ejercida por el medio ambiente; al respecto, en Brasil, Varzea y Marques (2005) consideran que la presencia de genes de resistencia de algunas variedades en el hospedero, incrementan el número de mutantes virulentos en la población. Otro factor que podría explicar el surgimiento frecuente y rápido de nuevas variantes es la criptosexualidad (Carvalho et al., 2011), la cual podría estar jugando un papel importante en la generación de variabilidad genética de H. vastatrix.
Comparación de haplotipos de H. vastatrix del Perú con los de Colombia
Al análisis de las secuencias ITS de las poblaciones de H. vastatrix peruanas se incluyeron 15 secuencias de ADN de la región ITS del hongo que se encuentran almacenadas en el GenBank, de las cuales 12 corresponden a H. vastatrix colombianas y 3 a las del CIFC, estas últimas son haplotipos de la raza II y XXII (Figura 3). En dicho análisis se hallaron un total de 73 haplotipos, de los cuales 65 fueron únicos (no presentaron repeticiones). Además, se evidenció que el haplotipo Hap_4 contiene a la raza II y XXII; se encuentra en mayor frecuencia y está distribuida en todas las zonas estudiadas (7 secuencias de Quillabamba, 13 de Villa Rica, 1 de Colombia y 2 de CIFC). Por lo tanto, a partir de este haplotipo ancestral, se podría suponer, que surgieron los demás, corroborando lo planteado por Zambolim et al. (2005) en Brasil, quien sugiere que a partir de la raza II surgieron las demás razas; y es la predominante en América Latina (Cristancho et al., 2007). Así mismo, se observa que los haplotipos Hap_36 y Hap_21 presentes en Colombia, Villa Rica y Quillabamba, son nodos que aparecieron en momentos posteriores. Además, el haplotipo Hap_8 se encuentra presente solamente en Quillabamba y Villa Rica, el cual luego de ingresar o surgir en el Perú se pudo adaptar mejor a las condiciones ambientales de las zonas cafetaleras peruanas. De acuerdo a lo observado en los principales nodos, en los cuales es reiterativa la presencia de secuencias de H. vastatrix colombianas y peruanas, se podría sugerir que en el Perú y Colombia la enfermedad pudo provenir de la misma zona llegando con cuatro años de diferencia (Scheiber y Zentmyer, 1984; Buriticá, 2010). Además, en la red no se observan nodos por origen de procedencia, lo que estaría indicando que las secuencias de H. vastatrix colombianas son similares a las secuencias de las peruanas. Existen reportes de estudios que se realizaron a inicios de 1970, que le atribuyen al viento un rol muy importante en la diseminación de la enfermedad. Martines et al. (1975) comprobaron la presencia de uredosporas a más de 1000 m de altura con una buena viabilidad de germinación, asimismo la dispersión por el viento fue sugerida como la causa de introducción y rápida diseminación del hongo a Sudamérica (Bowden et al., 1971); también se mencionan otros medios de dispersión de la enfermedad, por ejemplo la introducción de la enfermedad a Brasil a través de plantas procedentes de África, por personas (trabajadores migrantes) y por semillas (Schieber, 1972), por lo tanto, la dispersión antropogénica pudo tener un rol muy importante en la llegada del hongo al Perú.
Basados en el análisis de poblaciones del hongo de las regiones estudiadas, se podrían generar tres hipótesis acerca del origen de la epidemia de roya amarilla del café surgida en el año 2013 en el Perú. La primera postularía que existió la introducción de una nueva raza de H. vastatrix al Perú, la cual provino de la Raza II, y debió adaptarse rápidamente a las condiciones ambientales de las zonas cafetaleras, que se encuentran en altitudes superiores a los 1000 msnm. Además, por la mayor frecuencia observada de los principales haplotipos ancestrales en Villa Rica debió llegar antes a esta zona que a Quillabamba; sin embargo, en la red haplotípica existen más de un nodo principal considerados como ancestrales (Hap_4, Hap_8, Hap_21 y Hap_36). Estos cuatro haplotipos debieron llegar de una manera progresiva y en un corto periodo de tiempo al Perú. Así mismo, el haplotipo Hap_4 que corresponde a la raza II, está en mayor frecuencia en ambas zonas cafetaleras e incluyen en su conformación a la secuencia CT_2N colombiana.
La segunda hipótesis postularía que hubo una evolución de la población de H. vastatrix a partir de la raza II, generándose una variante capaz de infectar a los cafetos de altura; sin embargo, de acuerdo a lo observado en la red haplotípica (Figura 3), debieron formarse de manera casi simultánea al menos 4 variantes las que corresponden a los haplotipos ancestrales; no obstante, en dos de ellos (Hap_4 y Hap_36) se encuentran secuencias tanto de Colombia como de Perú, las cuales debieron generarse simultáneamente en ambos países o de lo contrario pudieron haber sido introducidos desde el Perú, lo cual es una posibilidad poco probable considerando que existen controles sobre el movimiento de todo material vegetal (Cristancho et al., 2007).
La tercera hipótesis, apunta a los cambios en las condiciones climáticas que habrían favorecido la reproducción del hongo y su expansión hacia las zonas por encima de los 1000 msnm. Esta hipótesis se sustenta con lo observado en la red haplotípica (Figura 3), en la cual, se han formado 4 nodos correspondientes a los haplotipos ancestrales (Hap_4, Hap_8, Hap_21 y Hap_36), posiblemente existentes desde hace años atrás, porque se encuentran en ambas zonas cafetaleras peruanas y están en proceso de expansión. Además, el haplotipo de mayor frecuencia es Hap_4 que corresponde a la raza II, la cual existe desde el año 1979 en el Perú. Esta hipótesis, es similar a la que propusieron, Cristancho et al. (2012) en Colombia para el origen de la epidemia de roya del café ocurrido en el año 2008 y que afectó los cafetos colombianos sobre los 1400 msnm.
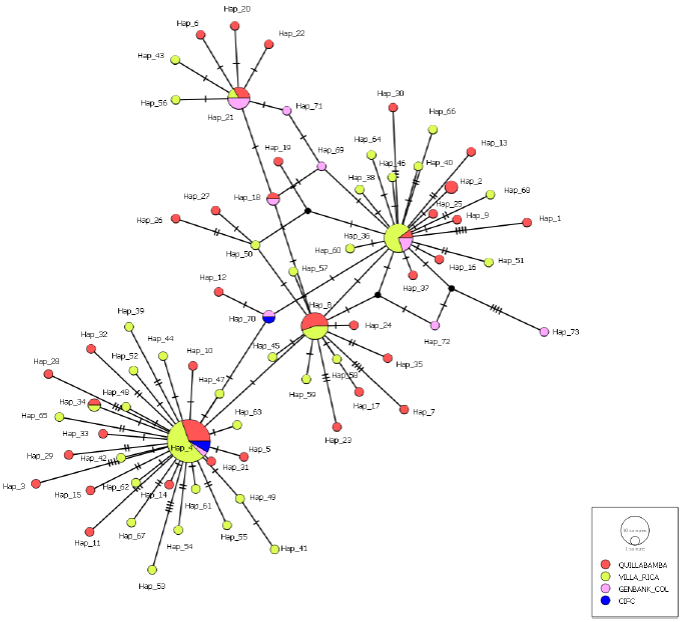
Figura 3 Red haplotípica por el método de unión por la mediana (Median-Joinig) de 73 haplotipos detectados para la región ITS de Hemileia vastatrix de Quillabamba, Villa Rica, Colombia y del CIFC. Los círculos representan un único haplotipo, el tamaño de los círculos es proporcional al número de secuencias que posee un haplotipo, eventos de mutaciones simples están indicados por líneas transversales y los haplotipos no muestreados están representados por nodos de color negro.
CONCLUSIONES
Mediante el análisis de la diversidad genética de Hemileia vastatrix a través de la secuenciación de la región ITS, se determinó que los valores de diversidad haplotípica para las zonas cafetaleras de Quillabamba y Villa Rica del Perú fueron altos (0.934 +/- 0.024 y 0.977 +/- 0.012 respectivamente); no obstante, la diversidad nucleotídica fue baja (0.00293 +/- 0.00026 y 0.00540 +/- 0.00038, respectivamente). Sin embargo, la mayor diversidad de H. vastatrix se encuentra en la zona cafetalera de Quillabamba. Entre las dos zonas evaluadas se ha encontrado una población grande no diferenciada por zona de procedencia, y sus haplotipos son similares a los de Colombia. Las secuencias de las razas II y XXII almacenadas en el GenBank que corresponden al haplotipo Hap_4, se encuentran tanto en Quillabamba como en Villa Rica. Además, se proponen tres hipótesis para explicar el incremento de la intensidad de la roya en el Perú.











 texto en
texto en