INTRODUCCIÓN
México produce aproximadamente 764,514 t de papaya (Carica papaya L.) al año (FAO, 2014). Debido a su volumen de producción y generación de recursos, es importante en mercado nacional e internacional; sin embargo, los frutos de papaya muestran susceptibilidad a distintos microorganismos entre los que destacan los hongos, los cuales ocasionan pérdidas entre 10 al 50% debido a daños en la calidad (Morton, 1987; Suárez-Quiroz et al., 2013). Dentro de los hongos mucorales reportados como fitopatógenos importantes se encuentran Gilbertella persicaria, Mucor spp.y Rhizopus spp. (Hyde et al., 2014), incluidos en las familias Choanephoraceae, Mucoraceae y Rhizopodaceae, respectivamente, los cuales ocasionan pudrición blanda de frutas como pera, manzana y durazno, y hortalizas como el tomate, son de rápida propagación y se han reportado ampliamente en zonas con climas tropicales y subtropicales (Michailides y Spotts, 1990).
Los mucorales son principalmente hongos saprofíticos que habitan suelo y en plantas en descomposición. Mucor spp. se consideran un grupo polifilético, por lo que es necesario realizar estudios en múltiples regiones conservadas del genoma para su correcta identificación (Hyde et al., 2014; Walther et al., 2013). Gilbertella persicaria es la única especie de su género (Benny, 1991; Walther et al., 2013; Hoffmann et al., 2013) y muestra un elevado grado de polimorfismo a nivel de morfología colonial y tasa de desarrollo micelial (Papp et al., 2001). Dentro de Rhizopus, destacan las especies R. oryzae (syn. R. arrhizus) y R. stolonifer debido al gran número de casos reportados (Hyde et al., 2014).
En México, los hongos mucorales ocasionan pudrición blanda de frutos de papaya en precosecha y poscosecha por lo que afectan el rendimiento y la calidad de los frutos en porcentajes aun no estimados. Recientemente, G. persicaria se reportó afectando frutos en el estado de Colima (Cruz-Lachica et al., 2016); sin embargo, los estudios de identificación de estas especies son escasos y están basados en características morfológicas del microorganismo y en síntomas de la enfermedad (Suárez-Quiroz et al., 2013), lo cual en estas especies es altamente susceptible a errores (Ginting et al., 1996). El objetivo de este estudio fue identificar las especies de hongos mucorales causantes de pudrición blanda en frutos de papaya mediante una identificación que incluyó caracterización morfológica y molecular.
MATERIALES Y MÉTODOS
Recolección de muestras
La recolección de frutos con síntomas de pudrición blanda (Figura 1A) se realizó con base en su incidencia en huertos de papaya cv. Maradol, durante diferentes muestreos en 2014: dos en Tecomán, Colima durante mayo y agosto, en el huerto El Trébol (18° 53’ 04.9” N, 103° 56’ 33.7” O); uno realizado en octubre en Oaxaca en la localidad de San José Río Verde Jamiltepec (16° 08’ 12’’ N, 97° 45’ 11’’ O), y dos más realizados durante octubre en Veracruz, en el huerto El Diamante en Cotaxtla (18° 52’ 25’’ N, 96° 12’ 2’’ O) y en Tlalixcoyan (21° 50’ 3’’ N, 96° 10’ 35’’ O).

Figura 1 Patogenicidad de cepas de mucorales en frutos de papaya Maradol. (A) Fruto con signos y sintomas en campo (B y G) Fruto control asperjado con agua destilada estéril sin síntomas, (C) Fruto con esporangios color café oscuro de Gilbertella persicaria morfotipo 1, (D) Fruto con esporangios de color gris a negro de Gilbertella persicaria morfotipo 2, (E) Fruto con esporangios café claro de Mucor irregularis, (F) Fruto con esporangios gris a negro de Rhizopus oryzae y, (H) Corte longitudinal de un fruto con visible pudrición blanda interna y desprendimiento de tejidos por acción enzimática.
Aislamiento, purificación y preservación de hongos mucorales
De cada fruto con síntomas, se dividió el área de las lesiones en cuadrantes y se tomó un trozo del borde de aproximadamente 0.5 cm de tejido por sección y se lavaron con alcohol etílico al 70% durante 1 min, con un lavado posterior con agua destilada; después, se sembraron en placas con PDA (Agar Papa Dextrosa, Bioxon) y se incubaron a 27 °C. La purificación se realizó por transferencia de puntas de hifas a nuevas placas de PDA y posteriormente se obtuvieron cultivos monospóricos (Chukwuka et al., 2010; Suárez-Quiroz et al., 2013). Los aislados se preservaron en tubos de vidrio con agua destilada estéril, para lo cual, se colocaron 20 discos de 6 mm de micelio en crecimiento activo en medio PDA de cultivos de 5 días de desarrollo, se selló la tapa con parafilm y se colocaron a 4 °C (Ryan et al., 2012).
Prueba de patogenicidad
Los postulados de Koch para los aislados seleccionados se efectuaron en frutos sanos de papaya cv. Maradol en estado de madurez comercial; inicialmente, los frutos se desinfestaron con alcohol etílico al 70% y después se lavaron con agua destilada. Cada aislado se inoculó en 3 frutos, seleccionado tres sitios de inoculación por cada uno, en donde se realizaron heridas con una aguja estéril y se inocularon por aspersión con una suspensión de esporangiosporas a una concentración de 5 x 105 esporangiosporas/mL; adicionalmente, se incluyeron 3 frutos sanos sin heridas y asperjados con la misma concentración de esporangiosporas y 3 frutos control, los cuales fueron heridos y se asperjaron sólo con agua destilada estéril. Todos los frutos se colocaron dentro de bolsas de polietileno (tres frutos por bolsa), con toallas absorbentes humedecidas con agua destilada estéril para generar aproximadamente 80% de humedad relativa y se incubaron a 25 °C durante 5 días. Finalmente, el hongo se reaisló de las lesiones y las características morfológicas se confirmaron con el hongo inoculado al inicio del experimento (Beales, 2012).
Caracterización morfológica
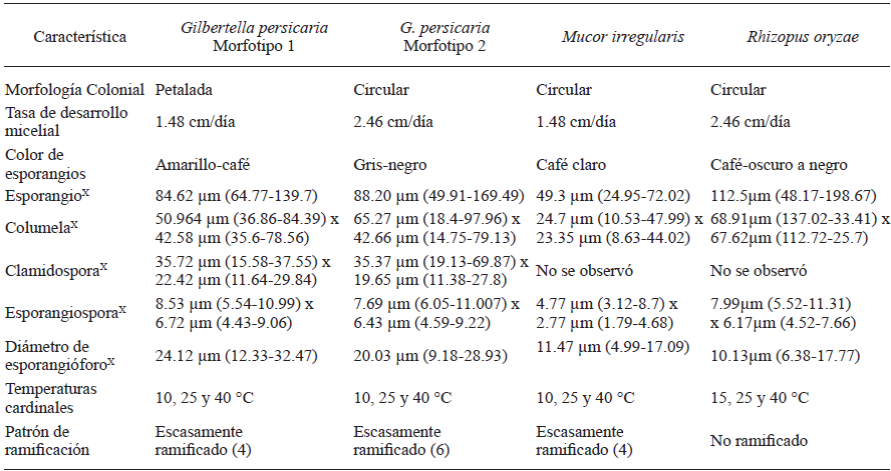
Se registraron las características culturales macroscópicas y microscópicas de los aislados crecidos en PDA; para lo cual se realizaron preparaciones teñidas con azul de lactofenol-algodón y se observaron en un microscopio óptico (Carl Zeiss Imager A2, Alemania). Los apéndices de esporangiosporas y espinas de esporangios se observaron en un microscopio de contraste de interferencia diferencial (Leica DMI 6000 B, Alemania) acoplado con una cámara (Leica DFC450C). Se registró la medida de 100 estructuras morfológicas, las cuales incluyeron: diámetro y patrón de ramificación de esporangióforo, tamaño y forma de esporangios y esporangiosporas, columela y clamidosporas, tamaño y presencia/ausencia de apéndices, así como el tipo de micelio (Campbell et al., 2013).
Tasa de desarrollo micelial
En el centro de cajas petri de 90 mm de diámetro con medio PDA, se colocó un disco de 6 mm proveniente del margen de colonias fúngicas de los aislados crecidos durante 3 días en PDA, y se incubaron a 4, 10, 15, 25 y 40 °C. El diámetro de la colonia se registró diariamente hasta que cubrió la totalidad de la placa, posteriormente, se efectúo la relación del diámetro con el número de días de la prueba (Michailides, 1991).
Caracterización molecular
Extracción de ADN, amplificación por PCR y secuenciación. El micelio de cada aislado se produjo en medio PDA durante dos días a 27 °C; posteriormente, se colectó con un portaobjetos estéril y el ADN genómico se extrajo de acuerdo al método de bromuro de cetiltrimetilamonio (CTAB) siguiendo el protocolo descrito por Voigt et al. (1999) con ligeras modificaciones: brevemente, se colocó 30 mg de micelio en tubos de microcentrífuga de 1.5 mL y se agregó 700 μL de solución amortiguadora CTAB [100 mM Tris-Cl (pH 8.0), 1.4 M NaCl, 25 mM de EDTA, 2% CTAB], se maceró y se colocó en vórtex por 10 s; posteriormente, se agregó 700 μL de cloroformo-alcohol isoamílico (24:1) (Sigma-Aldrich) por tubo, se sometió a vórtex por 10 s y se centrifugó a 12,300 x g durante 10 min. Se recuperó una porción de 500 μL de la fase superior y se colocó en un nuevo tubo; se agregó una cantidad igual de isopropanol (Sigma-Aldrich) a -20 °C, y se mezcló suavemente; posteriormente, se centrifugó de nuevo a 12,300 x g durante 2 min y se descartó el sobrenadante. La pastilla de ADN se lavó con 500 μL de etanol al 70%, se centrifugó a 12,300 x g durante 2 min. Se descartó el etanol y se permitió su evaporación por completo. Finalmente, la pastilla se resuspendió en 200 μL de solución amortiguadora Tris EDTA [10 mM Tris-Cl (pH 8.0), 1 mM EDTA (pH 8.0)].
La amplificación se realizó de acuerdo con Walther et al. (2013), en el cual el ADN ribosomal, incluyendo la región completa ITS1-5.8S-ITS2 y la región D1/D2 de la subunidad 28S (LSU), se amplificaron con el par de oligonucleótidos V9G (5’ TTACGTCCCTGCCCTTTGTA3’) (de Hoog y van den Ende, 1998) y LR3 (5’-GGTCCGTGTTTCAAGAC3’) (Vilgalys y Hester, 1990). La mezcla de reacción de PCR (25 μl), incluyó 20 ng de ADN, 0.4 μM de cada oligonucleótido, 0.185 mM de cada deoxinucleótido trifosfato (dATP, dTTP, dGTP, dCTP), 5X amortiguador de reacción, 1.5 mM de MgCl2 y 0.8 U de la enzima Taq ADN polimerasa utilizando el kit GoTaq® PCR Core Systems (Promega, USA). La reacción de amplificación se realizó en un termociclador marca BioRad T100™ (Singapur), con los siguientes ciclos: un paso de desnaturalización inicial de 5 min a 94 °C, seguido por 35 ciclos de 1 mina 94 °C, 1 min a 53 °C y 2 min a 72 °C, con una extensión final de 7 min a 72 °C. Para visualizar las bandas de los productos de ADN amplificados, los productos se corrieron en gel de agarosa al 0.8%, teñido con bromuro de etidio. La estimación de los pesos moleculares de los productos amplificados se realizó por comparación con un marcador molecular de 1 Kb (Promega, USA); posteriormente, la visualización de los productos amplificados se realizó en un fotodocumentador Molecular Imager Gel® Doc™ XR+ (BioRad, USA).
La purificación de los productos de PCR se realizó con el kit Wizard® SV Gel and PCR Clean-Up System (Promega, USA) siguiendo las indicaciones del fabricante. La secuenciación se llevó a cabo en el Laboratorio Nacional de Genómica para la Biodiversidad, CINVESTAV unidad Irapuato, utilizando para la región ITS, los oligonucleótidos ITS1 (5’-TCCGTAGGTGAACCTGCGG-3’) e ITS4 (5’TCCTCCGCTTATTGATATGC-3’)(White et al.,1990), y para la región D1/D2 28S (LSU) los oligonucleótidos NL1 (5′-GCATATCAATAAGCGGAGGAAAAG3’) (O’Donnell, 1993) y LR3 (Vilgalys y Hester, 1990), el proceso de secuenciación se realizó para ambos sentidos del amplicón.
Análisis filogenéticos de las secuencias
La edición de las secuencias se realizó con el programa Bioedit Sequence Alignment Editor, versión 7.2.5. (Hall, 1999). El alineamiento se llevó a cabo con el programa ClustalW y las secuencias consenso obtenidas se compararon con la herramienta básica de búsqueda de alineamientos locales BLASTN del NCBI (National Center for Biotechnology Information). Para realizar los análisis evolutivos, las secuencias obtenidas fueron guardadas en formato FASTA y el alineamiento se realizó con el programa ClustalW incluido en el software MEGA 6.0 (Tamura et al., 2013). La historia evolutiva fue inferida usando el método de máxima verosimilitud (Maximum Likelihood Method), basado en el modelo Tamura-Nei (Tamura y Nei, 1993). La representación gráfica del dendrograma con base en la región ITS y 28S (LSU) se realizó por la aplicación del método Neigborh-Joininig, con un análisis bootstrap de 1000 repeticiones para determinar los valores de confianza para los clados (Felsenstein, 1985), utilizando cepas de referencia disponibles en GenBank del NCBI.
RESULTADOS
De los 4 trozos sembrados por fruto enfermo, un 100% correspondió al hongo reportado para cada caso. Se obtuvieron 28 aislados asociados a pudrición blanda en frutos de papaya, 4 de Colima, 23 de Veracruz y 1 de Oaxaca. Todos los aislados se caracterizaron morfológicamente y se depositaron en el cepario de hongos del laboratorio de Fitopatología de CIAD. Los aislados de Colima, el de Oaxaca y 5 representativos de Veracruz se seleccionaron para evaluar patogenicidad y caracterizarlos molecularmente (Cuadro 1).
Cuadro 1 Especies de hongos mucorales causantes de pudrición blanda en frutos de papaya. Cada cepa (clave de cepario HP “Hongo Papaya”) corresponde al hongo obtenido de los 4 trozos de tejido sembrados en PDA por fruto enfermo.
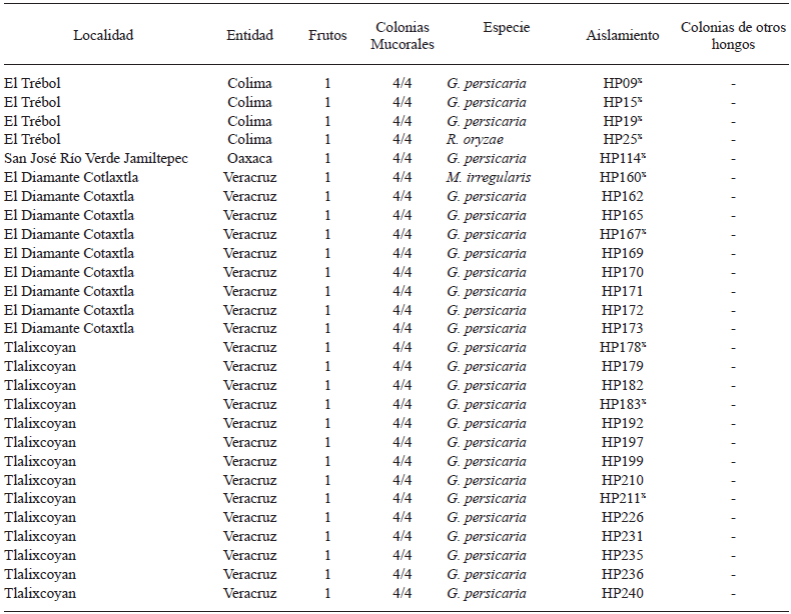
x aislados representativos utilizados en la caracterización molecular
Pruebas de patogenicidad
Todos los frutos de papaya cv. Maradol inoculados a través de heridas con las especies de mucorales presentaron, a las 24 h después de la inoculación, el desarrollo de síntomas que incluían lesiones de apariencia húmeda de aproximadamente 1 cm de diámetro; posteriormente, se desarrolló micelio de color blanco a las 48h, y a las 72h se produjeron esporangios; finalmente, a los 5 d después de inoculación, se observó el desarrollo de abundante micelio y esporangios de color café claro a oscuro (Figura 1E y 1C) y de gris a negro (Figura 1D y 1F). En contraste, los frutos sin heridas y testigo con heridas no mostraron síntomas de enfermedad (Figura 1B y 1G). El daño interno en los frutos infectados consistió en una pudrición blanda que ocasionó desprendimiento de tejidos (Figura 1H).
Características Morfológicas de Mucor irregularis
Las colonias del aislamiento HP160, identificado como M. irregularis, fueron circulares, uniformes y con abundante micelio aéreo de color amarillo pálido (Figura 2A); color que de igual manera se observó al reverso de la placa (Figura 2B). La tasa de desarrollo micelial fue de 1.48 cm/día a 25 °C, no se desarrolló a 4 °C, pero sí entre 10 y 40 °C (Cuadro 2). Los esporangióforos, en su mayoría no ramificados, presentaron en la punta esporangios globosos, menores a 80 μm, que contenían en su interior cientos de esporangiosporas elípticas (Figura 2E y 2C). La columela de apariencia hialina casi esférica presentó un collarete remanente de la pared esporangial (Figura 2D).

Figura 2 Características morfológicas de Mucor irregularis. (A) Desarrollo micelial circular de color amarillo pálido y abundante micelio aéreo, (B) Placa invertida mostrando una coloración amarillo claro, (C) Esporangio globoso con esporangiosporas, (D) Liberación de esporangiosporas y presencia de columella con forma casi esférica y collarete característico y, (E) Esporangióforos hialinos no ramificados.
Características morfológicas de Gilbertella persicaria
G. persicaria mostró dos tipos de morfología colonial (Figura 3A y 3E). En el primer tipo, la colonia es petalada con esporangios amarillo a café (Figura 3C) y con un desarrollo micelial de 1.48 cm/ día a 25 °C (cepas HP09 y HP114); mientras que, el segundo tipo presenta colonias circulares, con micelio más denso y coloración de esporangios de gris a negro (Figura 3G y 3H), con una tasa de desarrollo micelial de 2.46 cm/día (HP15, HP19, HP167, HP178, HP183 y HP211). Ninguna de las cepas se desarrollaron a 4 °C, pero si mostraron desarrollo entre 10 a 40 °C. Otras características en G. persicaria incluyen la liberación de esporangiosporas por medio de la ruptura de la pared esporangial en dos mitades (Figura 3I), la presencia de pequeñas espinas hialinas en la pared de los esporangios (Figura 3K) y la presencia de apéndices hialinos en las esporangiosporas (Figura 3J). En las estructuras microscópicas de los dos tipos de colonias, no se observaron diferencias morfológicas relevantes (Cuadro 2). Los esporangióforos son escasamente ramificados (de cuatro a seis por cada 100 analizados), la columela ovoide con un collarete residual y, clamidosporas cilíndricas (Figura 3D) abundantes en PDA en cultivos de 5 días.

Figura 3 Características morfológicas de Gilbertella persicaria. (A) Colonia en PDA con desarrollo micelial irregular en colonias petaladas, (B) Reverso del cultivo, (C) Esporangio globoso multiesporado de color amarillo-café, (D) Clamidospora cilíndrica, (E) Cultivo en PDA con desarrollo micelial circular, (F) Reverso del cultivo, (G) Esporangios negros, (H) Esporangio maduro globoso multiesporado de color oscuro, (I) Apertura de la pared esporangial en dos mitades mostrando la columela ovoide, (J) Esporangiospora con apéndices hialinos polares, (K) Pared esporangial cubierta de pequeñas espinas hialinas. Figuras (J y K) barra=20 μm. Figura 3
Características morfológicas de Rhizopus oryzae (syn. R. arrhizus)
Las colonias de la cepa HP25 identificada como Rhizopus oryzae mostró abundante micelio aéreo de color blanco en un inicio y posteriormente grisáceo (Figura 4A y 4B), esporangios de color café oscuro a negro que contenían esporangiosporas de apariencia estriada (Figura 4C y 4E). Al liberarse las esporangiosporas se observó la columela globosa de color café claro (Figura 4D). Los esporangióforos emergen a partir de rizoides basales (Figura 4F). R. oryzae mostró desarrollo micelial de 2.46 cm/ día a 25 °C, no se desarrolló a 4 °C, pero sí entre 15 y 40 °C (Cuadro 2).

Figura 4 Características morfológicas de Rhizopus oryzae. (A) Colonia en PDA con abundante micelio algodonoso, (B) Colonia con esporangios negros, (C) Esporangio globoso multiesporado de color negro, (D) Columela de subglobosa a globosa, (E) Esporangiosporas de apariencia estriada y (F) Rizoide basal.
Identificación molecular
La reacción de amplificación con los oligonucleótidos V9G y LR3, generó un fragmento aproximado de 1500 pb, que incluyó la región completa ITS1-5.8S-ITS2 y la región D1/D2 de la subunidad 28S (LSU) ribosomal. Las secuencias consenso obtenidas variaron para la región ITS1-5.8S-ITS2 de 543 a 751 pb y para la región parcial de la subunidad 28S (LSU) ribosomal de 632 a 711 pb.
La comparación en la base de datos mostró para la secuencia de la cepa HP160 (N° Accesos KR076763 y KR076764), un porcentaje de identidad de 99% y 96% para la región ITS con cepas identificadas como Mucor sp. (N° Acceso HM770967 y KP714393, respectivamente); mostrando 91% de identidad con la cepa de M. irregularis (N° Acceso JX976251) (Figura 5); sin embargo, para la región parcial 28S (LSU) ribosomal mostró un 99% de identidad con diferentes cepas de M. irregularis, las cuales muestran un alineamiento en un 100% (Figura 6).

Figura 5 Dendrograma basado en el método de máxima verosimilitud a partir de secuencias obtenidas para la región ITS1-5.8S-ITS2 de aislados de especies de Mucorales y de cepas de referencia de NCBI, los N° de acceso se encuentran entre paréntesis. Los valores de probabilidad de los nodos para cada clado indican el porcentaje de las réplicas del análisis de 1000 bootstraps que soportan ese clado. Rhizomucor pusillos se utilizó como outgroup. La escala representa el número de sustituciones por sitio.

Figura 6 Dendrograma basado en el método de máxima verosimilitud a partir de la región 28S (LSU) para la cepa HP160 y cepas de referencia de NCBI, los N° de acceso se encuentran entre paréntesis. Los valores de probabilidad de los nodos para cada clado indican el porcentaje de las réplicas del análisis de 1000 bootstraps que soportan ese clado. Rhizomucor pusillos se utilizó como outgroup. La escala representa el número de sustituciones por sitio.
La comparación de las secuencias de la región ITS1-5.8S-ITS2 de las cepas HP09, HP15, HP19, HP114, HP167, HP178, HP183 y HP211, mostraron 100% de identidad con secuencias depositadas en el NCBI (N° de Acceso JN206224 y KC683539) de la especie G. persicaria, donde se observa en el análisis filogénetico la formación de dos grupos que corresponden a los dos morfotipos encontrados (Figura 5). La confirmación de esta especie se realizó con el análisis de las secuencias de la región parcial 28S (LSU) ribosomal (Cuadro 1), que de igual manera mostró 100% de identidad con las cepas (N° Acceso JN206517 y JN939197) de G. persicaria. Finalmente, la comparación de la secuencia de la cepa HP25 de la región ITS1-5.8S-ITS2 y parcial 28S (LSU) (N° de Acceso KT899481 y KT899482) mostró 100% de identidad con secuencias publicadas en el NCBI (N° de Acceso KJ417552 y AY213624), respectivamente, de la especie R. oryzae (Figura 5). La cobertura de las consultas de todas las secuencias consenso en la base de datos de NCBI varió de 91 a 100%.
DISCUSIÓN
En México, la identificación de especies de mucorales fitopatógenos es escasa. Los resultados de este estudio enfocado en utilizar técnicas morfológicas y moleculares para identificación de hongos evidenció la presencia de tres especies de mucorales asociados a pudrición blanda de frutos de papaya. Los síntomas causados en frutos de papaya fueron similares a los reportados en frutos de durazno, pera y manzana (Michailides y Spotts, 1990), con lesiones iniciales suaves y de apariencia acuosa, y que en un corto periodo de tiempo se cubren de masas de micelio. El síntoma de pudrición blanda observada en frutos se debe principalmente, a la producción de enzimas como amilasas, lipasas, poligalacturonasas y proteasas, las cuales contribuyen en la degradación de los polisacáridos estructurales y de almacenamiento (Alves et al., 2002; Krisch et al., 2010).
Las características morfológicas de M. irregularis concuerdan con lo descrito por diversos autores (Walther et al., 2013; Peng et al., 2015). Es necesario mencionar que, considerando que el género Mucor es polifilético, la region ITS fue insuficiente para discriminar entre especies estrechamente relacionadas, por lo que la confirmación de especie se realizó con el análisis filogénetico de la región 28S (LSU) (Walther et al., 2013; Hoffmann et al., 2013; Hyde et al., 2014). M. irregularis recientemente fue reportado como fitopatógeno en maíz (Peng et al., 2015). Éste se considera un primer reporte de M. irregularis afectando papaya.
La morfología de las estructuras de G. persicaria es similar a la reportada por diversos autores (Benny, 1991; Guo et al., 2012; Pinho et al., 2014); es considerada una especie monotípica en su género, el cual se ubica en la subfamilia Gilbertelloideae y familia Choanephoraceae (Hyde et al., 2014). En este estudio se observó el desarrollo de dos morfotipos, los cuales se han definido por Lacap et al. (2003) como cepas de una misma especie que presentan diferencias en morfología colonial y tasa de desarrollo micelial, lo cual se corroboró con la separación en clados dentro de los dendogramas tanto de la región ITS como 28S (Figuras 5 y 6). El morfotipo 1 (colonias petaladas, esporangios café) se observó en el 31% de los aislamientos; mientras que el resto de aislamientos mostró el morfotipo 2 (69%). G. persicaria es un patógeno común en regiones tropicales y subtropicales causando la pudrición blanda de tomate, pera, durazno, pitahaya, jambolán (Ginting et al., 1996; Guo et al., 2012; Pinho et al., 2014) y recientemente, se reportó causando enfermedad en frutos de papaya en el estado de Colima, México (Cruz-Lachica et al., 2016); en este estudio se reporta la presencia de esta especie en los estados de Oaxaca y Veracruz con lo que se demuestra que está presente en otras regiones de producción de papaya y debería considerarse su distribución como un factor de importancia.
Las características morfológicas de R. oryzae, concuerdan con las reportadas por diversos autores y su identidad se confirmó mediante el análisis filogenético. R. oryzae se ha reportado causando pudrición blanda en banana y cidra (Kwon et al., 2012; Hakim et al., 2015), por lo que se considera a este trabajo como el primer reporte afectando papaya en México.
De acuerdo con el origen de las muestras analizadas, M. irregularis fue detectado en el estado de Veracruz, R. oryzae en el estado de Colima y G. persicaria en Colima, Oaxaca y Veracruz. En México, Mucor hiemalis y M. circinelloides se han descrito afectando papaya en Veracruz (Suárez-Quiroz et al., 2013); sin embargo, la identificación de especies en ese estudio sólo involucró características morfológicas, por lo que se recomienda realizar análisis moleculares debido a lo siguiente:
M. hiemalis y M. irregularis se encuentran en un grupo de especies estrechamente relacionadas que requieren del uso de análisis moleculares en al menos dos regiones del genoma para su identificación (Walther et al., 2013); en el caso de M. circinelloides, la identificación en base a síntomas en fruto y características de esporangios concuerda con lo obtenido para G. persicaria, por lo que características distintivas de esta última especie como apertura de pared esporangial en dos mitades y apéndices hialinos en esporangiosporas pueden pasar desapercibidas.
CONCLUSIONES
En este estudio se demostró que Gilbertella persicaria, Mucor irregularis y Rhizopus oryzae son agentes causales de pudrición blanda en frutos de papaya. Éste es el primer reporte de Mucor irregularis y de Rhizopus oryzae afectando este cultivo en México y además demuestra que G. persicaria se encuentra afectando frutos en huertos de los tres estados. Finalmente, este estudio evidencia que la caracterización fenotípica y genotípica en conjunto es una estrategia adecuada para identificar especies, en particular las que pertenecen a taxones estrechamente relacionados. Los números de acceso de M. irregularis y R. oryzae en el genbank NCBI fueron KR076763 y KR076764, y KT899481 y KT899482, respectivamente.











 texto en
texto en