El género Phytophthora, dentro de los oomicetes, incluye un grupo diverso de microorganismos fitopatógenos destructivos que infectan un amplio rango de hospedantes y causan considerables pérdidas económicas en ambientes naturales y agrícolas en todo el mundo (Erwin y Ribeiro, 1996). Más de 100 especies de Phytophthora han sido descritas (Martin et al., 2012); algunas especies, son altamente especializadas a un hospedante y otras especies, como por ejemplo P. nicotianae, tienen un amplio rango de hospederos en cultivos agrícolas y plantas ornamentales.
Vinca rosea (L.) es una planta herbácea, también identificada como Catharanthus roseus (L.) G. Don. Es ornamental, resistente a altas temperaturas, humedad, sequía e infertilidad de suelos (Schubert y Leahy, 1989). Es ampliamente utilizada como planta de jardín y actualmente existen más de 100 híbridos desarrollados (McGovern y Begeman, 1996); adicionalmente, las plantas de vinca se han identificado como hospedantes secundarios para muchos microorganismos fitopatógenos como virus, hongos y oomicetes (Hagan, 2004), los cuales afectan cultivos hortofrutícolas como papaya, tomate, chile y pepino, entre otros (Haider et al., 2007).
La enfermedad conocida como tizón y marchitamiento en plantas de V. rosea es causada por Phytophthora spp., progresa rápidamente y se presenta en climas cálidos y húmedos especialmente en el verano cuando la humedad es alta (Hao et al., 2010). Los síntomas de la enfermedad inician con una coloración gris en las puntas de los tejidos infectados, los cuales gradualmente se necrosan. Las lesiones progresan rápidamente y ocasionan un marchitamiento general de la planta (McGovern y Begeman, 1996). Varias especies, entre las que se incluyen P. nicotianae, P. citrophthora, P. cryptogea y P. tropicalis, se han descrito como causantes de tizón y marchitez en plantas de vinca (McGovern y Begeman, 1996; McMillan y Garofalo, 2004; Orilowski et al., 2011; Hao et al., 2010).
La identificación y caracterización de Phytophthora spp. se basa principalmente en características morfológicas (Waterhouse, 1963): tipo de micelio, forma, tamaño y desarrollo de esporangios, producción de estructuras de reproducción sexual, así como la formación de clamidosporas (Pintos et al., 2004). Debido a la variación morfológica intraespecífica, la identificación exacta de aislados es con frecuencia difícil, incluso para los especialistas. Por esta razón, las técnicas moleculares que analizan secuencias genéticas han ayudado a la identificación y delimitación de especies. En oomicetes, las regiones genómicas más comúnmente utilizadas para la identificación, a nivel de especie, son la región espaciadora interna transcrita (ITS) (Cooke et al., 2000) y algunas regiones codificantes de genes nucleares, que incluyen el factor de elongación 1-α (TEF-1α) y beta tubulina (β-Tub) (Kroon et al., 2004). Otra región prometedora para desarrollar oligonucleótidos específicos para especies de Phytophthora es el gen YPT1 relacionado con una proteína RAS (Meng y Wang, 2010), ya que las regiones no codificantes de este gen han mostrado suficiente variación para fundamentar el desarrollo de marcadores moleculares para casi todas las especies de Phytophthora (Schena y Cooke, 2006).
En Culiacán, Sinaloa, las plantas ornamentales de vinca son comunes en parques y/o camellones urbanos, jardines en casas y viveros de plantas ornamentales. Durante la época de lluvia en verano (junio-agosto) es común observar en las plantas síntomas de tizones de hoja y marchitez. Un reporte previo menciona la presencia de P. nicotianae en vinca causando tizón (Alvarez-Rodriguez et al., 2013); sin embargo, se desconoce si es la única especie por lo que los objetivos de este estudio fueron obtener y caracterizar, mediante técnicas morfológicas y moleculares, las especies de Phytophthora asociadas al tizón en plantas de vinca, determinar los tipos de compatibilidad sexual y evaluar la patogenicidad en los aislados obtenidos.
Materiales y métodos
Aislamiento del agente causal de tizón y marchitez en vinca
Se colectaron muestras de plantas enfermas creciendo en jardines urbanos y viveros de plantas ornamentales en Culiacán. Muestras de tejidos infectados se colocaron en bolsas de plástico y se trasladaron al laboratorio para realizar el aislamiento de las especies de Phytophthora asociadas a la enfermedad. En el laboratorio, pequeñas porciones de tejido vegetal infectado se lavaron con agua destilada, se desinfestaron con hipoclorito de sodio al 0.5 % por 1 min y finalmente se colocaron en cajas Petri con medio de cultivo agar V8 (840 ml de agua destilada, 163 ml de jugo V8, 3 g de CaCO3 y 18 g de agar bacteriológico), las cuales se mantuvieron a temperatura ambiente por 72 h.
Caracterización morfológica
Los aislados se reactivaron en medio de cultivo agar V8, una vez que se desarrolló el micelio, se realizaron preparaciones permanentes con lactofenol y fueron observadas en un microscopio (Carl Zeiss Axiostar Imager A2). La purificación de cada aislado se realizó mediante la técnica de transferencia de puntas de hifa (Donahoo y Lamour, 2008). Para inducir el desarrollo de esporangios, se realizaron cortes en la colonia en crecimiento y la placa se inundó con agua destilada estéril. Las placas se incubaron por 3 días a temperatura ambiente (Martin et al., 2012). La presencia de esporangios fue revisada en un microscopio biológico adaptado con una cámara, una vez que se produjeron los esporangios se retiró el agua y se agregó a las placas fucsina acida al 2% para teñirlos. Después de 24 h se tomaron muestras y se observaron al microscopio para registrar las medidas de esporangios y otras estructuras.
La presencia de clamidosporas se observó en medio a base de zanahoria (200 g de zanahoria, 20 g de agar bacteriológico y 1 L de agua destilada). Los aislados crecieron en el medio a base de zanahoria y se incubaron por 15 días a 25 °C en la oscuridad (Mirsoleimani y Mostowfizadeh-Ghalamfarsa, 2013); después de este tiempo, las observaciones se realizaron bajo el microscopio para registrar la presencia y tamaño de las clamidosporas.
Para determinar el tipo de compatibilidad y registrar la formación de estructuras de reproducción sexual, cada aislado se confrontó con cepas de referencia A1 y A2 de P. nicotianae en medio de cultivo V8; un disco de 0.5 cm de diámetro de agar con micelio en crecimiento activo de cada aislado se colocó a una distancia aproximada de 1 cm de distancia de discos de agar con micelio en crecimiento activo de las cepas de referencia A1 y A2 de P. nicotianae. Las placas se incubaron por 15 días a 25 °C y entonces se registró la presencia/ausencia de oosporas, así como las características de oogonios y anteridios (Javier-Alva y Mattos, 2006; Waterhouse, 1963).
Identificación molecular
El micelio de los aislados se desarrolló en medio líquido PD (papa-dextrosa), y se almacenó a -20 °C. El ADN se extrajo utilizando el protocolo propuesto por Zelaya-Molina et al. (2011), el cual consiste en una serie de lavados con cloroformo-alcohol isoamílico. Un grupo de 10 aislados representativos que comparten características morfológicas se utilizaron para amplificar el ADN con los pares de oligonucleótidos ELONGF1 (5´-TCACGATCGACATTGCCCTG-3´) y ELONGR1 (5´-ACGGCTCGAGGATGACCATG-3´) para TEF-1α, TUBUF2 (5´-CGGTAACAACTGGGCCAAGG-3´) y TUBUR1 (5´-CCTGGTACTGCTGGTACTCAG-3´) para beta tubulina (Kroon et al., 2004) y los oligonucleótidos DC6 (5´-GAGGGACTTTTGGGTAATCA-3´) e ITS4 (5´-TCCTCCGCTTATTGATATGC-3´) para la región ITS (Bonants et al., 1997).
Las condiciones para efectuar la PCR usando los oligonucleótidos para TEF-1α y β-Tub consistieron de una desnaturalización inicial a 95 °C por 2 min, después 35 ciclos de desnaturalización a 95 °C por 1 min, alineamiento a 60 °C por 1 min, elongación a 72 °C por 1 min y una extensión final a 72 °C por 10 min. Las condiciones de amplificación con los oligonucleótidos para ITS fueron 94 °C por 3 min, después 30 ciclos a 94 °C por 1 min, 55 °C por 1 min, 72 °C por 1 min y una extensión final a 72 °C por 10 min.
Los oligonucleótidos específicos para la especie P. nicotianae, Pn1 (5´-GACTTTGTAAGTGCCACCATAC-3´) y Pn2 (5´-CTCAGCTCTTTTCCTTGGATCT-3´) (Meng y Wang, 2010), se utilizaron bajo las siguientes condiciones de PCR: 94 °C por 5 min, 35 ciclos a 94 °C por 1 min, alineamiento a 58 °C por 1 min, elongación a 72 °C por 1 min y una extensión final a 72 °C por 10 min.
Secuenciación y análisis filogenético
Los productos de PCR obtenidos de los genes ITS, TEF-1α y β-Tub se purificaron con el kit Wizard SV Gel and PCR Clean-Up System (Promega), siguiendo las especificaciones descritas por el proveedor. La secuenciación se realizó por electroforesis capilar con un secuenciador automático ABI PRISM 310 (Genetic Analizer PE Applied Biosystems), en la unidad de Biología Molecular del Instituto de Fisiología Celular de la UNAM. Las secuencias fueron editadas con el programa CHROMAS Pro 1.6 (Technelysium Pty Ltd, South Brisbane, Qld, Australia), y comparadas con secuencias del Banco de Genes (GenBank, siglas en inglés) del Centro Nacional para la Información Biotecnológica (NCBI, siglas en inglés) utilizando el software alineamiento local básico de nucleótidos (BLAST-N, siglas en inglés) y el algoritmo Megablast. Para el alineamiento múltiple y análisis filogenético de cuatro secuencias de aislados representativos se utilizó el software MEGA 6.06 (Tamura et al., 2013). Las secuencias fueron alineadas con otras 22 pertenecientes a diferentes especies de Phytophthora (Cuadro 1) utilizando el programa MUSCLE integrado en MEGA (Edgar, 2004), usando los valores por default para la penalización por apertura y extensión de gaps y con posterior corrección manual del alineamiento. Los alineamientos fueron sometidos al programa de selección del modelo de sustitución nucleotídica, para seleccionar el modelo evolutivo que mejor se ajustara a los datos. Para la reconstrucción filogenética se utilizó el método de máxima verosimilitud y el modelo Tamura-Nei. La variación de tasas entre sitios fue modelada por una distribución gamma más sitios invariables (4 categorías). El soporte de la topología interna de los dendrogramas fue determinado por análisis de bootstrap con 1000 remuestreos (Felsenstein, 1985). Se realizaron alineamientos concatenados de los 3 marcadores utilizando el software DAMBE 6.1.23 (Xia, 2013) y un árbol filogenético fue construido como se describió previamente.
Evaluación de patogenicidad
Plantas de vinca de 10 semanas de edad se inocularon al asperjar una suspensión de zoosporas de 1 x 104 zoosporas/ml de 10 aislados representativos provenientes de distintos sitios de colecta. Tres macetas que contenían 5 plantas cada una, se inocularon con cada aislado. Plantas inoculadas sólo con agua destilada estéril se utilizaron como un tratamiento testigo. Todas las plantas se mantuvieron en un área de malla sombra a 25 °C y 80-90 % de humedad relativa. Una vez que se desarrollaron los síntomas en las plantas inoculadas, se realizó el reaislamiento del patógeno y se cuantificó la incidencia de la enfermedad.
Resultados y Discusión
Identificación morfológica
Se obtuvieron 39 aislados de Phytophthora sp. provenientes de plantas de vinca con síntomas de tizón y marchitez (Figura 1A); 77 % provinieron de viveros de plantas ornamentales y 23 % de plantas en jardines y áreas urbanas. Los aislados produjeron micelio cenocítico y colonias algodonosas de color blanco en agar V8; crecieron entre 7 y 30 °C (temperatura óptima 25 °C); desarrollaron esporangios persistentes, terminales de forma oval o alimonada, principalmente papilados (Figura 1B) que midieron en promedio 34.16 μm de longitud y 26.35 μm de ancho (n=24). Waterhouse (1963) menciona que el rango del tamaño de esporangios de P. nicotianae se encuentra alrededor de 25-50 x 20-40 μm; mientras que, Erwin y Ribeiro (1996) reportaron rangos de 11-60 x 20-45 μm. Una proporción largo/ancho de 1.29 μm (n=24) se observó en los esporangios, similar a la proporción de 1.3 descrita por Erwin y Ribeiro (1996) para P. nicotianae. Todos los aislados mostraron la presencia de clamidosporas intercalares y terminales (Figura 1D), lo cual es una característica de P. nicotianae (Waterhouse, 1963; Erwin y Ribeiro, 1996).
Compatibilidad sexual
Los aislados fueron heterotálicos, 21 de tipo de compatibilidad A1 y 18 tipo de compatibilidad A2 (Cuadro 2). La interacción entre los tipos de compatibilidad A1 y A2 produjeron oosporas apleróticas (Figura 1C), con oogonios esféricos y lisos, y anteridios anfíginos. Estas características son consistentes con P. nicotianae según Waterhouse (1963) y Erwin y Ribeiro (1996).
En algunos sitios de muestreo se encontraron los dos tipos de compatibilidad, lo cual sugiere que existe recombinación sexual; esto incrementaría la variabilidad genética de la población y podría causar la aparición de aislados altamente patogénicos (Rodríguez-Tovar et al., 2004; Blaya et al., 2015). Una observación importante fue la presencia de los dos tipos de compatibilidad en tres de los cuatro viveros muestreados, lo que significa que podría ocurrir recombinación en este sitio y que al venderse algunas plantas con infecciones iniciales, éstas representarían un factor de dispersión en la región.
Identificación molecular
Un fragmento de aproximadamente 1300 pb se obtuvo cuando se utilizaron los oligonucleótidos DC6/ITS4 en los diez aislados seleccionados. Los productos se secuenciaron y se compararon con secuencias depositadas en el NCBI. Los porcentajes de homología fluctuaron entre 98-100 % con la especie P. nicotianae. Los oligonucleótidos ELONGF1/ELONGR1 generaron un fragmento de aproximadamente 1000 pb, los productos se secuenciaron y cuando se compararon las secuencias se obtuvo una homología entre 99-100 % con P. nicotianae. Los oligonucleótidos TUBUF2/TUBUR1 generaron un fragmento de aproximadamente 1000 pb, y sus secuencias mostraron una homología entre 98-100 % con P. nicotianae. Las secuencias de las distintas regiones amplificadas fueron depositadas en el NCBI (Cuadro 3).
Cuadro 3. Accesiones de secuencias de tres loci depositadas en GenBank de aislados de P. nicotianae obtenidos de V. rosea de zonas urbanas en Culiacán e incidencia de infección en experimento de patogenicidad.
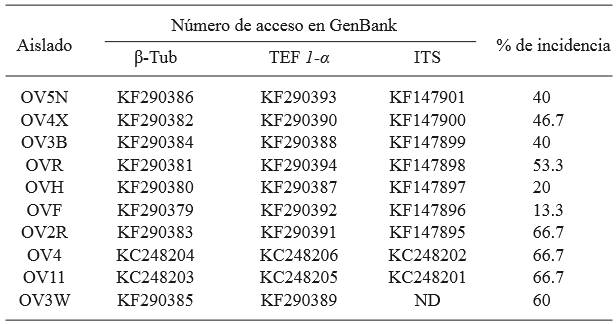
Β-Tub = beta tubulina, TEF 1-α = factor de elongación, ITS= región espaciadora interna transcrita ND= no determinado
Los oligonucleótidos Pn1/Pn2 específicos para P. nicotianae amplificaron un fragmento de aproximadamente 400 pb en todos los aislados (Figura 2). Estos resultados fueron los esperados para P. nicotianae (Meng y Wang, 2010). La utilización de estos oligonucleótidos específicos puede ser una estrategia rápida para identificar esta especie.
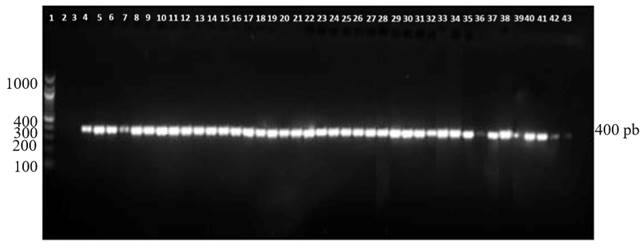
Figura 2. Fragmento amplificado de ADN con los oligonucleótidos Pn1-Pn2 específicos para P. nicotianae. Línea 1, Marcador molecular 100 bp; Línea 2, agua; Línea 3, control negativo (P. capsici); Línea 4, P. nicotianae control positivo, Líneas 5-43 aislados de P. nicotianae de Vinca rosea.
La reconstrucción filogenética resultante de la concatenación de los tres marcadores mostró que los cuatro aislados analizados en el presente estudio se agruparon en el mismo clado que las cepas de referencia de P. nicotianae (Figura 3; 100 % de bootstrap), perteneciente al clado 1 de especies de Phytophthora (Cooke et al., 2000; Kroon et al., 2004; Blair et al., 2008). Los análisis de las secuencias parciales individuales de cada marcador mostraron bajos porcentajes de soporte de la topología interna de los dendrogramas comparados con los valores resultantes de la concatenación de los 3 loci bajo análisis. Con las secuencias obtenidas en el presente estudio, se realizó el alineamiento individualmente por cada marcador utilizado (ITS, β Tub y TEF 1-α), posteriormente se concatenaron los 3 alineamientos (1589 pb) y se encontró una similitud entre el 99.6 y 100 % entre las secuencias, por lo que no todas las secuencias son iguales, se encontraron 11 diferencias entre nucleótidos y 2 gaps por inserciones de nucleótidos (Datos no mostrados). En un estudio futuro se podría estimar la diversidad genética de poblaciones de Phytophthora nicotianae al analizar un mayor número de aislados, de diferentes regiones y años de colecta para enriquecer el estudio y correlacionarla con las características fenotípicas del patógeno.
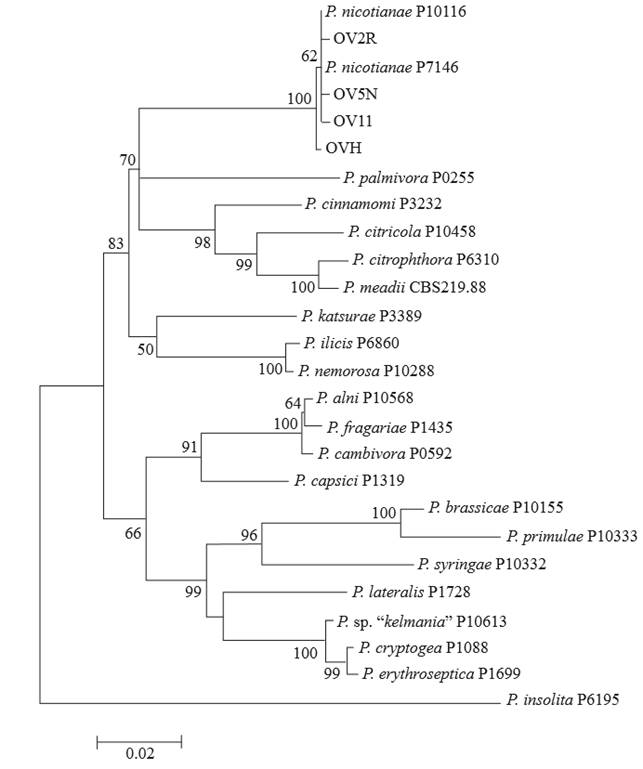
Figura 3. Árbol filogenético de aislamientos representativos del presente estudio (OV2R, OV5N, OV11 y OVH) y 22 cepas de referencia de Phytophthora derivado de las secuencias parciales de tres loci (ITS, β Tub y TEF 1-α). El árbol fue construido con el software MEGA 6.06 (bootstrap=1000), utilizando el modelo de sustitución Tamura-Nei (TN93 G+I). Los valores bootstrap están mostrados sobre las ramas.
Evaluación de patogenicidad
Los 10 aislados evaluados en los ensayos de patogenicidad causaron los síntomas de la enfermedad en plantas de V. rosea. Los síntomas iniciales de la enfermedad se observaron a los 4 días posteriores a la inoculación e incluyeron tizón de hojas. Después, las plantas desarrollaron síntomas de marchitez y murieron a los 12 días posteriores a la inoculación. Todos los aislados fueron virulentos, infectaron plantas sanas de vinca; sin embargo, existió una variación en el nivel de incidencia, ya que el porcentaje de plantas con síntomas fluctuó entre 13 y 66.7 % (Cuadro 3). Las cepas inoculadas fueron reaisladas de plantas inoculadas.
Es importante mencionar que P. nicotianae infecta más de 300 hospedantes diferentes entre los que se incluyen Solanum lycopersicum y Capsicum annuum (Erwin y Ribeiro 1996). Por ello, no podemos descartar que la especie P. nicotianae encontrada en plantas de vinca en el área urbana, también esté causando algunas infecciones en el área hortícola del valle de Culiacán, la cual se basa principalmente en la producción de tomate y chile. Aunque diversas especies de Phytophthora han sido reportadas como causantes de tizón y marchitez en vinca (McGovern y Begeman, 1996; McMillan y Garofalo, 2004; Orilowski et al., 2011; Hao et al., 2010), el análisis morfológico, filogenético y patogénico realizado en esta investigación demuestra que P. nicotianae es la responsable de los daños observados en esta planta ornamental en las muestras colectadas en la ciudad de Culiacán.
Conclusiones
Phytopthora nicotianae fue la única especie asociada al tizón y muerte de plantas de vinca en la ciudad de Culiacán. La caracterización molecular indicó que las secuencias nucleotídicas en las regiones TEF 1-α, β-Tub e ITS mostraron 98-100 % de homología con la especie P. nicotianae y se agruparon filogenéticamente con cepas de referencia de esta especie. Todos los aislados fueron detectados por los oligonucleótidos Pn1/Pn2 específicos para P. nicotianae. Los postulados de Koch demostraron la patogenicidad de los aislados sobre vinca. Este es uno de los pocos estudios enfocados en enfermedades de plantas ornamentales en áreas urbanas en el Noroeste de México.











 text in
text in 





