El trompillo, Solanum elaeagnifolium es una planta perenne de la familia Solanaceae, endémica del noreste de México y el sureste de EE.UU. (Texas y Florida) (Cuda et al., 1988), se considera una planta invasora y se distribuye en casi todo el mundo (EPPO, 2007). Se considera a la región de Monterrey, Nuevo León, México, como centro de origen y evolución de esta planta con base a la gran diversidad, distribución y frecuencia de enemigos naturales que presenta (Wapshere, 1988). Es una maleza de importancia económica en varios cultivos sobre todo en agricultura intensiva del norte de la República Mexicana. También, se considera una especie ruderal como resultado de los habitats alterados por el hombre y comúnmente se puede encontrar en orillas de carreteras, campos de cultivo abandonados, inclusive en zonas urbanas (Vibrans, 2009). Debido a la capacidad que tiene como reservorio de plagas y enfermedades, se han establecido medidas de manejo cultural como parte de la regulación fitosanitaria para restringir su establecimiento y dispersión de insectos plaga y otros fitófagos (DOF, 1997). Dentro de los organismos asociados a S. elaeagnifolium y que pudieran ser utilizados como posibles agentes de control biológico destacan diferentes órdenes y familias de insectos, ácaros eriófidos y nematodos. El nematodo agallador foliar Orrina phyllobia (Thorne, 1934) Brzeski, 1981 fue reportado inicialmente como Ditylenchus phyllobius sin. Nothanguina phyllobia (Orr.) y está distribuido en el sur de EE.UU. y sur de la India afectando esta maleza (Brzeki, 1991). Es un parásito obligado e induce agallas sobre las hojas, por lo que puede utilizarse como un potencial método de control biológico de S. elaeagnifolium (Cuda et al., 1988, Wapshere, 1988, Sforza y Jones, 2007, Kwong y Sagliocco, 2012). La identificación de O. phyllobia está basada principalmente en caracteres morfológicos de hembras y machos (Siddiqi, 2000, Subbotin y Riley, 2012), sin embargo, debido a que existe una gran variedad de especies de nematodos anguinidos que afectan las partes aéreas, es necesario confirmar su identidad mediante procedimientos de mayor precisión basados en su información genética. En México se carece de evidencia objetiva de la presencia de este nematodo afectando esta maleza por lo que en esta investigación se propuso identificar por morfotaxonomía, su confirmación molecular y la reconstrucción filogenética de poblaciones del nematodo agallador foliar O. phyllobia de Guanajuato, México.
En el mes de julio del 2015 se realizó un muestreo dirigido a plantas de S. elaeagnifolium con deformaciones o agallas en hojas (Figura 1) presentes en parcelas aleatorias de maíz (< de 5 ha) y puntos localizados de zonas urbanas como orillas de carreteras y caminos. Se colectaron un total de 12 muestras en el municipio de San Luis de la Paz, Guanajuato, México entre las coordenadas geográficas: 21° 16' 24.39" N, 100° 29' 35.17" W y 21° 18' 33.41" N, 100° 31' 39.32" W. Las muestras se conservaron en papel humedo para conservar la humedad e integridad de los especímenes y mantenidas en refrigeración a 4 °C ± 2. Las hojas deformadas y con agallas de S. elaeagnifolium se disectaron e incubaron en agua destilada estéril a 22 °C ± 2 para favorecer la emergencia de los diferentes estadios biológicos (huevos, juveniles de segundo estadio (J2), machos y hembras). Para el estudio morfológico, los nematodos (machos y hembras) se transfirieron a una gota de agua sobre un porta objetos, se relajaron en calor con la ayuda de un mechero de alcohol y se montaron en medio agua-agar 2 % (Esser, 1986). Los especímenes se examinaron en un microscopio compuesto con contraste de fases Carl Zeiss adaptado con un micrómetro y cámara digital, se tomaron mediciones de los principales caracteres morfométricos de machos y hembras. Se consideraron los siguientes caractéres: L=longitud del cuerpo; longitud del estilete, espícula, bursa y gobernáculo así como los índices De Man: a=longitud del cuerpo/anchura máxima del cuerpo; b=longitud del cuerpo/longitud del esófago; b'=longitud del cuerpo/distancia de la región anterior al post corpus; c= longitud del cuerpo/longitud de la cola; c'=longitud de la cola/ancho del cuerpo a nivel del ano; V=distancia región anterior-vulva; V'=distancia de la región anterior a la vulva/región anterior al ano X 100. Se realizó una comparación morfotaxonómica de los valores obtenidos con los reportados por Brzeski, 1991; Nickle, 1991; Subbotin and Riley, 2012. Para la realización de Microscopía Electrónica de Barrido (MEB), machos y hembras se fijaron en solución formol-glicerol 8:2, por un periodo mínimo de 24 h y se realizaron cinco lavados con solución sorensen (cacodilato de sodio 0.1 M y pH=7.0) cada 15 min. En una campana de extracción se colocaron 500 μl de tetraóxido de osmium 1 % por 2 h, se decantó y se volvieron a efectuar cinco lavados con solución sorensen (Eisenback, 1985). Posteriormente se deshidrataron en gradientes de etanol (30, 40, 50, 60, 70, 80, 90, 100 %). Se colocaron los especímenes en sobres de papel filtro y se introdujeron a una cámara de secado de punto crítico Quorum-Emitech K850 (Eisenback, 1986). Una vez terminado el proceso de secado se colocan los especímenes en discos porta muestra y se observaron en un microscopio de barrido marca Zeiss Sigma VP.

Figura 1 Síntomas causados por el nematodo agallador foliar Orrina phyllobia en trompillo Solanum elaeagnifolium. (A), (B) y (C) distorsión y agallamiento de hojas; (D) Agallamiento en la inflorescencia.
La extracción de ADN a partir de especímenes individuales de J2, machos y hembras se realizó a través del método propuesto por Williams et al., 1992 y Thomas et al., 1997 utilizando los especímenes previamente analizados por morfotaxonomía. Los nematodos se colocaron individualmente en 10 μl de solución amortiguadora de lisis (10 mM Tris-HCl, pH 8.0, 2.5 mM MgCl2, 50 mM KCl, 0.45 % Tween 20; 0.05% gelatina) sobre un cubreobjetos. Se maceró el especímen con una punta de micropipeta con ayuda del microscopio estereoscópico y se mezcló la solución amortiguadora de extracción-nematodo. Se recuperó la solución y se trasfirió a un tubo de microcentrifuga de 200 μl y congeló a -40 °C por 30 min. Pasado el tiempo de congelación, las muestras se incubaron a 65 °C por 1 h agitando al menos una vez y se agregó 0.1 μl de Proteinasa K (60 μg/m) en los últimos 10-15 min. Se incubó a 95 °C por 15 min para inactivar la Proteinasa K y se conservó a -20 °C. Para la reacción de la cadena de polimerasa (PCR) se amplificaron dos marcadores moleculares: 1. La región del ITS1-5.8S-ITS2 del ADNr utilizando los iniciadores AB28 (5'-GTTTCCGTAGGTGAACCTGC-3') (Joyce et al., 1994) y TW81 (3'-ATATGCTTAAGTTCAGCGGGT-5') (Howlett et al., 1992). 2. La amplificación de los segmentos de expansión D2-D3 del gen 28S con los iniciadores D2A (5'-ACAAGTACCGTGAGGGAAAGTTG-3') y D3B (3'-TCGGAAGGAACCAGCTACTA-5') (Nunn, 1992). La reacción de la PCR para los iniciadores AB28 y TW81 se realizó en un volumen final de 50 μl con los siguientes componentes: 5 μl de solución amortiguadora de PCR 10X, 3 μl de MgCl2 50 mM, 1 μl mix de DNTP's 10 mM, 2 μl de cada iniciador a 10 μM, 0.5 μl de Taq polimerasa 5 U/μl, 5 μl de extracción de ADN y 31.5 μl de H2O libre de ADNAsas y ARNasas. Las condiciones de termociclaje incluyeron una etapa de desnaturalización inicial de 9 5°C durante 3 min seguido de 35 ciclos de 95 °C durante 0.45 min, 57 °C durante 0.30 min y 72 °C durante 1.30 min, y finalizando con un ciclo a 72 °C durante 5 min (Skantar et al., 2007). La reacción de la PCR para los iniciadores D2A y D3B comprendió 5 μl de solución amortiguadora de PCR 10X, 3 μl de MgCl2 50mM, 1 μl mix de DNTP's 10mM, 2 μl de cada iniciador 10 μM, 0.5 μl de Taq polimerasa 5U/μl, 5 μl de extracción de ADN y 31.5 μl de H2O libres de ADNasas y ARNasas. Las condiciones de la amplificación fueron las siguientes: desnaturalización inicial de 95 °C durante 3 min, seguido de 35 ciclos de 95 °C durante 0.45 min, 55 °C durante 0.45 min y 72 °C durante 1 min, y finalmente con un ciclo a 72 °C durante 5 min (Vovlas et al., 2011). Se utilizó un termociclador i-Cycler (BIO-RAD) para todas las reacciones. Los productos amplificados se analizaron mediante electroforesis horizontal utilizando geles de agarosa al 1.4 % en solución tampón 1X TAE, teñidos con GelRed y visualizados en un fotodocumentador Gel Doc EZ (BIORAD). Los productos de la PCR fueron secuenciados mediante tecnología Sanger, ensamblados y editados con el programa CodonCode Aligner 4.2.7 (CodonCode Corporation, Centerville, Massachusetts). Durante la edición de los electroferogramas fue importante revisar manualmente los posibles errores en la lectura de las bases nitrogenadas de la secuencia de ADN y se eliminaron los iniciadores.
Obtenidas las secuencias de poblaciones mexicanas, se realizó la búsqueda de homología con secuencias del GenBank (NCBI), utilizando el algoritmo BLAST para comprobar si las secuencias generadas de los nematodos que causan agallas en S. elaeagnifolium pudieran ser similares o iguales a otras secuencias reportadas. Las secuencias de ambos marcadores moleculares de las poblaciones mexicanas junto con otras secuencias del GenBank se sometieron a una alineación múltiple con la opción de Clustal W (Thompson et al. 1994). La búsqueda del mejor modelo de sustitución para ambos marcadores se realizó a través de Mr Modeltest 2.3 (Nylander, 2004) y PAUP 4.0b10 (Swofford, 1998). El análisis filogenético para los dos marcadores se llevó a cabo mediante inferencia bayesiana con MrBayes v3.2 (Ronquist y Huelsenbeck, 2003), con el modelo GTR+G, con un árbol de arranque aleatorio y se corrió con la Cadena de Markov de Monte Carlo (MCMC) para 1x106 generaciones, tomando muestras a intervalos de 100. Dos corridas se realizaron para cada análisis. Después de desechar muestras burn-in y la evaluación de la convergencia, las muestras restantes se retuvieron para análisis posteriores. Las topologías se utilizaron para generar un árbol con la regla de la mayoría de consenso 50 % de probabilidades posteriores para determinar los valores de soporte de los clados obtenidos. Para ambas filogenias se utilizó Radopholus similis (GQ281456) para la región ITS15.8S-ITS2 y Radopholus spp. (HQ823572) para la región D2D3 del gen 28S como grupos externos. Los árboles filogenéticos se visualizaron utilizando Fig Tree (Rambaut, 2009).
Las características y valores morfotaxonómicos de hembras y machos coinciden parcialmente con las descripciones hechas por Brzeski (1991), y las reportadas por Subbotin y Riley (2012) para el nematodo agallador foliar Orrina phyllobia (=Ditylenchus phyllobius). Hembra: Cuerpo corto y recto 692 μm (637-785); cápsula cefálica plana en forma de tapón (Figura 2A y Fig. 2D), estilete pequeño 9 μm (8-9) con nódulos grandes; esófago alargado, ausencia del bulbo medio; glándula esofágica sobrepuesta al intestino; campo lateral con cuatro incisuras (Figura 2E); Ovario extendido, oocitos en una o dos filas; saco post-uterino corto; cola aguda con punta redondeada. Macho: Cuerpo corto y delgado 700 μm (672-737), estilete delicado 9 μm (8-9) con nódulos grandes; capsula cefálica muy parecida a la de la hembra (Figura 2B); espícula corta 19.8 μm (18-21); bursa sub-terminal 58.3 μm (48-66); cola con proyección con terminación en punta (Figura 2F). El Cuadro 1 muestra la identificación morfométrica de la población de O. phyllobia del estado de Guanajuato, México y su comparación con las poblaciones de Ditylenchus phyllobius de Arizona, EE.UU., citado por Nickle (1991) y la población de Texas, EE.UU. Brzeski (1991). Existen diferencias en los índices: a (longitud del cuerpo/el diámetro mayor del cuerpo) y b ́ (longitud del cuerpo/distancia de las glándulas esofágicas al extremo anterior del cuerpo cuando está sobrepuesta) ya que la población mexicana posee valores más altos que los reportados. Los valores morfométricos relativamente más altos puede deberse a que los especímenes analizados no fueron sometidos al efecto de ningún agente químico fijador, y sólo fueron anestesiados en calor.

Figura 2 Imágenes en MEB de Orrina phyllobia; (A) Vista lateral de la cabeza de la hembra; (B) Vista lateral de la cabeza del macho; (C) Vista lateral de la cabeza del juvenil J2; (D) Región labial de la hembra; (E) Campos laterales; (F) Cola del macho.
Cuadro 1 Características morfométricas comparativas de hembras y machos de Orrina phyllobia de la población de Guanajuato, México con especies reportadas por Nickle, (1991) y Brzeski, (1991). Todas las medidas están en μm, media (rango).
| Carácter |
Orrina phyllobia
(Guanajuato, México) |
Ditylenchus phyllobius
(Nickle, 1991) |
Ditylenchus phyllobius
(Brzeski,1991) |
||||
| Hembra | Macho | Hembra | Macho | Hembra | Macho | ||
| n | 20 | 20 | - | - | - | - | |
| L | 696.2 (637-785) | 700 (672-737) | (590-840) | (670-800) | 684 (592-838) | 750 (672-799) | |
| a | 31.6 (22.5-39) | 32.8 (30-36.8) | (20-32) | (27-34) | 25 (20-32) | 29 (27-34) | |
| b | 7.8 (7.5-10.1) | 8.6 (8.1-9) | (7.4-10.5) | (7.0-9.7) | 8.6 (7.4-10.5) | 8.5 (7-9.7) | |
| b' | 7.7(6.6-9.8) | 8.4 (7.9-8.7) | (4.1 -6.5) | (4.7-7.5) | 5.5 (4.1-6.5) | 5.8 (4.7-7.5) | |
| c | 18.3 (17-19.6) | 20.4 (19.7-22.4) | (11.4-17.6) | (16.3-19.2) | 14.6 (11.4-17.6) | 18.1 (16.3-19.2) | |
| c' | 4.4 (3.5-5.6) | 4 (3.7-4.5) | (2.9-4.5) | (2.5-3.1) | 3.7 (2.9-4.5) | 2.8 (2.5-3.1) | |
| Estilete | 9 (8-9) | 9 (8-9) | (9-10) | (9-10) | (9-11) | (9-10) | |
| Espícula | - | 19.8 (18-21) | - | - | - | 20.5 (19-22) | |
| Bursa | - | 58.3 (48-66) | - | - | - | - | |
| Gubernáculo | - | - | - | - | - | - | |
| V | 79.2 (78-81) | - | (78-85) | - | 81 (78-84) | - | |
| V' | 89.5 (85-92) | - | (85-89) | - | 87 (85-89) | - | |
L=longitud del cuerpo; a=longitud del cuerpo/anchura máxima del cuerpo; b=longitud del cuerpo/longitud del esófago; b'= longitud del cuerpo/distancia de la región anterior al post corpus; c= longitud/longitud de la cola; c'= longitud de la cola/ancho del cuerpo a nivel del ano; V= distancia región anterior-vulva; V'= distancia de la región anterior a la vulva/región anterior al ano X 100
La amplificación de las regiones ITS1-5.8SITS2 del ADNr y segmentos de expansión D2-D3 del gen 28S produjeron un fragmento de 850 pb y 900 pb, respectivamente basado en electroforesis en gel de agarosa (Figuras 3 y 4). La búsqueda de homología por BLAST con secuencias del NBCI para la región ITS1-5.8S-ITS2 del ADNr muestra un 99 % de similitud (E=0.0) con D. phyllobius (AF363112) lo que confirma la identidad de las poblaciones mexicanas. Respecto a los segmentos de expansión D2-D3 del gen 28S, el número de secuencias genéticas de este grupo de nematodos anguinidos en la base de datos del NCBI es muy limitado sin embargo, con los pocos que existen se obtuvo un blasting de 78 % de similitud con secuencias de Ditylenchus gallaeformans (KF494346), un nematodo que ocasiona síntomas similares en maleza de zonas tropicales del género Miconia en Brasil y Costa Rica (Oliveira et al., 2012). Si bien D. gallaeformans fue la especie que más similitud tuvo con las secuencias de las poblaciones mexicanas de O. phyllobia, el porcentaje de similitud fue muy bajo y la única relación de parentesco que tienen es que ambos, pertenecen a la familia Anguinidae y que ocasionan alteraciones en las partes aéreas de sus hospedantes. Las secuencias de ambos marcadores moleculares del ADNr se depositaron en la base de datos genético del NCBI (GenBank) con números de acceso KT192615 y KT192616 para ITS1-5.8S-ITS2 y KT192617 y KT192618 para los segmentos de expansión D2-D3 del gen 28S como aportación al conocimiento de las poblaciones mexicanas de este grupo de nematodos. La reconstrucción filogenética bayesiana a partir de la región ITS1-5.8S-ITS2 de un alineamiento múltiple que incluyó 37 secuencias ubicó a la población mexicana de O. phyllobia en un clado independiente con un valor de soporte de 96.96 % junto con la población americana de D. phyllobius (=O. phyllobia) AF363112 (Figura 5). Esta diferencia se debe en parte a la variabilidad intraespecífica existente entre poblaciones de la misma especie y por otro lado a la región secuenciada del ADNr de O. phyllobia de E.E.U.U. ya fue parcialmente diferente pues sólo incluyó un fragmento parcial del gen 18S, la región del ITS1 y el gen 5.8S. En lo que respecta a la inferencia de los segmentos de expansión D2-D3 del gen 28S, el alineamiento múltiple incluyó 21 secuencias. El conjunto de datos incluyó 807 caracteres nucleotídicos. Debido a que no existen secuencias de este marcador molecular en GenBank para este nematodo, la población mexicana de O. phyllobia y las secuencias del GenBank de otras especies de anguinidos formaron agrupaciones independientes con un valores altos de soporte (Figura 6). Es importante destacar que con ambos marcadores moleculares, O. phyllobia se concentra en un clado monofiletico (alto valor de soporte 100 %) y está relacionada filogenéticamente con Ditylenchus destructor y D. arachidis (ITS1-5.8S-ITS2) y con D. gallaeformans (D2-D3) todos, miembros de la familia Anguinidae (Figura 5 y 6), resultados que coinciden con los reportados por Douda et al., 2013.

Figura 3 Productos de PCR usando los iniciadores AB28 y TW81; 1-2: Control (+) Ditylenchus dipsaci; 3-4 control (-) Agua destilada estéril; 5-7 población de O. phyllobia. M: Marcador molecular 100 pb.

Figura 4 Productos de PCR usando los iniciadores D2A y D3B; 1-2: Control (+) Ditylenchus dipsaci; 3-4 control (-) Agua destilada estéril; 5-8 población de O. phyllobia. M: Marcador molecular 100 pb.
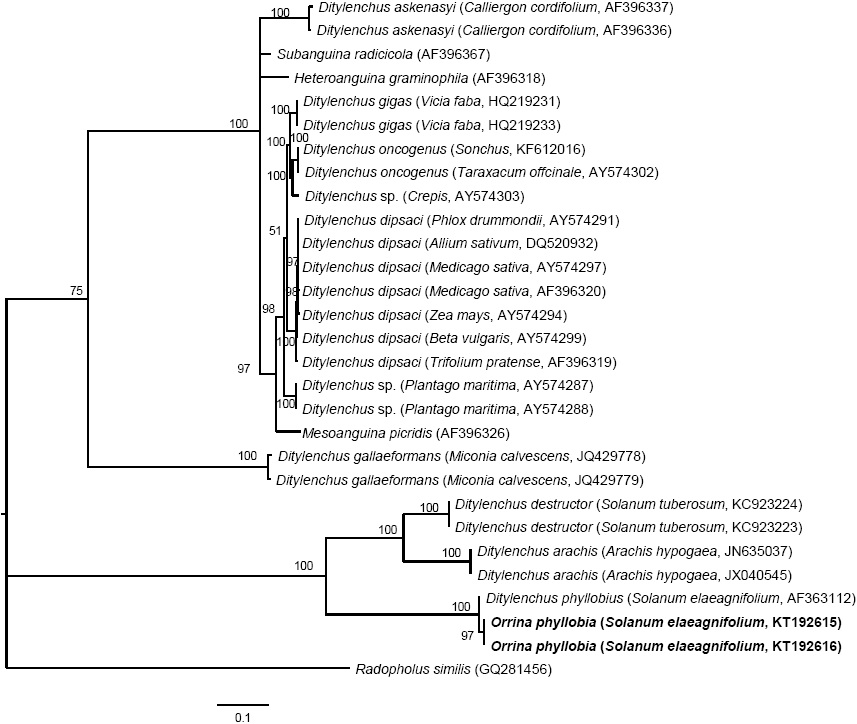
Figura 5 Inferencia bayesiana de nematodos afectando S. elaeagnifolium. Árbol consenso de probabilidades superiores al 50 % de la alineación de secuencias de ITS1-5.8S-ITS2 generados a partir del modelo complejo: GTR + G con secuencias de anguinidos depositadas en el GenBank.

Figura 6 Inferencia bayesiana de secuencias de nematodos afectando S. elaeagnifolium. Árbol consenso de probabilidades superiores al 50 % de la alineación de los segmentos de expansión D2-D3 generados a partir del modelo complejo: GTR + G con secuencias de anguinidos depositadas en el GenBank.
Las secuencias de la región ITS1-5.8S-ITS2 del ADNr permitieron confirmar genéticamente la identidad de poblaciones mexicanas de O. phyllobia como el agente causal del agallamiento foliar de S. elaeagnifolium, utilizando como referencia la secuencia de D. phyllobius de EE.UU. (Powers et al., 2001).
Si bien existe evidencia de O. phyllobia como un prospecto agente de control biológico para el trompillo, también existe la posibilidad de que pueda afectar algunas otras especies de solanáceas nativas y algunas cultivadas como la berenjena Solanum melongena (Field et al., 2009). Se sugiere para estudios futuros determinar cuál es el comportamiento de O. phyllobia al ser inoculado en diferentes especies cultivadas para determinar la gama de hospedantes. El empleo de la taxonomía con un enfoque integrativo para la identificación de nematodos en S. elaeagnifolium fue de gran importancia ya que fortaleció las bondades de la taxonomía tradicional. El estudio morfotaxonómico y molecular permitió determinar la presencia del ne-matodo foliar Orrina phyllobia atacando trompillo Solanum elaeagnifolium; maleza de importancia económica y ruderal en el municipio de San Luis de la Paz, Guanajuato, México.











 texto em
texto em 


