Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista mexicana de fitopatología
versão On-line ISSN 2007-8080versão impressa ISSN 0185-3309
Rev. mex. fitopatol vol.34 no.1 Texcoco 2016
https://doi.org/10.18781/R.MEX.FIT.1507-1
Notas fitopatológicas
Identificación y actividad antagónica in vitro de aislados de Bacillus spp. y Trichoderma spp. contra hongos fitopatógenos comunes
1Centro de Investigación en Alimentación y Desarrollo, A.C. Campus Cuauhtémoc, Cuauhtémoc, Chihuahua, México
2Facultad de Ciencias Agrotecnológicas, Universidad Autónoma de Chihuahua, Campus Cuauhtémoc, Cuautémoc, Chihuahua, México
3Centro de Investigación y de Estudios Avanzados (CINVESTAV, Campus Irapuato) del Instituto Politécnico Nacional, Irapuato, Guanajuato, México
Muchas enfermedades en cultivos causadas por hongos patógenos representan un problema económico debido a las pérdidas que éstas causan. Del mismo modo microorganismos antagonistas tales como Trichoderma spp. y Bacillus spp., entre otros, han sido usados como agentes de control biológico como una alternativa eficiente para reducir el uso de fungicidas químicos en el control de enfermedades de plantas. El objetivo de este estudio fue identificar taxas de microorganismos patógenos y antagonistas basado en sus caracteres moleculares y evaluar la actividad antagónica in vitro de dos aislados de Bacillus y dos aislados de Trichoderma contra cinco hongos patogénicos comunes: Fusarium oxysporum, Botrytis cinerea, Penicillium crustosum, Aspergillus nidulans y Alternaria alternata. Para la identificación molecular, los nueve microorganismos fueron usados para la extracción del ADN genómico y amplificación del gen del ADN ribosomal 18S y del Espaciador Transcrito Interno con los iniciadores ITS (ITS5 e ITS4) para los hongos y el gen del 16S del ADNr, usando los iniciadores universales EU(F) y EU(R) para bacterias. Ambos aislados de T. asperellum mostraron una actividad antagonista significativa contra los hongos fitopatógenos probados, donde los porcentajes de inhibición del crecimiento radial (PICR) de las colonias de hongos fitopatógenos fluctuó de 43 a 71 %, mientras los PICR inducidos por Bacillus fueron significativos, con valores de hasta 69 % cuando se probaron in vitro contra B. cinerea, mientras que el efecto más bajo se observó con F. oxysporum, P. crustosum y A. nidulans. Ambas especies de Bacillus, indujeron la presencia de un halo de inhibición, con valores de 5 y 11 mm, cuando se probaron in vitro contra F. oxysporum y B. cinerea, respectivamente. Los efectos antagonistas de los aislados de bacterias y hongos, muestran que estos microorganismos pueden ser usados como agentes de control biológico de varios hongos fitopatógenos de cultivos.
Palabras clave: Bacillus methylotrophicus; Bacillus amyloliquefaciens; Trichoderma asperellum; fitopatógenos; región ITS; identificación molecular
Several diseases in crop plants caused by pathogenic fungi represent an economic problem due the losses they cause. Likewise, antagonistic microorganisms have been used as biological control agents such as Trichoderma spp. and Bacillus spp., among others as an efficient alternative to reduce the use of chemical fungicides in the control of crop diseases. The aim of this study was to identify the taxa of pathogens and antagonists microorganisms based on their molecular characters and to evaluate the antagonistic activity in vitro of two Bacillus isolates and two Trichoderma isolates, against five common pathogenic fungi: Fusarium oxysporum, Botrytis cinerea, Penicillium crustosum, Aspergillus nidulans, and Alternaria alternata. The nine microorganisms were used for the gDNA extraction and to amplify the 18S ribosomal DNA gene and the Internal Transcribed Spacer with the ITS primers ITS5 and ITS4 of fungi and gene of the 16S of the rDNA, using the universal primers EU(F) and EU(R) for bacteria, for molecular identification. Both T. asperellum isolates showed a significant antagonistic activity against the phytopathogenic fungi tested where the percentage of radial growth inhibition (PRGI) of the fungi colonies ranged from 43 to 71 %, whereas the PRGI induced by Bacillus species, were significant, with values up to 69 % when tested in vitro against B. cinerea, while the lowest effect was observed with F. oxysporum, P. crustosum, and A. nidulans. Both Bacillus species induced the presence of an inhibition halo, with values of 5 and 11 mm, when tested in vitro against F. oxysporum, and B. cinerea, respectively. The antagonistic effect of the bacterial and fungal isolates showed that these microorganisms could be used as biological control agents of several phytopathogenic fungi of crop plants.
Key words: Bacillus methylotrophicus; Bacillus amyloliquefaciens; Trichoderma asperellum; phytopathogens; ITS region; molecular identification
Una gran diversidad de microorganismos patogénicos tales como Oomicetos, hongos, bacterias, nematodos, entre otros, pueden causar enfermedades en cultivos, siendo un serio problema ocasionando pérdidas económicas en todo el mundo. Por consiguiente, los problemas y limitaciones del control de enfermedades en plantas, ha sido manejado mediante el uso de plaguicidas químicos. Además, su uso inapropiado se ha relacionado con varios problemas tales como contaminación ambiental, residuos en alimentos, también el desarrollo de resistencia a los fungicidas sintéticos por los patógenos y la pérdida de la biodiversidad natural en los agroecosistemas (Harris et al., 2001; Fisher et al., 2012). Ésta problemática ha conducido a la búsqueda de nuevas alternativas para el manejo de enfermedades de plantas que sean ecológica y económicamente viables, con un impacto mínimo en el ambiente.
Una alternativa al control químico de hongos fitopatogénicos es su supresión por el uso de agentes de control biológico con énfasis en el uso de antagonistas nativos (Zeilinger and Omann, 2007; Vinale et al., 2008), siendo una herramienta que no afecta al ambiente. Se ha considerado la posibilidad de controlar patógenos por el uso de varios microorganismos con actividad antagónica tales como hongos, bacterias y actinomicetos. Sin embargo, los dos primeros han sido los más usados, principalmente los géneros Trichoderma y Bacillus. Bacillus subtilis ha sido la especie de bacterias más usada, sin embargo, existen otras especies de antagonistas tales como B. methylotrophicus y B. amyloliquefaciens, las cuales han sido pobremente estudiadas, pero han sido consideradas como colonizadores eficientes que se diseminan ampliamente en diferentes hábitats, debido a su capacidad de formar esporas, su crecimiento en un amplio rango de temperaturas, producción de antibióticos que inhiben el crecimiento de fitopatógenos, además de ser promotores del crecimiento vegetal (Leelasuphakul et al., 2008). Ambos antagonistas, hongos y bacterias, constituyen la mayoría de la población microbiana en el suelo, que ha sido el principal reservorio natural, donde el equilibrio entre la diversidad de microorganismos es dinámico, contribuyendo en la reducción de enfermedades en plantas cultivadas y no cultivadas (Gajera and Vakharia, 2010; Singh and Islam, 2010). Este balance ecológico esta apoyado por diferentes interacciones complejas para inhibir el crecimiento de hongos fitopatogénicos a través de varios modos de acción tales como: competencia directa por nutrientes y espacio, micoparasitísmo directo, producción de antibióticos por la excreción de compuestos tóxicos como citoquininas y auxinas, inactivación de enzimas patógénicas, además pueden ser compatibles con las prácticas de manejo, incluyendo el uso de plaguicidas, producción de compuestos volátiles orgánicos e inorgánicos. Además de su rápido crecimiento, son fácilmente adaptables, inducen resistencia sistémica en las plantas hospederas hacia los patógenos y contribuyen a una mejor asimilación de nutrientes (Ezziyyani et al., 2004; Harman, 2006; Leelasuphakul et al., 2008; Vinale et al., 2008; Arguelles, 2009; Correa et al., 2009).
Sin embargo, las poblaciones de microorganismos antagonistas han sido reducidas por actividades antropogénicas, especialmente por los plaguicidas químicos usados en el manejo de cultivos (Gajera and Vakharia, 2010). Los agentes de control microbiano se utilizan comúnmente para el manejo de enfermedades bacterianas y fúngicas en cultivos y estos han comenzado a ser ampliamente utilizados, con una posible reducción en el uso de plaguicidas sintéticos. Estos agentes microbianos son identificados generalmente basado en sus caractéres morfológicos microscópicos. Aunque los microorganismos pueden ser identificados basados en sus caracteres morfológicos, las técnicas moleculares son hoy en día las más ampliamente utilizadas y en general son herramientas más aceptadas para la identificación que ofrecen información rápida y fiable para el estudio de identidad (Gajera and Vakharia, 2010). Por lo tanto, los objetivos de este estudio fueron identificar los taxones y evaluar la actividad antagónica in vitro de dos aislados de Bacillus y dos aislados de Trichoderma asperellum, contra cinco hongos fitopatogénicos comunes.
Sitio experimental y muestras biológicas
El experimento se realizó in vitro, en el Laboratorio de Patología Vegetal y Control Biológico del Centro de Investigación en Alimentación y Desarrollo, A.C., Campus Cuauhtémoc, Chihuahua, México. Los microorganismos utilizados, se obtuvieron del cepario de microorganismos del mismo Campus. Los patógenos se aislaron de frutos y tejido enfermo de árboles de manzano y de muestras de suelo cercano a la rizosfera, recolectados de diferentes huertos de manzano y regiones geográficas de Chihuahua, México, en diferentes periodos. Así mismo, los antagonistas se obtuvieron de muestras de suelo cercano a la rizosfera de árboles de manzano en las mismas regiones geográficas y fueron seleccionados por su capacidad antagónica in vitro contra Oomicetos (Rios-Velasco et al., 2014). Los hongos fueron activados en papa-dextrosa-agar (PDA, BioxonMR Becton Dickinson de México, S.A. de C.V.) e incubados a 26 °C en una cámara ambiental (Precision Scientific, Model 6LM, Winchester, VA, USA), por 5-10 d.
Identificación morfológica y molecular
Nueve microorganismos: cinco aislados de hongos fitopatógenicos, dos de hongos antagonistas y dos de bacterias antagonistas, usados en este estudio, fueron identificados hasta género con la ayuda de claves taxonómicas, de acuerdo a sus caracteres morfológicos, así como observados en un microscopio óptico (Carl Zeiss, Jena, Germany) (Dugan, 2006; Watanabe, 2010). Subsecuentemente, fueron identificados molecularmente en el Centro de Investigación y de Estudios Avanzados del Instituto Politécnico Nacional, CINVESTAV, Campus Irapuato, Guanajuato, México.
Para identificar los aislados de hongos por sus caracteres moleculares, se extrajo su ADN genómico (ADNg). Por lo tanto, un explante de cada hongo purificado se colocó y cultivó en una caja de Petri con PDA cubierto con celofán estéril para facilitar la recolecta de la biomasa fúngica e incubado a 26 °C por 7 d. El micelio se recolectó en un mortero de porcelana, junto con un amortiguador [200 mM Tris-HCl a pH = 8, 250 Mm NaCl, 25 mM EDTA, 0.5 % SDS] para la extracción de ADNg de hongos filamentosos a 70 °C, congelado con nitrógeno líquido y macerado, siguiendo el protocolo descrito por Raeder and Broda (1985). El ADNg obtenido de los hongos, fue examinado por electroforesis en un gel de agarosa al 1 %, el cual se utilizó para amplificar el gen 18S del ADN ribosomal (ADNr) y el Espaciador Transcrito Interno (ITS, por sus siglas en inglés), usando los iniciadores universales ITS5 (5'-GGAAGTAAAAGTCGTAACAAGG-3') e ITS4 (5'-TCCTCCGCTTATTGATATGC-3'), donde los fragmentos esperados fueron de aproximadamente 600 a 710 pb (White et al., 1990). Las condiciones de amplificación fueron divididas en varios pasos: un paso inicial de desnaturalización a 94 °C por 5 min, un segundo paso comprendiendo 30 ciclos, incluyendo una desnaturalización a 94 °C por 30 s, un paso de alineamiento a 60 °C por 30 s, un paso de extensión a 72 °C por 45 s y cuando se completaron los ciclos, una extensión final a 72 °C por 10 min.
Para las bacterias antagonistas, se tomó un explante de la colonia purificada de 72 h de crecimiento, para la extracción del ADNg descrita por Heddi et al. (1999). El ADNg obtenido, se examinó por electroforesis en un gel de agarosa al 1 %, el cual se utilizó para amplificar el gen del 16S del ADNr por PCR, usando los iniciadores universales EU(F) (5'-AGAGTTTGATCATGGCTCAG-3') y EU(R) (5'-TACCTTGTTACGACTTCACC-3'). Las condiciones de amplificación se dividieron en varios pasos: un paso inicial de desnaturalización a 94 °C por 5 min, un segundo paso comprenidendo 35 ciclos incluyendo una desnaturalización a 94 °C por 30 s, un paso de alineamiento a 53 °C por 45 s, un paso de extensión a 72 °C por 45 s y cuando los ciclos se completaron, una extensión final a 72 °C por 10 min (Heddi et al., 1999).
Los productos de PCR de ambos taxones de microorganismos, se examinaron por electroforesis en un gel de agarosa al 1 %. Subsecuentemente, fueron secuenciados por la compañía Macrogen (Rockville, Maryland, EUA). Las secuencias obtenidas se compararon con la base de datos del NCBI usando el algoritmo de BLAST (Altschul et al., 1990), para verificar el porcentaje de identidad y que correspondíeran a las especies identificadas. Las secuencias de nucleótidos obtenidas, no han sido depositadas en el NCBI.
Preparación del inóculo
El inóculo se preparó mediante una suspensión de esporas fúngicas y bacterias, respectivamente, en agua peptonada estéril que contenía cloruro de sodio al 0.85 % y peptona al 0.1 % en microtubos de 2 mL. La concentración de esporas se estimó usando una cámara de Neubauer (Neubauer Improved bright-line, Marienfield, Alemania) y las bacterias se cuantificaron por la técnica de diluciones seriadas y conteo de colonias en placa. Ambas suspensiones se almacenaron a 0 °C hasta su uso. La concentración de la suspensiones donde los círculos de papel filtro fueron sumergidas para la evaluación de la actividad antagónica in vitro, fluctuó de 1.0 × 107 a 9.5 × 107 esporas de hongos o UFC-bacteria por mililitro.
Evaluación del antagonismo In vitro
Las confrontaciones in vitro de T. asperellum contra hongos fitopatogénicos, se realizaron usando la técnica de cultivo dual (cajas de Petri, 90 × 15 mm) con PDA, que consistió en colocar en un lado de la caja, un círculo de papel filtro de 6 mm de diámetro, impregnado con una suspensión de conidias y micelio del inóculo del patógeno y en el lado opuesto de la caja de Petri, se colocó otro círculo de papel filtro con el micelio y/o conidias del antagonista. Se realizaron diez repeticiones por triplicado para cada evaluación. También se consideraron diez testigos (cajas de Petri) por triplicado, tanto de los fitopatógenos como de los antagonistas, crecidos por separado. Las cajas de Petri inoculadas, se incubaron a 28 °C por 15 d en una cámara ambiental (Precision Scientific, Model 6LM, Winchester, EUA). El crecimiento radial de las colonias de hongos se midió cada 24 h para ambos microorganismos patógenos y antagonistas. El halo de inhibición, entre las colonias de fitopatógenos y antagonistas en confrontación, se midió hacia los puntos cardinales de la caja Petri a los 14 d post-incubación (Aquino-Martínez et al., 2008).
Se evaluó el tiempo cuando las colonias patógeno-antagonista entraron en contacto, la inhibición del crecimiento radial de la colonia y el tipo de antagonismo de acuerdo con la escala de Bell (Bell et al., 1982). Donde la actividad antagónica se midió en cinco niveles, siendo el nivel uno de la escala cuando el antagonista sobrecreció completamente al patógeno y llenó por completo la superficie del medio de cultivo, y el nivel cinco fue cuando el patógeno sobrecreció por completo al antagonista y llenó por completo la superficie del medio de cultivo. El antagonismo de T. asperellum se evaluó registrando las siguientes variables: crecimiento radial del antagonista (CRA), crecimiento radial del patógeno (CRP) y el porcentaje de inhibición del crecimiento radial (PICR). El PICR se determinó al sexto día de post-incubación usando la fórmula propuesta por Ezziyyani et al. (2004), PRGI = [(R1 - R2) / R1] × 100, donde R1 fue el crecimiento radial de la colonia testigo (patógeno) y R2 el crecimiento radial de la colonia del patógeno en la confrontación in vitro.
La confrontación in vitro de las especies de Bacillus contra patógenos se realizó colocando un círculo de papel filtro de 6 mm de diámetro del patógeno en el centro de la caja de Petri, que contenía PDA, mientras la bacteria se inoculó en los puntos cardinales de la caja de Petri, usando círculos de papel filtro de 6 mm de diámetro con el inóculo. Se realizaron diez repeticiones (cajas de Petri) por triplicado, para cada evaluación in vitro bacteria-hongo. Cada uno de los los tratamientos testigo de hongos fitopatógenos se sembraron por separado, e incubados a 28 °C por 14 d. Se midió diariamente, el crecimiento radial del patógeno en la confrontación, hacia la colonia del antagonista y en los testigos se midió el crecimiento radial desde el centro de la caja de Petri hacia los puntos cardinales.
Análisis estadístico
Ambos bioensayos se realizaron por triplicado, con diez cajas de Petri por cada repetición, para cada evaluación in vitro de bacteria-hongo, usando un diseño completamente al azar, con cinco tratamientos: dos aislados de hongos antagonistas y dos aislados de bacterias antagonistas, confrontados contra cinco patógenos, además de 1 control, donde cada tratamiento fue un microorganismo antagonista con 30 cajas de Petri (ej., un total de 600). Los datos del PICR y halo de inhibición se analizaron usando el Statistical Analysis System versión 9.0 (SAS, 2002), para el análisis de varianza (ANVA) y las medias fueron separadas usando la prueba de Tukey (P ≤ 0.05).
Patógenos y antagonistas identificados
Los hongos fitopatógenos identificados morfológicamente y de las secuencias de los productos de PCR fueron: Fusarium oxysporum cepa LCF32, Botrytis cinerea cepa G409, Penicillium crustosum cepa 06CK005, Aspergillus nidulans cepa UOA/HCPF, Alternaria alternata cepa HMY 2-1, los cuales tuvieron un 99-100 % de identidad, excepto P. crustosum, que mostró un 78 % de identidad, y una máxima similitud con las máximas puntuaciones y las claves taxonómicas, correspondiendo a cada cepa y de acuerdo con las secuencias disponibles en la base de datos del GenBank (NCBI), obtenidas por el algoritmo de BLAST (Altschul et al., 1990). Así mismo, los datos de la secuenciación mostraron que los aislados de los hongos y bacterias antagonistas identificados fueron: Trichoderma asperellum TC1 cepa LAHC-FFPK-M16- y Trichoderma asperellum TC2 cepa BHU-BOT-RYRL16, Bacillus methylotrophicus cepa IS04, Bacillus amyloliquefaciens cepa Abk-2, tuvieron 99-100 % de identidad y las máximas puntuaciones, al ser comparadas con las secuencias del GenBank, obtenidas por el algoritmo de BLAST en la base de datos del NCBI (Altschul et al., 1990).
Actividad antagónica
I in vitro . El crecimiento radial de los cinco hongos fitopatogénicos evaluados, se redujo significativamente al ser evaluado in vitro contra T. asperellum TC1 y TC2, donde ambos hongos antagonistas mostraron una eficiencia significativa al inhibir el crecimiento de los hongos fitopatógenos a los 6-8 d post-evaluación (Cuadro 1, Figura 1). Las cajas de Petri de los testigos de ambos aislados de T. asperellum, fueron completamente cubiertas por el crecimiento de los hongos al tercer día de post-inoculación. Mientras tanto, al sexto día post-inoculación, ambos aislados TCI y TC2, entraron en contacto con F. oxysporum, B. cinerea, P. crustosum, and A. Alternata, excepto con A. nidulans. Los valores más altos de PICR se mostraron en B. fabae con ambos aislados de T. asperellum TC1 y TC2 con 71.6 y 69.8 %, respectivamente (Cuadro 1), mientras que los valores más bajos se observaron en P. crustosum y F. oxysporum con 52.3 y 43.3 %, respectivamente (Cuadro 1).
Cuadro 1 Crecimiento radial in vitro de hongos fitopatógenos, al ser inhibidos por especies de Bacillus a los 14 d post-inoculación y por especies de Trichoderma a los seis días de incubación.
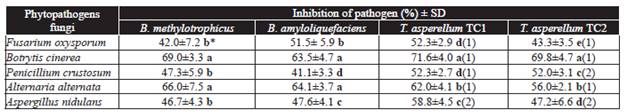
Las letras en la misma columna, indican diferencias significativas en los tratamientos donde las medias seguidas por la misma letra, no son significativamente diferentes (ANVA) de acuerdo con la prueba de Tukey (P ≤ 0.05).
La actividad antagónica se midió de acuerdo con la escala de Bell et al. (1982) donde: (1) el antagonista (Trichoderma) sobrecrece completamente la colonia del patógeno y cubre completamente la superficie del medio, y (2) el antagonista (Trichoderma) sobrecrece dos tercios de la superficie del medio. DE = Desviación estándar.
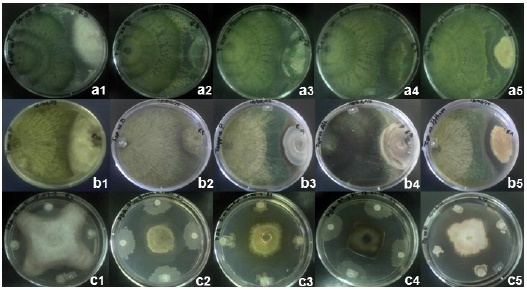
Figura 1 Micoparasitismo de Trichoderma asperellum TC1 (a1-a5) y Trichoderma asperellum TC2 (b1-b5), y halos de inhibición de Bacillus amyloliquefaciens (c1-c5) al ser confrontado in vitro contra Fusarium oxysporum, Botrytis cinerea, Penicillium crustosum, Alternaria alternata y Aspergillus nidulans, respectivamente, a los 14 d post-inoculación.
En las evaluaciones in vitro contra A. nidulans, se observaron halos de inhibición de 2.6 y 4.5 mm con los aislados de T. asperellum TC1 y TC2, respectivamente (Cuadro 2, Figura 1), esto puede deberse a la producción de compuestos volátiles que inhibieron el crecimiento de Trichoderma spp., aún así, ambos aislados fueron eficientes en reducir el crecimiento de los hongos fitopatógenos al sexto día de post-inoculación (Duffy et al., 2003; Harman, 2006; Zeilinger and Omann, 2007; Vinale et al., 2008). Al respecto, Cooney et al. (2001), mencionan que T. harzianum produce el antibiótico 6-pentil-α-pirone, el cual tiene un efecto dual en la inhibición del crecimiento del patógeno y también puede regular los genes por la biosíntesis de tricotecenos y micotoxinas con actividad antimicrobiana de amplio espectro. Asimismo, Gajera and Vakharia (2010) reportaron una alta inhibición del crecimiento de A. niger causada por T. viride y T. harzianum durante un estudio realizado in vitro. Además de tener la habilidad para producir enzimas que degradan la pared celular tales como la quitinasa, β-1,3 glucanasa y proteasa en el medio de cultivo durante el antagonismo in vitro. Por otra parte, el antagonista o sus antibióticos purificados, pueden reducir la producción de deoxinivalenol por debajo del 80 % y los genes antagónicos están regulados por la presencia del patógeno (Duffy et al., 2003). Por lo tanto, la eficiencia de Trichoderma spp. para inhibir hongos fitopatógenos puede deberse a su competencia por espacio y nutrientes, producción de antibióticos (compuestos volátiles y no volátiles), micoparasitismo e inactivación de las enzimas de los patógenos (Ezziyyani et al., 2004).
Cuadro 2 Halo de inhibición entre las colonias de hongos fitopatógenos y las especies antagónicas de Bacillus a los 14 d post-inoculación y aislados de Trichoderma asperellum a los 6 d de incubación.
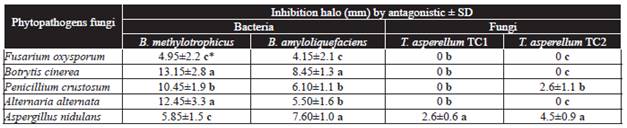
Las letras en la misma columna, indicant diferencias significativas en los tratamientos donde las medias seguidas por la misma letra, no son significativamente diferentes (ANVA) de acuerdo con la prueba de Tukey (P ≤ 0.05). DE = Desviación estándar.
Además, un halo de inhibición se observó en el aislado T. asperellum TC2 cuando se evaluó in vitro contra P. crustosum (Cuadro 2), esto puede ser debido a la producción de patulina, un metabolito secundario producido por este fitopatógeno (Leelasuphakul et al., 2008).
La inhibición del crecimiento de las colonias de hongos patogénicos, inducida por B. methylotrophicus y B. amyloliquefaciens, fue significativamente diferente, donde el mayor efecto se observó en B. cinerea con PICRs de 69 y 63.5 %, respectivamente, A. alternata mostró PICRs de 66 y 64.1 %, respectivamente; donde las menores reducciones del crecimiento se registraron para F. oxysporum, P. crustosum y A. nidulans (Cuadro 1). Estos resultados son similares a los obtenidos por Madhaiyan et al. (2010) y Zhang et al. (2012) quienes encontraron una alta capacidad de algunas cepas de Bacillus de las mismas especies para inhibir el crecimiento de diversos hongos fitopatógenos, lo cual es debido a la producción de compuestos volátiles y antibióticos pertenecientes a la familia de las iturinas y subtilinas, que actuan en la pared celular de los hongos. También, la producción de enzimas hidrofílicas para destriuir los polisacáridos, ácidos nucléicos y lípidos, los cuales son usados como fuente de energía y que podrían estar involucrados.
Bacillus amyloliquefaciens y B. methylotrophicus indujeron un halo de inhibición de la colonia del patógeno a los 14 d post-incubación (Cuadro 2). Las medias por antagonista, mostraron que B. methylotrophicus tuvo mayor actividad antagónica que B. amyloliquefaciens, ya que el promedio de los halos de inhibición fue significativo, de 4.95 a 13.15 mm y 4.15 a 8.45 mm, respectivamente, en las colonias de hongos fitopatógenos (Cuadro 2). Los mayores halos de inhibición producidos por B. amyloliquefaciens y B. methylotrophicus se observaron con B. cinerea con 8.45 y 13.25 mm, respectivamente (Cuadro 2). Bacillus methylotrophicus mostró los mayores efectos antagónicos contra fitopatógenos, que puede deberse a la producción de antibióticos y/o compuestos orgánicos volátiles como cianuro de hidrógeno, el cual inhibe el crecimiento de hongos fitopatógenos y ejerce efectos nocivos en el crecimiento in vitro de varios fitopatógenos, estos compuestos pueden tener efectos directos e indirectos en la actividad de enzimas fúngicas específicas (Wheatley, 2002; Duffy et al., 2003; Correa et al., 2009; Zhang et al., 2012), así como también lo reportado por Madhaiyan et al. (2010) y Zhang et al. (2012), quienes encontraron que las cepas de B. methylotrophicus tienen una alta actividad antagónica contra una gran diversidad de hongos fitopatógenos.
De acuerdo con Guillén-Cruz et al. (2006), B. amyloliquefaciens fue el más eficiente contra Phytophthora spp. al ser evaluado in vitro, además de tener un efecto antagonista e inducir el crecimiento vegetal. Al respecto, Souto et al. (2004) mencionan que la cepa de B. amyloliquefaciens adiciona péptidos y lipopéptidos al medio de cultivo, tales como fungicina, iturina, bacilomicina, entre otros, teniendo propiedades antifúngicas al ser confrontada in vitro contra hongos fitopatógenos tales como Fusarium, Rhizoctonia y Sclerotinia.
Ambos antagonistas, bacterias y hongos, inhibieron significativamente el crecimiento in vitro de F. oxysporum, B. cinerea, P. crustosum, A. alternata, y A. nidulans, donde B. methylotrophicus y T. asperellum TC1, fueron los más eficientes, lo cual sugiere la presencia de compuestos bioactivos. Por lo tanto, pueden ser considerados como agentes de control biológico viables. Por lo que, es importante llevar a cabo más estudios para identificar esos compuestos bioactivos y la forma en que suprimen el crecimiento de los patógenos.
Literature cited
Altschul SF, Gish W, Miller W, Myers EW and Lipman DJ. 1990. Basic local alignment search tool. Journal of Molecular Biology 215: 403-410. http://dx.doi.org/10.1016/S0022-2836(05)80360-2. [ Links ]
Aquino-Martínez JG, Vázquez-García LM and Reyes-Reyes BG. 2008. Biocontrol in vitro e in vivo de Fusarium oxysporum Schlecht. f. sp. dianthi (Prill. y Delacr.) Snyder y Hans con hongos antagonistas nativos de la zona florícola de Villa Guerrero, Estado de México. Revista Mexicana de Fitopatología 26: 127-137. Available online: http://www.scielo.org.mx/pdf/rmfi/v26n2/v26n2a5.pdf [ Links ]
Arguelles-Arias A, Ongena M, Halimi B, Lara Y, Brans A, Joris B and Fickers P. 2009. Bacillus amyloliquefaciens GA1 as a source of potent antibiotics and other secondary metabolites for biocontrol of plant pathogens. Microbial Cell Factories 8: 1-12. http://dx.doi.org/10.1186/1475-2859-8-63. [ Links ]
Bell D, Well H and Markham C. 1982. In vitro antagonism of Trichoderma species against six fungal plant pathogens. Phytopathology 72: 379-382. Available online: https://www.apsnet.org/publications/phytopathology/backissues/Documents/1982Articles/Phyto72n04_379.PDF. [ Links ]
Cooney JM, Lauren DR and di-Menna M.E. 2001. Impact of competitive fungi on trichothecene production by Fusarium graminearum. Journal of Agricultural and Food Chemistry 49: 522-526. http://dx.doi.org/10.1021/jf0006372. [ Links ]
Correa OS, Montecchia MS, Berti MF, Fernández-Ferrari M.C, Pucheu NL, Kerber NL and García AF. 2009. Bacillus amyloliquefaciens BNM122, a potential microbial biocontrol agent applied on soybean seeds, causes a minor impact on rhizosphere and soil microbial communities. Applied Soil Ecology 41: 185-194. http://dx.doi.org/10.1016/j.apsoil.2008.10.007. [ Links ]
Duffy B, Schouten A and Raaijmakers JM. 2003. Pathogen self-defense: Mechanism to counteract microbial antagonism. Annual Review Phytopathology 41: 501-538. http://dx.doi.org/10.1146/annurev.phyto.41.052002.095606. [ Links ]
Dugan FM. 2006. The Identification of fungi. An illustrated introduction with keys, glossary, and guide to literature. The American Phytopathological Society. U.S Department of Agriculture, Agricultural Research Service, Washington State University, Pullman. APS Press. USA. St. Paul, Minnesota USA. 176p. [ Links ]
Ezziyyani M, Pérez-Sánchez C, Requena ME, Rubio L and Candela-Castillo ME. 2004. Biocontrol por Streptomyces rochei -Ziyani-, de la podredumbre del pimiento (Capsicum annuum L.) causada por Phytophthora capsici. Anales de Biología 26: 69-78. Available online: https://www.um.es/analesdebiologia/numeros/26/PDF/08-BIOCONTROL.pdf [ Links ]
Fisher MC, Henk DA, Briggs CJ, Brownstein JS, Madoff LC, McGraw SL and Gurr SJ. 2012. Emerging fungal threats to animal, plant and ecosystem health. Nature 484: 186-194. http://dx.doi.org/10.1038/nature10947. [ Links ]
Gajera HP and Vakharia DN. 2010. Molecular and biochemical characterization of Trichoderma isolates inhibiting a phytopathogenic fungi Aspergillus niger Van Tieghem. Physiological and Molecular Plant Pathology 74: 274-282. http://dx.doi.org/10.1016/j.pmpp.2010.04.005. [ Links ]
Guillén-Cruz R, Hernández-Castillo FD, Gallegos-Morales G, Rodríguez-Herrera R, Aguilar-González CN, Padrón-Corral E and Reyes-Valdés MH. 2006. Bacillus spp. como biocontrol en un suelo infestado con Fusarium spp., Rhizoctonia solani Kühn y Phytophthora capsici Leonian y su efecto en el desarrollo y rendimiento del cultivo de chile (Capsicum annuum L.). Revista Mexicana de Fitopatología 24: 105-114. Available online: http://www.redalyc.org/pdf/612/61224204.pdf [ Links ]
Harman GE. 2006. Overview of mechanisms and uses of Trichoderma spp. Phytopathology 96: 190-194. http://dx.doi.org/10.1094/PHYTO-96-0190. [ Links ]
Harris CA, Renfrew MJ and Woolridge MW. 2001. Assessing the risks of pesticide residues to consumers: recent and future developments. Food Additives & Contaminants 18: 1124-1129. http://dx.doi.org/10.1080/02652030110050122. [ Links ]
Heddi A, Grenier AM, Khatchadourian C, Charles H and Nardon P. 1999. Four intracellular genomes direct weevil biology: Nuclear, mitochondrial, principal endosymbiont and Wolbachia. Proceedings of the National Academy of Sciences of the United States of America 96: 6814-6819. http://dx.doi.org/10.1073/pnas.96.12.6814. [ Links ]
Leelasuphakul W, Hemmanee P and Chuenchitt S. 2008. Growth inhibitory properties of Bacillus subtilis strains and their metabolites against the green mold pathogen (Penicillium digitatum Sacc.) of citrus fruit. Postharvest Biology and Technology 48: 113-121. http://dx.doi.org/10.1016/j.postharvbio.2007.09.024. [ Links ]
Madhaiyan M, Poonguzhali S, Kwon SW and Sa TM. 2010. Bacillus methylotrophicus sp. nov., a methanol-utilizing, plant-growth-promoting bacterium isolated from rice rhizosphere soil. International Journal of Systematic and Evolutionary Microbiology 60: 2490-2495. http://dx.doi.org/10.1099/ijs.0.015487-0. [ Links ]
Raeder U and Broda P. 1985. Rapid preparation of DNA from filamentous fungi. Letters in Applied Microbiology 1: 17-20. http://dx.doi.org/10.1111/j.1472-765X.1985.tb01479.x [ Links ]
Rios-Velasco C, Ruiz-Cisneros MF, Berlanga-Reyes DI, Acosta-Muñiz CH, Ornelas-Paz JJ, Romo-Chacón A, Salas-Marina MA and Ibarra-Rendón JE. 2014. Enfermedades radiculares en árboles de manzano y su manejo en huertos del estado de Chihuahua. Boletín técnico generado en el proyecto 362, CIAD, A.C. Fundación Produce Chihuahua. 54p. Available online: http://siproduce.sifupro.org.mx/seguimiento/archivero/8/2013/anuales/anu_362-25-2014-05-1.pdf [ Links ]
SAS Institute. 2002. SAS User's Guide. Version 9.0. SAS Institute, Cary, NC. [ Links ]
Singh A and Islam MN. 2010. In vitro evaluation of Trichoderma spp. against Phytophthora nicotianae. International Journal of Experimental Agriculture 1: 20-25. Available online: http://ggfjournals.com/assets/uploads/IJEA_20-25_(Nazrul_vi)_Report_soil_biol_final.pdf [ Links ]
Souto GI, Correa OS, Montecchia MS, Kerber NL, Pucheu NL, Bachur M and García AF. 2004. Genetic and functional characterization of a Bacillus sp. strain excreting surfactin and antifungal metabolites partially identified as iturin-like compounds. Journal of Applied Microbiology 97: 1247-1256. http://dx.doi.org/10.1111/j.1365-2672.2004.02408.x [ Links ]
Vinale F, Sivasithamparam K, Ghisalberti EL, Marra R, Barbetti MJ, Li H, Woo SL and Lorito M. 2008. A novel role of Trichoderma secondary metabolites in the interactions with plants. Physiological and Molecular Plant Pathology 72: 80-86. http://dx.doi.org/10.1016/j.pmpp.2008.05.005. [ Links ]
Watanabe T. 2010. Pictorial atlas of soil and seed fungi. Morphologies of cultured fungi and key to species. Third edition. CRS Press, Taylor & Francis Group. Boca Raton London New York. 404 p. [ Links ]
Wheatley R.E. 2002. The consequences of volatile organic compound mediated bacterial and fungal interactions. Antonie Van Leeuwenhoek 81: 357-364. http://dx.doi.org/10.1023/A:1020592802234. [ Links ]
White TJ, Bruns T, Lee S and Taylor J. 1990. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. : Innis M.A., Gelfand, D.H., Sninsky, J.J. and White, T.J. (Eds.), pp. 315-322. PCR protocols: A guide to methods and applications. San Diego: Academic Press. http://dx.doi.org/10.1016/B978-0-12-372180-8.50042-1. [ Links ]
Zeilinger S and Omann M. 2007. Trichoderma biocontrol: Signal transduction pathways involved in host sensing and mycoparasitism. Gene Regulation and Systems Biology 1: 227-234. Available online: http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2759141/pdf/grsb-2007-227.pdf [ Links ]
Zhang QL, Liu Y, Ai GM, Mia LL, Zhen HY and Liu ZP. 2012. The characteristics of a novel heterotrophic nitrification aerobic denitrification, Bacillus methylotrophicus strain L7. Bioresource Technology 108: 35-44. http://dx.doi.org/10.1016/j.biortech.2011.12.139. [ Links ]
Recibido: 01 de Julio de 2015; Aprobado: 17 de Noviembre de 2015











 texto em
texto em 


