Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista mexicana de fitopatología
versão On-line ISSN 2007-8080versão impressa ISSN 0185-3309
Rev. mex. fitopatol vol.29 no.2 Texcoco 2011
Artículos científicos
Identificación de Razas y Biovares de Ralstonia solanacearum Aisladas de Plantas de Tomate
Identification of Races and Biovars of Ralstonia solanacearum Isolated From Tomato Plants
José Misael Perea Soto, Raymundo Saúl García Estrada, Raúl Allende Molar, José Armando Carrillo Fasio, Josefina León Félix, Benigno Valdez Torres y Fabiola Sary Mell López Soto
Centro de Investigación en Alimentación y Desarrollo, A.C., Coordinación Culiacán, km 5.5 Carr. Culiacán-El Dorado, Culiacán, Sinaloa, CP 80110, México. Correspondencia: rsgarcia@ciad.mx
Recibido: Junio 07, 2011
Aceptado: Septiembre 02, 2011
Resumen
La marchitez bacteriana de las solanáceas causada por Ralstonia solanacearum, es una de las enfermedades con daño más devastador en cultivos de importancia como tomate, papa, berenjena, chile y tabaco. El objetivo de este trabajo fue identificar a nivel de raza y biovar, cepas de R. solanacearum aisladas de plantas de tomate, cultivadas en Valles de Sinaloa, San Quintín, B.C. y de Jalisco en México. Se aislaron un total de ocho cepas de R. solanacearum a partir de plantas de tomate afectadas por la bacteria. Todos los aislamientos se realizaron en el medio de cultivo SMSA y fueron identificados como R. solanacearum, mediante estudios morfológicos, fisiológicos y bioquímicos, inmunotiras y confirmación por PCR, utilizando un par de oligonucleótidos universales 759/760, se amplificó un fragmento de 280 pb del ADN ribosomal de R. solanacearum. La identificación de la raza se realizó con el par de primers 630/631 específicos para raza 3. Mediante la producción de ácidos a partir de disacáridos, se determinó que todas las cepas pertenecen al biovar 2 (Bv2).
Palabras clave: Marchitez bacteriana, tomate, Ralstonia solanacearum, Raza 3, Biovar 2.
Abstract
Bacterial wilt caused by Ralstonia solanacearum is one of the most devastating diseases in economically important crops such as tomatoes, potatoes, eggplant, pepper and tobacco. The objective of this study was to identify strains of R. solanacearum isolated from tomato plants grown in Culiacan Valley, San Quintin, B.C. and Jalisco in Mexico to race and biovar levels. Eight strains of R. solanacearum from wilted tomato plants were isolated. Isolations were made on SMSA culture medium, and identified as R. solanacearum using morphological, physiological and biochemical studies, immunoassay strip and polymerase chain reaction (PCR) by using the universal primers 759/760 to amplify a 280 bp fragment from the ribosomal DNA. Race identification was performed with R. solanacearum race 3 specific primers. Based on the ability of isolates to produce acid from a panel of disacarides and sugar alcohols, all isolates belong to biovar 2 (Bv2).
Keywords: Bacterial wilt, tomato, Ralstonia solanacearum, Race 3, Biovar 2.
La marchitez bacteriana de las solanáceas causada por Ralstonia solanacearum, está presente en el mundo en regiones tropicales, subtropicales y templadas, atacando a más de 200 especies de plantas (Hayward, 2000; Elphistone, 2005). La bacteria invade a las plantas hospederas a través de la raíz y coloniza los vasos del xilema en el sistema vascular. Las plantas infectadas muestran disminución de crecimiento, amarillamiento, marchitamiento repentino y mueren rápidamente (Sánchez etal., 2008).
Debido a su diversidad genética, R. solanacearum es una especie heterogénea que se considera como un grupo complejo de aislamientos relacionados (Fegan y Prior, 2005). Por más de cuatro décadas, R. solanacearum se ha subdividido en cinco razas (R1, R2, R3, R4y R5), en base a su rango de hospederos y en seis biovares (Bv1, Bv2, Bv3, Bv4, Bv5 y Bv6), según su capacidad metabólica para la utilización de diversas fuentes de carbono (Hayward, 1964; 1991; Denny y Hayward, 2001). Dentro de este esquema de clasificación, las cepas que normalmente afectan tomate pertenecen a la raza 1 (Bv1, Bv3 y Bv4) y a la raza 3 [Bv2 y Bv2Tropical (Bv2T)]. Las cepas Bv2 son menos activas metabólicamente que las cepas Bv2T (Hayward, 1994; Denny, 2006).
La R3Bv2 de R. solanacearum es una bacteria cuarentenada en Norteamérica y la Unión Europea (Lambert, 2002). La R3Bv2 tiene como principales hospederos papa y tomate, aunque también puede afectar a berenjena, plantas de ornato y malezas (Allen et al., 2001; Janse et al., 2004; Swanson et al., 2005). Las cepas R3Bv2 pueden ser identificadas biológicamente mediante pruebas fisiológicas; tales como las pruebas para determinación de biovar y rango de hospedante, a diferencia de varias cepas de la R1. Además, las cepas R3Bv2 no inducen reacción de hipersensibilidad en tabaco (Robertson et al., 2004; Ji et al., 2007).
Las técnicas de identificación y caracterización molecular de R. solanacearum son útiles para conocer los aspectos evolutivos de este patógeno (Seal y Elphinstone, 1994; Cook y Sequeira, 1994; Villa et al., 2005; Fegan y Prior, 2005). La identificación de la especie de R. solanacearum puede lograrse mediante la técnica de reacción en cadena de la polimerasa (PCR), utilizando un par de oligonucleótidos universales (759 y 760) que amplifica parte del gen IpxC (Opina et al., 1997). La PCR para identificar la Raza 3, se basa en amplificar parte de un profago presente en el genoma de todas las cepas R3Bv2 probadas (Fegan et al., 1998).
Aunque la bacteria R. solanacearum, se encuentra distribuida en diversas partes del mundo y en toda América, en México se ha informado la presencia de la R2Bv1 en plantas de tomate en Guerrero (Dr. Fucikovsky, comunicación personal). Sin embargo, no se ha reportado la presencia de la R3Bv2 de manera oficial afectando cultivos de tomate. Los primeros aislamientos de R. solanacearum R1Bv1 y R3Bv2 (reportados en tabaco y papa, respectivamente), se encuentran registrados en la década de los setenta (Ji et al., 2007; Sánchez et al., 2008); aunque no se reporta el lugar de origen de estos aislamientos.
En el Valle de Culiacán, durante la década pasada, la marchitez bacteriana de las solanáceas no se consideraba de importancia económica (Ramírez y Sainz, 2006), pero si fue en el cultivo de papa en el norte de Sinaloa. En los últimos cinco años, R. solanacearum ha sido causante de pérdidas económicas importantes en diferentes campos agrícolas. Existe poca información en la región sobre este patógeno y no se ha documentado la clasificación taxonómica de las cepas presentes en los cultivos del Valle de Culiacán y de otras regiones productoras de tomate. Debido a que la identificación del patógeno es el primer paso para diseñar estrategias en el manejo de la enfermedad, el objetivo de este estudio fue caracterizar e identificar biológica y genéticamente a nivel de raza y biovar las cepas de R. solanacearum aisladas de plantas de tomate cultivadas en diferentes campos agrícolas del Valle de Culiacán, San Quintín, B.C. y Autlán, Jalisco, mediante pruebas inmunológicas, bioquímicas, moleculares y fisiológicas.
MATERIALES Y MÉTODOS
Aislamiento de cepas de R. solanacearum. Las cepas de R. solanacearum que se utilizaron en esta investigación (Cuadro 1), fueron aisladas a partir de muestras de plantas de tomate con síntomas típicos de la marchitez bacteriana de las solanáceas. Las plantas enfermas colectadas se sometieron a pruebas rápidas de identificación; la primera fue la de flujo bacteriano, la cual consistió en cortar un trozo de tallo y sumergirlo en un vaso de precipitado con agua destilada, después de unos segundos se observó la precipitación del flujo bacteriano en forma de un hilo continuo de color blanco (Denny, 2006); y la segunda técnica consistió en el uso de inmunotiras (Agdia Inc.), específicas para R. solanacearum, como lo recomienda el fabricante (Ji et al., 2007). Las muestras positivas se colocaron en una bolsa de plástico estéril y se transportaron al Laboratorio de Fitopatología del Centro de Investigación en Alimentación y Desarrollo, A.C. (CIADA.C.).

Aislamiento y preservación de R. solanacearum. El aislamiento de la bacteria se realizó a partir de tejido vascular necrosado de la base del tallo de plantas enfermas. Para ello se seleccionaron trozos de tejido infectado y se depositaron en un tubo que contenía 2 mL de agua destilada estéril y se mantuvieron en reposo por 5 min. La suspensión se sembró con un asa bacteriológica esterilizada con fuego, sobre placas de medio de cultivo agar cloruro de tetrazolium (TZC) (Kelman, 1954) y en medio semiselectivo Sur de África modificado por Elphinstone et al. (1996) (SMSA), el cual contiene cristal violeta (violeta de genciana), sulfato B de polimixina, bacitracina, cloromicetina y ciclohexamida, todos ellos al 1%, además de bencilpenicilina al 0.1%. Las cajas sembradas se incubaron a28oC por un período de tres a cuatro días.
Para preservar las cepas, se seleccionaron las colonias con morfología típica de R. solanacearum (mucoide y con una coloración rosa en el centro), se incrementaron en tubos con 5 mL de medio de cultivo líquido de ácido casamino peptona glucosa (CPG) con agitación a 150 rpm y temperatura de 28oC durante 24 h (Hendrick y Sequeira, 1984). De cada una de las cepas, se tomó una alícuota de 600 μL de cultivo bacteriano y se depositaron en un tubo Eppendorf, al que posteriormente se le adicionaron 600 μL de glicerol a una concentración de 30% y se almacenaron a -80°C.
Características de colonias de R. solanacearum. Las características de tamaño, forma, superficie, bordes, color y producción de pigmento de cada una de las cepas o colonias bacterianas detectadas, se observaron en placas con medio de cultivo TZC (Kelman, 1954; Champoiseau et al., 2009). Estas placas se incubaron a temperatura de 28°C por 48h (Figura 1).

Pruebas bioquímicas para la identificación de R. solanacearum. La tinción de Gram se realizó de acuerdo a lo descrito por Suslow et al. (1982). En la prueba de oxidasa se siguió la metodología descrita por Hildebrand et al. (1988). La producción de dihidrolasa de arginina se realizó mediante la metodología propuesta por Thornley (1960). La determinación del metabolismo oxidativo/fermentativo de los carbohidratos se realizó de acuerdo a lo señalado por Hughy Leifson (1953). La reducción de nitratos a nitritos y producción de gas fue realizada como lo señalan Hayward et al. (1990). La hidrólisis de almidón y licuefacción de gelatina se realizó de acuerdo a lo propuesto por Hayward (1964). Todas estas pruebas se realizaron por triplicado.
Obtención de ADN. La extracción del ADN se realizó de colonias bacterianas de R. solanacearum desarrolladas por 24 h a 28oC en medio de cultivo TZC. La extracción se realizó mediante lisis por calor a 80oC por 5 min.
PCR para confirmación de especie y de raza. La confirmación de la especie en todas las cepas de R. solanacearum, se realizó por PCR mediante la utilización del par de oligonucleótidos universales 759/760 (5' GTC GCC GTC AAC TCA CTT TCC 3' y 5' GTC GCC GTC AGC AAT GCG GAA TCG 3') (Opina et al., 1997). La identificación de todas las cepas R3Bv2 se realizó con la utilización del par de oligonucleótidos 630/631(5' ATA CAG AAT TCG ACC GGC AC 3' y 5' ATT CAC ATG CAA TTC GCC TAC 3') en la PCR específica para Raza 3 (Fegan et al., 1998).
Las reacciones de PCR se realizaron en un termociclador Eppendorf Mastercycler personal, utilizando el sistema de detección PCR core Systems 1 (Promega). El volumen total de la mezcla de reacción fue de 25 μL, para todas las reacciones. El contenido de la mezcla de reacción fue: 30 ng de DNA genómico, 2.5 μL de buffer de PCR 10x, 2.5 mM de MgCl2, 0.25 mM de cada dNTP, 20 pmol de cada primer y 2.5 mM de ADN Taq polimerasa. La amplificación del ADN se realizó a 94oC por 15 s, seguido por 30oC por 58 s, 72oC por 30 s 30 ciclos, 72oC por 5 min, la reacción se mantuvo a 4oC.
Una alícuota de 10 μL de los productos de PCR, se analizaron en geles de agarosa al 1.5%, se tiñeron con 1 μL de bromuro de etidio (10 mg L-1). La electroforesis se realizó por 40 min a 70 V cm-1. El gel se visualizó bajo luz UV. La información fue registrada con una cámara digital (Olympus). La respuesta positiva se definió como la presencia de una banda visible del tamaño esperado (280pb) para la confirmación de la especie y de 304 pb, para la identificación de la Raza 3, mientras que una respuesta negativa se definió como la ausencia del fragmento esperado. En la electroforesis se usó un marcador de 100 pb como estándar de peso molecular.
Determinación de biovares. La clasificación de las cepas en biovares se realizó de acuerdo a la prueba fisiológica desarrollada por Hayward (1964; 1991). Ésta se basa en la capacidad de la cepa para oxidar tres disacáridos (celobiosa, lactosa y maltosa), tres alcoholes hexosa (manitol, dulcitol y sorbitol) y la capacidad para utilizar trehalosa, mio-inositol y D-ribosa. El medio base "Hayward" (Hayward, 1964) se esterilizó en autoclave a 121°C a 20 lb de presión por 20 min, se distribuyó en alícuotas de 90 mL y se adicionó una concentración de 1% de cada carbohidrato cuando la temperatura del medio descendió aproximadamente a 55°C. Los carbohidratos se esterilizaron por filtración con filtros Millipore® con poro de 0.22 μm. Se distribuyeron alícuotas de 2 mL de medio en tubos de 10 cm. Se preparó una suspensión bacteriana en agua deionizada estéril, a una concentración de 1 x 108 UFC mL-1 a partir de cultivo bacteriano desarrollado en caldo CPG y con agitación por toda la noche (He et al., 1983). Cada tubo se inoculó con una alícuota de 3 mL de la suspensión bacteriana, cada prueba se realizó tres veces con un control sin inocular. Los tubos se incubaron a 28oC por tres semanas. Se observaron las reacciones después de 1,3, 7,14 y 28 días de incubación a 28°C. Se evaluó el cambio de color de verde olivo a amarillo, evidenciando la producción de ácido a partir de los disacáridos y oxidación de los alcoholes hexosa (Denny y Hayward, 2001).
Prueba de hipersensibilidad en tabaco. Se preparó una suspensión bacteriana con cada una de las cepas aisladas de R. solanacearum obtenidas, la cual se ajustó a una concentración de 1 x 108 UFC mL-1 en agua destilada esterilizada y se infiltró entre las nervaduras en hojas gruesas de tabaco (Nicotiana tabacum cv. 'Burley') con la ayuda de una aguja hipodérmica de acuerdo con la metodología propuesta por Lozano y Sequeira (1970). Como control negativo, se infiltró solamente agua destilada esterilizada. Las plantas se mantuvieron a 28oC bajo condiciones de invernadero y se evaluó, tejido muerto, tejido colapsado y amarillamiento después de 24 h de la inoculación.
Pruebas de Patogenicidad. La patogenicidad de las cepas identificadas se evaluó en plantas de papa ('Alfa'), tomate ('Imperial'), berenjena ('ES4013'), pimiento ('Roble') y tabaco ('Burley'). Para realizar la inoculación, se incrementó el cultivo bacteriano en placas con medio de cultivo CPG agar a 28oC por 48 h, se lavó con agua deionizada estéril y ajustó a una concentración de 1 x 108 UFC mL-1 (absorbancia fue de 0.3 con una longitud de onda de 600 nm) (He et al., 1983). De la suspensión bacteriana preparada de cada una de las cepas, se tomó una alíquota de 5 mL para cada planta y se vertió en la cavidad de trasplante a cinco plantas por cultivo con cada una de las cepas. Tres días después, las plantas se trasplantaron en macetas de 15 cm de alto, las cuales contenían una mezcla en relación 1:1 de suelo y sustrato estéril. Las macetas se colocaron en un invernadero tipo malla-sombra sobre platos hondos y a éstos se les depositó agua para mantener alta humedad y facilitar la infección y desarrollo de la marchitez. Las plantas se fertilizaron de acuerdo a los principios de la solución de Steiner, por intervalos de 10 días y se mantuvieron bajo condiciones de invernadero de 23-28oC. Las plantas inoculadas se distribuyeron de acuerdo a un diseño experimental de dos factores (cepas y tiempo), con las plantas anidadas (cinco réplicas) en cepas y medidas repetidas en el tiempo con bloques (cultivos) aleatorizados completos. el experimento se repitió dos veces. La marchitez en las plantas se evaluó semanalmente por cuatro semanas después de la inoculación.
RESULTADOS Y DISCUSIÓN
Identificación mediante pruebas bioquímicas. De las plantas enfermas de tomate, se obtuvieron un total de ocho cepas aisladas en medio de cultivo TZC y SMSA, las que fueron seleccionadas por las pruebas de flujo bacteriano e inmunotiras. Todas las cepas produjeron colonias típicas mucoide, superficie brillosa, borde irregular y color blanco con rosa en el centro en el medio TZC (Figura 1). Después de 72 h, todas las cepas presentaron un color café alrededor de las colonias. Sólo la cepa CRs2 mostró una pigmentación menos marcada que el resto de las cepas. Lo anterior sugiere la obtención de colonias virulentas de acuerdo con lo reportado por Kelman (1954) y Champoiseau et al. (2009), que establecen diferencias entre colonias virulentas con las características anteriores y colonias avirulentas; no mucoides, borde circular y de color opaco.
Las pruebas bioquímicas (Cuadro 2) mostraron resultados similares para todas las cepas, semejante a lo reportado en estudios de identificación de cepas de R. solanacearum realizados por Hayward (1964; 1994) y por Nouri et al. (2009).
Identificación de R. solanacearum por PCR.
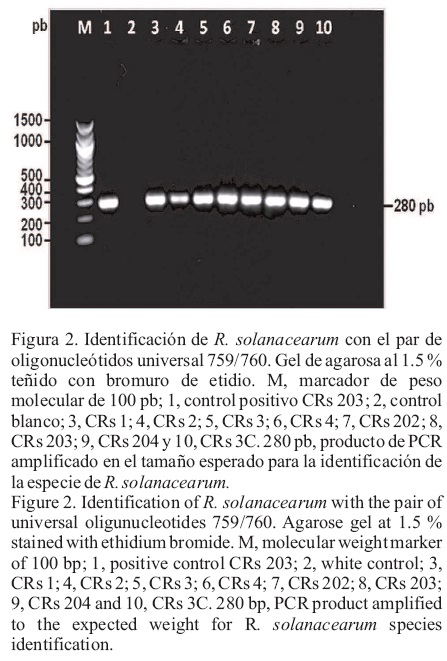
Todas las cepas identificadas como R. solanacearum mediante pruebas bioquímicas e inmunológicas, amplificaron por PCR con el par de oligonucleótidos universal 759/760 (Opina et al., 1997). La reacción amplificó el fragmento de tamaño esperado de 280, lo que permitió identificarlas como R. solanacearum (Figura 2).
Determinación de biovar. Todos los aislamientos en este estudio fueron caracterizados e identificados de acuerdo con su habilidad para utilizar diferentes fuentes de carbono (Cuadro 3). Las pruebas fisiológicas propuestas por Hayward (1964; 1994), para determinar biovares, mostraron un perfil similar para todas las cepas. Todas las cepas utilizaron lactosa, maltosa, celobiosa y mio-inositol pero no manitol, dulcitol, sorbitol y trehalosa, redujeron nitratos a nitritos y produjeron gas a partir de nitratos. Por lo que se puede determinar, de acuerdo al mismo autor, que estas cepas analizadas pertenecen al Bv2.

Estos resultados coinciden con estudios previos, en los cuales se realizaron aislamientos de R. solanacearum en papa, tomate y geranio y fueron identificados mediante las pruebas fisiológicas desarrolladas por Hayward (1964; 1991), en biovar 2 (Jeong et al., 2007; Sánchez et al., 2008; Nouri et al., 2009). Los resultados en este estudio muestran que todas las cepas detectadas se ubican en un sólo grupo de R. solanacearum como responsables de causar la marchitez bacteriana del tomate en el Valle de Culiacán, B.C. y Jalisco.
Reacción de hipersensibilidad en hojas de tabaco. En la prueba de reacción de hipersensibilidad en tabaco, todas las hojas infiltradas con la suspensión de cada una de las cepas de R. solanacearum, no mostraron reacción de hipersensibilidad a las 24 h. Después de 36 h, mostraron en el área de la infiltración, una necrosis parcialmente desecada y con un halo de color amarillo alrededor de la lesión.
Estudios de reacción de hipersensibilidad en tabaco, realizados en Florida por Ji et al. (2007) con cepas R1Bv1 y con una cepa R3Bv2, mostraron variabilidad en la respuesta de la reacción de hipersensibilidad entre cepas R1Bv1, ya que algunas cepas R1Bv1 mostraron reacción a las 24 h, mientras que otras fueron negativas a esta prueba al igual que la cepa R3Bv2 (Cuadro 4). Lo anterior demuestra la variabilidad existente entre cepas R1Bv1 ante esta prueba; sin embargo, las cepas R3Bv2 no presentan dicha variabilidad. Por lo anterior, algunos investigadores sugieren que la prueba de reacción de hipersensibilidad, no presenta rasgos exitosos en la diferenciación de cepas Bv1 (Ji et al., 2007). Lo anterior no incluye al grupo de cepas R3Bv2, ya que de acuerdo con los resultados en este estudio, las cepas aisladas en el Valle de Culiacán, como las cepas originarias del Valle de San Quintín, B.C. y de Jalisco, presentan características similares ante pruebas bioquímicas y fisiológicas.

Pruebas de patogenicidad. Todas las cepas utilizadas en esta investigación fueron avirulentas en tabaco y altamente virulentas en tomate. La cepa CRs2 afectó tomate y papa. La cepa CRs202 afectó tomate, papa y chile pimiento. La cepa CRs204 afectó tomate y chile pimiento y la cepa CRs3C unicamente tomate. Las cepas capaces de afectar un rango más amplio de hospedantes fueron la cepa CRs3 y CRs203, las cuales afectaron principalmente tomate, además de papa, berenjena y chile pimiento; lo cual coincide con las características típicas de rango de hospedantes reportadas para las cepas R3Bv2 (Pradhanang et al., 2000; Janse et al., 2004; Norman et al., 2009; Champoiseau et al., 2009).
Dentro del grupo de cepas que comunmente afectan tabaco se han identificado a la raza 1 Bv1, Bv3 y Bv4 aisladas de plantas de tabaco infectadas naturalmente. Por otro lado, cepas Bv1 aisladas de tomate y geranio y cepas Bv3 aisladas de pimiento han mostrado capacidad para afectar tabaco (Ji et al., 2007; Xu et al., 2009); sin embargo, algunas cepas R1Bv1 aisladas en Florida, Estados Unidos, han mostrado ser avirulentas en tabaco, dicha característica se le ha atribuido a la presencia del gen funcional avrA (factor de inducción de HR positiva en tabaco) (Robertson et al., 2004; Ji et al., 2007).
Identificación molecular de cepas de R. solanacearum R3Bv2. El grupo de cepas de R. solanacearum R3Bv2 ha sido reportado en diferentes regiones agrícolas del mundo (Swanson et al., 2005; Elphinstone, 2005; Sánchez et al., 2008). En la identificación a nivel de raza de R. solanacearum, la PCR específica para cepas R3Bv2 con los oligonucleótidos 630/631 (Fegan et al., 1998) amplificó un fragmento de 304 pb en todas las cepas utilizadas (Figura 3).

Estos resultados son similares a los obtenidos por Sánchez et al. (2008) en la identificación de cepas R3Bv2 aisladas de papa, geranio y tomate en un estudio de diversidad y distribución de R. solanacearum en Guatemala, donde obtuvo un fragmento de 304 pb. Nouri et al. (2009) identificaron cepas R3Bv2 aisladas de papa, ellos utilizaron la PCR específica para raza 3 y encontraron a tres cepas de un total de 40 diferentes ya que no amplificaron el fragmento esperado de 304 pb. Dichas cepas mediante pruebas fisiológicas fueron identificadas como Bv2-T. Lo anterior confirma la capacidad del par de primer 630/631 para diferenciar cepas Bv2 y cepas Bv2-T (cepas biovar 2T son fisiológicamente más activas) en la identificación de cepas R3Bv2 el cual fue utilizado en nuestro estudio.
Aunque se ha reportado que el grupo de cepas R3Bv2 afecta cultivos en regiones templadas (Swanson et al., 2005), estudios de distribución geográfica de R. solanacearum en diferentes regiones agrícolas muestran la presencia del grupo de cepas R3Bv2 en climas tropicales, subtropicales y templados (Sánchez et al., 2008).
La detección de las cepas de R. solanacearum raza 1 y 3, es importante por su diferente agresividad en los cultivos susceptibles (Ji et al., 2007). En México se reportó la presencia de la R2BV1 de R. solanacearum en plantas de tomate (Dr. Fucikovsky, comunicación personal), sin embargo en relación a la presencia de la R3BV2, no existen informes sobre la identificación y caracterización de este patógeno en cultivos de tomate.
CONCLUSIONES
Las pruebas morfológicas, bioquímicas, fisiológicas, inmunotiras y PCR, indican que todas las cepas de Ralstonia solanacearum aisladas corresponden a la Raza 3 (R3). De acuerdo a la utilización de diferentes fuentes de carbono, todas las cepas estudiadas pertenecen al Biovar 2 (Bv2). Todas las cepas de R. solanacearum fueron altamente patógenas en tomate. Ninguna de las cepas estudiadas fue patogénica en plantas de tabaco. En Sinaloa, este es el primer trabajo de identificación y caracterización de cepas de R. solanacearum en tomate.
LITERATURA CITADA
Allen C, Kelman A and French ER. 2001. Brown rot of potatoes. Pp:11-13. In: Stevenson WR, Loria R, Franc GD and Weingartner DP (eds.). Compendium of Potato Diseases, 2nd ed. American Phytopathological Society, St. Paul, MN. 125p. [ Links ]
Champoiseau PG, Jones JB and Allen C. 2009. Ralstonia solanacearum race 3 biovar 2 causes tropical losses and temperate anxieties. Online. Plant Health Progress doi:10.1094/PHP-2009-0313-01-RV. [ Links ]
Cook D and Sequeira L. 1994. Strains differentiation of Pseudomonas solanacearum by molecular genetic methods. Pp:77-93. In: Hayward AC and Hartman GL (eds.). Bacterial Wilt: The Disease and Its Causative Agent, Pseudomonas solanacearum. CAB International, Wallingford, United Kingdom. 268p. [ Links ]
Denny TP. 2006. Plant pathogenic Ralstonia species. Pp:573-644. In: Gnanamanickam SS (ed.). Plant-Associated Bacteria. Springer. Printed in the Netherlands. 710p. [ Links ]
Denny TP and Hayward AC. 2001. Gram-negative bacteria. Pp:151-173. In: Schaad NW, Jones JB and Chun W (eds.). Laboratory Guide for Identification of Plant Pathogenic Bacteria. American Phytopathological Society. St Paul, MN, USA. 398p. [ Links ]
Elphinstone JG. 2005. The current bacterial wilt situation: A global view. Pp:9-28. In: Allen C, Prior P and Hayward AC (eds.). Bacterial Wilt Disease and the Ralstonia solanacearum Species Complex. American Phytopathological Society. St. Paul, Minnesota. 528p. [ Links ]
Elphinstone JG, Hennessy J, Wilson JK and Stead DE. 1996. Sensitivity of different methods for the detection of Ralstonia solanacearum in potato tuber extracts. EPPO Bulletin 26:663-678. [ Links ]
Fegan M and Prior P. 2005. How complex is the Ralstonia solanacearum species complex? Pp:449-461. In: Allen C, Prior P and Hayward AC (eds.). Bacterial Wilt: The Disease and the Ralstonia solanacearum Species Complex. American Phytopathological Society. St. Paul, Minnesota. 528p. [ Links ]
Fegan M, Holoway G, Hayward AC and Timmis J. 1998. Development of a diagnostic test based on the polymerase chain reaction to identify strains of Ralstonia solanacearum exhibiting the biovar 2 genotype. Pp:34-43. In: Prior P, Allen C and Elphinstone J (eds.). Bacterial Wilt Disease: Molecular and Ecological Aspects. Springer-Verlag, Berlin. 461p. [ Links ]
Hayward AC. 1964. Characteristics of Pseudomonas solanacearum. Journal Applied Bacteriology 27:265-277. [ Links ]
Hayward AC, El-Nashaar H M, Nydegger U and De Lindo L. 1990. Variation in nitrate metabolism in biovars of Pseudomonas solanacearum. Journal of Applied Bacteriology 69:269-280. [ Links ]
Hayward AC. 1991. Biology and epidemiology of bacterial wilt caused by Pseudomonas solanacearum. Annual Review of Phytopathology 29:65-87. [ Links ]
Hayward AC. 1994. Systematics and phylogeny of Pseudomonas solanacearum and related bacteria. Pp:123-135. In: Hayward AC and Hartman GL (eds.). Bacterial Wilt: The Disease and Its Causative Agent, Pseudomonas solanacearum. CAB International, Wallingford, United Kingdom. 268p. [ Links ]
Hayward AC. 2000. Ralstonia solanacearum. Pp:32-42. In: Encyclopedia of Microbiology, Vol. 4, 2nd ed. Academic Press, London (GB). 1142p. [ Links ]
He LY, Sequeira L and Kelman A. 1983. Characteristics of strains of Pseudomonas solanacearum of China. Plant Disease 67:1357-1361. [ Links ]
Hendrick CA and Sequeira L. 1984. Lipopolysaccharide-defective mutants of the wilt pathogen Pseudomonas solanacearum. Applied and Environmental Microbiology 48:94-101. [ Links ]
Hildebrand DC, Schroth MN and Sands DC. 1988. Laboratory guide for identification of plant pathoenic bacteria. Pp:60-81. In: Schaad NW (ed.). Pseudomonas. The American Phytopathological Society. St. Paul, Minnesota. 398p. [ Links ]
Hugh R and Leifson E. 1953. The taxonomic significance of fermentative versus oxidative metabolism of carbohydrates by various Gram-bacteria. Journal Bacteriology 66:24-26. [ Links ]
Janse JD, Van Den Beld HE, Elphinstone J, Simpkins S, Tjou-Tam-Sin NNA and Van Vaerenbergh J. 2004. Introduction to Europe of Ralstonia solanacearum biovar 2, race 3 in Pelargonium zonale cuttings. Journal of Plant Pathology 87:147-155. [ Links ]
Jeong Y, Kim J and Kang Y. 2007. Genetic diversity and distribution of Korean isolates of Ralstonia solanacearum. Plant Disease 91:1277-1287. [ Links ]
Ji P, Allen C, Sánchez-Pérez A, Yao J, Elphinstone JG, Jones JB and Momol MT. 2007. New diversity of Ralstonia solanacearum strains associated with vegetable and ornamental crops in Florida. Plant Disease 91:195-203. [ Links ]
Kelman A. 1954. The relationship of pathogenicity of Pseudomonas solanacearum to colony appearance in a tetrazoliummedium. Phytopathology 44:693-695. [ Links ]
Lambert CD. 2002. Agricultural bioterrorism protection act of 2002: Possession, and transfer of biological; agents and toxins; interim and final rule (7 CFR Part 331). Federal Register 67:76908-76938. [ Links ]
Lozano J and Sequeira L. 1970. Differentiation of races of Pseudomonas solanacearum by a leaf infiltration tecnique. Phytopathology 60:833-838. [ Links ]
Norman DJ, Zapata M, Gabriel DW, Duan YP, Yuen JMF, Mangravita-Novo A and Donahoo RS. 2009. Genetic diversity and host range variation of Ralstonia solanacearum strains entering North America. Phytopathology 99:1070-1077. [ Links ]
Nouri S, Bahar M and Fegan M. 2009. Diversity of Ralstonia solanacearum causing potato bacterial wilt in Iran and the first record of phylotipe II/biovar 2T strains outside South America. Plant Pathology 58:243-249. [ Links ]
Opina N, Tavner F, Hollway G, Wang JF, Li TH, Maghirang R, Fegan M, Hayward AC, Krishnapillai V, Hong WF, Holloway BW and Timmis J. 1997. A novel method for development of species and strain specific DNA probes and PCR primers for identifying Burkholderia solanacearum (formerly Pseudomonas solanacearum). Asia Pacific Journal Molecular Biology and Biotechnology 5:19-30. [ Links ]
Pradhanang PM, Elphinstone JG and Fox RTV. 2000. Identification of crop and weed hosts of Ralstonia solanacearum biovar 2 in the hills of Nepal. Plant Pathology 49:403-413. [ Links ]
Ramírez VJ y Sainz RRA. 2006. Manejo integrado de las enfermedades del tomate. Pp:25-27. 1a ed. Once Rios Editores. México. 360p. [ Links ]
Robertson AE, Wechter WP, Denny TP, Fortnum BA and Kluepfel DA. 2004. Relationship between avirulence gene (avrA) diversity in Ralstonia solanacearum and bacterial wilt incidence. Molecular Plant-Microbe Interactions 17:1376-1384. [ Links ]
Sánchez AP, Mejía L, Fegan M and Allen C. 2008. Diversity and distribution of Ralstonia solanacearum strains in Guatemala and rare occurrence of tomato fruit infection. Plant Pathology 57:320-331. [ Links ]
Seal SE and Elphinstone JG. 1994. Advances in identification and detection of Pseudomonas solanacearum. Pp:35-57. In: Hayward AC and Hartman GL (eds.). Bacterial Wilt: The Disease and Its Causative Agent, Pseudomonas solanacearum. CAB International. Wallingford, United Kingdom. 268p. [ Links ]
Suslow TV, Schroth MN and Isaka M. 1982. Application of a rapid method for Gram differentiation of plant pathogenic and saprophytic bacteria without staining. Phytopathology 72:917-918. [ Links ]
Swanson JK, Yao J, Tans-Kersten J and Allen C. 2005. Behavior of Ralstonia solanacearum race 3 biovar 2 during latent and active infection of geranium. Phytopathology 95:136-143. [ Links ]
Thornley MJ. 1960. The differentiation of Pseudomonas from other Gram-negative bacteria on the basis of arginine metabolism. Journal Applied Bacteriology 23:37-52. [ Links ]
Villa JE, Tsuchiya K, Horita H, Natural M, Opina N and Hyakumachi M. 2005. Phylogenetic relationships of Ralstonia solanacearum species complex strains from Asia and other continents based on 16S rDNA, endoglucanase, and hrpB gene sequences. Journal of General Plant Pathology 71:39-46. [ Links ]
Xu J, Pan ZC, Prior P, Xu JS, Zhang Z, Zhang H, Zhang LQ, He LY and Feng J. 2009. Genetic diversity of Ralstonia solanacearum strains from China. European Journal Plant Pathology 125:641-653. [ Links ]














