Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de fitopatología
versión On-line ISSN 2007-8080versión impresa ISSN 0185-3309
Rev. mex. fitopatol vol.26 no.2 Texcoco ene. 2008
Artículos científicos
Caracterización de Aislamientos Mexicanos de Pochonia chlamydosporia var. chlamydosporia (Goddard) Gams y Zare para el Control Biológico de Nacobbus aberrans (Thorne) Thorne y Allen
Characterization of mexican isolates of Pochonia chlamydosporia var. chlamydosporia (Goddard) Gams and Zare for biological control of Nacobbus aberrans (Thorne) Thorne and Allen
Rosalinda Flores–Camacho1, Simon D. Atkins2, Rosa Helena Manzanilla–López3, Ignacio Cid del Prado–Vera4, y Ángel Martínez–Garza4
1 Dirección General de Inspección Fitosanitaria, Municipio Libre 377 Piso 7–B, Col. Santa Cruz Atoyac, Deleg. Benito Juárez, México, D.F. 03310.
2 Plant Pathology and Microbiology Department, Rothamsted Research, Harpenden, Herts, AL5 2JQ, UK.
3 Nematode Interactions Unit, Rothamsted Research, Harpenden, Herts, AL5 2JQ, UK. Correspondencia: rosa.manzanilla–lopez@bbsrc.ac.uk
4Colegio de Postgraduados (CP), Programa de Fitosanidad, km 36.5 Carr. México–Texcoco, Montecillo, Edo. de México 56230.
Recibido: Octubre 30, 2006
Aceptado: Agosto 11, 2008
Resumen
Cinco aislamientos nativos del hongo Pochonia chlamydosporia, agente de control biológico de nematodos agalladores y formadores de quistes, se obtuvieron de muestras colectadas en parcelas infestadas con Nacobbus aberrans sensu stricto, en localidades de los estados de México, Morelos, Puebla y Tlaxcala (México). Se describen las metodologías para la obtención y el cultivo de los aislamientos SMB3A, SC1, SMB3, SM4 y MHCH, y las pruebas de efectividad del hongo en el control biológico del nematodo (i. e. porcentaje de huevos parasitados y colonización de la rizósfera), así como la caracterización molecular con β–tubulina–PCR, ERIC–PCR (amplificación de secuencias intergénicas de concenso repetitivas de enterobacterias), ITS–PCR (espaciadores internos transcritos), RFLP–PCR (polimorfismo de tamaño de los fragmentos de restricción) y RAPD–PCR (ADN polimórfico amplificado al azar). Los aislamientos mexicanos produjeron un parasitismo superior al 60% en los huevos del nematodo y una colonización del 100% de la rizósfera. Todos los aislamientos se identificaron molecularmente como pertenecientes a Pochonia chlamydosporia var. chlamydosporia usando cebadores específicos (β–tubulina–PCR) para esta especie.
Palabras claves: Nematodo falso–agallador, hongos nematófagos, RFLP, ERIC, ITS, β–tubulina.
Abstract
Five native isolates of the fungus Pochonia chlamydosporia, a biological control agent of root–knot and cyst–forming nematodes, were collected from Nacobbus aberrans sensu stricto infested plots in different localities in the states of Mexico, Morelos, Puebla and Tlaxcala (Mexico). Isolation and culturing methods are described for isolates SMB3A, SC1, SMB3, SM4, MHCH, and evaluation of the parasitism of nematode eggs and rhizosphere colonization by the fungus. Molecular characterization of the isolates was made using β–tubulin–PCR, ERIC–PCR (enterobacterial repetitive intergenic consensus sequence), ITS–PCR (internally transcribed spacer regions), RFLP–PCR (restriction fragment length polymorphism), RAPD–PCR (random amplified polymorphic DNA) and sequencing of DNA fragments. More than 60% of nematode eggs were parasitized by the Mexican isolates. All isolates colonised the complete extent of the rhizosphere. All isolates were identified using species–specific primers (β–tubulin–PCR) as belonging to Pochonia chlamydosporia var. chlamydosporia.
Keywords: False root–knot nematode, nematophagous fungi, RFLP, ERIC, ITS, β–tubulin.
Pochonia chlamydosporia (Goddard) Gams y Zare (2001) es un parásito facultativo que se ha aislado de nematodos formadores de quistes (Globodera spp., Heterodera spp.) y agalladores (Meloidogyne spp.) (Kerry y Jaffee, 1997; Kerry, 2001; Morgan–Jones y Rodríguez–Kabana, 1988). El hongo coloniza el rizoplano como saprótrofo hasta que infecta los huevos del nematodo y forma redes de hifas con órganos especializados que penetran la capa vitelina mediante lisis enzimática (Kerry, 1997). La posibilidad de cultivarlo en medio artificial líquido o sólido (Kerry y Jaffee, 1997), su abundancia en raíces infestadas con nematodos, su fácil dispersión y colonización en la rizósfera así como su especificidad hospedatoria y virulencia (De Leij y Kerry, 1991; Gams, 1988; Mauchline et al., 2004; Morton et al., 2003), lo han hecho un agente de uso potencial en el control biológico de Meloidogyne spp. (Kerry y Bourne, 1996, 2002). La selección de aislamientos depende de pruebas de laboratorio e invernadero en las que se evalúa: 1) la capacidad para colonizar la rizósfera, 2) la producción y viabilidad de las clamidosporas y 3) la mortalidad que causan a los huevos. La planta hospedante y las condiciones ambientales tienen un efecto significativo en el crecimiento del hongo (Bourne et al., 1996), por lo que es preferible identificar aislamientos con potencial colonizador de la rizósfera de los cultivos locales (Hidalgo–Díaz etal., 2000). Otra característica importante del hongo es que puede sobrevivir en ausencia del nematodo. Antes del uso de técnicas moleculares, la caracterización de las especies del género Pochonia se basaba en criterios morfológicos, el análisis de compatibilidad vegetativa, características bioquímicas y diferencias en virulencia (Butterfield y De Vary, 1977; Gams, 1988; Joaquim y Rowe, 1990). Hirsch et al. (2000) han reportado distintos métodos de PCR (reacción en cadena de la polimerasa) y el uso de cebadores (secuencias iniciadoras) específicos, diseñados con base en el gen de la β–tubulina para discriminar entre especies estrechamente relacionadas. Este ha sido un avance importante pues la universalidad de la β–tubulina permite emplearla tanto en el análisis filogénetico (Edlind et al., 1996) como en el diseño de cebadores. La especificidad de los cebadores de la β–tubulina (Tub1f y Tub1r) para P. chlamydosporia var. chlamydosporia ha sido confirmada en Rothamsted Research usando aislamientos provenientes de distintos nematodos (e.g. Globodera spp., Heterodera spp., Meloidogyne spp.), de países latinoamericanos (Hidalgo–Díaz et al., 2000), de la Comunidad Europea (Morton et al., 2003), Australia y Nueva Zelanda (Arora et al., 1996) entre otros. Los resultados han demostrado la importancia de contar con una herramienta de diagnóstico rápida para la identificación precisa del hongo en muestras de suelo, raíces y huevos (Atkins et al., 2003; Hirsch et al., 2000, 2001). Nacobbus aberrans (Thorne) Thorne y Allen incluye a la mayor parte de poblaciones del nematodo falso–agallador (N. aberrans sensu lato) estudiadas en Norte y Sudamérica, aunque las poblaciones mexicanas se ubican en N. aberrans sensu stricto (s.s.) (Reid et al., 2003). Al igual que Meloidogyne spp., Nacobbus spp. induce agallas en las raíces del hospedante. Ambos géneros producen hembras endoparásitas sedentarias que depositan huevos en una matriz gelatinosa, la cual es generalmente expulsada de la agalla quedando expuesta en la rizósfera junto con los huevos. Estos huevos pueden ser parasitados y destruidos por el hongo. Los estudios sobre hongos nematófagos en el control de Nacobbus aberrans sensu lato (s.l.) son escasos (Manzanilla–López et al., 2002), pero es posible que algunas especies de hongos que han sido efectivas en el control biológico de Meloidogyne spp. sean bio–controladores de N. aberrans también. Considerando que la especie P. chlamydosporia es un parásito facultativo que habita naturalmente en suelos supresivos (Kerry, 1995) y que es un agente promisorio para el control biológico de los nematodos agalladores (Kerry y Bourne, 2002), se realizó el presente trabajo con el objetivo de aislar y caracterizar molecularmente aislamientos nativos mexicanos de P. chlamydosporia var. chlamydosporia del suelo y las raíces de jitomate (Lycopersicon esculentum Mill.), chile (Capsicum annuum L.) y frijol (Phaseolus vulgaris L.) infectados por N. aberrans y que pudiesen ser usados como agentes potenciales en el control biológico del nematodo.
MATERIALES Y MÉTODOS
Muestreo. Se colectaron muestras de raíces y suelos en zonas productoras de jitomate, frijol y chile infestados en forma natural con N. aberrans, ubicadas en los estados de México, Morelos, Tlaxcala y Puebla (Cuadro 1). El muestreo se realizó en la temporada de lluvias de verano (1999–2000). Los muestreos se realizaron con un patrón aleatorio (Barker y Campbell, 1981). El suelo se recogió en la periferia de la raíz, eliminando los primeros 5 cm de suelo, antes de colectar muestras de 100 g entre los 15 y 25 cm de profundidad, además de 50 g de raíces agalladas por punto de muestreo. Las muestras se depositaron en bolsas de polietileno, se etiquetaron y se mantuvieron en una hielera hasta su procesamiento en el laboratorio de nematología del Instituto de Fitosanidad del Colegio de Posgraduados (CP).

Aislamiento de Pochonia sp. a partir de muestras de suelo. Un gramo de suelo de cada muestra se depositó en un tubo conteniendo 9 mL de agua–agar (AA) al 0.05%. La muestra se agitó en un vortex de 10 a 15 seg y luego se tomó 1 mL para hacer diluciones de 10–1, 10–2 y 10–3. Posteriormente, 0.2 mL de las diluciones 10–2 y 10–3 se depositaron en placas de Petri con medio de cultivo semi–selectivo (harina de maíz + rosa de Bengala + antibióticos) (Kerry y Bourne, 2002). Las placas se incubaron 10 días a 25°C, al término de los cuales se identificaron y cuantificaron las colonias correspondientes. Para la producción de clamidosporas, las colonias se cultivaron en harina de maíz–agar (HMA) o papa–dextrosa–agar (PDA).
Aislamiento de Pochonia sp. a partir de raíces. Las raíces infectadas con N. aberrans fueron cortadas en segmentos de 1 cm. Se separó al azar un 1 g de raíces y se maceró con 9 mL de AA al 0.05% en un mortero estéril. Enseguida se tomó 1 mL de esta mezcla (10–1) para diluirlo en otro tubo con 9 mL de AA al 0.05%. Este procedimiento se repitió dos veces más para obtener diluciones 10–2 y 10–3. Después se tomaron 0.2 mL de cada dilución y se sembraron en placas de Petri con medio semi–selectivo, esparciéndolos uniformemente con una asa de vidrio. Las colonias se identificaron 10 días después de mantener en incubación las placas Petri a 25°C.
Aislamiento de Pochonia sp. a partir de masas de huevos de N. aberrans. Se siguieron dos métodos: En el primero, con ayuda del microscopio estereoscópico, se separaron 600 masas de huevos (mh) a partir de las raíces agalladas de cada cultivo muestreado. Aquéllas que presentaron micelio, se enjuagaron tres veces con agua destilada estéril antes de agitarlas para dispersar los huevos. Después de 3 h en reposo, se tomaron alícuotas de 0.2 mL de la suspensión que se depositaron en placas de Petri con AA (8 g de agar en 1 L de agua sin antibióticos) e incubaron a 25°C por dos a tres días. En el segundo método, los huevos de 400 mh se separaron de la matriz gelatinosa y se sembraron en placas de Petri con HMA. Posteriormente, bajo el microscopio compuesto se identificaron los huevos parasitados por el hongo y se sembraron en HMA.
Incremento y selección de aislamientos de Pochonia sp. Todos los aislamientos se incrementaron masivamente en dos sustratos: A) Trigo molido–arena de cuarzo. Discos de HMA con el hongo fueron depositados en matraces de 250 mL conteniendo trigo molido y arena de cuarzo previamente esterilizada. Después de una semana en reposo a temperatura ambiente, cada matraz se agitó vigorosamente y se dejó dos semanas más en reposo hasta obtener sustratos cubiertos por una masa algodonosa. La presencia de clamidosporas se verificó antes de proceder a su extracción. El sustrato de cada matraz se lavó en una serie de tamices de 150 μm, 53 μm y 10 μm de apertura de malla. El residuo del tamizado final se mezcló con arena de cuarzo fina en proporción de 10:1 (10 de arena: 1 de clamidosporas p:p) (De Leij y Kerry, 1991). B) Arroz. Este proceso involucró dos fases de fermentación: a) una líquida y otra sólida. Para la fermentación líquida, se lavaron 50 g de arroz que se pusieron a cocer en 1 L de agua hasta llevarla a punto de ebullición durante 45 min; de esta mezcla se tomaron y filtraron 50 mL que se vertieron en un matraz de 250 mL antes de ser esterilizados. Luego se agregaron cinco discos de HMA con el hongo. El matraz con su contenido se mantuvo a 28°C en agitación constante a 150 rpm durante tres días. Después se realizó la fermentación sólida. En un matraz de 250 mL, se añadieron 50 g de arroz remojado estéril y se agregaron 25 mL del líquido con conidios y clamidosporas dejándolos en incubación a 28°C por 21 días. El sustrato se lavó enseguida en los tamices de 150 μm, 53 μm de apertura; las clamidosporas se colectaron en un tamiz de 10 μm de apertura y se mezclaron con arena fina de cuarzo en una proporción de 10:1.
Producción y viabilidad de clamidosporas. El sustrato con las clamidosporas se mezcló antes de tomar una muestra de 1 g y depositarla en un tubo con 9 mL de AA al 0.05%, agitando para formar una suspensión. Las clamidosporas se cuantificaron en un hemacitómetro (dos repeticiones por alícuota). Para evaluar la viabilidad de las clamidosporas, se pesó 1 g de la mezcla del sustrato de arena fina de cuarzo y se agregó a tubos con AA al 0.05%, para hacer diluciones de 101, 10–2, 10–3, 10–4 y 10–5. Se sembraron 0.2 mL de cada dilución en placas de Petri con sorbosa (2 g L–1), agar (12 g L–1), con antibióticos (50 mg L–1 de sulfato de estreptomicina, cloramfenicol e hidrocloruro de clorotetraciclina). Las placas se incubaron dos días a 25°C. El porcentaje de germinación se estimó cuantificando aleatoriamente un total de 100 clamidosporas germinadas y no germinadas (Kerry y Bourne, 2002). Los aislamientos mexicanos se compararon con un aislamiento estándar procedente de Brasil (VC10), para evaluar su efectividad en la colonización de la rizósfera y huevos del nematodo. El aislamiento VC10 fue provisto por Rothamsted Research.
Colonización de la rizósfera en condiciones estériles. La superficie de semillas de trigo (Triticum aestivum L.) se esterilizó en hipoclorito de sodio al 7% con unas gotas de detergente líquido, manteniendo las semillas y la solución esterilizadora en agitación durante 2 h. Después se enjuagaron 4 ó 5 veces con agua estéril y se transfirieron a placas de Petri (12 g de agar, 10 g de glucosa, 0.1 g peptona, 0.1 g levadura) y se incubaron para su germinación de 2 a 3 días a 25°C. Varios tubos de ensayo se llenaron con vermiculita hasta ¾ partes de su volumen, enseguida se les agregó agua hasta el nivel de la vermiculita y se taparon con algodón antes de esterilizarlos. Cuatro discos de HMA con los aislamientos del hongo y las semillas de trigo previamente esterilizadas, lavadas y germinadas, se colocaron dentro de los tubos de ensayo. Los tubos se sellaron y cubrieron con papel aluminio antes de incubarlos por tres semanas a 25°C. Posteriormente, en condiciones asépticas, se cortaron fragmentos de raíz del trigo de 1 cm de largo que se sembraron en AA. Tres días después se observaron al microscopio para verificar la colonización de la raíz con el micelio del hongo. Los fragmentos positivos se cuantificaron y se estimaron los porcentajes de colonización.
Parasitismo del hongo en los huevos de N. aberrans s.s. Se agregaron 0.2 mL de suspensión de huevos de N. aberrans s.s. de las poblaciones Montecillo, Tecamachalco y Zacatecas (Flores–Camacho et al., 2007) en placas de Petri con los aislamientos ya desarrollados del hongo en HMA. Después de tres días a 25°C, se examinaron aleatoriamente 100 huevos bajo el microscopio para determinar el porcentaje de huevos parasitados (i.e. infectados con hifas) en tres poblaciones de N. aberrans s.s. El testigo consistió de huevos del nematodo colectados a partir de masas de huevos producidas en plantas de jitomate inoculadas con los nematodos y sin el hongo.
Análisis estadístico. Los resultados obtenidos de todas las variables excepto el porcentaje de parasitismo de huevos, se transformaron a rangos para hacer una aproximación del análisis estadístico por ser variables no paramétricas. Con los valores transformados se hizo un análisis de varianza (ANAVA) y una comparación de medias (Tukey) con un nivel de significancia de 0.05 utilizando el paquete estadístico Statistical Analysis System (SAS) de acuerdo a la metodología citada por Martínez–Garza (1980).
Identificación de los aislamientos del hongo. Los aislamientos purificados se identificaron con claves taxonómicas (Barnett y Hunter, 1987; Domsch y Gams, 1980). Una parte de cada aislamiento se liofilizó y otra parte se cultivó en HMA antes de almacenarlos a 4°C para su identificación molecular en Rothamsted Research (Reino Unido).
Extracción del ADN genómico. Previo a la extracción del ADN genómico los aislamientos se cultivaron por 7 días en HMA en placas de Petri. Una porción de micelio de cada aislamiento se transfirió a microtubos (vol = 1.5 mL) conteniendo 40 μL de sosa cáustica (NaOH al 0.25 M). Posteriormente, los microtubos se colocaron en baño maría a punto de ebullición durante 30 seg. Las muestras se neutralizaron con la adición de 40 μL de HCl al 0.25 M y 20 μL de tampón Tris–HCl al 0.5 M con pH de 8.0, 0.25% (p/v) de Nonidet® P–40 (Sigma), y se colocaron en un baño maría a punto de ebullición 2 min, al término de los cuales los microtubos se colocaron en hielo inmediatamente (Klimyuk et al., 1993). Los testigos negativos para las PCR fueron Paecilomyces lilacinus (Thom) Samson y Acremonium sp., los cuales junto con el aislamiento VC10 de P. c. var. chlamydosporia (testigo positivo), fueron proporcionados por Rothamsted Research.
Amplificación del ADN. Las PCR se realizaron en un volumen de 20 μL. Cada solución constó de: 2 μL de 10x tampón de PCR (100 mM Tris–HCl, pH 8.3, 500 mM KCl, MgCl2 1.5 mM), 2 μL (0.02 pM) de cada cebador, 0.4 μL de dNTPs (deoxinucleótido trifosfatos), 0.08 μL de Taq polimerasa (5 U μL–1) (Taq polimerasa Roche®), 1 μL de DNA y 12.88 μL de agua de alta pureza. La amplificación se hizo en un termociclador (TRIO–Thermoblock TBI, Biometra®). Las condiciones de PCR se describen a continuación: β–tubulina–PCR (Hirsch et al., 2000). Cebadores β–tub 1:5'– ATG CAA GAA AGC CTT GCG AC–3'; β–tub 2: 5'–TTT GCA GTA TCT CAG TGT TC–3'. Condiciones de PCR: 95°C pausa, 35 ciclos [94°C 1 min, 55°C 1 min, 72°C 1 min]; 72°C 5 min. ERIC–PCR (White et al., 1990). Cebadores ERIC (amplificación de secuencias intergénicas de concenso repetitivas de enterobacterias): R1CIRE: 5' – CAC TTA GGG GTC CTC GAA TGT A –3'; ERIC2: 5' – AAG TAA GTG ACT GGG GTG AGC G –3'. La concentración de cada cebador fue de 10 pM y la concentración de MgCl2 de 2.5 mM. Las condiciones de amplificación fueron: 95°C pausa, 35 ciclos [1 min a 94°C, 42°C 1 min, 72°C por 1 min]; 72°C 5 min (Atkins y Clark, 2005). ITS–PCR (White et al., 1990). Cebadores ITS (espaciadores internos transcritos): ITS 4: 5'– GGA AGT AAA AGT CGT AAC AAG G–3'; ITS 5: 5'– TCC TCC GCT TAT TGA TAT GC–3'. Condiciones de PCR: 95°C pausa, 94°C 3 min, 35 ciclos [ 94°C 1 min, 42°C 1.5 min, 72°C 1.5 min]; 72°C 5 min. RFLP–PCR (Carder et al., 1993). Las enzimas de restricción para los RFLP–PCR (polimorfismo de tamaño de los fragmentos de restricción) fueron Hae III (10 U μL1), Hin F1 (10 U μL1), CfoI (10 U μL1), XbaI (10 U μL1), Nar1 (10 U μL1) y Sau 3A (4 U μL1) (BRL Ltd). La digestión de los productos de ITS–PCR se hizo en un volumen de 20 μL. Se adicionaron 2.3 μL del tampón 10x, la enzima(s) de restricción (1 μL) y se incubó 1 h en un baño maría a la temperatura adecuada, de acuerdo a las instrucciones del proveedor. Los productos de la digestión enzimática se corrieron en un gel de agarosa (Helena Biosciences, Sunderland) al 1.5%. RAPD–PCR (Folkertsma et al., 1994). Cebador para RAPD–PCR (ADN polimórfico amplificado al azar) V17 (Operon Technologies, Alameda, CA): 5'–ACC GGC TTG T–3'. Las condiciones para PCR fueron: 92°C pausa, 92°C 2 min, 35°C 1 min, 72°C 1 min, 39 ciclos [92°C 1 min, 35°C 1 min, 72°C 1 min ciclo], 92°C 1 min, 35°C 1 min, 72°C 5 min.
Electroforesis. El ADN se visualizó mediante electroforesis en geles de agarosa (Ultrapure ADN Grade Agarose–Bio–Rad®) al 1.5 % en tampón TBE (0.089 M Tris–HCl, pH 8.6, 0.089 M de ácido bórico y 0.002 M de EDTA). Los geles se tiñeron con bromuro de etidio (0.5 mg mL–1) y se tomaron fotografías a 302 nm en un transiluminador con UVP imagestore con software 7500 (WP®, UK). El tamaño de los fragmentos de ADN se cuantificó en geles de agarosa. El tamaño del marcador fue de múltiplos de 123 pares de bases (pb) (Life Technologies Ltd®, UK). Los productos de PCR y los marcadores se tiñeron con bromuro de etidio y se determinaron aquéllos aislamientos diferentes o similares entre sí.
Clonado y secuenciado. Muestras de los aislamientos almacenados en HMA a 4°C en las placas de Petri fueron cultivadas nuevamente en HMA e incubadas a 28°C. El DNA se extrajo de los aislamientos (Klimyuk et al., 1993) y las condiciones para ITS–PCR fueron las ya descritas. Los fragmentos de ITS–PCR se cortaron del gel de agarosa usando un "kit" de elución (lab Qiagen®, "elution kit") y se clonaron en el vector TA® (In Vitro Gel®), siguiendo en ambos casos recomendaciones del proveedor y se secuenciaron usando los cebadores universales M13 (en sentido directo y reverso).
RESULTADOS
Aislamientos del hongo. A partir de las muestras de suelo se obtuvieron cuatro aislamientos de P. chlamydosporia a los cuales se asignaron las claves: SC1, SMB3A, SMB3 y SM4. El aislamiento MHCH se obtuvo a partir de masas de huevos del nematodo. Los aislamientos se encuentran depositados en Rothamsted Research con los siguientes números que corresponden a las claves mexicanas entre paréntesis: 242 (SMB3), 472 (SMB3A), 240 (SC1), 241 (SM4), 476 (MHCH).
Sustrato de cultivo. En el sustrato de cultivo trigo molido–arena de cuarzo, SMB3A produjo el mayor número (log10) de clamidosporas (8.36 g–1) en comparación con el arroz (Cuadro 2).
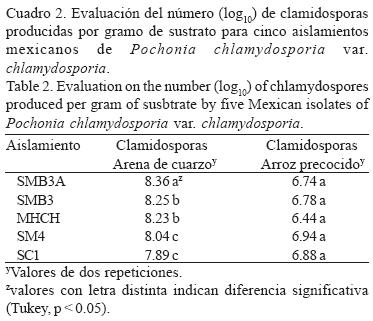
Viabilidad y producción de clamidosporas. El mejor aislamiento fue SMB3 A con un 93.3% de germinación, seguido del SMB3 con un 92.3 y VC10 con 92.3%. El que mostró menor viabilidad de las clamidosporas (80%) fue MHCH. La mayor producción de clamidosporas por g de sustrato se obtuvo con la arena de cuarzo (Cuadro 2). Ambos sustratos han sido reportados en la reproducción masiva del hongo para ser usado en pruebas de campo (Atkins et al., 2003; Hidalgo–Díaz etal, 2000; Verdejo–Lucaseía/., 2003).
Colonización de la rizósfera. Todos los aislamientos produjeron un 100% de colonización.
Porcentaje de parasitismo en los huevos. El aislamiento VC10 o aislamiento estándar ejerció el mayor porcentaje de parasitismo (89%) en la población Zacatecas del nematodo, seguido del MHCH (86%) y SMB3 (59%). Para la población Tecamachalco, el mayor porcentaje de parasitismo se obtuvo con el MHCH (88%); y SMB3A (72%), el de menor porcentaje de huevos parasitados (Cuadro 3). Para la población Montecillo, el aislamiento que produjo el mayor porcentaje de parasitismo fue SM4 (82%) y el menor SCI (67%).

Identificación de aislamientos de P. chlamydosporia con PCR. En el β–tubulina–PCR los cinco aislamientos mexicanos y el testigo positivo (VC10) generaron un solo producto de PCR de 270 pares de bases, confirmándose que corresponden a la especie P. c. var. chlamydosporia (Fig. 1), Usando el ERIC–PCR (Fig. 2), el perfil de bandas mostró distintos grupos: uno formado por MHCH y SMB3 y otro por SMB3A y SM4. El aislamiento SCI parece ser diferente a estos dos grupos. Los cebadores ITS4 y ITS5 indicaron que todos los aislamientos son similares. Los RFLP no mostraron diferencias (datos no presentados). Los perfiles generados con el cebador VI7 sugieren que con RAPD–PCR pueden identificarse tres grupos: SCI y SMB3A; SMB3 y MHCH; y SM4 (Fig. 3). Las secuencias de dos de los aislamientos se depositaron en la base de datos GenBank. Los números de acceso son: AY903605 (clave para 476), AY912487 (clave para 472). El análisis de la secuencia del producto de ITS–PCR mostró homologías con otras secuencias de P. c. var. chlamydosporia depositadas en GenEmbl.



DISCUSIÓN
Los estudios de control biológico de N. aberrans con hongos nematófagos son escasos (Manzanilla–López et al., 2002). Por ello, para hacer más rápida y eficiente la búsqueda de aislamientos en localidades mexicanas, se decidió obtener aislamientos de P. chlamydosporia, tomando en consideración su efecto bio–controlador en Meloidogyne spp. La acción parasítica de tos cinco aislamientos mexicanos del hongo sobre los huevos del nematodo fue superior al 60%, lo cual tos coloca como candidatos para ser usados en el control biológico de N. aberrans. Todos los aislamientos mexicanos generaron un solo producto de 270 pares de bases para β–tubulina–PCR. Las otras especies de hongos utilizados como testigos negativos (P. liiacinus y Acremonium sp.) confirmaron la especificidad de los cebadores para P. chlamydosporia var. chlamydosporia. La región ITS ha sido utilizada en otros ensayos para diferenciar entre aislamientos de P. chlamydosporia (Arora et al., 1996; Atkins et al., 2003). Aún cuando el producto amplificado del ITS–RFLP no permitió separar entre sí a los aislamientos mexicanos, éstos mostraron diferencias en la presencia de una secuencia repetitiva extragénica como ERIC (Fig. 2). La variación entre los aislamientos de P. chlamydosporia ha sido mostrada en otros estudios usando cebadores para ERIC–PCR (Arora et al., 1996; Mauchline et al., 2004; Morton et al., 2003). Este método molecular se ha utilizado para examinar rápidamente la variación genética entre poblaciones del hongo dando resultados más reproducibles que el uso de los cebadores estándares para RAPD–PCR (Arora et al., 1996). Debido a que el clonado y secuenciado de algunos fragmentos del DNA de los aislamientos mexicanos se realizó con un número pequeño de éstos, no se procedió a elaborar un árbol para establecer similitudes taxonómicas. Sin embargo, la información generada podría facilitar el diseño de cebadores específicos para estos aislamientos mexicanos, abriendo la posibilidad para identificar diversidad genética y monitorear los aislamientos del hongo en estudios aplicados (e.g. ecología, invernadero, laboratorio) de control biológico (Mauchline et al., 2002), y diseñar estrategias de manejo del nematodo y del cultivo más compatibles con el medio ambiente.
CONCLUSIONES
Los cinco aislamientos mexicanos fueron positivos a los cebadores β–tubulina específicos para la especie P. chlamydosporia var. chlamydosporia y cumplen con los requisitos básicos para ser considerados como agentes potenciales en el control biológico de N. aberrans. La producción de clamidosporas en el sustrato de trigo molido–arena de cuarzo fue mayor que en el de arroz precocido. Todos los aislamientos produjeron clamidosporas con germinación superior al 80%. Los métodos ERIC–PCR y RAPD–PCR detectaron diferencias entre aislamientos. El uso de cebadores específicos reduce el tiempo en las fases de detección y aislamiento del hongo en muestras de suelo y del nematodo (Atkins et al., 2005). Es necesario continuar la búsqueda de aislamientos de Pochonia spp. en distintas localidades y cultivos donde ocurre N. aberrans en la República Mexicana para evaluar su diversidad y eficiencia en el control biológico con base en criterios como los descritos en el presente trabajo.
AGRADEDIMIENTOS
El primer autor agradece al CONACYT (México) el apoyo brindado en sus estudios de maestría y al Consejo Británico (Educational Links) para realizar parte del presente estudio en el Reino Unido. Al Dr. Ken Evans por su ayuda en la revisión del trabajo y a la Dra. Joana Bourne por su ayuda en la primera fase del trabajo. Al Dr. Ángel Martínez Garza (in memoriam). Rothamsted Research recibe fondos de apoyo del Consejo de Investigación de Biotecnología y Ciencias Biológicas del Reino Unido (BBSRC).
LITERATURA CITADA
Arora, D.K., Hirsch, P.R., and Kerry, B.R. 1996. PCR–based molecular discrimination of Verticillium chlamydosporium isolates. Mycological Research 100:801–809. [ Links ]
Atkins, S.D., and Clark, I.M. 2005. Molecular Microbial Ecology Manual. Rothamsted Research (Internal Document), Harpenden, U.K. 22 p. [ Links ]
Atkins, S.D., Clark, I.M., Pande, S., Hirsh, P.R., and Kerry, B.R. 2005. The use of real–time PCR and species–specific primers for the identification and monitoring of Paecilomyces lilacinus. Federation of European Microbiological Societies, Microbiology Ecology 51:257–264. [ Links ]
Atkins, S.D., Hidalgo–Díaz, L., Clark, I.M., Morton, O., Montes de Oca, N., Gray, P., and Kerry, B.R. 2003. Approaches for monitoring the release of Pochonia chlamydosporia var. catenulata, a biological control agent of root–knot nematodes. Mycological Research 107:206–212. [ Links ]
Barker, K.R., and Campbell, C.L. 1981. Sampling nematode populations. pp. 451–474. In: B.M. Zuckermann, and R.A. Rhode (eds.). Plant Parasitic Nematodes Vol. III. Academic Press. New York, USA. 508 p. [ Links ]
Barnett, H.L., and Hunter, B.B. 1987. Illustrated Genera of Imperfect Fungi. Burgess Publishing Company. Minneapolis, MN, USA. 241 p. [ Links ]
Bourne, J.M., Kerry, B.R., and De Leij, F.A.A.M. 1996. The importance of the host plant on the interaction between root–knot nematodes (Meloidogyne spp.) and the nematophagous fungus, Verticillium chlamydosporium Goddard. Biocontrol Science and Technology 6:539–548. [ Links ]
Butterfield, E.J., and De Vary, J.E. 1977. Reassessment of soil assays for Verticillium dahliae. Phytopathology 67:1073–1078. [ Links ]
Carder, J.H., Segers, R., Butt, T.M., Barrara, D.J., Mende, N.V., and Coosemans, J. 1993. Taxonomy of the nematophagous fungi Verticillium chlamydosporium and V. suchlasporium based on secreted enzyme activities and RFLP analysis. Journal of Invertebrate Pathology 62:178–184. [ Links ]
De Leij, F.A.A.M., and Kerry, B.R. 1991. The nematophagous fungus Verticillium chlamydosporium Goddard, as a potential biological control agent for Meloidogyne arenaria (Neal) Chitwood. Revue de Nématologie 14:157164. [ Links ]
Domsch, K.H., and Gams, W. 1980. Compendium of Soil Fungi. Vol. I. Academic Press. London, UK. 859 p. [ Links ]
Edlind, T.D., Li, J., Visvesvara, G.S., Vodkin, H.H., McLaughlin, GL., and Katiyar, S.K. 1996. Phylogenetic analysis of β–tubulin sequences from a mitochondrial protozoa. Mycological Phylogenetics and Evolution 5:359–367. [ Links ]
Flores–Camacho, R., Manzanilla–López, R.H., Cid del Prado–Vera, I. y Martínez–Garza, A. 2007. Control of Nacobbus aberrans (Thorne, 1935) Thorne y Allen, 1944 with Pochonia chlamydosporia (= Verticillium chlamydosporium) (Goddard) Zare and W. Gams. Revista Mexicana de Fitopatología 25:26–34. [ Links ]
Folkertsma, R.T., Rouppe Van Der Voort, J.N.A.M., Van Gent–Pelzer, M.P.E., De Groot, K.E., Van Den Bos, W.J., Schots, A., Bakker, J., and Gommers, F.J. 1994. Inter and intra specific variation between populations of Globodera rostochiensis and G. pallida revealed by random amplified polymorphic DNA. Phytopathology 84:807–811. [ Links ]
Gams, W. 1988. A contribution to the knowledge of nematophagous species of Verticillium. Netherlands Journal of Plant Patholology 94:123–148. [ Links ]
Gams, W., and Zare, R. 2001. A revision of Verticillium sect. Prostrata. III. Generic classification. Nova Hedwigia 72:329–337. [ Links ]
Hidalgo–Díaz, L., Bourne, J.M., Kerry, B.R., and Rodríguez, M.G. 2000. Nematophagous Verticillium spp. in soils infested with Meloidogyne spp. in Cuba: isolation and screening. International Journal of Pest Management 46:277–284. [ Links ]
Hirsch, P.R., Mauchline, T.H., Mendum, T.A., and Kerry, B.R. 2000. Detection of the nematophagous fungus Verticillium chlamydosporium in nematode–infested plant roots using PCR. Mycology Research 104:435–439. [ Links ]
Hirsch, P.R., Atkins, S.D., Mauchline, T.H., Morton, C.O., and Kerry, B.R. 2001. Methods for studying the nematophagous fungus Verticillium chlamydosporium in the root environment. Plant and Soil 232:21–30. [ Links ]
Joaquim, T.R., and Rowe, R.C. 1990. Re–assessment of vegetative compatibility relationships among strains of Verticillium dahliae using nitrate non–utilizing mutants. Phytopathology 80:1160–1166. [ Links ]
Kerry, B.R. 1995. Ecological considerations for the use of the nematophagous fungus, Verticillium chlamydosporium, to control plant parasitic nematodes. Canadian Journal of Botany 73:565–570. [ Links ]
Kerry, B.R. 2001. Exploitation of the nematophagous fungal Verticillium chlamydosporium Goddard for the biological control of root–knot nematodes (Meloidogyne spp.). pp. 155–167. In: T.M. Butt, C. Jackson, and N. Magan (eds.). Fungi as Biocontrol Agents. Progress, Problems and Potential. CABI Publishing, Wallingford, United Kingdom. 309 p. [ Links ]
Kerry, B.R., and Bourne, J.M. 1996. The importance of rhizosphere interactions in the biological control of plant parasitic nematodes a case study using Verticillium chlamydosporium. Pest Science 47:69–75. [ Links ]
Kerry, B.R., and Bourne, J.M. 2002. A Manual for Research on Verticillium chlamydosporium, a Potential Biological Control Agent for Root–Knot Nematodes. IOBC/SORP. Darmstadt, Germany. 84 p. [ Links ]
Kerry, B.R., and Jaffee, B.A. 1997. Fungi as biological control agents for plant parasitic nematodes. The Mycota 4:203–218. [ Links ]
Klimyuk, V.I., Carroll, B.J., Thomas, C.M., and Jones, J.D.G. 1993. Alkali treatment for rapid preparation of plant material for reliable PCR analysis. Plant Journal 3:493–494. [ Links ]
Martínez–Garza, A. 1980. Introducción al SAS (Statistical Analysis System). Sistema para análisis estadístico. Centro de Estadística y Cálculo. Colegio de Postgraduados. Chapingo, Edo. de México. 176 p. [ Links ]
Mauchline, T.H., Kerry, B.R., and Hirsch, P. 2004. The biocontrol fungus Pochonia chlamydosporia shows nematode host preference at the infraspecific level. Mycological Research 108:161–169. [ Links ]
Mauchline, T.H., Kerry, B.R., and Hirsch, P. 2002. Quantification in soil and the rhizosphere of the nematophagous fungus Verticillium chlamydosporium by competitive PCR and comparison with selective plating. Applied and Environmental Microbiology 68:1846–1853. [ Links ]
Manzanilla–López, R.H., Costilla, M.A., Doucet, M., Franco, J., Inserra, R.N., Lehman, P.S., Cid del Prado–Vera, I., Souza, R.I., and Evans, K. 2002. The genus Nacobbus Thorne and Allen, 1944 (Nematoda: Pratylenchidae): Systematics, distribution, biology and management. Nematropica 32:149–227. [ Links ]
Morgan–Jones, G., and Rodríguez–Kabana, R. 1988. Fungi colonizing cysts and egg. pp. 39–58. In: G.O. Poinar, and H.B. Jansson (eds.). Diseases of Nematodes. Vol II. Press Inc. Boca Raton, Florida, USA. 150 p. [ Links ]
Morton, C.O., Mauchline, T.H., Kerry, B.R., and Hirsch, P. 2003. PCR–based DNA fingerprinting indicates host–related genetic variation in the nematophagous fungus Pochonia chlamydosporia. Mycological Research 107:198–205. [ Links ]
Reid, A., Manzanilla–López, R.H., and Hunt, D.J. 2003. Nacobbus aberrans (Thorne, 1935) Thorne and Allen, 1944 (Nematoda: Pratylenchidae); a nascent species complex revealed by RFLP analysis and sequencing of the ITS–rDNA region. Nematology 5:441–551. [ Links ]
Verdejo–Lucas, S., Sorribas, F.J., Ornat, C., and Galeano, M. 2003. Evaluating Pochonia chlamydosporia in a double–cropping system of lettuce and tomato in plastic houses infested with Meloidogyne javanica. Plant Pathology 52:521–528. [ Links ]
White, T.J., Bruns, T., Lee, S., and Taylor, J. 1990. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. pp. 315–322. In: M.A. Innes, D.H. Gelfand, J.S. Sninsky, and T.J. White. (eds). PCR Protocols. Academic Press. London, UK. 482 p. [ Links ]





![Reacción de Germoplasma Mejorado de Soya [Glycine max (L.) Merr.] a Macrophomina phaseolina (Tassi) Goidanich y Déficit Hídrico](/img/es/next.gif)








