Introducción
México se ubica entre los cinco países considerados megadiversos por su alta diversidad de especies, que representa aproximadamente el 12% del total mundial (Mittermeier et al. 1997). Sin embargo, la biodiversidad se ha visto amenazada por la deforestación de bosques y selvas, producto del cambio de uso de suelo con fines pecuarios y agrícolas, así como para el desarrollo urbano (FAO 2010). Al deforestarse grandes extensiones de selvas y bosques, se provoca la fragmentación o pérdida de hábitats de muchas especies (Wilcox & Murphy 1985). En este sentido, se entiende por fragmentación al proceso de división de un hábitat en una serie de parches más pequeños, aislados unos de otros (Wilcove et al. 1986), donde el grado de la fragmentación determina el tamaño y el aislamiento de los parches o fragmentos (Fahrig 2003).
La fragmentación o pérdida de hábitats tiene grandes efectos negativos, no solo en la riqueza y composición de especies (Wilcove et al. 1986; Fahrig 2003), pero también en la abundancia y distribución de muchas especies (Fahrig 2003). La magnitud de estos efectos dependerá del tiempo del aislamiento del fragmento, de la configuración y la estructura del paisaje, de la distancia y el grado de conectividad entre los fragmentos, del tamaño y forma del fragmento, de la posición de los fragmentos en el paisaje (Saunders et al. 1991), y de las características específicas de cada especie (Bicca-Marques 2003), que les permitirá su permanencia o extinción en el paisaje (Galindo-González 2007). Por ejemplo, los efectos de la fragmentación sobre las comunidades de murciélagos son la pérdida de especies con requerimientos especializados de hábitat, debido a que no pueden salir de los remanentes del bosque, y un incremento en la abundancia de especies generalistas, por su alta capacidad de desplazamiento en todo el paisaje (Galindo-González 2004; Galindo-González 2007; Sánchez 2009).
La fragmentación del hábitat también puede provocar el aislamiento poblacional a largo plazo, por la reducción en el tamaño poblacional y el debilitamiento de relaciones entre individuos (Kunz et al. 2007; Sánchez 2009), generando discontinuidades en el flujo génico entre poblaciones, e impactando en la estructura y diversidad genética (Holderegger & Di Giulio 2010). El aislamiento poblacional dependerá de la capacidad de respuesta de las especies a los disturbios en su hábitat (Galindo-González 2004). Por ejemplo, algunos murciélagos filostómidos frugívoros (subfamilias Carollinae y Stenodermatinae), frecuentemente aumentan su abundancia en los bosques perturbados (Fenton et al. 1992; Galindo-González 2004; Peters et al. 2006), pero son severamente afectados cuando ocurren cambios drásticos en sus refugios y sitios de forrajeo (Jones et al. 2001; Soriano & Ochoa 2001). Sin embargo, el murciélago zapotero Artibeus jamaicensis Leach, 1821 (subfamilia Stenodermatinae), bajo condiciones de perturbación del hábitat, puede mostrar una alta selectividad por el bosque tropical y baja abundancia en hábitats perturbados (Castro-Luna 2007). Así mismo, el huracán Georges (Puerto Rico, 1998) ocasionó el decline poblacional de A. jamaicensis, debido no solo a la muerte de muchos individuos por los fuertes vientos, sino también a la destrucción de sus refugios y sitios de forrajeo (Jones et al. 2001).
Artibeus jamaicensis es una de las especies más abundantes de murciélagos neotropicales (Ortega & Arita 1999), de amplia distribución geográfica en el continente americano (Eisenberg & Redford 1999), con alta movilidad (vuelo de larga distancia), se adapta fácilmente a cualquier tipo de hábitat y ocupa una amplia variedad de refugios (Ortega & Castro-Arellano 2001; Larsen et al. 2007). Es esencialmente frugívoro, y se alimenta de una amplia variedad de especies vegetales (Studier et al. 1983); pero si estos recursos son escasos puede consumir néctar, insectos y polen (Ortega & Castro-Arellano 2001). Lo anterior permite que generalmente A. jamaicensis no se vea afectado por la perturbación y fragmentación de sus hábitats, lo cual se refleja en su alta diversidad genética y alto flujo génico (Carstens et al. 2004; Vázquez-Domínguez et al. 2013).
Sin embargo, se han reportado poblaciones de A. jamaicensis con alta diferenciación genética dentro y entre ellas, a una escala geográfica fina (Landaverde-González et al. 2012). La escala geográfica juega un papel muy importante en el aislamiento poblacional, principalmente en murciélagos que pueden volar kilómetros de distancia entre fragmentos de vegetación (Rossiter et al. 2000), como A. jamaicensis. A pesar de la alta vagilidad de algunas especies de murciélagos, es posible encontrar alta diferenciación genética cuando se evalúan a gran escala geográfica (Miller-Butterworth et al. 2003; Juste et al. 2009).
Los murciélagos son considerados altamente móviles y, por lo tanto, son menos susceptibles a la fragmentación del hábitat que otros animales (Ripperger et al. 2013). Sin embargo, la capacidad de movilidad de cada especie determina su vulnerabilidad a la fragmentación de su hábitat, y su estructura genética (Meyer et al. 2009). Ripperger et al. (2013) estudiaron a escala geográfica fina (de aproximadamente 20 km), poblaciones de Artibeus watsoni Thomas, 1901 (Chiroptera: Phyllostomidae), y encontraron correlación de la diversidad genética con el tamaño de los parches. Así mismo, Carollia castanea Allen, 1890 (Chiroptera: Phyllostomidae) responde de mejor manera a la fragmentación del hábitat a pequeña escala que otras especies de murciélagos filostómidos, y su flujo génico disminuye a menudo que aumenta la distancia geográfica (Ripperger et al. 2014). Ambas especies, son frugívoras y comunes en bosques primarios y secundarios (Melo et al. 2009; Reid 2009).
En general, A. jamaicensis es de los filostómidos más estudiados a nivel molecular, con estudios enfocados en la filogenia (Lim et al. 2004), la filogeografía de especies (Phillips et al. 1991; Larsen et al. 2007; Fleming et al. 2009), y la genética de poblaciones (Pumo et al. 1996; Carstens et al. 2004; Landaverde-González et al. 2012). En México, A. jamaicensis también ha sido de los murciélagos más estudiados. En este sentido, los trabajos han mostrado alta diversidad genética y nula estructura poblacional de A. jamaicensis (Vargas-Miranda 2009; Vázquez-Domínguez et al. 2013); mientras Ortega et al. (2003) analizaron las relaciones entre machos dominantes y subordinados, y encontraron una relación de parentesco padre-hijo, y tampoco encontraron estructura poblacional. Ruiz et al. (2013) analizaron poblaciones de A. jamaicensis distribuidas desde América Central hasta el oeste de la península de Yucatán, de las cuales encontraron dos haplogrupos con una amplia diversidad genética y fuerte estructura genética. Los análisis de la historia demográfica de los dos haplogrupos, señalan que ambos sufrieron eventos sucesivos de expansión poblacional.
Aunque, A. jamaicensis ha sido de los murciélagos más estudiados genéticamente en México, solo un trabajo se ha enfocado en entender el efecto de las características ecológicas del hábitat sobre la diversidad y estructura genética de A. jamaicensis (Vázquez-Domínguez et al. 2013). Por lo tanto, es importante ampliar esta información, debido a que la diversidad genética de poblaciones locales de A. jamaicensis podría verse afectada por la modificación y fragmentación del hábitat. En el estado de Chiapas (México) la deforestación debida a la ganadería es muy frecuente (Covaleda et al. 2014), lo que podría afectar las poblaciones de murciélagos. Sin embargo, no se han realizado estudios genéticos de A. jamaicensis¸ para evaluar el impacto de diferentes hábitats en las poblaciones de A. jamaicensis. Por lo tanto, los objetivos de esta investigación fueron: (I) evaluar la diversidad genética, (II) determinar la estructura y la diferenciación genética, y (III) evaluar la historia demográfica de A. jamaicensis en dos hábitats diferentes en Chiapas.
Material y métodos
Área de estudio. Los individuos de A. jamaicensis se capturaron en dos localidades de selva mediana subperennifolia en la región fisiográfica Llanura Costera del Pacífico, Chiapas, con una distancia entre ellas de 29 km (Fig. 1). Se consideraron a priori como dos poblaciones por la distancia y condiciones contrastantes del hábitat. La primera localidad se conoce como Pantaleón (Mpio. de Mapastepec; 15°31´33.0” N - 92°49´21.6” W; 585 msnm), este sitio comprende vegetación densa y nativa, en la cual se observó la presencia de especies raras de murciélagos, vulnerables a la perturbación de su hábitat, y reportadas en bosques maduros y densos. La segunda localidad es Manacal (Mpio. de Pijijiapan; 15°38´24.8” N - 93°04´23.8” W; 630 msnm), comprende aproximadamente 8 ha de vegetación modificada (selva con platanar, árboles ornamentales y comestibles) y rodeada de grandes extensiones de potreros.
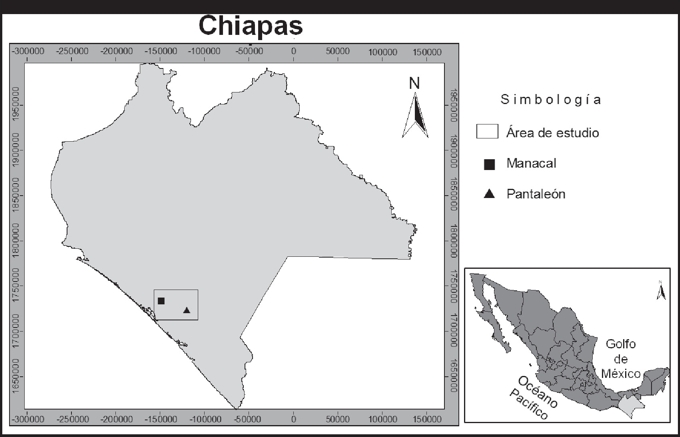
Figura 1 Área de muestreo de Artibeus jamaicensis, en dos hábitats contrastantes en Chiapas, México.
Colecta de muestras. Se utilizaron dos redes de niebla de 2 × 12 m colocadas entre senderos de las 18:00 a 24:00 horas y revisadas cada media hora. Los individuos de A. jamaicensis se identificaron con base a la clave de identificación de Medellín et al. (2008). Se capturaron 34 individuos de A. jamaicensis en abril de 2015: 19 individuos en Manacal y 15 individuos en Pantaleón. El ADN de A. jamaicensis se obtuvo de una pequeña porción de la membrana alar, cortando aproximadamente 3 mm del uropatagio con un bisturí estéril. Las muestras se colocaron en viales de 1.5 ml con alcohol al 96% hasta su posterior análisis genético.
Extracción, amplificación y secuenciación de ADN. La extracción de ADN se realizó siguiendo el protocolo de Fenol-Cloroformo-Alcohol isoamílico (Sambrook et al. 1989). Se amplificó un fragmento de ADN de la región mitocondrial D-loop con los iniciadores E (5´-CCT GAAGTA GGA ACC AGA TG-3´; Wilkinson & Chapman 1991) y P (5´-CCC CAC CAT CAA CAC CCA AAGCTGA-3´; Wilkinson et al. 1997). En cada amplificación se utilizó la técnica de reacción en cadena de la polimerasa (PCR), con un volumen total de 39 µl constituido por: 18 µl de mezcla maestra (Mastermix, PROMEGA), 15 µl de agua ultrapura, 2 µl iniciador E, 2 µl iniciador P, y 2 µl de ADN molde. Las amplificaciones se realizaron en un termociclador de gradiente MJ Mini (BIO-RAD) con el siguiente programa: ciclo inicial de 94 °C por 4 minutos, seguidos de 35 ciclos de 94 °C por 60 segundos, 55 °C por 90 segundos, 72 °C por 60 segundos, y un ciclo final de 72 °C por 10 minutos. El producto de las reacciones de PCR fueron visualizadas en electroforesis (100 V; 50 minutos), en gel de agarosa al 2% en medio TAE 1X con tinción de Bromuro de Etidio (1 g/100 ml en TAE 1X), con un marcador control de 100 pb (DNA ladder, PROMEGA). La purificación y secuenciación por electroforesis capilar (Sanger) de los productos de PCR se realizaron en la empresa Macrogen (Seúl, Corea del Sur).
Alineamiento y análisis de secuencias. Las 34 secuencias de A. jamaicensis se compararon con secuencias depositadas en el banco de genes (GenBank) para confirmar la identificación de la especie, con la ayuda de BLAST (Basic Local Alignment Search Tool; Altschul et al. 1990) de la base de datos NCBI (National Center for Biotechnology Information). Las secuencias y sus electroferogramas fueron revisadas y corregidas manualmente con el programa ChromasPro versión 1.5 (Technelysium Pty Ltd 2003-2009). Su alineación se realizó en ClustalX versión 2.0.11 (Larkin et al. 2007) con los parámetros por defecto.
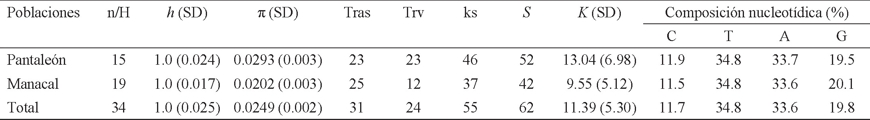
Diversidad genética. La diversidad genética total y por localidad se estimó con la diversidad haplotípica (ḣ = probabilidad de que dos haplotipos seleccionados al azar de una muestra sean diferentes) y la diversidad nucleotídica (π = número promedio de diferencias nucleotídicas por sitio entre dos secuencias escogidas al azar; Nei 1987), usando el programa DnaSP versión 5 (Librado & Roza 2009). También se estimaron por localidad y en total, el número de sitios segregados o sitios polimórficos (S), el número de transiciones y transversiones observadas, el número de sustituciones sinónimas por sitio (ks), el número promedio de las diferencias nucleotídicas entre pares de secuencias (K) y la composición nucleotídica. Todos estos parámetros se determinaron con el programa Arlequín versión 3.1 (Excoffier et al. 2006).
Diferenciación y estructura genética. La diferenciación genética se estimó con los estadísticos de Hudson et al. (1992) y el índice de fijación FST (Wright 1951). Los primeros se basan en el valor de HST = 1 - (HS / HT), donde HS es el promedio ponderado de la diversidad haplotípica de las subpoblaciones y HT es la estimación de la diversidad haplotípica de la población total. Adicionalmente, se estimó el valor de KST = 1 - (KS / KT), donde KS es el promedio ponderado de K1 y K2 (número promedio de las diferencias entre las secuencias en subpoblaciones 1 y 2, respectivamente) y KT es el promedio de las diferencias entre dos secuencias de la muestra total (Hudson et al. 1992). La significancia de los estadísticos de Hudson et al. (1992) se evaluó con la Chi-cuadrada y con 1,000 repeticiones en el programa DnaSP versión 5 (Librado & Rozas 2009), y el FST con 10,000 repeticiones en el programa Arlequín 3.1 (Excoffier et al. 2006). Para determinar cómo se encuentra repartida la variación genética en distintos niveles jerárquicos (entre y dentro poblaciones), se realizó un análisis de varianza molecular (AMOVA) con 10,000 repeticiones para las pruebas de significancia e intervalo de confianza al 95 %, en el programa Arlequín 3.1 (Excoffier et al. 2006).
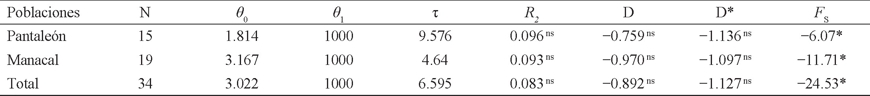
Historia demográfica. Se examinó la historia demográfica de A. jamaicensis por localidad y en total, con base en la distribución de diferencias nucleotídicas pareadas (distribución mismatch) (Slatkin & Hudson 1991) y el modelo de neutralidad. El primero se basa en tres parámetros: teta inicial (θ 0 = tamaño poblacional antes del proceso de expansión), teta final (θ 1 = tamaño poblacional después del proceso de expansión) y τ (tau = tiempo de expansión en unidades de mutaciones) (Rogers & Harpending 1992). La distribución mismatch multimodal indica equilibrio demográfico, y la unimodal una expansión demográfica poblacional relativamente reciente (Slatkin & Hudson 1991; Rogers & Harpending 1992). La significancia estadística se evaluó con 10,000 repeticiones, bajo la hipótesis nula del modelo de expansión demográfica repentina reciente, mediante la suma de desviaciones al cuadrado (SSD; por sus siglas en inglés) en el programa Arlequín versión 3.1 (Excoffier et al. 2006), y el estadístico R2 (Ramos-Onsins & Rozas 2002) en el programa DnaSP versión 5 (Librado & Rozas 2009).
El modelo de neutralidad tiene como hipótesis nula que la variación molecular es el resultado de un balance entre las mutaciones neutrales o al azar, y la extinción de mutaciones por deriva génica, manteniendo las frecuencias génicas en equilibrio (Kimura 1991; Perfectti et al. 2009). Para buscar desviaciones en este modelo se utilizaron los estadísticos D (Tajima 1989), D* (Fu & Li 1993) y F S (Fu 1997). Los valores negativos de estos estadísticos son esperados en poblaciones que han experimentado una expansión demográfica poblacional relativamente reciente o efecto de selección purificadora. Los valores positivos indican procesos de selección balanceadora (o equilibrada) o reducción del tamaño poblacional (Kaplan et al. 1989; Tajima 1989). El nivel de significancia estadística de las tres estimaciones se evaluó con 10,000 repeticiones (con simulación coalescente) en el programa DnaSP versión 5 (Librado & Rozas 2009).
Análisis genealógicos. Las relaciones a nivel intraespecífico de A. jamaicensis se analizaron a partir de la construcción de una red de haplotipos, a través del método de Median-Joining de acuerdo al criterio de Bandelt et al. (1999) y máxima parsimonia, implementado en el programa Network versión 4.6.1.3. (Fluxus Technology Ltd 2012). Los parámetros empleados fueron: épsilon de 0, peso transiciones- transversiones 1/1, peso de caracteres 5/10 y el criterio de costo de conexión.
Resultados
Se analizaron 34 individuos de A. jamaicensis (18 machos y 16 hembras): 19 individuos (9 machos y 10 hembras) de Manacal y 15 individuos (9 machos y 6 hembras) de Pantaleón. El alineamiento de las secuencias arrojó una matriz de 396 (bp) por secuencia, incluyendo los eventos de inserción-delección y los huecos producidos por el alineamiento (gaps). El análisis de los 396 pb reveló 62 sitios polimórficos y 34 haplotipos únicos.
Diversidad genética. La diversidad haplotípica (h) en ambas poblaciones tuvo un valor de 1 (Cuadro 1). La población Pantaleón presentó el mayor número de sustituciones y sitios segregados, lo que originó un valor de π = 0. 0.0293, ligeramente mayor que el observado en Manacal (π = 0.0202; Cuadro 1). La composición nucleotídica total y en ambas localidades, estuvo representada mayormente por Timinas (T) y Adeninas (A; Cuadro 1).
Cuadro 1 Diversidad genética por localidad, y en total, de Artibeus jamaicensis provenientes de dos hábitats contrastantes en Chiapas (México), basada en un segmento de 396 bp del D-loop. Las abreviaturas son: tamaño de la muestra (n), número de haplotipos (H), diversidad haplotípica (h), diversidad nucleotídica (π), número de transiciones observadas (Tras), número de transversiones observadas (Trv), número de sustituciones sinónimas (ks), número de sitios polimórficos o número de segregados (S), número promedio de las diferencias nucleotídicas (K), composición nucleotídica (C = Citocina, T = Timina, A = Adenina, G = Guanina), y desviación estándar (SD).
Diferenciación y estructura genética. De acuerdo con los estadísticos de Hudson et al. (1992) las poblaciones de Pantaleón y Manacal no presentaron diferenciación genética y subdivisión (H ST = 0.00; χ2 = 34.0, P = 0.42, g.l. = 33). Sin embargo, basadas en las diferencias nucleotídicas, se pudo observar diferenciación genética significativa, con los dos tipos de parámetros determinados, los parámetros de Hudson et al. (1992) (K S = 9.33, K ST = 0.03; P = 0.019), y el índice de fijación (FST = 0.054; P = 0.00079). La prueba de AMOVA (para 62 sitios polimórficos) reveló una ligera diferenciación con el 94.62 % de la variación genética repartida entre individuos dentro de las localidades, mientras que el 5.38 % se debe a variaciones entre las localidades (Cuadro 2).
Cuadro 2 Análisis de Varianza Molecular (AMOVA) de 34 secuencias de Artibeus jamaicensis de dos hábitats contrastantes en Chiapas (México), realizado a través de 10,000 repeticiones (P < 0.01) en el programa Arlequín 3.1 (Excoffier et al. 2006).

Historia demográfica. La distribución mismatch, con varios picos ligeramente marcados, difiere de lo esperado bajo el modelo de expansión poblacional repentina, dando lugar a una distribución multimodal (Fig. 2). Sin embargo, la desviación no fue significativa en Pantaleón (SSD = 0.0106; P = 0.39), Manacal (SSD = 0.005; P = 0.64) y en total (SSD = 0.001; P = 0.90). Así mismo, R2 no es estadísticamente significativo para Pantaleón (P = 0.06), Manacal (P = 0.08) y en total (P = 0.13). Los valores negativos de los estadísticos D y D* no son estadísticamente significativos (P > 0.10). No obstante, el estadístico F S es significativamente negativo (P < 0.02) en ambas localidades y en total (Cuadro 3).

Figura 2 Distribución mismatch de los haplotipos de Artibeus jamaicensis de dos hábitats contrastantes en Chiapas (México), sobre un modelo de expansión poblacional. La línea sólida indica la distribución esperada y la punteada la observada. A) Localidad Pantaleón, B) Localidad Manacal, y C) Total de los individuos.
Cuadro 3 Pruebas estadísticas de la distribución de las diferencias pareadas (distribución mismatch) e índices de neutralidad de Artibeus jamaicensis de dos hábitats contrastantes en Chiapas (México), basadas en un segmento de 396 pb de la región mitocondrial D-loop: tamaño de la población (N), teta inicial (θ 0 = 2µN0), teta final (θ 1 = µN1), tau (τ = 2µt), estadístico R2 (Ramos-Onsins & Rozas 2002), estadístico D (Tajima 1989), estadístico D* (Fu & Li 1993) y estadístico F S (Fu 1997). Nivel de significancia estadística: ns P > 0.05 (no significativo); *P ≤ 0.05 (significativo).
Análisis genealógicos. Los valores de parsimonia obtenidos para la reconstrucción de la red de haplotipos fueron altos en todos los pasos (P > 0.95). Se identificaron 34 haplotipos únicos, los cuales se conectaron entre sí parsimoniosamente entre 1 y 9 pasos mutacionales (Fig. 3). No se observaron haplotipos agrupados de acuerdo a la ubicación geográfica, los haplotipos de Pantaleón se mezclan con los de Manacal.

Figura 3 Red de haplotipos de Artibeus jamaicensis de dos hábitats contrastantes en Chiapas (México), construida en Network versión 4.6.1.3, con el método de máxima parsimonia usando un segmento de 396 pb de la región mitocondrial D-loop. Los rectángulos representan los haplotipos de la localidad Pantaleón (Pan), y las elipses representan los haplotipos de la localidad Manacal (Man). Los puntos negros representan un haplotipo hipotético ancestral y las líneas representan los pasos mutacionales (hembras = ♀; machos = ♂).
Discusión
Artibeus jamaicensis se adapta fácilmente a diferentes tipos de hábitat, posee una alta movilidad y alta abundancia, lo cual le facilita un flujo génico alto entre sus poblaciones (Rossiter et al. 2012; Vázquez-Domínguez et al. 2013). Estos aspectos de la historia natural de A. jamaicensis dan lugar a los altos niveles de diversidad genética, y la baja diferenciación genética encontrados en las poblaciones de Pantaleón y Manacal. Además, la distancia geográfica entre los dos hábitats podría no ser suficiente para aislar las poblaciones de A. jamaicensis, por su alta capacidad de vuelo.
Los valores de diversidad genética observados en la presente investigación son consistentes con lo reportado en estudios moleculares basados en el ADNmt de A. jamaicensis, los cuales señalan que esta especie posee una alta diversidad genética con valores de h cercanos a 1.0 (Carstens et al. 2004; Ruíz et al. 2013; Del Real-Monroy et al. 2014). El impacto de la fragmentación del hábitat en la conectividad genética, a escala geográfica fina, se ha evaluado también en otras especies frugívoras y de hábitos generalistas, como Artibeus watsoni, mostrando una h de 0.90 a 0.99 (Ripperger et al. 2013), y Carollia castanea con una h de 0.86 a 0.97 (Ripperger et al. 2014), valores similares a los reportados en este estudio para A. jamaicensis. Es posible que la tolerancia a los hábitats perturbados y la alta movilidad de estos murciélagos, los hace menos susceptibles a la fragmentación del hábitat, y les permiten mantener el intercambio genético entre las poblaciones, en comparación con otros murciélagos (Ripperger et al. 2013; Ripperger et al. 2014).
En el presente estudio se encontró un haplotipo por individuo en ambas poblaciones, lo cual se refleja en su diversidad muy alta (h = 1). Esto podría deberse al tamaño del muestreo (34 individuos), el cual es pequeño comparado con otros estudios (de 49 a 193 individuos; Carstens et al. 2004; Fleming et al. 2009; Ruíz et al. 2013; Del Real-Monroy et al. 2014), y a la ubicación de las redes de colecta en sitios con disponibilidad de alimentos. Además, el D-loop tiene una alta tasa de evolución, de sustitución y polimorfismo (Lunt et al. 1998), que promueve altos valores de diversidad genética.
Los bajos valores de diversidad nucleotídica (π < 0.03) reportados en este estudio son congruentes con lo registrado en otros estudios de A. jamaicensis, los cuales oscilan entre 0.009 y 0.1086 (Carstens et al. 2004; Fleming et al. 2009; Ruíz et al. 2013; Del Real-Monroy et al. 2014). La diversidad nucleotídica de la localidad Pantaleón (π = 0.029) es ligeramente mayor a la de Manacal (π = 0.020), debido a un mayor número de sitios polimórficos en Pantaleón y a las pocas mutaciones presentes en diferentes sitios polimórficos. Una alta diversidad haplotípica acompañada de una baja diversidad nucleotídica, significa que los haplotipos difieren en un número reducido de nucleótidos, lo cual es característico de efectos de expansión demográfica poblacional en un tiempo relativamente reciente (Hamilton 2009).
La localidad Pantaleón comprende una amplia extensión de vegetación densa y continua, donde se observaron especies raras de murciélagos, vulnerables a la perturbación de su hábitat y reportadas en bosques maduros y densos como Centurio senex Gray, 1842 (Chiroptera: Phyllostomidae), Lonchorhina aurita Tomes, 1863 (Chiroptera: Phyllostomidae) y Rhogeessa sp. (Chiroptera: Vespertilionidae; Schulze et al. 2000; Gómez-Nísino 2006; Ávila-Cabadilla et al. 2009). La cantidad de árboles y la variedad de especies florísticas que conforman esta vegetación, podrían proporcionar mayor cantidad y calidad de refugios, y mayor disponibilidad de alimento para A. jamaicensis en comparación con lo que ofrece la poca cobertura vegetal de la localidad Manacal, la cual es un fragmento de vegetación aislado, resultado de la ganadería extensiva de la zona. No obstante, Manacal alberga árboles ornamentales y frutales, como la almendra (Terminalia catappa Linneo, 1767) y el mango (Mangifera indica Linneo, 1876); de los cuales sus frutos son frecuentemente consumidos por A. jamaicensis (Kunz & Díaz 1995). Por lo tanto, ambas localidades proporcionan los recursos necesarios para la persistencia de estos murciélagos, lo cual mantiene sus altos niveles de diversidad genética.
Los recursos que ofrece Manacal para A. jamaicensis son suficientes para que sus individuos no busquen recursos en otros sitios lejanos como Pantaleón. Cuando estos murciélagos tienen refugios con condiciones adecuadas para alimentarse, y tener a sus crías, se mueven a cortas distancias (Ortega et al. 2008). Si hay alimento se mueven máximo a 600 m de los refugios, pero en hábitats con poca disponibilidad de alimento pueden volar hasta 8 km por noche (Morrison 1978; Ortega & Castro-Arellano 2001; Ortega et al. 2002). Aunque algunos estudios genéticos sugieren que la distancia geográfica parece no influir en la estructura genética de A. jamaicensis (Vargas-Miranda 2009; Vázquez-Domínguez et al. 2013), la distancia geográfica entre Pantaleón y Manacal es suficiente para dar lugar a cierta estructura genética de A. jamaicensis, como se ha reportado en zonas núcleo de la Reserva de la Biosfera Maya en Petén, Guatemala (Landaverde-González et al. 2012).
No obstante, existen otros factores que contribuyen o facilitan el flujo génico entre poblaciones de A. jamaicensis en Pantaleón y Manacal, como la dinámica en sus relaciones sociales bajo el sistema de apareamiento poligínico, donde la reproducción no se restringe a las cópulas entre miembros del mismo grupo social (Clutton-Broock 1988), y los individuos se mueven constante y temporalmente entre los grupos sociales dentro de una misma población o entre diferentes poblaciones (Ortega et al. 2004). La aclimatación a distintos tipos de hábitats (Clutton-Broock 1988), y la alta vagilidad de A. jamaicensis, le facilita un flujo génico alto entre sus poblaciones, lo que reduce su diferenciación genética (Carstens et al. 2004; Rossiter et al. 2012; Vázquez-Domínguez et al. 2013; Del Real-Monroy et al. 2014). La conexión entre las poblaciones de A. jamaicensis es sostenida por el resultado del AMOVA, el cual indica que el mayor porcentaje de variación genética se reparte entre los individuos dentro de las poblaciones (94.62%).
El análisis de las distribuciones mismatch muestra para ambas localidades y en total, curvas multimodales, las cuales sugieren poblaciones en equilibrio demográfico. Sin embargo, la forma de la distribución no es suficiente para rechazar la hipótesis nula de expansión demográfica poblacional relativamente reciente (Yun-Dong et al. 2015). Un proceso de expansión poblacional reciente produce un patrón unimodal sesgado hacia la izquierda de la distribución mismatch, debido a las comparaciones entre secuencias idénticas o similares (Rogers & Harpending 1992). Esto es observado en las distribuciones mismatch de ambas localidades, sugiriendo que A. jamaicensis experimentó una expansión demográfica poblacional repentina y relativamente reciente en su historia evolutiva, lo cual también es apoyado por la R2 y SSD.
Los valores negativos de las pruebas de neutralidad D, D* y F S para ambas localidades, y en total, también se asocian a una rápida expansión poblacional. Los valores negativos en las pruebas de neutralidad se dan porque los alelos raros son más numerosos de lo esperado, señalando poblaciones que han experimentado una expansión reciente (Ramos-Onsins & Rozas 2002). Los valores no fueron significativos para D y D*, pero si para F S. De acuerdo a Fu (1997), la prueba F S es más potente para detectar crecimiento poblacional y expansión demográfica reciente o selección, que el estadístico D (Tajima 1989), y generalmente arroja grandes valores negativos, como los obtenidos en este estudio.
Otros estudios también han señalado que A. jamaicensis experimentó una expansión demográfica repentina y reciente (Carstens et al. 2004; Larsen et al. 2007; Vargas-Miranda 2009; Ruíz et al. 2013), asociado a la glaciación del Pleistoceno (Phillips et al. 1991; Grant & Bowen 1998). Durante la última máxima glaciación, las regiones templadas fueron cubiertas por grandes extensiones de hielo, obligando a las especies a desplazarse hacia refugios en diferentes áreas geográficas (Hewitt 2000). Se asume que el crecimiento poblacional y la expansión geográfica produjeron, probablemente, una rápida diversificación de haplotipos con mínimas diferencias nucleotídicas (Grant & Bowen 1998). Es por ello, que se esperan poblaciones actuales con alta diversidad haplotípica y baja diversidad nucleotídica, las cuales están asociados a eventos de crecimiento poblacional y colonización, durante y después, de las glaciaciones del Pleistoceno (Shiang-Fan et al. 2006).
El patrón de diversidad haplotídica y nucleotídia de A. jamicensis en Pantaleón y Manacal coincide con lo esperado en poblaciones que han experimentado una reciente expansión demográfica. No obstante, los tiempos de divergencia y expansión inferidos a través de datos moleculares son hipotéticos y los estimadores poseen errores grandes (Hillis et al. 1996). Por otro lado, el tamaño de muestra y poblaciones pueden generar un sesgo en la estimación de parámetros poblacionales, por lo que la expansión demográfica y geográfica reciente de A. jamaicensis deber tomarse como una hipótesis que requiere ser sometida a prueba en estudios futuros.
La topología de la red de haplotipos mostrada en el presente estudio, donde a partir de un haplotipo central surgen varios haplotipos separados por un único paso mutacional, también son indicios de una reciente expansión demográfica poblacional (Forster 2004). En la red de haplotipos la distribución geográfica no se relaciona con la distribución de los haplotipos. En el interior como en los extremos de la red, los haplotipos de ambas localidades se mezclan. La ausencia de una división geográfica en la red de haplotipos puede deberse a la escala geográfica fina del estudio y a la alta vagilidad de A. jamaicensis. Sin embargo, se ha observado la mezcla de haplotipos entre poblaciones insulares de A. jamaicensis separadas por grandes distancias (> 100 km de océano; Carstens et al. 2004).
Por tratarse de una especie con una historia evolutiva reciente (Ruíz et al. 2013) hace falta incluir poblaciones de diferentes lugares de México, para identificar qué procesos demográficos históricos han dejado marcas evolutivas en la distribución geográfica actual de los haplotipos de A. jamaicensis. Para futuros trabajos genéticos se recomienda ampliar el área de estudio, aumentar el tamaño de muestra y el tamaño del fragmento de ADN, así como complementar la información de un gen mitocondrial con un gen nuclear, para tener un mejor panorama de las implicaciones de la modificación del hábitat en la diversidad genética de A. jamaicensis.
Conclusiones
Las poblaciones de A. jamaicensis en Manacal y Pantaleón presentan altos niveles de diversidad genética, lo cual es indicio de que los hábitats de ambas localidades ofrecen los recursos necesarios para la permanencia de los individuos dentro de las poblaciones, y para el mantenimiento de su alta diversidad genética. Al tener los recursos necesarios en ambas localidades, da lugar al desplazamiento reducido de estos murciélagos en el área de estudio, lo cual se refleja en cierta diferenciación genética. A pesar de lo anterior, existen otros factores que permiten la conexión e interacción entre ambas poblaciones de A. jamaicensis como sus atributos de especie generalista, su dinámica social bajo el sistema poligínico, y su alta capacidad de desplazamiento.











 nueva página del texto (beta)
nueva página del texto (beta)


