Introducción
La apertura de México al libre comercio ha incrementado el riesgo de la introducción de plagas exóticas, que por sus características fisiológicas causan pérdidas económicas, de salud y ambientales (FAO-IPPC 2007). Actualmente, en diversos cultivos de interés económico, se tienen bajo vigilancia 31 plagas identificadas como de alto riesgo, entre las que se encuentran el complejo de escarabajos ambrosiales o escolítidos del género Xyleborus (Coleoptera: Curculionidae: Scolytinae) (SENASICA 2015). La información relativa a especies de Xyleborus en México ha ido en aumento debido a que algunas especies representan riesgo para algunos árboles de importancia económica, tal es el caso de Xyleborus affinis (Eichhoff, 1868), X. ferrugineus (Fabricius, 1801), X. volvulus (Fabricius, 1775) y X. glabratus (Eichhoff, 1877). Esta última, aunque no se encuentra reportada en México, es considerada plaga potencial debido a que actúa como vector primario del hongo patógeno Raffaelea lauricola T.C. Harr., Fraedrich & Aghayeva que causa la “marchitez del laurel”, una enfermedad vascular letal para las plantaciones de aguacate Persea americana Mill. Lauraceae (Pérez et al. 2015b). La rápida dispersión de la plaga en EUA, aunado a la amplia disposición de hospederos susceptibles en varias entidades de México (Loera 2014), permiten prever que esta especie puede representar a corto plazo una importante amenaza fitosanitaria (SENASICA 2015). Por tanto, contar con diagnóstico preciso y oportuno, será crítico en la prevención de invasión de la especie exótica X. glabratus.
El diagnóstico tradicional por caracteres morfológicos específicos de especies del género Xyleborus generalmente es difícil, debido a la similitud morfológica existente entre las especies, o porque los ejemplares no cuentan con la calidad requerida para su determinación (Kuerová et al. 2009, Pérez-De La Cruz et al. 2009, Pérez et al. 2015a). Dados estos inconvenientes se han desarrollado técnicas moleculares que permiten determinar con rapidez y sensibilidad la identidad de los organismos (Cognato et al. 2011), mediante el uso de marcadores moleculares. En este sentido, Chang et al. (2013) identificaron 32 especies del género Xyleborus al desarrollar códigos de barras mediante una PCR dirigida a dos regiones del gen mitocondrial Citocromo Oxidasa subunidad I (CO1). También se han empleado técnicas moleculares para definir la filogenia de Xylosandrus (Dole et al. 2010), y se ha hecho uso de la secuenciación de proteínas de la subunidad grande del ribosoma (LSU rADN), para la determinación de los hongos asociados con X. glabratus (Harrington et al. 2010).
La PCR ha ganado aceptación debido a su rapidez, sensibilidad y reproducibilidad en la determinación específica por lo que en este estudio se propuso como objetivo validar y estandarizar esta herramienta como elemento de diagnóstico de especies de Xyleborus.
Materiales y métodos
Material biológico. Se utilizaron individuos donados a partir de la colección entomológica del Centro Nacional de Referencia Fitosanitario (CNRF) y del Colegio de Postgraduados, Campus Montecillo, México, previamente identificados mediante caracteres taxonómicos por el personal del CNRF, Dr. Armando Equihua Martínez y M.C. Mauricio Pérez Silva. Las muestras incluían individuos pertenecientes al género Xyleborus, colectados en diferentes trampas con luz instaladas en plantaciones de cacao y aguacate en Tabasco y Michoacán, México y Florida, EUA (Cuadro 1) y preservados en seco o etanol a 70%. En todos los individuos se documentaron las características morfológicas distintivas de cada ejemplar utilizando un fotomicroscopio Tesovar (Carl Zeiss), para posteriormente realizar la extracción de ADN.
Cuadro 1 Especies del género Xyleborus, utilizados para la extracción de ADN y amplificación del gen CO1, colectados en trampas de luz instaladas en plantaciones de cacao y aguacate en Tabasco y Michoacán, México y Florida, EUA.
Extracción de ADN a partir de especímenes de Xyleborus. Previo a la extracción de ADN, los especímenes se lavaron, tres veces con 500 µL de TAE 1X durante 5 min, para eliminar el etanol. Cada espécimen se colocó en una sanita estéril, y se dejó secar por 48 h. El ADN se extrajo individualmente de hembras adultas del género Xyleborus, empleando el kit de Qiagen DNeasy® mericom Food [69514] (DMF) (Qiagen 2010) siguiendo las instrucciones marcadas por el fabricante, con las siguientes modificaciones: 1) se utilizó un solo espécimen por tubo de reacción, macerando el insecto con 100 µL de buffer de lisis y con la ayuda de un micropistilo (Diagger Company); 2) el volumen de los reactivos fue de 0.3 µL de Proteinasa K; 100 µL de cloroformo, buffer PB y AW2; y 3) el ADN se recuperó con 20 µL de buffer EB. En esta primera etapa se realizaron extracciones de dos ejemplares de X. affinis y dos de X. volvulus. Una vez verificada la presencia de ADN mediante la medición por espectrofotometría (Nanodrop®), se realizó la extracción con ejemplares representantes de siete especies.
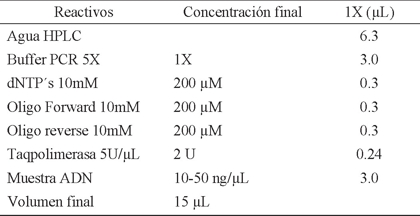
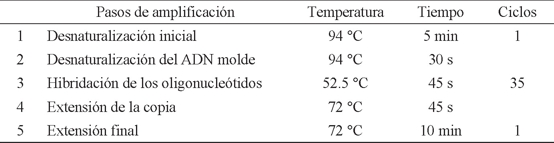
Estandarización y validación del PCR. La estandarización y validación se realizó con ejemplares del género Xyleborus y para comprobar que se obtuvo la calidad mínima de ADN extraído para las reacciones de PCR se realizaron reacciones de amplificación con dos pares de oligos dirigidos al gen CO1, mediante el uso de los oligos externos reportados por Cognato y Sperling (2000): CI-J-2183 5´-CAACATTT ATTTTTGATTTTTTGG-3´ y TL2-N-3014 5´-TCCAATGCACTAAT CTGC CATATTA-3´ y los oligos internos reportados por Chang et al. (2013) J2210 5´-TCGCAT ATTATTAGGCAAGAAAGAG-3´ y N2739 5´-AGAAAT GTTGTG GGAAGAAAG-3. La primera amplificación generó un fragmento de aproximadamente 1300 pb, y la segunda amplificación un fragmento de ~500 pb. En la estandarización de la amplificación se evaluaron dos polimerasas comerciales de Promega (Go Taq® Flexi (M8295) y Go Taq® (M3005)) (Promega 2015), se ajustaron las temperaturas de alineamiento, así como la concentración de los componentes de la reacción (Bolívar et al. 2014). La reacción de PCR se optimizó para realizar en un volumen final de entre 10 y 15 µL por reacción.
Límite de detección. Fue pertinente establecer el límite de detección (LD) de las muestras de ADN obtenido considerando que el tamaño de los especímenes de Xyleborus es muy pequeño (2.3 mm) excepto X. horridus (4 mm) (Pérez et al. 2015a), y el peso no llega al mínimo sugerido por el kit comercial (200 mg), por lo que existía la posibilidad de extraer ADN insuficiente para la reacción de PCR. Para establecer el límite de detección se realizaron diluciones seriales con un factor de 10 a partir de las muestras de PCR amplificadas. Así se realizaron diluciones desde 10-1 hasta 10-5 (Jiménez et al. 2014), que correspondieron a 780 ng/µL (ADN sin diluir) y 0.078 pg/µL, respectivamente.
Posteriormente los productos de PCR se fraccionaron en un gel de agarosa a 1.5 % que se tiño con bromuro de etidio (0.5 µL). La electroforesis se realizó a 90 V por 40 min en una solución amortiguadora TAE 1X (4.68g de Tris Base 1M pH 8.0; 1.14 mL de ácido acético glacial y 2 g de EDTA 0.5M). Para su visualización se comparó el tamaño del amplicon con el marcador de peso molecular (MM) de 100 pb.
Análisis molecular. Los productos de la PCR obtenidos a partir de 26 individuos del género Xyleborus se limpiaron siguiendo el proceso establecido en el kit Wizard®SV Gel y PCR Clean-Up System (Promega® Cat: a9282) de acuerdo con las recomendaciones del proveedor. Este producto fue enviado a secuenciar al Instituto de Biotecnología de la UNAM (IBT UNAM) con los oligos internos (J2210 y N2739). Los electroferogramas se editaron y analizaron mediante un alineamiento por homología utilizado el programa Clustal W (http://www.ebi.ac.uk/Tools/msa/clustalo/) implementado en la versión 6.0 del programa Mega (Tamura et al. 2013).
Resultados
Identificación morfológica. Se corroboró la identidad de los ejemplares de acuerdo a la clave propuesta por Pérez et al. (2015a). La diferenciación de especies del género se hizo con base en caracteres morfológicos como las crenulaciones del pronoto, forma del mazo antenal, forma y ornamentaciones del margen anterior del pronoto, dientes en la protibia y declive elitral, entre otros (Fig. 1) (Wood 1986, Rabaglia et al. 2006, Pérez et al. 2015a). Además, las características distintivas de Xyleborus affinis, X. ferrugineus, X. glabratus, X. horridus, X. intrusus, X. spinulosus y X. volvulus (Fig. 1) fueron validadas por el M. en C. Pérez, especialista en el grupo, y que coincidieron con las reportadas en la literatura (Pérez-De La Cruz et al. 2009, Atkinson et al. 2013, Pérez et al. 2015a).

Figura 1 Vista lateral de especies de Xyleborus. A) Xyleborus glabratus, B) Xyleborus affinis, C) X. ferrugineus, D) X. intrusus, E) X. volvulus, F) X. spinulosus y G) X. horridus. Escala: 1mm.
Extracción de ADN a partir de especímenes de Xyleborus. Aun cuando el kit DFM de Qiagen® no es específico para insectos y no estaba documentado su uso para este grupo de organismos, resultó adecuado en la extracción de ADN a partir de un individuo de Xyleborus, de manera sencilla, rápida, reproducible con calidad y cantidad suficiente para realizar reacciones de amplificación del ADN. El total de las extracciones amplificaron un fragmento de ~500 pb correspondientes a una sección del gen CO1 (Fig. 2). Una utilidad práctica del kit DMF Qiagen® fue el hecho de que la mayoría de las muestras procesadas tenían una antigüedad de ocho años de conservación en etanol a 70% o en seco, como el caso de X. glabratus, por lo que su aplicación en material almacenado es evidente, sin embargo, se observó mejor rendimiento de ADN en las muestras con menor tiempo de preservación (Cuadro 2).
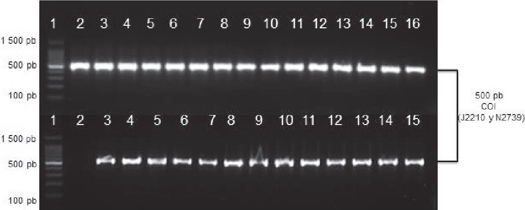
Figura 2 Amplificación del ADN extraído de diferentes muestras de Xyleborus con el Kit DMF Qiagen® dirigido al gen COI. Arriba: 1) Marcador de peso molecular (100 pb Ladder), 2-7) X. affinis, 8-13) X. ferrugineus, 14-16) X. horridus. Abajo: 1) MPM, 2) control negativo, 3) X. glabratus, 4-6) X. intrusus, 7-9) X. spinulosus, y 10-15) X. volvulus.
Cuadro 2 Rendimiento promedio de ADN extraído de diferentes especies de Xyleborus con el Kit DMF Qiagen® dirigido al gen CO1.

Estandarización y validación de la PCR. El ADN obtenido mediante el kit DMF Qiagen® fue evaluado en su calidad para realizar una reacción de amplificación. Antes de realizar dicho procedimiento se probaron las condiciones de amplificación debido a que algunas amplificaciones iniciales no tuvieron la reproducibilidad esperada, por lo que se procedió a estandarizar las condiciones de amplificación.
Se evaluaron dos polimerasas comerciales, varias temperaturas de alineamiento y diferentes concentraciones de cada uno de los componentes de la mezcla de reacción. Una vez evaluados los diferentes parámetros se definieron las condiciones que dieron como resultado alta reproducibilidad en las amplificaciones (Fig. 3) (Cuadro 3). La reacción de amplificación se estandarizó para un volumen final de 15 µL (Cuadro 4).
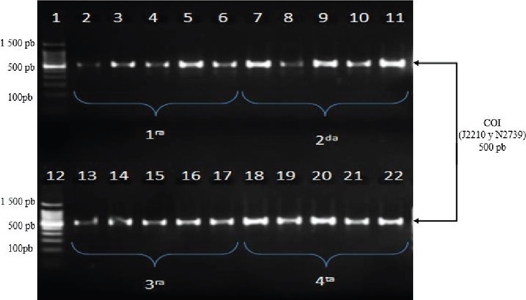
Figura 3 Reproducibilidad de la PCR-anidada, de acuerdo a las condiciones previamente estandarizadas y validadas. 1 y 12) MM 100 pb; 2-6) 1er ensayo del PCR con cinco repeticiones; 7-11) 2do ensayo del PCR con cinco repeticiones; 13-17) 3er ensayo del PCR con cinco repeticiones; 18-22) 4to ensayo del PCR con cinco repeticiones.
Cuadro 3 Programa de amplificación para especies del género Xyleborus, citado por Chang et al. (2013), con modificación en la temperatura de alineación.
Análisis molecular. El segmento amplificado correspondió a ~500 pb del gen CO1. Se observó 100 % de homología entre las secuencias de la misma especie, asimismo, se determinaron los especímenes con base en las características morfológicas distintivas. Las secuencias consenso fueron analizadas por homología y depositadas en el GenBank a través de la aplicación BLAST (Cuadro 5).
Cuadro 5 Número de acceso de secuencias consenso para especies del género Xyleborus derivadas de este estudio y depositadas en el GenBank.
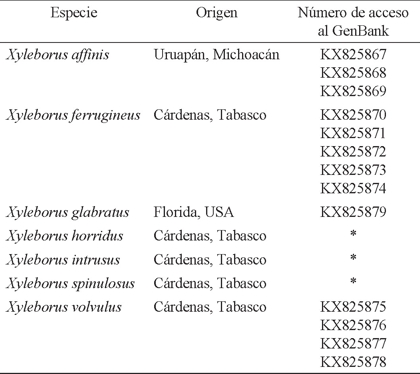
* En proceso de liberación por ser las primeras secuencias del gen CO1 depositadas en el GenBank para las especies.
Puesto que las amplificaciones se realizaron a partir de especies identificadas taxonómicamente para dar certidumbre, se realizaron las secuenciaciones de cada amplificación por triplicado. Con la secuencias consenso obtenidas de las tres secuencias, el análisis BLAST indicó que para X. affinis se obtuvo una homología de 92% con una de las secuencias para X. affinis presentes en el GenBank (AF187138.1); las secuencias generadas para X. spinulosus, X. horridus e X. intrusus, presentaron baja homología contra otras especies por no haber secuencias para CO1 disponibles para estas especies. Por tanto, estas son las primeras secuencias del gen CO1 depositadas en el GenBank para estas tres especies. La secuencia generada para X. ferrugineus presentó una identidad de 79% contra la secuencia JX424246.1 de la misma especie.
Discusión
El diagnóstico tradicional de especies del género Xyleborus por caracteres morfológicos específicos generalmente es difícil, debido a la similitud morfológica existente entre las especies. No obstante, mediante el uso de claves específicas como la propuesta por Pérez et al. (2015a) fue posible reconocer las características distintivas de las especies estudiadas.
En este estudio, se confirma que el uso del kit de extracción Qiagen DNeasy® mericon Food (69514) para obtener material genético de especies del género Xyleborus (preservados hasta por ocho años, en seco ó en alcohol a 70%) fue confiable y sensible pues el ADN extraído de un ejemplar de Xyleborus fue suficiente (10.46 a 61.1 ng/µL) para obtener el producto de la amplificación esperado. El método permitió también obtener ADN de calidad, partiendo incluso de solo una parte del individuo (X. horridus tamaño promedio 4.0 mm) debido a que se obtuvo buen producto de amplificación de la cabeza, abdomen y apéndices. Este resultado coincide con lo reportado por autores quienes señalan que la calidad del ADN extraído mediante el uso del kit DFM se debe principalmente a la utilización de CTAB (Bromuro de Cetil-Trimetil-Amonio); reactivo que ayuda a la eliminación de polisacáridos proteínas y polifenoles de la muestra, lo cual mejora la calidad del ADN extraído (Marzachi et al. 1998, Reineke et al. 1998, Kušec et al. 2015, Schwerer et al. 2015). Contrario a ello Volk et al. (2014), señalan que el bajo rendimiento de ADN (cantidad y calidad) obtenido con el kit DMF, junto con el alto costo, en comparación con otras metodologías, pone en duda el uso práctico del kit. No obstante, se debe considerar que la obtención de material genético para un análisis molecular, ya sea para confirmar identidad como en este trabajo, o para estudios filogenéticos, de diversidad o ecología molecular depende, con frecuencia, del tipo, tamaño y tiempo de preservación del material (Barrio-Caballero 2012, Martínez de la Puente et al. 2013), debido a que generalmente se obtienen mejores resultados con ejemplares grandes, frescos y correctamente conservados (Cornejo et al. 2014).
Otro factor que repercutió en la calidad de la amplificación fue la temperatura de alineamiento (Tm). Si bien se observó amplificación de calidad en las temperaturas de 49.1, 50.7 y 52.5 °C, valores cercanos a la Tm (47 °C) utilizada por Chang et al. (2013) en la PCR para el diagnóstico de Xyleborus, los mejores resultados se lograron con una Tm de 52.5 °C debido a que se generó un amplicon de mejor calidad en comparación con el resto de las temperaturas evaluadas (Bolívar et al. 2014, Cornejo et al. 2014).
Asimismo, los ensayos realizados para determinar la concentración de los dNTP´s y oligos indicaron que la amplificación de mejor calidad se logró con la concentración de 200 µM. Estos valores coinciden con los recomendados por diversos autores, quienes señalan que la concentración tanto de dNTP´s y oligos debe usarse con precisión debido a que un exceso o déficit puede ocasionar errores en la PCR (Bolívar et al. 2014, Suzuki y Yao 2014). Al respecto Cornejo et al. (2014), y Bolívar et al. (2014), reportan que la presencia de dímeros, ocasionados por exceso de reactivos (MgCl2, dNTP´s y oligos) compromete el éxito en la amplificación del ADN molde.
Los ensayos realizados revelaron la estandarización y validación de la técnica al observarse una alta reproducibilidad en la amplificacion de las repeticiones de la PCR; es decir, la existencia de 100 % de probabilidad de encontrar el mismo resultado entre las muestras. Estos hallazgos coinciden con autores quienes refieren la alta reproducibilidad de la técnica de PCR y sus variantes como una herramienta confiable en la detección de diferentes organismos, además de su alta sensibilidad y rapidez; características que le confieren gran ventaja frente a los métodos convencionales (Villarreal et al. 2008, Cornejo et al. 2014, Hernández y Guzmán 2014).
Aun cuando la identificación morfológica de las especies en este estudio, coincide con los caracteres diacríticos para cada especie de acuerdo a las claves taxonómicas y se dio certidumbre a las secuencias obtenidas, ya que se realizaron al menos tres réplicas de secuenciación para cada especie, se presentaron algunas inconsistencias y bajo porcentaje de homología respecto a la información reportada en el GenBank. Por ejemplo, las secuencias de este estudio correspondientes a X. ferrugineus presentaron homología con Xyleborus schaufussi (Blandford, 1894) (AB588939.1) con una identidad y cobertura de 83% y 99%, respectivamente, especie que de acuerdo con Pérez et al. (2015a y b) no está reportada para el país, aspecto que pone en duda la identidad taxonómica de la especie. Por otro lado, en la base de datos solo esta reportada una secuencia para X. ferrugineus (JX424246.1) para el gen CO1, sin embargo, la información no es homologa a la secuencia obtenida para esta especie en el presente estudio. La no coincidencia en secuencias quizás se deba a una determinación errónea, de origen, de la especie y secuencia disponible en el GenBank, puesto que los especímenes que se trabajaron en este estudio fueron determinados con base en clave específica para especies mexicanas, las características distintivas de la especie fueron corroboradas por los especialistas y se revisaron varios especímenes. Algo similar, se presentó con las secuencias para X. affinis, X. volvulus y X. glabratus reportadas por Cognato et al. (2011), pues no hubo coincidencia con las reportadas en el presente estudio. La única secuencia donde se encontró concordancia en 93% y 100% de cobertura entre la información encontrada en este trabajo con la reportada en el GenBank fue para X. affinis con numero de acceso AF187138.1; sin embargo, no se encontró artículo publicado relacionado que permitiera conocer si previo al análisis molecular se corroboró la determinación morfológica de la especie. Asimismo, no se encontraron secuencias en la base de datos NCBI para confrontar la información de las especies de X. horridus, X. intrusus y X. spinulosus, de modo que las secuencias derivadas de este estudio se constituyen en los primeros registros fiables para dichas especies. Los números de acceso al GenBank de las mismas se anotan en el Cuadro 5.
Aun cuando se tiene un avance en el conocimiento y se cuenta con la técnica de PCR anidada para la detección rápida, sensible y confiable de especies del género Xyleborus es necesario ampliar las pruebas a otras especies para conocer con precisión los alcances de la herramienta y obtener información que permita la identificación veraz y oportuna de especies del género Xyleborus en cualquier estadio de su ciclo de vida.
Conclusiones
La implementación del kit DMF fue apta para la obtención de material génico de especies del género Xyleborus en un menor tiempo y con la calidad adecuada, lo que permite su amplificación mediante PCR-anidada.
El método permitió procesar un solo insecto por extracción, y obtener material genético de muestras de varios años de preservación. Se estandarizó, validó y optimizó la técnica de PCR-anidada con alta reproducibilidad, aportando resultados de mayor confiabilidad permitiendo la correcta identificación molecular de las especies evaluadas. Se obtuvo la secuenciación parcial del gen CO1 para Xyleborus affinis, X. ferrugineus, X. glabratus, X. horridus, X. intrusus, X. spinulosus y X. volvulus. De esta manera el implementar esta metodología para el diagnóstico de especies Xyleborus constituye una alternativa de diagnóstico complementaria a la tradicional.











 nueva página del texto (beta)
nueva página del texto (beta)