Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Acta zoológica mexicana
versión On-line ISSN 2448-8445versión impresa ISSN 0065-1737
Acta Zool. Mex vol.24 no.1 Xalapa abr. 2008
Artículos originales
Identificación molecular de especies crípticas de Trichogramma Westwood (Hymenoptera: Trichogrammatidae) de importancia agrícola en México
Martha Patricia ESPAÑA-LUNA1, Alejandro GONZÁLEZ-HERNÁNDEZ1, Omar G. ALVARADO-GÓMEZ2 & Julio LOZANO-GUTIÉRREZ3
1 Facultad de Ciencias Biológicas, Universidad Autónoma de Nuevo León. Av. Universidad y Pedro de Alba s/n Ciudad Universitaria. San Nicolás de los Garza, N. L. MÉXICO. mpesp24@yahoo.com, agonzale@fcb.uanl.mx
2 Facultad de Agronomía de la Universidad Autónoma de Nuevo León. omarguadalupe@intercable.net
3 Unidad Académica de Agronomía de la Universidad Autónoma de Zacatecas, MÉXICO. jlozano_75@yahoo.mx
Recibido: 14 de junio de 2006
Aceptado: 10 de octubre de 2007
RESUMEN
La identificación de las avispas parasitoides del género Trichogramma es difícil debido a su tamaño pequeño y a la plasticidad de sus características morfológicas. La morfología de genitalias y antenas de los machos es de utilidad para determinar las especies, sin embargo esto representa un problema en particular cuando las especies se reproducen por partenogénesis. El objetivo del presente trabajo fue identificar a nivel molecular especies crípticas de Trichogramma de importancia agrícola en México. Se identificó a las especies crípticas T. pretiosum, T. fuentesi y T. exiguum, así como a las no crípticas T. atopovirilia y T. pintoi. Se construyó una clave dicotómica de identificación, basada en el tamaño del espaciador transcrito interno 2 (ITS2) del DNA ribosomal amplificado por PCR, y el polimorfismo en la longitud de los fragmentos de restricción entre las especies. Las regiones ITS2 fueron secuenciadas y comparadas con secuencias de GenBank, se calcularon los porcentajes de similitud y divergencia entre las especies. Esta herramienta molecular puede resolver la identificación de especies crípticas presentes en México.
Palabras clave: Trichogramma, especies crípticas, identificación, PCR-RFLP, ITS2.
ABSTRACT
Trichogramma parasitic wasps often are cryptic species and can not be distinguished because of their small size and the plasticity of their morphological traits. The species identity of Trichogramma has been based on the morphology of the male genitalia and flagellar structures, this presents a particular problem for parthenogenetic forms. The objective here was to identify by molecular methods cryptic species of Trichogramma occurring in agricultural fields of Mexico. We identified the cryptic species T. pretiosum, T. fuentesi y T. exiguum, and the non cryptic species T. atopovirilia y T. pintoi. A identification dichotomous key to species was constructed using as taxonomic characters the size of the internal transcribed spacer 2 (ITS2) region of ribosomal DNA and the difference in restriction length polymorphism of species. Also the ITS2 regions were sequenced coupled with ITS2 sequences from GenBank and the percents of similarity and divergence were calculated between species. This molecular tool can solve identification of cryptic species of Trichogramma from Mexico.
Key words: Trichogramma, cryptic species, identification, PCR-RFLP, ITS2.
INTRODUCCIÓN
Los parasitoides del género Trichogramma son avispas diminutas de aproximadamente 0.5-1.5 mm de longitud, que parasitan huevos de diversas ordenes de insectos (Pinto 1998). Actualmente, las especies de Trichogramma son los parasitoides más usados como agentes de control biológico de plagas agrícolas en el mundo, debido a su fácil reproducción y su rango amplio de hospederos (Knutson 1998), entre los que destacan las plagas del orden Lepidoptera en diversos cultivos agrícolas (Sappal et al. 1995). Nueve especies de Trichogramma se reproducen en insectarios privados o gubernamentales alrededor del mundo, y se estima que son liberadas sobre 33 millones de hectáreas de cultivos agrícolas y forestales en 30 países. Los países de la Unión Soviética ocupan el primer lugar como productores y liberadores de Trichogramma, seguidos por China y México (Knutson 1998).
En México estas avispitas se liberan anualmente en 1.5 millones de hectáreas (van Lenteren & Bueno 2003). Al respecto, Rodríguez-del-Bosque & Arredondo (1999) reportaron que las principales especies que se reproducen masivamente bajo condiciones de laboratorio son T. pretiosum, T. exiguum, T. minutum, T. atopovirilia, T. platneri y T. pintoi; mientras que García-González et al. (2005) mencionan que las especies que se comercializan actualmente son T. exiguum T. fuentesi, T. pintoi y T. pretiosum.
La identificación de las especies de Trichogramma es difícil debido a su tamaño pequeño, a la existencia de numerosos ecotipos, además de la carencia de características de diagnóstico suficientes (Pinto 1998). Nagarkatti & Nagaraja (1971) identificaron a las especies a través de la genitalia de los machos, sin embargo, estas características no son confiables debido a que existen especies con genitalias similares, además esta técnica no procede cuando las poblaciones se reproducen por partenogénesis (Pinto 1998). Entre las especies crípticas más frecuentes está T. pretiosum que comúnmente se confunde con T. ostriniae, T. interius, T. deion, T. platneri y T. minutum. Asimismo, T. exiguum es comúnmente identificada como T. fuentesi, T. minutum y T. platneri.
Vanlerberghe-Massuti (1994) señala la importancia de contar con métodos rápidos y confiables para la caracterización de especies crípticas de Trichogramma y enfatiza la utilidad de los métodos basados en marcadores moleculares. Así mismo, Pinto (1998) menciona que la identificación de las especies crípticas de Trichogramma, mediante métodos moleculares, es de gran utilidad ya que asegura la liberación de las especies adecuadas para cada plaga en particular.
Un marcador importante usado para diferenciar especies y biotipos de Trichogramma, son los espaciadores transcritos internos (ITS 1 y 2) del DNA ribosomal (DNAr) (Pinto et al. 1997, Stouthamer et al. 1999, Ciciola Jr. et al. 2001, Pinto et al. 2002, Thompson et al. 2003, Li et al. 2004), debido principalmente a que el DNAr está conservado en la naturaleza a través de la evolución, en tanto que los espacios intergénicos son regiones que evolucionan más rápido (Coleman 2003), lo que los hace variables entre especies (Pinto et al. 1997). La reacción en cadena de la polimerasa (PCR) y el polimorfismo en la longitud de los fragmentos de restricción (PCR-RFLP) se pueden utilizar para distinguir especies y biotipos de Trichogramma (Sappal et al. 1995, Thompson et al. 2003). Esta técnica se basa en el uso de una o más enzimas de restricción que cortan los enlaces fosfodiester en secuencias específicas de productos amplificados por PCR, posteriormente se detectan los fragmentos de DNA de distinto peso molecular en geles de agarosa o poliacrilamida (Avise 2004), Pinto et al. (2002) y Silva et al. (1999) emplearon este procedimiento, para elaborar claves dicotómicas para identificar especies de Trichogramma.
La secuenciación del ITS2 del DNAr se ha empleado para identificar especies de Trichogramma (van Kan et al. 1997, Stouthamer et al. 1999, Pinto et al. 2002, Ciciola Jr et al. 2001, Li et al. 2004), así como en otros organismos: hongos, plantas y animales, incluyendo otros insectos (Loxdale &? Lushai 1998). El objetivo de esta investigación fue Identificar a nivel genético especies crípticas de importancia agrícola en México.
MATERIAL Y MÉTODOS
Colecta de muestras. Se obtuvieron 45 muestras de campo de regiones agrícolas localizadas en los siguientes estados: Aguascalientes, Chiapas, Coahuila, Durango, Guanajuato, Nayarit, Nuevo León, San Luis Potosí, Sinaloa, Sonora, Tamaulipas, Veracruz y Zacatecas. Asimismo se analizaron 23 muestras procedentes de laboratorios de reproducción masiva, de los estados: Chihuahua, Coahuila, Colima, Guanajuato, Jalisco, Michoacán, Morelos, Nayarit, Nuevo León, Sinaloa, Sonora, Tamaulipas, Veracruz y Zacatecas.
Las muestras de campo fueron tomadas mediante la colecta manual de masas huevos de lepidopteros. Además se tomaron muestras con red de golpe y los especímenes se colocaron en alcohol etílico 100%. Para las muestras de los laboratorios se solicitaron 20 pulgadas cuadradas de cartoncillo con huevos de la palomilla Sitotroga cerealella (Lepidoptera: Gelechiidae) parasitados por Trichogramma. Las muestras se colocaron en cámaras de emergencia durante 48 horas, posteriormente los adultos se conservaron en alcohol al 100% para preservarlos.
Identificación morfológica de las especies. Con el objetivo de determinar la presencia de especies crípticas en las muestras de campo y de insectarios de reproducción comercial, se hizo una identificación inicial por análisis morfológico en la Colección de Insectos Benéficos Entomófagos (CIBE) de la Facultad de Ciencias Biológicas de la Universidad Autónoma de Nuevo León, utilizando la clave de Pinto (1998), y fue confirmada por el Dr. Richard Stouthamer de la Universidad de California en Riverside, quien utilizando especímenes de las mismas muestras, amplificó por PCR la región ITS2 del DNAr e hizo la secuenciación y alineamientos con las secuencias de especies conocidas.
Extracción de DNA. El DNA se extrajo mediante el procedimiento descrito por Pinto et al. (2002), modificado de la siguiente manera: un espécimen de cada muestra se agitó en 0.5 ml de TAE durante 30 minutos, posteriormente se incubó en 100 µl de Chelex-100 al 5% (Bio-Rad Corp.), con 3 µl de proteinasa K (Promega) (20 mg/ml) para desintegrar las proteínas extracelulares. La incubación se llevó a cabo durante una hora a 56ºC, con agitación en vortex cada 15 minutos. Finalmente se incubó a 95ºC durante de 10 min.
Amplificación por PCR. La reacción en cadena de la polimerasa (PCR) se realizó en volúmenes de 25 µl. Se utilizó un termociclador marca ThermoHybaid, 2.5 µl de DNA, 2.5 µl de buffer PCR, 0.5 µl de una mezcla de dNTP's (desoxinucleótidos trifosfatados) (10 mM cada uno), 0.3 µl de cada oligo (10 ng), 0.3 µl de enzima Taq DNA polimerasa (5 unidades/ µl), y 17.5 µl de agua destilada estéril (Pinto et al. 2002). La región ITS2 se amplificó mediante los oligos citados por Campbell et al. (1993) con la modificación de Ciciola Jr et al. (2001), por ser específicos para Trichogramma: 5'-TGTGAACTGCAGGACACATG-3' localizado en la región 5.8S del DNAr, y 5'-GTCTTGCCTGCTCTGAG-3' localizado en la región 28S del DNAr. El programa térmico usado fue de 3 min a 95ºC seguidos por 33 ciclos de 45 s a 92ºC, 45 s a 53ºC, y 45 s a 72ºC con 3 min a 72ºC después del último ciclo. Los productos de PCR fueron separados por electroforesis en geles de agarosa 1%, con un marcador de peso molecular ladder 100 (Promega). Los geles fueron teñidos con bromuro de etidio, visualizados en un transiluminador de luz UV y fotografiados con una cámara Polaroid.
Análisis de restricción. El análisis se realizó con las enzimas EcoRI y AluI (Promega). Las digestiones se realizaron en volúmenes de 20 µl conteniendo 15 µl de producto de PCR, 2 µl de bufer de restricción, 0.1 mg/ml de albúmina de suero de bovino (BSA) y 1 µl de cada enzima. Las reacciones se incubaron a 37°C durante toda la noche. Los fragmentos se separaron por electroforesis en geles de Poliacrilamida 5%, utilizando un marcador de peso molecular ladder 100 (Promega), teñidos con bromuro de etidio, visualizados en un transilumninador de luz UV y fotografiados con una cámara Polaroid. Los fragmentos de restricción se compararon por la distancia de migración en el gel.
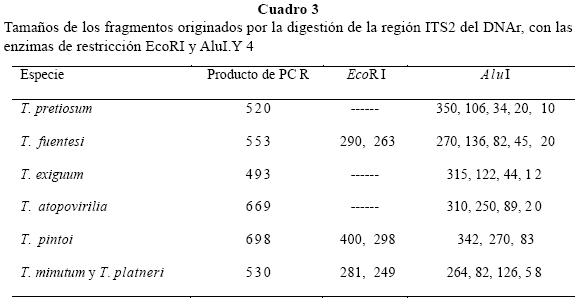
Clave molecular. La clave molecular para la identificación de las especies de Trichogramma en estudio fue construida con base en el tamaño de los productos de PCR de la región ITS2 del DNAr, las especies con tamaños similares en los productos de PCR fueron distinguidas por los patrones de bandeo de los fragmentos de restricción originados con las enzimas EcoRI y AluI. Además, debido a su presencia en México, en la clave se incluyeron a las especies T. minutum y T. platneri, las secuencias del ITS2 fueron obtenidas de Genbank (claves de acceso AY374439 y AF408657 respectivamente), y analizadas en el paquete DNA strider 1.0 (Christian Marck).
Análisis de secuencias. Se enviaron productos de PCR de todas las muestras, para ser secuenciados en el Centro de Investigaciones y Estudios Avanzados (CINVESTAV) Unidad Irapuato. Los productos de PCR fueron directamente purificados con el sistema de Wizard® PCR prep, (Promega) y secuenciados por el método de Sanger, "Método del Dideoxi Terminal", en un secuenciador ABI PRISM, 3700 DNA ANALIZER de Applied Biosystems. Las secuencias se editaron con el programa Editseq y alinearon con el programa MegAlign, ambos del paquete Winstar (DNASTAR Inc., Madison, Wis.). Se calcularon los porcentajes de similitud y divergencia entre ellas y con las secuencias reportadas para especies de Trichogramma en GenBank (http://www.ncbi.nih.gov/) (Cuadro 1).

RESULTADOS
Las especies crípticas obtenidas tanto de campo como de laboratorios de reproducción masiva, fueron T. pretiosum, T. fuentesi y T. exiguum. Asimismo, se encontró a las especies no crípticas T. atopovirilia solamente en campo y T. pintoi de una muestra de laboratorio (Cuadro 2).

Las especies fueron identificadas por un primer criterio que fue la longitud de los productos amplificados por PCR de la región ITS2 del DNAr, T. pintoi presentó el tamaño mayor con un producto amplificado de 698 pb, mientras que el producto menor fue para la especie T. exiguum con 493 pb. Las especies T. pretiosum, T. fuentesi y T. atopovirilia presentaron productos de PCR de 520, 553 y 669 respectivamente (Fig. 1).

El segundo criterio de identificación de las especies, fue la discriminación basada en el análisis de restricción con las enzimas EcoRI, y AluI cuyos perfiles de mostraron que T. pretiosum, T. exiguum y T. atopovirilia no presentan sitio de corte con EcoRI, mientras que los ITS2 de T. fuentesi y T. pintoi son cortados originando dos fragmentos de tamaños diferentes, la digestión con la enzima AluI originó fragmentos de diferentes tamaños para cada una de las especies (Fig. 2).
El análisis de restricción con el paquete DNA strider 1.0, de las secuencias de T. minutum y T. platneri, evidenció un sitio de corte con EcoRI en las secuencias de ambas especies, con fragmentos de 281 y 249 pb, mientras que con AluI se obtuvieron cuatro fragmentos (Cuadro 3). En el cuadro 4 se muestra la clave de identificación de las especies de Trichogramma en estudio; la discriminación que se originó con el tamaño de los productos amplificados por PCR, y por la longitud de los fragmentos en el análisis de restricción.

Las secuencias obtenidas de T. fuentesi fueron 100% similares a la única secuencia reportada para esta especie en Genbank, mientras que las demás especies variaron de 85.7 a 99.8% de similitud con las secuencias correspondientes. Los porcentajes de divergencia fueron de 0% con T. fuentesi, hasta 6.2% con T. atopovirilia (Cuadro 5)

Al analizar las secuencias de la región ITS2 del DNAr, se observó que a nivel intra-especie T. fuentesi fue la más similar con un 100% de similitud entre las secuencias comparadas, para T. pretiosum los porcentajes variaron de 96.0 a 99.8, para T. exiguum los valores fueron de 97.5 a 99%, mientras que T. atopovirilia presentó los porcentajes mayores de divergencia, que variaron desde 1.0 a 6.2%, con una similitud de 84.8 a 97.4%.
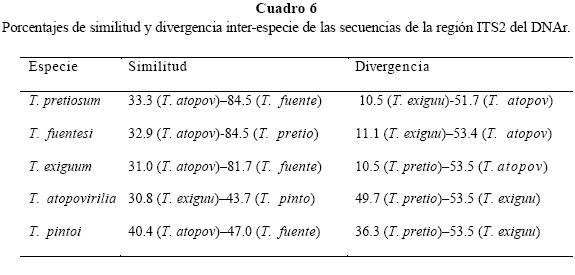
Los porcentajes de similitud inter-especie variaron, T. atopovirilia presentó la menor similitud con respecto a las demás especies con porcentajes de 30.8 a 43.7%, lo que coincide al ver que esta especie presentó datos de divergencia superiores al 50% con T. pretiosum, T. fuentesi, T. exiguum y en tanto que T. pretiosum y T. fuentesi son las más similares entre ellas, con un 84.5% (Cuadro 6).
DISCUSIÓN
La región ITS2 del DNAr es de utilidad para la identificación de especies crípticas (Stouthamer et al. 1999), las secuencias y el análisis de restricción resuelven las limitaciones que existen en la identificación morfológica, principalmente porque se puede llevar a cabo en machos y hembras, sin importar la dificultad que tiene el observar las diminutas características morfológicas (Pinto et al. 2002). En este estudio se identificó mediante la comparación de las secuencias y análisis de restricción del ITS2 del DNAr a las especies crípticas T. pretiosum, T. fuentesi y T. exiguum, así mismo, se propone una clave para identificar a estas especies, y donde se incluye a T. minutum y T. platneri. La reproducción comercial de T. minutum y T. platneri en México, así como el reporte de Pinto (1998) sobre la presencia bajo condiciones naturales de T. minutum en nuestro país, fue importante en la decisión de incluir a estas dos especies en la clave. Pinto (1998) menciona que T. minutum y T. platneri son morfológicamente iguales. Con respecto a la identificación molecular de estas especies, Stouthamer et al. (2000) no encontraron diferencias en las secuencias del ITS2, razón por la cual en la clave propuesta no se pueden separar. Borghuis et al. (2004) pudieron diferenciarlas gracias a la variación que presenta la secuencia del gen Citocromo Oxidasa II del DNA mitocondrial.
Durante esta investigación, también se identificó a T. pintoi y T. atopovirilia. Ambas especies presentan características morfológicas relativamente fáciles de diferenciar, aunque T. pintoi puede ser confundida con las especies europeas T. buesi y T. bourarachae, mientras que T. atopovirilia es morfológicamente distinta y fácilmente diferenciada de todas las especies descritas (Pinto 1998), sin embargo, fue importante incluir a estas dos especies en los análisis moleculares realizados, debido a su presencia en México.
El uso de la clave molecular elaborada con base en los tamaños de los productos de PCR de la región ITS2 del DNAr, así como su digestión con las enzimas de restricción EcoRI y AluI puede incrementar la precisión en las especies liberadas. Pinto et al. (2002) elaboraron una clave dicotómica basada en los tamaños del ITS2 amplificados por PCR, y digeridos con las enzimas de restricción EcoRI y MseI, logrando identificar a diez especies entre las que incluye a T. pretiosum y T. exiguum. Para la elaboración de la clave aquí presentada, fue suficiente utilizar EcoRI, para identificar a T. fuentesi y T. pintoi, mientras que T. pretiosum, T. exiguum y T. atopovirilia no presentan el sitio de digestión de esta enzima, lo que se convierte en una característica mas de identificación, en combinación con el tamaño del producto inicial de PCR.
Almeida & Stouthamer (2004) emplearon los tamaños de los productos de PCR del ITS2, y las enzimas de restricción EcoRI, MseI y MaeI para elaborar la clave molecular de identificación de las especies T. esalqueanum, T. exiguum, T. pretiosum, T. dendrolimi, T. brassicae, T. fuentesi, T. galloi, T. cacoeciae, T. lasallei, T. lopezandinensis, T. iracildae, T. rojasi, T. bruni, T. nerudai, T. pintoi, T. acaocioi y T. atopovirilia, sin embargo resulta de poca aplicación debido a que incluye diez especies que no están presentes en México, y el uso de más enzimas de restricción, lo que la hace poco práctica para su aplicación con especies mexicanas.
Investigación similar fue realizada por Silva et al. (1999), quienes elaboraron una clave molecular con los tamaños del ITS2 del DNAr, y la digestión con la enzima de restricción Mn1I para identificar a las especies presentes en Portugal T. cordubensis, T. evanescens, T. turkestanica, T. pintoi y T. bourarachae. Silva et al. (1999) mencionaron que los análisis de restricción originaron resultados confiables y repetibles, lo que facilita el uso de las claves dicotómicas basadas en productos de PCR y fragmentos de restricción.
Otro de los criterios de identificación de las especies de Trichogramma, fueron los porcentajes de divergencia y similitud entre las secuencias del ITS2 del DNAr, al respecto Ciciola et al. (2001), mencionaron la importancia de tener disponibles las secuencias de las especies, para determinar la presencia de especies nuevas por la comparación de secuencias de la población. En este estudio los porcentajes de divergencia inter e intra especie (10.5 a 53.5% y 0.0 a 6.2% respectivamente) fueron de utilidad para identificar a las especies de Trichogramma. Los porcentajes de divergencia entre las secuencias, inter e intra-especie obtenidos en esta investigación fueron similares al resultado de Walker et al. (2005), quienes analizaron las secuencias del ITS2 del DNAr de los tricogramatidos Aphelinoidea anatolica, A. turanica, A. roja y A. zarehi, los porcentajes de divergencia inter-especies que obtuvieron fueron de 31 a 54%, mientras que a nivel intra-especie no hubo divergencia .
Thompson et al. (2003) mencionaron que el ITS2 es de utilidad para identificar especies de Trichogramma debido a que la variación en las secuencias intra-especie es relativamente pequeña en comparación con la variación inter-especie, y además porque la mayoría de las especies crípticas presentan variación suficiente en el ITS2 para ser distinguidas.
Ciciola et al. (2001) enfatizaron la importancia de realizar la identificación morfológica de las especies de Trichogramma, previo a la identificación a través de métodos moleculares; Silva et al. (1999), mencionan al respecto que los métodos moleculares no reemplazan a los métodos de identificación tradicionales basados en la morfología, sin embargo, son importantes como herramientas útiles para identificar a las especies principalmente cuando son crípticas; cuando se tiene el conocimiento de cuales especies están presentes en cierta área; cuando es necesario complementar la identificación con otros métodos; así mismo, son de utilidad para determinar posibles contaminaciones o mezclas de especies en los laboratorios de reproducción masiva y estudios sobre dispersión y evaluación de parasitoides liberados en campo o nativos de una región.
AGRADECIMIENTOS
Este trabajo se realizó con apoyo financiero de SEP-CONACYT a través del proyecto No. UA 5527-Z, así como del Programa de Apoyo a la Investigación Científica y Tecnológica (PAICYT) Clave CN889-04 de la Universidad Autónoma de Nuevo León. A la Dra. Adriana E. Flores Suárez y al Dr. José María Viader Salvadó por sus observaciones y comentarios a este escrito.
LITERATURA CITADA
Almeida, R.P. & R. Stouthamer. 2004. ITS-2 sequences-based identification of Trichogramma species in South America. In: Trichogramma and its relationship with Wolbachia: Identification of Trichogramma species, phylogeny, transfer and costs of Wolbachia symbionts. Almeida, R.P. de (2004) Ph.D. Thesis. Chapter 3. Wagenignen University, The Netherlands. [ Links ]
Avise, J.C. 2004. Molecular Markers, Natural History, and Evolution. Second Edition. Sinauer Associates, Inc. Publishers. Sunderland, Massachusetts. University of Georgia. [ Links ]
Borghuis, A., J.D. Pinto, G.R. Platner, & R. Stouthamer 2004. Partial cytochrome oxidase II sequences distinguish the sibling species Trichogramma minutum Riley and Trichogramma platneri Nagarkatti. Biological Control 30:90-94. [ Links ]
Campbell, B.C., J.D. Steffen-Campbell, & J.H. Werren. 1993. Phylogeny of the Nasonia species complex (Hymenoptera: Pteromalidae) inferred from an internal transcribed spacer (ITS2) and rDNA sequences. Insect Mol. Biol. 2:225-237. [ Links ]
Ciciola, A. I. Jr., R.B. Querino, R.A. Zuchi, & R. Stouthamer. 2001. Systematics, morphology and physiology. Molecular tool for identification of closely related species of Trichogramma (Hymenoptera: Trichogrammatidae): T. rojasi Nagaraja & Nagarkatii and T. lasallei Pinto. Neotr.l Entomol. 30(4):575-578. [ Links ]
Coleman, A.W. 2003. ITS2 is a double-edged tool for eukaryote evolutionary comparisons. Trends Genet. 19(7): 370-375. [ Links ]
García-González, F., A. González-Hernández, & M.P. España-Luna. 2005. Especies de Trichogramma Westwood (Hymenoptera: Trichogrammatidae) presentes en Centros reproductores de México. Acta Zool. Mex. (n.s.) 21(3): 125-135. [ Links ]
Knutson, A. 1998. The Trichogramma manual. Texas Agricultural Extension Service report no. B-6071; 5-98. Agricultural Comunications, The Texas A & M University System, College Station, TX. [ Links ]
Li, Z. X., L. Zheng, & Z. R. Shen. 2004. Using internally transcribed spacer 2 sequences to re-examine the taxonomic status of several cryptic species of Trichogramma (Hymenoptera: Trichogrammatidae). Europ. J. Entomol. 101(3):347-358. [ Links ]
Loxdale, H.D. & G. Lushai. 1998. Molecular markers in entomology. Review article. Bull. Entomol. Res. 88:577-600. [ Links ]
Nagartti, S. & H. Nagaraja. 1971. Redescription of some known species of Trichogramma (Hym., Trichogrammatidae), showing the importance of the male genitalia as a diagnostic character. Bull. Entomol. Res. 61:13-21. [ Links ]
Pinto, J.D. 1998. Systematics of the North American species of Trichogramma Westwood (Hymenoptera: Trichogrammatidae). In: Memoirs of the Entomological Society of Washington. Number 22. [ Links ]
Pinto, J.D., A.B. Koopmanschap, G.R. Platner, & R. Stouthamer. 2002. The North American Trichogramma (Hymenoptera: Trichogrammatidae) parasitizing certain Tortricidae (Lepidoptera) on Apple and Pear, with ITS2 DNA Characterizations and description of a new species. Biol. Cont. 23:134-142. [ Links ]
Pinto, J.D., R. Stouthamer, & G.R. Platner 1997. A new cryptic species of Trichogramma (Hymenoptera: Trichogrammatidae) from the Mojave dessert of California as determined by morphological, reproductive and molecular data. Proc. of the Entomol. Soc. of Wash. 99:238-247. [ Links ]
Rodríguez-del-Bosque, L.A. & H.C.B. Arredondo. 1999. Quien es quien en el control biológico en México. INIFAP-CIRNE. Campo Experimental Río Bravo. Folleto Técnico No. 23 Tamaulipas, México. 147 p. [ Links ]
Sappal, N.P., R.S. Jeng, M. Hubbes, & F. Liu. 1995. Restriction fragment length polymorphisms in polymerase chain reaction amplified ribosomal DNAs of three Trichogramma (Hymenoptera, Trichogrammatidae) species. Genome 38:419-425. [ Links ]
Silva, I.M.M.S., J. Honda, F. Van Kan, J. Hu, L. Neto, B. Pintureau, & R. Stouthamer. 1999. Molecular differentiation of five Trichogramma species occurring in Portugal. Biol. Cont. 16:177-184. [ Links ]
Stouthamer, R., J. Hu, F. Van Kan, G.R. Platner & J.D. Pinto. 1999. The utility of internally transcriber spacer 2 DNA secuences of nuclear ribosomal gene for distinguishing sibling species of Trichogramma. BioCont. 43:421-440. [ Links ]
Stouthamer, R., Y. Gai, A.B. Koopmanschap, G.R. Platner & J.D. Pinto. 2000. ITS-2 sequences do not differ for the closely related species Trichogramma minutum and T. platneri. Entomol. Exp. Et Applicata 95:105-111. [ Links ]
Thompson, J.L., J.R. Bradley, M.E. Carew, & A.H. Ary. 2003. Identification and characterization of Trichogramma species from south-eastern Australia using the internal transcribed spacer (ITS-2) region of the ribosomal gene complex. Entomol. Exp. Et Applicata 106:235-240. [ Links ]
Van Kan F.J.P.M., J. Honda, J.D. Pinto, & R. Stouthamer. 1997. Molecular based techniques for Trichogramma identification. Proc. Exper. and Appl. Entomol. N.E.V. Amsterdam Vol 8:59-62. [ Links ]
Van Lenteren, J. & V.H.P. Bueno. 2003. Augmentative biological control of arthropods in Latin America. Biocont. 48(2):123-139. [ Links ]
Vanlerberghe-Masutti, F. 1994. Molecular identification and phylogeny of parasitic wasp species (Hymenoptera: Trichogrammatidae) by mitochondrial DNA RFLP and RAPD markers. Insect Mol. Biol. 3:229-237. [ Links ]
Walker, G.P., I.M. Bayoun, S.V. Triapitsyn, & J.Y. Honda. 2005. Taxonomy of Aphelinoidea (Hymenoptera: Trichogrammatidae) species attacking eggs of the beet leafhopper, Circulifer tenellus (Hemiptera: Cicadellidae), in California. Zootaxa 1068:1-25. [ Links ]














