El diagnóstico microbiológico ofrece pautas acerca del establecimiento y aplicación de criterios microbiológicos a los alimentos en cualquier punto desde el origen, la planificación y la formulación, así como la implementación de sistemas que garanticen la inocuidad y calidad de los productos.1,2 Las bacterias responsables de enfermedades provocadas por alimentos pertenecen a una gran variedad de géneros.3
En las zonas costeras, el consumo humano de productos del mar está directamente relacionado con aspectos laborales y deportivos. Sin embargo, esta práctica representa un riesgo epidemiológico debido a que la contaminación de las aguas costeras por los vertidos de aguas residuales de las ciudades es una constante fuente epidemiológica de salmonelosis, cólera, hepatitis y otras infecciones, además de las deficiencias en el manejo de los productos pesqueros.4, 5
En la región sureste de México, uno de los principales productos pesqueros es el Octopus maya, mejor conocido como pulpo rojo. Esta especie ha sido descrita como endémica y bentónica, que se distribuye en aguas someras de la plataforma continental de la península de Yucatán. La pesca de pulpo rojo se encuentra posicionada en el décimo lugar del volumen de producción de especies marinas del país y ocupa el quinto lugar en valor comercial. En los últimos tres años, la producción nacional promedio se ha mantenido por encima de las 25 000 toneladas; en 2011 superó las 27 500 toneladas.6
En los últimos 10 años, la tasa media de crecimiento anual de la producción fue positiva, con 0.78% a nivel nacional. Los estados de Campeche y Yucatán contribuyen con 94.2% de la producción nacional total de pulpo, cuyo valor es de 557 370 000 pesos. La calidad de este recurso puede verse afectada por una gran variedad de agentes patógenos, pues diversos estudios realizados en muestras representativas de alimentos marinos han mostrado la presencia de bacilos marinos halófilos pertenecientes a los géneros Vibrios y Aeromonas.7
Las bacterias pueden descomponer los alimentos marinos, lo que ocasiona pérdidas económicas al comercio de alimentos y provoca enfermedades a los consumidores.8 En este trabajo se evaluó la calidad microbiológica del pulpo rojo (Octopus maya) a partir de diversos parámetros de acuerdo con las normas oficiales mexicanas. Asimismo, se realizó una comparación con el Sistema BAx (análisis de ADN) con la finalidad de proporcionar alternativas en la detección de microbiota alterante o patógena.
Material y métodos
Toma de muestras
Las muestras se recolectaron durante los meses de octubre a diciembre de 2013 en los puertos pesqueros con mayor volumen de captura reportada en el estado de Campeche, México. Las localidades fueron Campeche, Champotón, Seybaplaya, Villa Madero, Isla Arena y Sabancuy. Cada muestra contenía un pulpo rojo de entre 200 g y 1 kg, que fue colocado en bolsas herméticas de nylon (Deltalab PLA-01117) rotuladas y transportadas dentro de un contenedor con hielo hasta el laboratorio. En total se recolectaron 30 muestras por triplicado de cada uno de los puertos seleccionados, lo que hizo un total de 540 unidades. El estudio fue observacional descriptivo, de corte transversal sin direccionalidad. El cálculo del tamaño de la muestra se hizo por la estimación de una proporción poblacional (Epidat 3.1).
Métodos de detección con las Normas Oficiales Mexicanas (NOM)
Para la detección de los microorganismos patógenos en las muestras de pulpo rojo se emplearon las estrategias y metodología de análisis para aislamiento e identificación establecidas por las Normas Oficiales Mexicanas: Vibrio,9Salmonella,10 coliformes fecales y Escherichia coli,11,12 y Staphylococcus aureus.13
Método alternativo BAX
La detección de microorganismos patógenos en las muestras se realizó a través del Sistema BAx, basado en la reacción en cadena de polimerasa. Se utilizaron 25 g de muestra y se inocularon en los medios de enriquecimiento específico para Salmonella, Vibrio cholerae, Vibrio parahaemolitycus, Vibrio vulnificus y E. coli O157:H7, y Staphylococcus aureus. Se realizó la extracción del ácido desoxirribonucléico (ADN) en cada cultivo, utilizando 5 μl de la solución de pre-enriquecimiento y 200 μl de una solución de proteasa 10 mg/ml. Después de la extracción se tomaron 50 μl de la solución y se colocaron en un microtubo para la reacción en cadena de la polimerasa (PCR), el cual contenía la mezcla de reacción con los iniciadores (primers) polimerasa y oligonucleótidos BAX. Las reacciones de amplificación de los fragmentos de ADN para las bacterias Salmonella, Vibrio cholerae, Vibrio parahaemolitycus, Vibrio vulnificus y E. coli O157:H7 se realizaron de acuerdo con las especificaciones del proveedor (Sistema BAX). Los resultados se expresaron como positivo/negativo a la presencia de amplificación de fragmentos específicos para cada bacteria.
Análisis estadístico
Los indicadores de calidad microbiológica fueron determinados como lo establece la NOM-029-SSA1-1993,14 incluyendo E. coli O157:H7 como mesofílicos aerobios. En el índice de calidad se asignó a cada indicador (microorganismos) un valor de 0 y a la ausencia, de 0.25; por lo tanto, si una muestra resulta negativa a todos los análisis (Salmonella, Vibrio cholerae, Vibrio parahaemolitycus y Vibrio vulnificus) tendrá un índice de 1. Por otra parte, si resulta positiva a los indicadores bacterianos tendrá un índice de 0.
El análisis de la comparación y concordancia entre métodos se llevó a cabo confrontando los resultados de 50 muestras procesadas por las dos técnicas por triplicado. Los datos fueron contrastados mediante una metodología gráfica de análisis Bland y Altman15 que permite evaluar la concordancia entre métodos de análisis determinando las diferencias entre los valores obtenidos en las pruebas contra las medias de ambos valores. Asimismo, se utilizó la prueba no paramétrica McNemar16 del paquete estadístico SPPS para comparar la distribución de proporciones obtenidas por los dos métodos y determinar si existe diferencia significativa entre ellos. Todas las pruebas estadísticas fueron realizadas con un nivel de significancia de p< 0.05 y de 95% para el nivel de confianza.
Resultados
Presencia de patógenos
Los análisis microbiológicos realizados a las 540 muestras del producto pesquero pulpo rojo revelaron que las bacterias con mayor presencia (82%) fueron las del género Vibrio, con la presencia de las especies V. cholerae, V. parahaemolyticus y V. vulnificus. Del total de las muestras, 11.7% fueron positivas para la bacteria Salmonella. En cuanto al grupo indicador "coliformes fecales", sólo se obtuvieron valores de 4.4%, mientras que E. coli O157:H7 mostró una presencia muy baja (1.1%). Se observó que S. aureus no rebasó los límites permitidos por la Norma Oficial Mexicana en ninguna de las muestras, por lo tanto su presencia se consideró poco relevante (Figura 1).

Figura 1 Presencia de los diferentes microorganismos en las muestras de pulpo rojo durante los meses de octubre a diciembre de 2013. Campeche, México
Al analizar la distribución de los patógenos por localización geográfica (Cuadro I), los resultados mostraron que el género Vibrio es el que presenta mayor incidencia en todas las localidades analizadas, seguido del grupo de los coliformes fecales. Asimismo, se evidenció que la bacteria S. aureus se encuentra por debajo de los límites permisibles por la NOM-115-SSA1-199413 en todos los sitios evaluados.
Índice de calidad
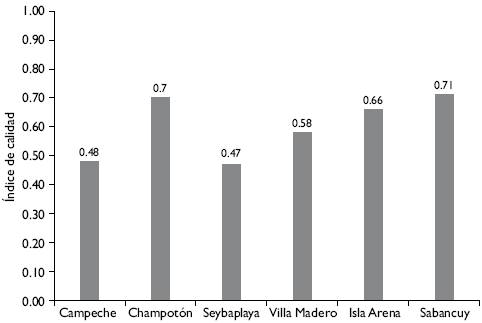
En el análisis de los indicadores de calidad microbiológica por localidad, de acuerdo con la NOM-129-SSA1-1995, incluido E. coli O157:H7 como mesófilo aerobio, las localidades de Sabancuy y Champotón tuvieron los mejores resultados con índices cercanos a 1, mientras que las muestras tomadas en Seybaplaya y Campeche mostraron índices de calidad más bajos (cercanos a 0.5), causados por la alta incidencia de V. cholerae y V. parahaemolyticus, que fueron positivas para ambas localidades (Figura 2).
Análisis Sistema BAX vs NOM
En la Figura 3 se muestra la comparación del total de muestras detectadas por ambos métodos y se observa que no existen diferencias en la aplicación de las dos técnicas. Estos resultados fueron analizados mediante la prueba de McNemar; se comprobó que no existen diferencias significativas en el uso de ambos métodos para la identificación de patógenos (valor de la estadística x2=0.5 y p=0.4795).

Figura 3 Comparación de métodos establecidos en las normas oficiales mexicanas contra el Sistema BAX en la detección de microorganismos patógenos en el pulpo rojo
El análisis de concordancia entre los métodos usados para la detección de patógenos, por el método gráfico de Bland-Altman, indicó que las medias aritméticas para ambos métodos fueron 1.25±0.76, por lo que la distribución de los datos se mantiene dentro de los límites de confianza, lo que demuestra que los dos métodos se pueden usar indistintamente (Figura 4).
Discusión
Los análisis realizados al producto pesquero pulpo rojo revelaron la presencia de bacterias pertenecientes al género Vibrio como V. cholerae, V. parahaemolyticus y V. vulnificus; esto puede deberse a que los ambientes marinos costeros y estuarios de las zonas tropicales son considerados ambientes naturales para estas bacterias.7,17,18 Sin embargo, estos microorganismos representan un riesgo sanitario eminente cuando están presentes en alimentos; tal es el caso de V. vulnificus, que se ha reportado en Estados Unidos, Dinamarca, Hong Kong, Japón, África y varias ciudades de América del Sur,19,20 y presenta un alto índice de mortalidad.
Vibrio parahaemolyticus es un patógeno entérico que se transmite a humanos, primordialmente a través del consumo. También se ha encontrado en camarones, cangrejos, jaibas y, ocasionalmente, en pescados,21 y se considera la principal causa de gastroenteritis trasmitida por mariscos a nivel mundial.22, 23
Vibrio cholerae es un habitante autóctono de los ecosistemas acuáticos, tanto en ríos, como en estuarios, por lo que el medio acuático se ha reconocido como reservorio y vehículo para la transmisión.24,25,26 La infección se debe al consumo de productos marinos crudos o con un mínimo de cocción, en los cuales V. cholerae puede ser contaminante de superficie o estar presente en el tracto intestinal.27-29
Los análisis para identificar bacterias coliformes totales y fecales ponen de manifiesto que el manejo del producto pesquero es deficiente, por lo tanto, existe riesgo de contaminación ambiental y fecal. Esto se observó principalmente en las localidades de Isla Arena y Seybaplaya. Las bacterias coliformes son un grupo heterogéneo compuesto por varios géneros, del cual existe poca evidencia que indique que pertenezcan a un solo género taxonómico.30 La falta de certeza en cuanto a su filiación taxonómica y la imprecisa correlación entre los métodos recomendados para la detección de coliformes han presentado problemas. Escherichia coli es aceptada como bacteria coliforme.11
Por otra parte, el género Salmonella spp, que se encuentra distribuido a nivel mundial y es un agente zoonótico importante que ha sido aislado a partir de una gran variedad de peces, crustáceos y moluscos bivalvos, también fue encontrado en las muestras tomadas en el puerto de Campeche, con una incidencia de 33%. Estos resultados superan datos registrados por la Food and Drug Administration (FDA) que reportó la incidencia de Salmonella en 1.3% de productos del mar, de 1990 a 1998.31 La alta incidencia de este microorganismo en la bahía de Campeche puede estar asociada con las descargas de aguas residuales domésticas que se conectan directamente con el mar, pues ya se ha demostrado la existencia de esta situación en la zona costera.32
Finalmente, los resultados obtenidos mostraron que el Stapphylococcus aureus no rebasó los niveles permitidos, lo que indica una escasa contaminación por manipulación humana. La reproducción de S. aureus en alimentos tiene gran importancia por tratarse de un microorganismo capaz de producir toxinas termorresistentes que, al ingerirse, causa intoxicaciones alimentarias.33 Se debe poner especial atención a esta bacteria, ya que la mayoría de los individuos pueden ser portadores y pueden trasmitirla a los alimentos durante su elaboración.
El análisis microbiológico del pulpo permitió evaluar problemas de almacenamiento, abuso de temperatura, vida útil, así como el potencial de contaminación fecal o posible presencia de patógenos (Escherichia coli, coliformes fecales, Salmonella spp). La presencia de estas bacterias puede estar asociada con las descargas de las redes domésticas sobre los efluentes pluviales en las poblaciones analizadas y con contaminación desde la obtención hasta el almacenamiento y el transporte a los puntos de venta.
Sistemas de análisis para detección e identificación de patógenos
Las técnicas para la identificación de patógenos a partir de su desarrollo en un cultivo específico suelen detectar un rango de numerosas especies; no requieren de equipo muy sofisticado y son pruebas sencillas de realizar. Sin embargo, presenta desventajas con respecto al tiempo y se requiere de un gran número de pruebas bioquímicas para la identificación confirmativa del patógeno, lo cual incrementa el costo del análisis.
Por otra parte, también se ha implementado el uso de anticuerpos para la identificación de microorganismos de manera simple y rápida. Sin embargo, la detección no suele ser precisa cuando existen mínimas cantidades de microorganismos que no hacen posible distinguir entre infecciones activas y latentes. Además, se carece de anticuerpos y antígenos para muchos microorganismos, que suelen tener un costo muy elevado, o bien, en algunos casos, pueden ofrecer falsos positivos.34
Los métodos moleculares para la detección de microorganismos utilizan análisis basados en la reacción en cadena de la polimerasa para la amplificación de fragmentos de ADN bacteriano específicos para cada especie.35 El equipo automatizado está acoplado a un detector de fluorescencia, lo que ofrece rapidez en la obtención de resultados y, debido a que es un método molecular, cuenta con alta especificidad y sensibilidad en la detección de las especies bacterianas. Estos métodos también abren la posibilidad de estudiar las poblaciones microbianas sin hacer aislamientos en medios selectivos, por lo tanto, se evitan los sesgos que pueden surgir con el cultivo de microorganismos.36,37 Asimismo, con esta nueva metodología también ahora es posible diferenciar las cepas virales y no virales, con base en su capacidad o incapacidad para producir sus factores virulentos. Comparar los métodos tradicionales de identificación de microorganismos con las técnicas moleculares de identificación, donde se contribuye al ahorro de tiempo, identificación determinante y disminución de costos, ha hecho que las técnicas moleculares ganen día a día más terreno en la investigación científica y diagnóstico de patógenos.38
Este trabajo comprobó que el sistema molecular BAx es tan eficiente como el uso de las pruebas bioquímicas establecidas por la NOM. Esta metodología molecular es considerada una técnica eficaz, por lo que puede ser empleada para una detección rápida y confiable en la evaluación microbiológica del pulpo.
Los centros de acopio del pulpo suelen ser establecimientos informales con buenas o regulares condiciones de higiene que pueden conducir a la reducción o pérdida de la calidad.
Conclusiones
La calidad e inocuidad de los alimentos debe ser una constante en todas las etapas de la producción del pulpo rojo (Octupus maya), sin importar lo simples o complejas que éstas sean. Con base en los presentes resultados, la calidad microbiológica del pulpo puede considerarse regular en general, lo que sugiere la necesidad de aplicar técnicas de identificación eficaz y rápida como el Sistema BAX. Estos métodos alternativos de análisis pueden coadyuvar a la implementación de estrategias efectivas que permitan cumplir con especificaciones sanitarias mínimas durante el manejo y procesamiento de los productos pesqueros, y así elevar los sistemas de control para disminuir los riesgos de brotes epidemiológicos en la región.











 nueva página del texto (beta)
nueva página del texto (beta)