Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Salud Pública de México
versión impresa ISSN 0036-3634
Salud pública Méx vol.44 no.4 Cuernavaca jul. 2002
Brote causado por Escherichia coli en Chalco, México
Iliana Alejandra Cortés-Ortiz, QFB,(1) Guadalupe Rodríguez-Angeles, M en C,(1) Eustolia Alejandra Moreno-Escobar, QFB,(1) Jesús Manuel Tenorio-Lara QFB,(1) Benita Pilar Torres-Mazadiego, QFB,(1) Edith Montiel-Vázquez, QFB.(2)
Cortés-Ortiz IA, Rodríguez-Angeles G, Moreno-Escobar EA, Tenorio-Lara JM, Torres-Mazadiego BP, Montiel-Vázquez E.
Brote causado por Escherichia coli en Chalco, México
Salud Publica Mex 2002;44:297-302.
El texto completo en inglés de este artículo está disponible en: http://www.insp.mx/salud/index.html
Resumen
Objetivo. Identificar el agente causal del brote de diarrea asociado con el desbordamiento del canal de aguas negras en Chalco. Material y métodos. Estudio retrospectivo y transversal, efectuado en el Instituto de Diagnóstico y Referencia Epidemiológicos (InDRE), de la Secretaría de Salud, con 1 550 hisopos rectales para el aislamiento e identificación bioquímica de V. cholerae y enterobacterias, obtenidos de la población del Valle de Chalco, que presentó diarrea y vómito durante el desastre natural acontecido el 31 de mayo de 2000. El análisis de los resultados se efectuó por la diferencia entre las proporciones de dos poblaciones (prueba de Ji cuadrada). Las cepas de E. coli se hibridaron por "colony blot" para los grupos ETEC, EIEC, EPEC y EHEC. Resultados. El 0.45% correspondió a Salmonella: S. agona, S. infantis, S. enteritidis, S. muenchen, S. typhimurium; 0.06% a Shigella flexneri 3a, y 76.6% a E. coli: 62.2% a ETEC (44.6 % con LT, 11.2% con ST, y 44.1% con ambas sondas), 0.84% a EIEC (sonda ial), 0.84% a EPEC (sonda bundle-forming pilus BFP), 0.08% a E. coli enterohemorrágica no-O157:H7 (sonda pCVD419), y 36.02% no hibridó. No se encontró asociación entre E. coli patógena con la edad y género. Conclusiones. Escherichia coli podría ser responsable del brote de diarrea. Es importante conocer el agente etiológico del brote para encaminar las estrategias en el estudio y control sanitario del mismo. El texto completo en inglés de este artículo está disponible en: http://www.insp.mx/salud/index.html
Palabras clave: Escherichia coli; brotes de enfermedades; sondas; AND; hibridación; México
Cortés-Ortiz IA, Rodríguez-Angeles G, Moreno-Escobar EA, Tenorio-Lara JM, Torres-Mazadiego BP, Montiel-Vázquez E.
Outbreak caused by Escherichia coli in Chalco, México
Salud Publica Mex 2002;44:297-302.
The English version of this paper is available at: http://www.insp.mx/salud/index.html
Abstract
Objective. To identify the etiologic agent responsible for a disease outbreak following an overflow of sewage water in Valle de Chalco, Mexico. Material and Methods. A retrospective cross-sectional study was carried out. Rectal samples were collected from the population of Chalco valley, who suffered from diarrhea and vomiting during a natural disaster that took place on May 31, 2000. The Instituto de Diagnóstico y Referencia Epidemiológicos (Epidemic Reference and Diagnosis Institute, InDRE, Ministry of Health), received 1521 rectal swab samples from diarrhea cases, to test for E. coli strains. Statistical analysis was performed to find a difference of proportions between cases and non-cases (chi-squared test). ETEC, EIEC, EPEC and EHEC pathogenic E. coli groups were hybridized by colony blot. Results. Strains isolated were ETEC (62.2%), EIEC (0.84%), EPEC (0.84%), and EHEC non-O157:H7 (0.08%); there was no hybridization in 36.02% of E. coli strains. Other isolated microorganisms were Salmonella spp (0.45%) and Shigella spp (0.06%). Conclusions. Enterotoxigenic E. coli was the most likely etiologic agent. Sanitary control strategies should be targeted to preventing outbreaks caused by this pathogenic agent. The English version of this paper is available at: http://www.insp.mx/salud/index.html
Key words: Escherichia coli; disease outbreaks; DNA probes; hybridization; Mexico
El municipio del Valle de Chalco Solidaridad se en cuentra en la parte oriente de la cuenca del Valle de México, sobre el lecho de un ex lago. Los ríos La Compañía y Amecameca sirven como límite natural al municipio; actualmente ambos confluyen en el Gran Canal, que drena aguas negras hacia la parte norte del Estado de México. Las viviendas están construidas predominantemente con paredes de tabique, los techos con láminas de asbesto, cartón o metal, y los pisos son de cemento. Cuentan con agua potable en 95.73% de los casos, pero sólo 57.74% de las viviendas tiene drenaje. De la superficie total del municipio 48.3% corresponde al área urbana y 51.7% es ocupado por áreas susceptibles de inundación en ciertas épocas del año, que se utiliza para el cultivo de algunos productos agrícolas. El 31 de mayo de 2000 se fragmentó la pared de contención del canal llamado "La Compañía," provocando el desbordamiento de las aguas negras que inundaron las colonias circunvecinas del Valle de Chalco. La mayoría de la gente se concentró en albergues, pero otros permanecieron en lo que quedaba disponible de sus viviendas. La inundación con las aguas negras en la zona residencial, el hacinamiento en los albergues, el difícil acceso al agua potable y a comida limpia, y el inadecuado manejo de las excretas, propiciaron el surgimiento de infecciones intestinales.1
Escherichia coli puede estar involucrada en problemas gastrointestinales, así como en infecciones urémicas y neonatales. Dentro de las infecciones intestinales, las E. coli se han clasificado en seis grupos: E. coli enterotoxigénica (ETEC), E. coli enteropatógena (EPEC), E. coli enteroinvasiva (EIEC), E. coli enterohemorrágica (EHEC), E. coli enteroagregativa (EAEC) y E. coli de adherencia difusa (DAEC).2 Escherichia coli enterotoxigénica se adquiere, al igual que los otros grupos patógenos de E. coli, por la ingestión de agua y alimentos contaminados. Las cepas de ETEC producen dos toxinas: LT (toxina termolábil) y ST (toxina termoestable), que actúan incrementando los niveles de Adenosina-5´- (AMPc) y Guanosina-5´- monofosfato cíclico (GMPc), respectivamente; esto provoca que las células de las criptas intestinales aumenten la secreción de agua y electrolitos, con la consecuente disminución en la absorción de las vellosidades, lo que clínicamente se manifiesta como diarrea acuosa.3 En EPEC, se ha involucrado al pilus BFP (bundle-forming pilus) como un factor de virulencia característico que participa en el proceso infeccioso4 al promover la adherencia íntima a enterocitos; posteriormente se produce la polimerización de la actina del citoesqueleto, seguida de la destrucción de las microvellosidades. Los cambios bioquímicos relacionados con este proceso son los que inducen probablemente la secreción de agua y electrolitos al espacio intraluminal, produciendo diarrea aguda.5,6 En EIEC, el mecanismo de patogenicidad es la invasividad que se inicia con la adherencia de la bacteria a las microvellosidades de la mucosa del intestino grueso, seguida por la entrada a la célula a través de una fagocitosis "no profesional," lo que afecta el borde de cepillo del enterocito. Ya libre en el citoplasma, se multiplica e invade a las células vecinas; la destrucción de las células, junto con la movilización de polimorfonucleares y macrófagos, desencadenan el proceso de inflamación y la aparición de diarrea con moco y sangre (disentería), muy similar a la producida por Shigella.7 En este mecanismo están implicados un plásmido de alto peso molecular (120-140MDa) y genes de invasión en el genoforo bacteriano, que codifican y regulan todo el proceso invasivo.8 En EHEC también se producen lesiones del tipo de "adherencia y esfacelación" como en EPEC, y además sintetiza dos citotoxinas, Toxina Shiga-like I y II (STX I y II), que producen la lisis en cultivo celular. Se ha reportado que la estructura de estas toxinas es similar a la LT de las cepas ETEC y su mecanismo de acción es en el nivel de la síntesis de proteínas. Además, poseen un plásmido de 90 Kb que codifica para la enterohemolisina. Este plásmido se encuentra presente en todos los aislamientos clínicos de O157:H7 asociados con colitis hemorrágica y síndrome urémico hemolítico.9 La producción de diarrea aguda y crónica en niños se debe a la acción de las toxinas y a la adherencia al enterocito por sus fimbrias.10 El grupo EAEC presenta un patrón de adherencia agregativa en cultivo celular.11 Este grupo está fuertemente asociado con diarrea persistente en niños.12 La patogénesis de EAEC está en estudio, aunque se han involucrado dos proteínas de alto peso molecular, Proteína codificada por un plásmido (Pet) y Proteína codificada en el genoma con actividad de proteasa (Pic), que producen acortamiento de las vellosidades, hemorragia, ulceración y necrosis en asa ligada de rata, además de la toxina ST de E. coli enteroagregativa (EAST 1) diferente de la que produce ETEC.11,13 DAEC es el grupo más reciente del cual aún no se conoce bien su mecanismo de patogenicidad.2
En México se ha aislado E. coli como agente etiológico de diarrea. Cravioto y colaboradores reportan, en un trabajo realizado en una localidad rural en el estado de Morelos, entre 1982 y 1985, a ETEC como el principal agente productor de diarrea (33.5%) y 8.1% para EPEC.14 Sepúlveda y colaboradores en 1984, reportaron 28% de aislamiento para EPEC y 13% para ETEC en un estudio de casos y controles, en una área del sur de la Ciudad de México.15 En 1990, Guerrant y colaboradores encontraron que ETEC es endémica en nuestro medio y que causa la diarrea del turista en todo el mundo.15 En 1991, Benítez y colaboradores comunicaron el aislamiento de cepas EHEC en 17% de una cohorte de niños que vivían en una comunidad rural.16
Este trabajo refiere los resultados obtenidos del estudio del brote que se presentó en el Valle de Chalco a raíz del desbordamiento del canal de aguas negras.
Material y métodos
El Instituto de Diagnóstico y Referencia Epidemiológicos (InDRE) recibió 1 550 muestras de hisopos rectales en medio de transporte Cary-Blair; cada hisopo provenía de un habitante del Valle de Chalco con problemas gastrointestinales. El plan de muestreo seleccionado, así como la ejecución del mismo, fue realizado por el personal de salud de la Dirección General de Epidemiología (DGE) de la Secretaría de Salud, quienes remitieron las muestras al laboratorio de Bacteriología Diagnóstica del InDRE, en donde las reciben de origen humano, para aislamiento e identificación bioquímica.
Las muestras se sembraron en tres medios de cultivo: directamente en una placa de agar MacConkey, para la búsqueda de enterobacterias, en un tubo con caldo tetrationato, y en agua peptonada alcalina como enriquecimiento, para la detección de Salmonella y Vibrio cholerae, respectivamente. Los cultivos obtenidos se resembraron en medios selectivos para cada grupo: se sembró en medio verde brillante y sulfito de bismuto para Salmonella, y en agar tiosulfato-citrato-bilis-sacarosa para Vibrio. Las colonias sospechosas se confirmaron bioquímicamente utilizando agar de triple azúcar hierro (TSI), agar lisina (LIA), medio movilidad, indol y ornitina (MIO), citrato de Simmons, urea de Christensen, caldo mucato y caldo sorbitol. Las cepas de Salmonella y Shigella aisladas se aglutinaron con sueros específicos para detectar su serotipo. De las cepas sospechosas a E. coli que se aislaron de la placa de agar MacConkey se seleccionaron cinco colonias por placa, las cuales fueron identificadas utilizando las pruebas bioquímicas anteriores, y se remitieron al laboratorio de Bacteriología Molecular, para confirmar su posible participación como agente patógeno.
Las 1 188 cepas de Escherichia coli se hibridaron en fase sólida (colony blot),17,18 lo que consistió en la siembra de las cepas en cinco placas de base de agar sangre (Difco) para transferirlas a membranas de nylon cargadas positivamente (MSI, Westboro, MA) después de 4h de incubación, ya que cada membrana se hibridó con una sonda diferente (cuadro I). A las 24h de incubación se retiraron las membranas y se procedió a la lisis de las colonias adheridas, para la obtención del DNA de la colonia, utilizando NaOH 0.5N, y el DNA obtenido se fijó por exposición a luz ultravioleta por tres minutos (Gene-Linker, BioRad). Las sondas para la detección de EHEC se obtuvieron a partir de la cepa pCVD419, que contiene un fragmento del gene que codifica para la enterohemolisina, la cual es un fragmento de 3400 bp cortado con la enzima de restricción Hind III. La sonda BFP se obtuvo de la cepa pMSD207, que contiene la sonda BFP para EPEC, un fragmento de 850 bp cortado con Eco RI.5,19 Las sondas para la enterohemolisina y BFP fueron marcadas por "random primed,'' con un kit de marcaje de DNA (Boheringer-Mannheim) con digoxigenina; siguiendo las instrucciones del fabricante, se preparó un "cocktail" con los hexanucleótidos y la mezcla de dNTP marcados con digoxigenina. El DNA molde se hirvió previamente para abrir las cadenas, y se le agregó la enzima Klenow para realizar la polimerización. A continuación, se dejó incubar a 37oC por 4 h. Las sondas IAL,20 ST y LT se marcaron mediante PCR utilizando dUTP marcado con digoxigenina (Boheringer-Mannheim).21 Los colony blots fueron hibridados a 65oC, y se revelaron mediante colorimetría utilizando NBT y BCIP (Sigma Chemical Co., St. Louis. Mo.).22 En el colony blot se incluyeron las cepas de referencia como testigos positivos: la cepa de E. coli H-10407 toxigénica, para el ensayo con las sondas LT y ST; la cepa pMS207, que tiene el gene codificador para la subunidad A del pili BFP, que identifica EPEC; la cepa O157:H7 para la sonda pCVD419 en la identificación de EHEC; y la cepa de E. coli E11 invasiva, para la sonda IAL para EIEC.

Resultados
De las 1 550 muestras de Cary-Blair, se obtuvo que 0.06% correspondió a Shigella (1/1 550), 0.45% a Salmonella (7/1 550), y 76.6% a E. coli (1 188/1 550). El 22.9% restante fueron bacterias sin importancia médica. El serotipo de Shigella fue S. flexneri 3a (1/1). Las cepas de Salmonella presentaron los siguientes serotipos: S. agona (2/7), S. infantis (1/7), S. enteritidis (2/7), S. muenchen (1/7), y S. typhimurium (1/7). No se aislaron colonias sospechosas de Vibrio cholerae.
En las 1 188 cepas de E. coli aisladas, 62.2% correspondió a ETEC (739/1 188), de las que 44.6% hibridó con la sonda para la enterotoxina LT, 11.2% con la enterotoxina ST, y 44.1% lo hizo con ambas sondas. El 0.84% de EIEC (10/1188) lo logró usando la sonda que detecta el locus asociado con la invasividad, ial; 0.84% a EPEC (10/1 188) con la sonda para la subunidad A del pili BFP, 0.08% a E. coli enterohemorrágica no O157:H7 (1/1 188) con la sonda para la enterohemolisina pCVD419, y 36.02% (428/1 188) no hibridó con ninguna de las sondas empleadas. Sin embargo, la única cepa que hibridó con la sonda pCVD419 no aglutinó con el antisuero O:157, por lo tanto, no se consideró en el grupo EHEC.
Las cepas que no hibridaron con ninguna de las sondas empleadas se consideraron E. coli no agrupadas, ya que dentro de ellas pudieron encontrarse E. coli enteroagregativas y E. coli de adherencia difusa que puedan estar involucradas en la producción de diarrea.
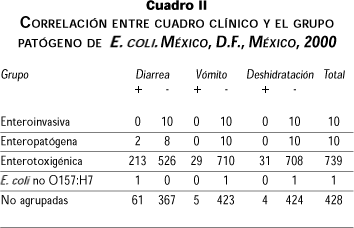
En el cuadro II se muestra la correlación entre el cuadro clínico (diarrea, vómito y deshidratación) y los grupos patógenos de E. coli. La presencia de diarrea varió, dependiendo del grupo de E. coli involucrado, con una diferencia estadísticamente significativa (p<0.0001), lo que indica claramente que ETEC presenta el mayor número de casos con diarrea, encontrándose 213 casos con ella en un total de 739 casos positivos a ETEC (28.8%). En contraste, los casos de diarrea causados por las cepas no agrupadas fueron 61, de un total de 428 (14.25%). El vómito fue un signo fuertemente asociado con la infección por cepas de ETEC presentándose en 4% de los casos positivos a esta cepa, mientras que en ninguno de los otros grupos tuvo la misma presencia. La deshidratación fue también más frecuente en ETEC, encontrándose una diferencia estadísticamente significativa (p=0.02) en comparación con los demás grupos de E. coli.
La infección por E. coli fue más frecuente en niños menores de cinco años entre el total de 1 188 muestras positivas. Estos datos se concentran en la figura 1. Sin embargo, no se encontró asociación estadísticamente significativa entre la edad y el grupo de E. coli involucrado.

En relación con el grupo de E. coli y el género de los pacientes, se observa que entre mujeres y hombres no hay diferencia significativa en la proporción del grupo de E. coli involucrado (p=0.1554). El grupo predominante que infecta tanto mujeres como hombres fue ETEC, con 62.3 y 62.08%, respectivamente. Estas diferencias tampoco son estadísticamente significativas (p=0.9386).
Discusión
Del total de muestras enviadas al Instituto se aisló Shigella en 0.06, Salmonella en 0.45 y E. coli en 76.6%. Del total de las cepas de E. coli aisladas, 63.96% fueron patógenas (ETEC, EIEC, EPEC y E. coli no O157:H7) y 36.02% comprendió a las cepas de E. coli que no pudieron asociarse con ningún grupo de E. coli con las sondas que se emplearon pero, presumiblemente, podrían encontrarse E. coli de importancia médica (E. coli enteroagregativa o de adherencia difusa). El alto porcentaje de aislamiento de E. coli patógena en este estudio, muestra claramente la importancia de E. coli como agente productor de gastroenteritis y como causa de brotes en la población. El grupo patógeno predominante de E. coli fue ETEC (62.2%), esto concuerda con lo descrito por Nataro y colaboradores (1988), quienes mencionan que ETEC es una importante causa de diarrea en zonas con condiciones sanitarias precarias y que en el ámbito mundial, es el grupo aislado con mayor frecuencia. Qadri y colaboradores (2000),23 en un estudio realizado durante dos años en Bangladesh, reportaron que ETEC es la causa principal de diarrea en niños, especialmente de 0 a 3 años. Además, en relación con la sintomatología presentada, se encontró que la asociación diarrea y grupo patógeno fue estadísticamente significativa (p<0.0001), y el grupo con más frecuencia de diarrea fue ETEC. La presencia de vómito y deshidratación también se asoció fuertemente con la infección por este grupo de E. coli.
Otro de los grupos de E. coli que se encontró fue EIEC (0.78%). Este grupo está asociado con cuadros de disentería bacilar semejantes al de la Shigella dysenteriae; sin embargo, en todos los casos en los cuales se aisló EIEC no se presentó diarrea; lo anterior podría deberse a que se hicieron aislamientos de portadores asintomáticos o a la resistencia natural del huésped. También es importante observar que en nuestro estudio el porcentaje de aislamiento de EIEC es diferente al reportado por Cravioto y colaboradores. (1985), quienes aislaron EIEC en 4% de los casos de una población infantil rural. Esta diferencia puede deberse a los tipos de población estudiada (rural vs. urbana) y a las diferentes condiciones epidemiológicas en las que fueron realizados estos estudios.
En este estudio se aisló EPEC en 0.84%, y sólo se encontraron dos casos de diarrea en un total de 10. Aunque se sabe que la diarrea causada por EPEC es similar a la causada por ETEC, se ha comunicado que en mayores de dos años su presencia como causante de diarrea no es estadísticamente significativa;2 esto podría explicar los casos de EPEC asintomáticos.
El 0.08% fue para una cepa que hibridó con la sonda pCVD419, pero por serología fue negativa para el antisuero O157. Por esta razón se consideró una E. coli no-O157:H7. Se ha hallado que el plásmido pO157 está presente prácticamente en todas las cepas O157:H7 y en otras de E. coli productoras de verotoxinas.9 Nataro y Kaper (1988) informaron que la E. coli no O157:H7 de los aislados no se encuentra implicada en brotes, ya que la expresión por estas cepas de las toxinas STX es aparentemente insuficiente para conferirles virulencia. Margall y colaboradores24 (1997) reportaron que los serotipos no O157:H7, además de producir toxina en menor cantidad, carecen de algún factor de patogenicidad como el gen E. coli attaching and effacing (eae) u otros, que codifica para la intimina que participa en la adherencia y pueden producir sólo enteritis y complicaciones menores. Por otra parte, Pebody y colaboradores25 (1999) reportaron un brote originado por una E. coli O157 verotoxigénica (no O157:H7), la que ocasionó en algunos pacientes síndrome urémico hemolítico.
El 36.02% correspondió a las cepas que no hibridaron con las sondas anteriores; este valor indica fuertemente la presencia de otros grupos de E. coli de importancia médica, como Escherichia coli enteroagregativa. Dicho grupo ha cobrado importancia en salud pública, principalmente en países desarrollados, y se ha visto implicado en gastroenteritis asociada con diarrea persistente (más de 14 días).12 Nataro y Kaper1 reportaron que respecto del grupo enteroagregativo de E. coli la forma más confiable para detectar EAEC, por biología molecular, es la PCR. Actualmente estamos evaluando el grupo de E. coli, que no pertenecieron a ningún grupo patógeno, para saber si EAEC se encuentra involucrada.
Es interesante el hecho de que, de todos los grupos de E. coli patógeno, ETEC sea el grupo bacteriano que se encuentra más frecuentemente relacionado con brotes y problemas gastrointestinales. La razón de esto no es clara, tal vez ETEC sea más resistente al medio ambiente, y por lo tanto se encuentre en mayor proporción, o que su capacidad para colonizar sea más efectiva. Nataro y Kaper (1988) confirmaron que la infección por EPEC en adultos no es frecuente, por la posible pérdida con la edad de receptores específicos.
En estudios como éste, es de suma importancia determinar el agente etiológico del brote, ya que las medidas que se deben tomar para el estudio y control sanitario son diferentes en el caso de infecciones por parásitos, bacterias o virus. En el caso de ETEC las estrategias específicas incluyen las medidas de intervención (rehidratación oral, uso de antibióticos) y de promoción de la higiene (agua y alimentos limpios, manejo adecuado de excretas). El aislamiento del agente etiológico permite, además, la vigilancia epidemiológica de microrganismos de notificación inmediata como E. coli O:157 H:7.
Agradecimientos: al Dr Esteban Puentes R, del Departamento de Epidemiología y Estadística, por su ayuda invaluable; a la QFB Patricia Frausto, por su colaboración en el cultivo de las bacterias, y al QFB Carlos Arturo Vázquez-Chacón, del Departamento de Biología Molecular, por su increíble apoyo en la redacción del artículo.
Referencias
1. Instituto Nacional de Estadística, Geografía e Informática. Anuario estadístico del Estado de México. México, D.F.: INEGI, 1996. [ Links ]
2. Nataro JP, Kaper J.Diarrheagenic Escherichia coli. Clin Microbiol Rev 1988;11:142-201. [ Links ]
3. Eslava C, Villaseca JM, Cravioto A. Cepas de Escherichia coli relacionadas con la diarrea. En: Diagnóstico de laboratorio de infecciones gastrointestinales. Giono-Cerezo S, Escobar-Gutiérrez A, Valdespino Gómez JL, ed. México, D.F., Instituto Nacional de Diagnóstico y Referencia Epidemiológicos, 1993:251-265. [ Links ]
4. Girón JA, Yue AS, Schoolnik GK. An inducible blundle-forming pilus of Enteropathogenic Escherichia coli. Science 1991;254:710-713. [ Links ]
5. Girón JA, Donnenberg MS, Martin WC, Jarvis KG, Kaper JB. Distribution of the bundle-forming filus structural gene (bfpA) among enteropathogenic Escherichia coli. J Infect Dis 1993;168:1037-1041. [ Links ]
6. Levine MM, Edelman R. Enteropathogenic Escherichia coli of classic serotypes associated with infant diarrhea: Epidemiology and pathogenesis. Epidemiol Rev 1984;6:31-51. [ Links ]
7. Rico-Martínez MG. Biología molecular en la patogenia de Shigella sp y Escherichia coli enteroinvasiva. Rev Latinoamericana Microbiol 1995;37: 367-385. [ Links ]
8. Buysse DM, Stover CK, Oaks EV, Venkatesan M, Kopecko DJ. Molecular cloning of invasion plasmid antigen (ipa) genes from Shigella flexneri: Analysis of ipa gene products and genetic mapping. J Bacteriol 1987;169: 2561-2569. [ Links ]
9. Levine MM, Xu J, Kaper J, Lior H, Prado V, Tall B et al. A DNA probe to identify enterohemorrhagic Escherichia coli of O157:H7 and other serotypes that cause hemorrhagic colitis and hemolytic uremic syndrome. J Infect Dis 1987;156:175-182. [ Links ]
10. Fukushima H, Hoshina K, Gomyoda M. Selective isolation of eae-positive strains of Shiga toxin-producing Escherichia coli. J Clin Microbiol 2000; 38:1684-1687. [ Links ]
11. Eslava C, Navarro F, Czeczulin JR, Henderson IR, Cravioto A, Nataro J. Pet, an autotransporter enterotoxina from enteroaggregative Escherichia coli. Infect Immun 1998;66:3155-3163. [ Links ]
12. Henry FJ, Udoy AS, Wanke CA, Aziz KM. Epidemiology of persistent diarrhea and etiologic agents in Mirzapur, Bangladesh. Acta Paediatr, 1996; 381 (Suppl):27-31. [ Links ]
13. Villaseca JM, Navarro F, Mendoza G, Nataro J, Cravioto A, Eslava C. Pet toxin from enteroaggregative Escherichia coli produces cellular damage associated with fodrin disruption. Infec Immun 2000;10:5920-5927. [ Links ]
14. Cravioto A, Ortega R, Rodríguez P, Reyes R, López D, Fernández G. Estudio longitudinal de colonización intestinal en una cohorte de niños rurales mexicanos. I. Diseño del estudio y hallazgos iniciales durante el periodo neonatal. Bol Med Hosp Infant Mex 1985;42:287-296. [ Links ]
15. Valdespino JL, García ML, Del Río A, Giono S, Salcedo RA, Sepúlveda J. Epidemiología y etiología de las diarreas Infecciosas. El caso de México. Rev Latinoam Microbiol 1994;36:307-324. [ Links ]
16. Benítez O, Uribe F, Navarro A, Hernández D, Ruiz T, Cravioto A. Etiología de diarrea con sangre en niños de una comunidad rural. Bol Med Hosp Infant Mex 1991;48:65-70. [ Links ]
17. Schultsz C, Pool GJ, Ketel R, De Wever B, Speelman P, Dankert J. Detection of enterotoxigenic Escherichia coli in stool samples by using nonradioactively labeled oligonucleotide DNA probes and PCR. J Clin Microbiol 1994;32;2393-2397. [ Links ]
18. Rademaker CM, Wolfhagen JJHM, Jansza M, Oteman M, Flutt A, Glerum JH et al. Digoxigenin labeled DNA probes for rapid detection of enterotoxigenic, enteropathogenic and vero cytotoxin producing Escherichia coli in fecal samples. J Microbiol Methods 1992;15:121-127. [ Links ]
19. Nagayama K, Bi Z, Ogachi T, Takarada Y, Shibata S, Honda T. Use of an alkaline phosphatase- conjugated oligonucleotide probe for the gene encoding the bundle-forming pilus of enteropathogenic Escherichia coli. J Clin Microbiol 1996;34:2819-2821. [ Links ]
20. Frankel G, Riley L, Girón JA, Valmassoi J, Friedmann A, Strakbine N et al. Detection of Shigella in feces using DNA amplification. J Infec Dis 1990; 61:1252-1256. [ Links ]
21. Jablonski E, Moomaw EW, Tullis RH, Ruth JL. Preparation of oligodeoxynucleotide-alkaline phosphatase conjugates and their use as hybridization probes. Nucleic Acids 1986;14:6115-6128. [ Links ]
22. Wachsmuth K. Laboratory detection of enterotoxins. En: Ellner PD, ed. Infectious diarrheal diseases: Current concepts and laboratoy procedures. CDC. Marcel Dekker New York 1984:93-115. [ Links ]
23. Qadri F, Das SK, Faruque AS, Fuchs GJ, Albert MJ, Sack RB et al. Prevalence of toxin types and colonization factors in enterotoxigenic Escherichia coli isolated during a 2-year period from diarrheal patients in Bangladesh. J Clin Microbiol 2000;38:27-31. [ Links ]
24. Margall N, Domínguez A, Prats G. Salleras L. Escherichia coli enterohemorrágica. Rev Esp Salud Publica 1997;71:437-443. [ Links ]
25. Pebody RG, Furtado C, Rojas A, McCarthy N, Nylen G, Ruutu P et al. An international outbreak of vero cytotoxin- producing Escherichia coli O157 infection amongst tourists; a challenge for the European infectious disease surveillance network. Epidemiol Infect 1999;123:217-223. [ Links ]
(1) Laboratorio de Bacteriología Molecular. Instituto de Diagnóstico y Referencia Epidemiológicos. México, D.F., México.
(2) Laboratorio de Bacteriología Enterica. Instituto de Diagnóstico y Referencia Epidemiológicos.
Fecha de recibido: 30 de enero de 2001 • Fecha de aprobado: 23 de enero de 2002
Solicitud de sobretiros: Guadalupe Rodríguez-Angeles. Laboratorio de Bacteriología Molecular, Departamento de Biología Molecular, Instituto de Diagnóstico y Referencia Epidemiológicos. Prolongación Carpio 470, colonia Santo Tomás, Delegación Miguel Hidalgo 11340. México, D.F.,México.
Correo electrónico: indre@cenids.ssa.gob.mx














