Introducción
Debido a que el pulmón es una de las principales vías de entrada de partículas y microorganismos patógenos, cuenta con un sistema inmunológico innato de defensa capaz de eliminarlos. Los macrófagos son las células encargadas de llevar a cabo un proceso continuo de eliminación de agentes externos a través de la fagocitosis (internalización de partículas o microorganismos), la degradación y la exocitosis (expulsión de las partículas o microorganismos degradados por medio de vesículas). Los productos de la exocitosis contienen material genético que puede ser indicativo de una patología. Debido a que el diagnóstico del agente patógeno en las enfermedades pulmonares requiere procedimientos invasivos que pueden comprometer la salud de los pacientes, en las últimas décadas se han realizado estudios en muestras biológicas no invasivas como la sangre periférica para identificar biomarcadores asociados a la enfermedad. Los biomarcadores o marcadores biológicos se definen como «una característica que mide y evalúa objetivamente como un indicador de procesos biológicos normales, procesos patológicos o respuestas farmacológicas a una intervención terapéutica».1 Aunque existe una gran cantidad de biomarcadores, las vesículas extracelulares de menor tamaño son de gran relevancia diagnóstica debido a que pueden migrar desde el órgano que los originó hacia la periferia, donde ejercen su función y pueden detectarse.
Los exosomas son microvesículas extracelulares de 30 a 100 nm de diámetro, derivadas de los cuerpos multivesiculares que se forman después de la ingestión de patógenos o partículas que se digieren dentro de los macrófagos y posteriormente son liberadas por exocitosis.2-4 Los exosomas pueden transportar moléculas tanto de la célula de origen como del patógeno, entre las que se encuentran: péptidos, proteínas, ARN mensajero (mARN) funcional y con mayor frecuencia, pequeños fragmentos de ARN conocidos como microARN (miARN).5
En esta revisión nos enfocaremos en conjuntar la información más relevante de los ARNs exosomales y su función en las enfermedades pulmonares causadas por agentes infecciosos y no infecciosos, enfocados en la comprensión de la enfermedad como biomarcadores para el diagnóstico o de pronóstico, y su posible uso terapéutico.
Biomarcadores exosomales en las enfermedades pulmonares causadas por agentes no infecciosos
ASMA
En México 8.5 millones de personas padecen asma, la cual es una afección respiratoria caracterizada por inflamación crónica6 en la que se produce una obstrucción respiratoria provocada por broncoconstricción, formación de edema así como una respuesta TH2 y secreción de IgE. Esta inflamación ocurre en respuesta a diversos estímulos tales como alérgenos, infecciones y contaminantes del aire.7-9 Durante la crisis asmática, los linfocitos T CD4+ reconocen los alérgenos ambientales y se diferencian en linfocitos TH2, los cuales producen citocinas como las interleucinas: IL-4, IL-5 e IL-13; además, también inducen la proliferación y la activación de las células cebadas y eosinófilos, lo que incrementa el proceso alérgico.8,10
En respuesta a los alérgenos, las células cebadas, los eosinófilos, las células dendríticas y las células epiteliales bronquiales generan exosomas, y el contenido intraexosomal incluye metabolitos activos y miARNs que regulan la activación o supresión de la respuesta inmunitaria y/o inducen la tolerancia inmunológica (Tabla 1).7,10-18 Estas moléculas y miARNs se han propuesto como biomarcadores de diagnóstico y como blancos terapéuticos para el control del asma.7,10-18
Tabla 1: Metabolitos y miARNs en asma.
| Metabolito | Origen | Modelo | Efecto | Uso | Referencia | |
|---|---|---|---|---|---|---|
| LTA4 hidrolasa y LTC4 sintasa | Células dendríticas derivadas de monocitos | In vitro, células dendríticas y macrófagos | Pueden metabolizar LTA4 a LT proinflamatorio B4 y C4, que inducen la migración de PMN | Objetivo terapéutico | (7),(10),(14) | |
| TGF-α1, IL-4 y GM-CSF | Células dendríticas | In vitro, células dendríticas | Modifican el fenotipo de las CPA, modulan la expresión y actividad de las enzimas de la vía LT en CPA | Diagnóstico y comprensión de la enfermedad | (7),(14) | |
| ETA y LTB4 | Células dendríticas | Modelos in vitro de células dendríticas | Quimiotácticos para eosinófilos y neutrófilos, inducen la migración de PMN y el reclutamiento de granulocitos a sitios inflamatorios | Comprensión de la enfermedad | (7),(10),(11) | |
| TGF-α1 | Macrófagos | In vitro, macrófagos | Disminuye la formación de exosomas | Objetivo terapéutico | (10),(11) | |
| Guanosina trifosfato fosfatasas de Rab | Macrófagos | In vitro, macrófagos | Reguladores del tráfico de membranas, están involucrados en el mecanismo de liberación de exosomas | Objetivo terapéutico | (11) | |
| IL-1α | Macrófagos | In vitro, macrófagos | Induce la diseminación microvesicular de los monocitos de sangre periférica | Objetivo terapéutico | (11) | |
| IFN-γ | Líquido de lavado broncoalveolar | Modelo de ratones | Incrementa la secreción de exosomas en macrófagos alveolares | Objetivo terapéutico | (11) | |
| POE, PBM y PCE | Exosomas de eosinófilos | Humano | Tienen funciones autócrinas y parácrinas, y mejoran la migración de eosinófilos e inducen la expresión de moléculas de adhesión como ICAM-1 y la integrina α2 | Comprensión de la enfermedad | (10),(11) | |
| Bet v1 | Células B | Humanos | Induce una respuesta de citocinas TH2, caracterizada por la liberación de IL-4, IL-5 e IL-13, y niveles bajos de IFN-γ y TNF-α | Objetivo terapéutico | (11),(17) | |
| IgE | Células cebadas | Humano | Induce la estimulación y la proliferación de linfocitos, e incrementa la producción de IL-2, IL-12, IFN-γ, IL-6 e IL-8. | Objetivo terapéutico | (10),(11),(12) | |
| CD63 y OX40L | Células cebadas derivadas de la médula ósea | Modelo de ratón | Se ligan con OX40 en la superficie de las células T e inducen la proliferación de células T y la diferenciación de las células TH2 | Objetivo terapéutico | (10),(18) | |
| FcεRI | Células cebadas | Humano | Se une a IgE libre, disminuyendo los niveles séricos de IgE, y limita los efectos de la activación de las células cebadas | Objetivo terapéutico | (10) | |
| miARNs | ||||||
| miR-150 | Células T citotóxicas CD8 + | Modelo de ratón | Suprime la dermatitis alérgica de contacto e induce tolerancia de antígenos específicos en ratones | Biomarcador diagnóstico y objetivos terapéuticos | Disminución de la expresión | (10),(16) |
| miR-221 | Células del músculo liso/tejido pulmonar | Humano/modelo de ratón | Regulación de la proliferación celular | Biomarcador diagnóstico | Incremento en la expresión | (11) |
| miR-21 | Suero | Humano | Regulación de la expresión de citocinas como la IL-12 | Biomarcador diagnóstico | Incremento en la expresión | (11) |
| miR-346 | Tejidos pulmonares | Modelo de ratón | Disminuye la respuesta TH2, regulando la expresión de IL-5 e IL-13 | Biomarcador diagnóstico y objetivos terapéuticos | Disminución de la expresión | (7),(15) |
| miR-1827 | Tejidos pulmonares | Modelo de ratón | Disminuye la respuesta TH2, regulando la expresión de IL-5 e IL-13 | Biomarcador diagnóstico y objetivos terapéuticos | Disminución de la expresión | (7),(15) |
| miR-574-5p | Tejidos pulmonares | Modelo de ratón | Disminuye la respuesta TH2, regulando la expresión de IL-5 e IL-13 | Biomarcador diagnóstico y objetivos terapéuticos | Disminución de la expresión | (7),(15) |
| miR-let-7 | Tejidos pulmonares | Modelo de ratón/CD4+ | Regula la secreción de IL-13 en células T | Biomarcador diagnóstico y objetivos terapéuticos | Disminución de la expresión | (11),(13) |
| miR-15a | Esputo y suero | Células T CD4+ | Regulación de la angiogénesis y la migración celular | Biomarcador diagnóstico | Disminución de la expresión | (11) |
| miR-146a y miR-146b | Células del músculo liso humanas | Humano | Regulación la expresión e inflamación de ciclooxigenasa-2 e IL-1α | Biomarcador diagnóstico | Disminución de la expresión | (11) |
| miR-19a | Esputo y suero | Humano | Regulación de la expresión de SOCS1 y A20 | Biomarcador diagnóstico | Incremento en la expresión | (11) |
| miR-126 | Tejido pulmonar | Modelo de ratón | Regulación de la inflamación | Biomarcador diagnóstico y pronóstico | Incremento en la expresión | (11) |
LTA4H hidrolasa = leucotrieno A4 hidrolasa, LTC4 sintasa = leucotrieno C4 sintasa, TGF-β1 = factor de crecimiento transformador de citocinas reguladoras beta 1, IL = interleucinas, GM-CSF = factor estimulante de colonias de granulocitos y macrófagos, LT = leucotrieno, CPA = células presentadoras de antígeno, ETA = ácido 5-cetoeicosatetraenoico, LTB4 = leucotrieno B4, PMN = leucocitos polimorfonucleares, IFN-γ = interferón-gamma, TNF-α = factor de necrosis tumoral, POE = peroxidasa de eosinófilos, PBM = proteína básica de mielina, PCE = proteína catiónica de eosinófilos, SOCS1 = supresor de la señalización de citosinas, Bet v1 = péptido de abedul, ICAM.1 = molécula de adhesión intercelular 1.
En los últimos años se ha profundizado el estudio de los exosomas para comprender el papel que tienen en los mecanismos fisiológicos y patológicos del asma. De manera general, se observó que las proteínas y algunos metabolitos están asociados con la patogenia de la enfermedad, mientras que los miARNs están asociados con la regulación y protección (Tabla 1).
EPOC
La enfermedad pulmonar obstructiva crónica (EPOC) es la tercera causa de defunción a nivel global;19 en México se reportaron 29,000 defunciones atribuibles a la EPOC en 2017.20 La EPOC es una enfermedad caracterizada por la inflamación crónica de las vías respiratorias que se produce por la inhalación de partículas o gases nocivos, y que limita el flujo del aire en los pulmones.21,22 Se ha identificado un amplio grupo de factores genéticos y ambientales como las causas del inicio y la progresión de la EPOC, siendo el tabaquismo una de las principales.21 Su patología se caracteriza por la destrucción de la estructura alveolar, la remodelación y el estrechamiento de las vías respiratorias pequeñas inducidas por inhalación de gases y partículas contaminantes.23,24
Aunque el papel de los exosomas en la EPOC ha sido poco estudiado, se ha reportado un incremento de los exosomas circulantes en pacientes con EPOC y durante la exacerbación aguda.25 El aumento en la producción de exosomas se correlaciona con marcadores inflamatorios sistémicos como el factor de necrosis tumoral (TNF) soluble y la IL-6, sugiriendo la relación entre los exosomas y el proceso inflamatorio en la EPOC.25 Por otro lado, en modelos de ratones se ha descrito que el mecanismo por el cual los exosomas contribuyen al desarrollo de la EPOC es a través de la elastasa transportada dentro de los exosomas, la cual une y degrada la matriz extracelular (MEC).26
En muestras de resección y de células pulmonares se han identificado miARNs exosomales, los cuales pueden ser útiles como biomarcadores para el diagnóstico y como objetivos terapéuticos,21,27-36 (Tabla 2). Aún se requiere buscar su expresión en los exosomas del suero de los pacientes para ser más accesible su uso diagnóstico.
Tabla 2: miARNs en la enfermedad pulmonar obstructiva crónica (EPOC).
| miARN | Muestra | Modelo | Expresión | Efecto | Uso | Referencia |
|---|---|---|---|---|---|---|
| EV-exosomas | ||||||
| miR-210 | Neumonectomía y lobectomía | Humano | Incremento en la expresión | Regula la autofagia a través de ATG7 en fibroblastos pulmonares | Objetivo terapéutico | (32) |
| Células pulmonares | ||||||
| miR-181c | Resección pulmonar | Humano/modelo en ratones | Disminución en la expresión | Regula la respuesta inflamatoria, quimiotaxis de neutrófilos, generación de especies reactivas de oxígeno | Objetivo terapéutico | (29) |
| miR-126 | Sangre/in vivo | Humano/modelo murino | Disminución en la expresión | Función en la angiogénesis y la homeostasis vascular | Objetivo terapéutico | (30) |
| miR-145 | Las células primarias del músculo liso de los pulmones | Humano | Incremento en la expresión | Regula la liberación de citocinas proinflamatorias (IL-6 y CXCL8) en las células del músculo liso por medio de la molécula SMAD3 | Objetivo terapéutico y biomarcador diagnóstico | (31) |
| miR-29c | Sangre | Humano | Incremento en la expresión | Es un regulador negativo de la expresión de la colágena | Diagnóstico, estadificación y pronóstico de la enfermedad | (33),(35) |
| miR-126 | Sangre | Humano | Disminución en la expresión | Regula la producción de CXCL12 | Diagnóstico, estadificación y pronóstico de la enfermedad | (33),(36) |
| miR-146a | Esputo | Humano | Disminución en la expresión | Induce la expresión de COX-2 y PGE2 | Diagnóstico, estadificación y pronóstico de la enfermedad | (27),(34) |
| miR-let- 7c | Esputo | Humano | Disminución en la expresión | Su expresión correlaciona inversamente con la concentración del TNF-RII | Diagnóstico, estadificación y pronóstico de la enfermedad | (27),(34) |
| miR-34a | Células microvasculares de pulmón | Humano | Incremento en la expresión | Controla la expresión de AKT, y la proteína p53 | Biomarcador diagnóstico | (28),(34) |
| miR-199a-5p | Células microvasculares de pulmón | Humano | Incremento en la expresión | Su expresión correlaciona inversamente con la expresión de la proteína HIF-1α | Biomarcador diagnóstico | (28),(34) |
IL = interleucinas, CXCL8 = por sus siglas en inglés C-X-C motif Chemiokine Ligand 8, SMAD3 = por sus siglas en inglés mothers against decapentaplegic homolog 3, CXCL-12 = por sus siglas en inglés C-X-C motif chemokine ligand 12, COX-2 = ciclooxigenasa-2, PGE2 = prostaglandina E2, RTFN-II = receptor del factor de necrosis tumoral tipo II.
Fibrosis pulmonar
No se cuenta con datos epidemiológicos de la fibrosis pulmonar idiopática (FPI) en nuestro país; se estima que en promedio cada neumólogo de México diagnostica ocho pacientes con FPI por año.37 Actualmente, la pirfenidona y el nintedanib son las únicas opciones de tratamiento aprobadas por la Administración de Medicamentos y Alimentos de los Estados Unidos (FDA), pero no detienen ni revierten la fibrosis, sino que simplemente retardan la progresión de la enfermedad. Al ser una enfermedad progresiva, incurable y mortal es de suma importancia su estudio con el fin de encontrar marcadores tempranos de la enfermedad o alternativas terapéuticas.39 La FPI es una enfermedad pulmonar intersticial fibrosante progresiva e irreversible de etiología desconocida que conduce a la muerte.39 En la FPI se observa formación de focos fibroblásticos y el depósito excesivo de proteínas de la MEC.38 Se caracteriza por un proceso de cicatrización anormal del tejido pulmonar que produce un exceso de tejido fibrótico; a medida que la fibrosis se engrosa, el tejido pulmonar va perdiendo la capacidad de facilitar el intercambio de gases y llevar el oxígeno a la sangre.39,40
Los exosomas y su contenido tienen un especial interés como objetivos terapéuticos y como biomarcadores diagnósticos en la FPI. Se han identificado miARNs involucrados en el desarrollo de fibrosis de órganos;38 en pacientes con FPI se han observado cambios significativos en la expresión de miARNs, como miR-21, miR-200 y Let-7, entre otros. Recientemente se informó que la disminución de la expresión de miR-30a podría estar asociada con la progresión de la FPI. Los estudios de los miARNs provenientes de muestras como el esputo, el tejido y células del pulmón se describen en la Tabla 3.36,41-50 Actualmente, la FPI se estudia de manera activa debido a la falta de tratamientos eficaces. Se ha puesto mayor interés en los miARNs, ya que pueden ser útiles como biomarcadores diagnósticos así como posibles objetivos terapéuticos, aunque sería de gran utilidad diagnóstica identificar esos miARNs en exosomas séricos.
Tabla 3: miARNs en la fibrosis pulmonar.
| miARN | Muestra | Modelo | Expresión | Efecto | Uso | Referencia |
|---|---|---|---|---|---|---|
| miR-142-3p | Esputo | Humano | Disminución en la expresión | Induce la proliferación de progenitores mesenquimales mediante el control de la señalización de WNT | Biomarcador diagnóstico | (41) |
| miR-let-7d-5p | Esputo | Humano | Disminución en la expresión | Induce la transición mesenquimal epitelial en las células epiteliales pulmonares | Biomarcador diagnóstico | (41) |
| miR-21 | Tejido pulmonar | Modelo murino | Incremento en la expresión | Reprime Smad7 promoviendo la activación de fibroblastos mediada por TGF-β | Biomarcador diagnóstico y objetivo terapéutico | (38),(46),(50) |
| miR-200 | Pulmones | Modelo murino | Disminución en la expresión | Inhibe la TEM inducida por TGF-β en células epiteliales alveolares mediante la represión de los factores de transcripción GATA3, ZEB1 o ZEB2 | Objetivo terapéutico | (38),(47) |
| miR-30 | Células pulmonares | Humano | Disminución en la expresión | Inhibe la expresión de Smad3 | Objetivo terapéutico | (39),(50) |
| miR-99 | Células pulmonares/tejido pulmonar | Humano/modelo murino | Disminución en la expresión | Activa la señalización de TGF-β, regula la expresión de CAV1 (mediador crítico del proceso de fibrosis pulmonar) y participa en los procesos fibróticos | Objetivo terapéutico | (39),(43) |
| miR-let-7 | Células pulmonares/tejido pulmonar | Humano/modelo murino | Disminución en la expresión | Se asocia con el proceso fibrótico | Objetivo terapéutico | (13),(34),(39) |
| miR-22 | Tejido pulmonar | Modelo murino | Incremento en la expresión | Induce la vía de señalización ERK en fibroblastos pulmonares humanos mediante la regulación de CTGF | Objetivo terapéutico | (38),(44),(45) |
WNT = Acrónimo del gen wingless de Drosophila y de su homólogo en vertebrados integrated o Int-1, TGF-β = factor de crecimiento transformante β, TEM, β-lactamasas TEM, GATA3 = , ZEB1 = factor transcripcional ZEB1, ZEB2 = factor transcripcional ZEB2, CAV1 = Caveolina 1, ERK = Extracellular signal-regulated kinase, CTGF = factor de crecimiento de tejido conectivo.
Cáncer pulmonar
Anualmente se diagnostican aproximadamente 6,000,000 de casos nuevos de cáncer de pulmón (CP) a nivel global.43,51 En México se diagnostican 9,000 casos nuevos de CP, siendo la primera causa de muerte por cáncer.52 El CP es un crecimiento anormal y descontrolado de células que invaden y destruyen el tejido pulmonar.53,54 Este tipo de CP se divide en células pequeñas, que tienen una incidencia de 25%, y en células no pequeñas que contienen dos subtipos patológicos principales: adenocarcinoma y carcinoma de células escamosas, con una incidencia de 80%.55,56 El CP es de mal pronóstico y la supervivencia es de cinco años.57
Los exosomas han sido ampliamente estudiados en cáncer, y se sabe que contribuyen a la progresión tumoral acarreando oncogenes,58 regulando la modulación del sistema inmunológico,59 que propicia el crecimiento tumoral60 y la formación de nichos premetastásicos,61 entre otros.62,63 Esto conduce a una red de interacciones muy complejas. Los exosomas en cáncer tienen un papel bimodal, es decir, pueden modificar el entorno local y/o sistémico y contribuir al desarrollo y esparcimiento del cáncer4 o pueden activar al sistema inmunológico para inducir una respuesta antitumoral.57 En la Tabla 4 se muestran los miARNs identificados en plasma y en tejido pulmonar y sus utilidades clínicas. Aún se requiere identificar una procedencia exosomal que sugiera una asociación directa con el cáncer pulmonar.
Tabla 4: miARNs en cáncer pulmonar.
| miARN | Muestra | Modelo | Expresión | Análisis | Uso | Referencia |
|---|---|---|---|---|---|---|
| miR-17-3p, miR-21, miR-106a, miR-146, miR-155, miR-199, miR-192, miR-203, miR-205, miR 210, miR-212, miR-214, let-7b-5p, let-7e-5p, miR-23a-3p, miR-486-5p, miR-151a-5p, miR-30a-3p, miR-200b-5p, miR-629, miR-100, miR-154-3p, miR-1246 | Tejido pulmonar | Humano | Incremento en la expresión | NSCLC versus donantes sanos | Biomarcador diagnóstico | (64),(65),(67) |
| miR-132-3p, miR-181b-5p, miR-27a-3p, miR-27b-3p, miR-320a, miR-361-5p, let-7b-5p, miR-24-3p, miR-3184-5p, miR-486-5p, miR-486-3p | Plasma | Humano | Incremento en la expresión | Adenocarcinoma versus donantes sanos | Biomarcador diagnóstico | (68) |
| let-7a-5p, let-7d-5p, let-7f-5p, miR-26b-5p, miR-30a-3p, miR-30e-3p, miR-744-5p, let-7e-5p, miR-191-5p, miR-206, miR-21-5p, miR-23a-5p, miR-23b-5p | Plasma | Humano | Disminución en la expresión | Adenocarcinoma versus donantes sanos | Biomarcador diagnóstico | (68) |
| miR-320b, let-7b-5p, miR-24-3p, miR-3184-5p, miR-486-5p, miR-486-3p | Plasma | Humano | Incremento en la expresión | Carcinoma de células escamosas versus donantes sanos | Biomarcador diagnóstico | (66) |
| miR-10b-5p, miR-15b-5p, let-7e-5p, miR-191-5p, miR-206, miR-21-5p, miR-23a-5p, miR-23b-5p | Plasma | Humano | Disminución en la expresión | Carcinoma de células escamosas versus donantes sanos | Biomarcador diagnóstico | (68) |
| miR-23b-3p, miR-10b-5p, miR-21-5p | Plasma | Humano | Incremento en la expresión | Mala supervivencia cáncer pulmonar | Biomarcador diagnóstico | (66) |
NSCLC = cáncer pulmonar de células no pequeñas.
Biomarcadores exosomales en las enfermedades pulmonares causadas por microorganismos patógenos
Influenza
Se estima que cada año entre 3,000,000 y 5,000,000 de personas se enferman gravemente por influenza y entre 290,000 y 650,000 personas fallecen a causa de la influenza a nivel global.69 La influenza es una infección viral que afecta la nariz, la garganta, los bronquios y los pulmones; se transmite mediante gotículas y pequeñas partículas expulsadas con la tos y/o los estornudos, y puede producir desde una enfermedad tipo gripal con síntomas leves hasta neumonías e incluso la muerte. Existen cuatro tipos de virus de influenza: A-D; sin embargo, los que representan un problema de salud pública son los virus de influenza A (IAV, por sus siglas en inglés) y B, siendo el IAV el causante de la epidemia de influenza en 2009 en México.70
El virus de influenza A (IAV) es parte de la familia Orthomyxoviridae, está compuesto por ocho segmentos de ARN monocatenario encerrados dentro de una matriz proteica.71 Algunas de las proteínas que codifican los segmentos de ARN son NS2, NS1, M1, M2, NP, PB1, PB2, PA, y HA, que es una proteína de unión al receptor, y NA, enzima que degrada ácido siálico, entre otras. En la infección por el IAV, la proteína HA se une a receptores celulares con residuos de ácido siálico, permitiendo la entrada del virus.72 El IAV se clasifica en subtipos de acuerdo con las combinaciones de dos proteínas de superficie: la hemaglutinina (HA), y la neuraminidasa (NA). Actualmente hay dos subtipos que circulan entre humanos: AH1N1 y AH3N2.
Los exosomas derivados de células infectadas con IAV pueden activar las células del sistema inmunológico a través de los epítopos virales de la HA H-2M cargados en las moléculas del complejo principal de histocompatibilidad clase II (MHCII), los cuales pueden ser presentados a las células T e inducir una respuesta inmunitaria en el hospedero.73 También se ha observado que los exosomas pueden unir las partículas virales por reconocimiento del ácido siálico, neutralizando la capacidad infectiva de los virus.74
Los miARNs contenidos en los exosomas tienen funciones reguladoras; se ha identificado que el miR-483-3p aislado de lavado broncoalveolar durante la infección por el IAV, inhibe los reguladores negativos de la vía de señalización RIG-I e induce la expresión de citocinas proinflamatorias y genes estimulados por IFN (interferón) , como IL-6, CCL2, TNF-α y SP110. En influenza aviar, el miR-483-3p de exosomas regula la producción de citocinas inflamatorias como el IFN-β, IL-6, CCL2 y TNF-α en células endoteliales vasculares, sugiriendo que podría ser un mecanismo por el que se produce una secreción exacerbada de citocinas proinflamatorias.75 Ambos miARNs podrían ser biomarcadores para el desarrollo de la inflamación excesiva en pacientes con influenza.
Al igual que en la EPOC, en la influenza se han reportado miARNs provenientes de tejidos y células pulmonares como posibles biomarcadores, por lo que su identificación en los exosomas séricos proporcionaría un uso más generalizado. Se ha identificado la expresión del miR-146a asociada a la replicación de IAV, mientras que la disminución en la expresión reprime la replicación viral; el mecanismo sugerido es a través de la disminución de la respuesta de los interferones tipo 1. Al inhibir el miR-146a, se observó que las lesiones pulmonares (LP) de ratones disminuyen y la tasa de supervivencia se incrementa, sugiriendo que el miR-146a podría ser un blanco terapéutico para disminuir la severidad de la infección por IAV.76 Otros miARNs de importancia clínica son el miR-302c que, al igual que el miR-146a, propicia la replicación viral a través de la disminución de la expresión IFN-β,77 y los miARNs: hsa-mir-127-3p, hsa-mir-486-5p, hsa-mir-593-5p y mmu-mir-487b-5p, los cuales en un tratamiento combinado suprimieron la replicación viral y mejoraron eficazmente la protección contra una dosis letal de IAV en ratones.78
Debido a la capacidad de los miARNs para neutralizar las partículas virales y su participación en la inducción de moléculas antivirales como los IFN-I, los miARNs podrían ser blancos terapéuticos en las infecciones causadas por IAV.
COVID-19
A finales de 2019, un coronavirus previamente desconocido, designado como SARS-CoV-2 y descubierto en la ciudad de Wuhan, China, provocó un brote de neumonía viral inusual denominada COVID-19.79,80 La COVID-19 es una enfermedad zoonótica que causa un síndrome respiratorio agudo que se ha extendido por todo el mundo, alcanzando proporciones pandémicas.81 Esta enfermedad tiene un espectro clínico muy variable, se presenta tanto como una enfermedad asintomática como con cuadros clínicos paucisintomáticos, leves, moderados, graves o críticos. En el caso de las condiciones clínicas leves se presenta comúnmente fiebre, tos seca, fatiga, dolor de cabeza y neumonía leve; mientras que la condición clínica grave se asocia con disnea, síndrome de dificultad respiratoria aguda (SDRA) e hipoxemia.82 Los casos más críticos requieren ventilación mecánica para controlar la insuficiencia respiratoria. En pacientes críticos, las manifestaciones sistémicas como el choque séptico, la disfunción multiorgánica o la insuficiencia multiorgánica a menudo se producen como consecuencia de la tormenta de citocinas inflamatorias desencadenada por el virus.83
El SARS-CoV-2 es un β-coronavirus de cadena positiva sencilla de ARN,84 el cual está constituido por la glicoproteína de envoltura pequeña (E), glicoproteína de membrana (M), proteína de nucleocápside (N), varias proteínas accesorias y por la glicoproteína spike (S), que facilita la entrada viral a las células del hospedero mediante el receptor de la enzima convertidora de angiotensina 2 (ACE2, por sus siglas en inglés). La serina proteasa TMPRSS2 interviene en la internalización de la proteína S, fusionándola con la membrana del hospedero.85 La relación de los exosomas con la infección por SARS-CoV-2 aún no se ha comprendido completamente; sin embargo, se ha demostrado que la respuesta de las células inmunes es regulada por los exosomas de las células infectadas por virus, aumentando la propagación y la infección del virus a través de la trasmisión del genoma viral a células sanas, además se ha reportado que los receptores exosomales pueden aumentar la entrada del virus a las células, y se piensa que en el caso del SARS-CoV-2, los exosomas pueden estar implicados en la propagación y la infección por los receptores CD9 y ACE2.86 Debido a que los virus SARS «secuestran» la maquinaria de biogénesis de las vesículas extracelulares, modificándola para crear un entorno apto para la infección viral, la generación de vesículas con partículas virales podría ser una estrategia para evadir su detección por parte del sistema inmunológico; sin embargo, no se han realizado estudios en profundidad sobre las vesículas extracelulares (EV) y su relación con los coronavirus.87
Mediante un análisis computacional se ha reportado que los miARNs del hospedero y los del virus pueden estimular la patogénesis viral a través de la desregulación de algunas de las principales vías de señalización inmune antiviral, como la autofagia y la señalización IFN-I, y también puede aumentar las complicaciones en los pacientes infectados.88,89 Se ha sugerido que miR-1207-5p es capaz de interactuar con el genoma de SARS-CoV-2, alterando la regulación del factor estimulante de colonias-1 (CSF-1), lo que propicia una respuesta inflamatoria en pacientes con COVID-19; es probable que esto sea un factor del desarrollo de la FPI debido a la estimulación de la transición epitelial a mesénquima.90 Por otro lado, se ha observado que la sobreexpresión de miR-200c inhibe la expresión de ACE2 en cardiomiocitos, lo que podría prevenir complicaciones cardiovasculares.91 Además, se ha reportado que las terapias basadas en EV podrían tener un potencial significativo para tratar LP ocasionadas por COVID-19 debido a su capacidad para reducir la inflamación, regenerar el epitelio alveolar, reparar la permeabilidad microvascular así como prevenir la FPI. Estas capacidades son mediadas por los miRs-126, 30b-3p, 145, 27a-3p, sindecan-1, factor de crecimiento de hepatocitos y angiopoyetina-1, los cuales son liberados de las EV92 y posiblemente sean transportadas por exosomas, por lo que se requiere identificarlos en exosomas séricos.
La información sobre el papel de los miARNs en la COVID-19 aún es escasa; sin embargo, los datos reportados están enfocados en su posible uso como biomarcadores y agentes terapéuticos.
Neumonía
La muerte por neumonía en niños a nivel mundial fue de 808,694 en 2017, lo que corresponde a 15% de todas las defunciones de niños menores de cinco años.93 En México se reportaron 117,731 casos nuevos de neumonía en 2018.94 La neumonía es una infección respiratoria aguda causada por una variedad de patógenos, siendo las más frecuentes las infecciones causadas por bacterias como Streptococcus pneumoniae, Haemophilus influenzae y Moraxella catarrhalis; mientras que las infecciones causadas por virus se deben principalmente al virus sincitial respiratorio (VSR).95,96 Los patógenos se acumulan en las vías respiratorias inferiores y los alvéolos provocando la entrada de líquido exudativo inflamatorio,97 lo que causa infiltrados pulmonares visibles en la radiografía de tórax.97,98 La neumonía es un factor de riesgo en individuos muy jóvenes o ancianos, siendo la principal causa individual de mortalidad infantil en todo el mundo.93,95
El papel de los exosomas se ha reportado en la infección por S. pneumoniae y Klebsiella pneumoniae, donde se observó que los macrófagos alveolares de ratones y de humanos generan exosomas que contienen miARN que regula la IL-36γ.99 También durante la infección con VSR se observó transporte de ARN y proteínas del hospedero así como ARN viral, los cuales pueden activar la respuesta inmunitaria innata e inducir la liberación de mediadores proinflamatorios como las quimiocinas MCP-1, IP-10 y RANTES en monocitos humanos y células epiteliales de las vías respiratorias.100
Por otro lado, el papel de los exosomas como posibles biomarcadores se ha sugerido como grupos de miARNs. En la neumonía grave adquirida en la comunidad se encontró que la expresión de un subconjunto de miARNs exosomales (miR-146a, miR-27a, miR-126 y miR-155) se incrementa en los exosomas del suero de pacientes con el síndrome de dificultad respiratoria aguda (SDRA).101 El miR-126 es de especial importancia, ya que regula la integridad vascular y la angiogénesis al mejorar las acciones del factor del crecimiento endotelial promoviendo la regeneración vascular,102-104 por lo que los autores postulan que el miR-126 es inducido en gran parte por una respuesta inflamatoria, lo que explica los niveles elevados del miR-126 en pacientes con SDRA, respaldando su posible uso como biomarcador dirigido para la detección temprana y el pronóstico de SDRA.101 También se ha reportado el uso de exosomas y miARNs como posible biomarcador para el diagnóstico de neumonía en niños causada por adenovirus.105 El estudio se realizó en suero de niños con neumonía causada por el virus, encontrando la expresión de dos pares de miARNs (miR-450a-5p/miR-103a-3p y miR-103b-5p/miR-98-5p), los cuales pueden proporcionar biomarcadores para el diagnóstico de la enfermedad.105 El miR-450a-5p controla la expresión del mARN del STAT 1; el miR-98-5p puede inhibir la activación de la señalización del IFN-α y el miR-103a-3p participa en la diferenciación de linfocitos T, como TH2 y TH17, en el asma equino grave.106-108 Estos miARNs regulan la vía de señalización de los interferones tipo 1, y pueden afectar la respuesta inmunitaria durante la progresión de la neumonía causada por adenovirus, por lo que se sugieren como posibles biomarcadores.105
Debido a que la neumonía es causada por diversos agentes patógenos, se desconoce el papel que tienen los exosomas inducidos por cada microorganismo en esta enfermedad. Los estudios realizados se han enfocado principalmente en la biomedicina para el uso de miARNs como biomarcadores en el diagnóstico de la enfermedad. Debido a la escasa investigación en esta área, se deben realizar más estudios respecto a los exosomas y su contenido molecular para contribuir a la comprensión de la patogenia, diagnóstico y tratamiento de la enfermedad.
Tuberculosis
De acuerdo con datos de la Organización Mundial de la Salud (OMS), en 2018 se registraron 10,000,000 de personas con tuberculosis (TB) y 1.4 millones murieron a causa de esta enfermedad. La TB es una de las 10 principales causas de defunción y la principal causa de muerte por un solo agente infeccioso a nivel global.109 En México se reportaron 1,800 defunciones por TB en 2018, de las cuales 90% fueron por TB pulmonar.110 La TB es una enfermedad infecto-contagiosa causada por Mycobacterium tuberculosis.111 La TB puede presentarse como un espectro dinámico desde una infección asintomática hasta una enfermedad potencialmente mortal.112 Los bacilos de M. tuberculosis se transmiten por aerosoles y, aunque en 90% de los individuos infectados causa una infección asintomática, M. tuberculosis es capaz de evadir la respuesta inmunitaria y permanecer en estado latente.113 Por otro lado, M. tuberculosis es un bacilo de difícil tratamiento y continuamente se están seleccionando cepas resistentes a los fármacos, generando TB farmacorresistente, lo que aumenta el reto para el control de la enfermedad,112,114 por lo que la búsqueda de biomarcadores para el diagnóstico y seguimiento de tratamiento es una de los principales metas.
Existe evidencia de que los exosomas pueden transportar componentes derivados de M. tuberculosis después de la infección.115,116 Se sabe que los exosomas derivados de macrófagos infectados aumentan la liberación de IFN-γ, TNF-α, RANTES e iNOS; estas citocinas y quimiocinas tienen la capacidad de estimular macrófagos naïve de una manera proinflamatoria y pueden activar células T CD4+ y CD8+, dependiendo de una ruta alternativa de presentación de antígenos a estas células.115,117 Además, se ha demostrado que la proteína de choque térmico 70 del huésped transportada en exosomas incrementa la respuesta proinflamatoria.117 Por otro lado, los exosomas provenientes de células infectadas por M. tuberculosis contienen un conjunto de miARNs específicos, los cuales son de importancia biomédica, ya que pueden tener un uso como biomarcadores para el diagnóstico de la enfermedad, e incluso diagnosticar entre TB activa y TB latente. Los principales miARNs, su origen, función y uso se muestran en al Tabla 5.118-131
Tabla 5: miARNs en tuberculosis pulmonar.
| miARN | Muestra | Modelo | Expresión | Efecto | Uso | Referencias |
|---|---|---|---|---|---|---|
| hsa-let-7e-5p y hsa-let-7d-5p | Suero sanguíneo | Humano | Incremento en la expresión | En la respuesta inmunológica hacia M. tuberculosis | Biomarcador diagnóstico para TBL | (119),(120),(125),(126) |
| hsa-miR-142-3p | Suero sanguíneo | Humano | Incremento en la expresión | Modula la fagocitosis interactuando con N-Wasp en las células hospedero | Biomarcador diagnóstico | (119),(127) |
| miR-144-3p | Suero sanguíneo | Humano | Incremento en la expresión | Inhibe la autofagia y facilita la infección por BCG induciendo ATG4a en células RAW264.7 | Biomarcador diagnóstico | (119),(128) |
| miR-23a-5p | Suero sanguíneo | Humano | Incremento en la expresión | Induce la autofagia y la supervivencia de M. tuberculosis | Biomarcador diagnóstico | (119),(129) |
| miR-484 | Sangre periférica | Humano | Incremento en la expresión | Regula las vías metabólicas activando la proteína de fisión mitocondrial 1 | Biomarcador diagnóstico | (121),(130),(131) |
| miR-5094 | Sangre periférica | Humano | Incremento en la expresión | Incrementa la infección por M. tuberculosis | Biomarcador diagnóstico | (130) |
| miR-1293 | Sangre periférica | Humano | Incremento en la expresión | Induce la expresión de mirtrones para facilitar la infección por M. tuberculosis | Biomarcador diagnóstico | (130) |
| miR-6848 | Sangre periférica | Humano | Incremento en la expresión | Induce la expresión de mirtrones para facilitar la infección por M. tuberculosis | Biomarcador diagnóstico | (130) |
| miR-6849 | Sangre periférica | Humano | Incremento en la expresión | Induce la expresión de mirtrones para facilitar la infección por M. tuberculosis | Biomarcador diagnóstico | (130) |
| miR-20b | Exosomas de plasma | Humano | Incremento en la expresión | Regula el factor de transcripción HIF1 y regula genes involucrados en la proliferación celular, migración, metabolismo energético, angiogénesis y apoptosis | Biomarcador diagnóstico | (122),(123) |
| miR-19 | Exosomas de plasma | Humano | Incremento en la expresión | Diferenciación y desarrollo celular | Biomarcador diagnóstico | (122),(124) |
NWASP-proteína = neural del síndrome de Wiskott-Aldrich, BCG = vacuna de Bacillus Calmette-Guerin, ATC4a = Autophagy Related 4a Cysteine Peptidase, RAW264.7 = línea celular de macrófagos de ratones, HIF-1 = factor 1 inducible por hipoxia.
Por último, se ha reportado que los exosomas generados durante la estimulación de macrófagos con proteínas de filtrado de cultivo (PFC) de M. tuberculosis activan las células T y reducen la carga bacteriana en ratones tratados con estos exosomas, similar a los ratones vacunados con bacilo de Calmette-Guérin (BCG); sin embargo, no está claro si esta respuesta se debe a un componente específico en los exosomas o a las proteínas micobacterianas como ESAT-6 (Rv3875), el complejo Ag85 (Rv3804c, Rv1886c, y Rv0129c), MPT64 (1980c), y MPT63 (1926c) acarreadas dentro de los exosomas, las cuales son altamente inmunogénicas.119 Por este motivo se han propuesto los exosomas de macrófagos infectados en el diseño de nuevas vacunas; sin embargo, se necesitan más estudios para comprender mejor el mecanismo de acción.132
Los miARNs y ARNs exosomales expresados o desregulados en las enfermedades pulmonares causadas por agentes infecciosos y no infecciosos para la comprensión de la enfermedad, o para su uso como biomarcadores en el diagnóstico y el pronóstico, y para su uso terapéutico se resumen en la Figura 1.
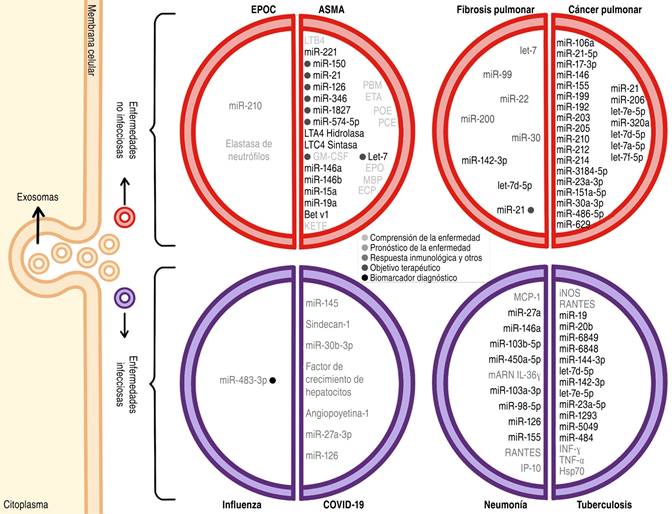
Figura 1: Contenidos exosomales que se inducen en las enfermedades pulmonares. miRNAs exosomales que se han identificado en las enfermedades no infecciosas como EPOC, asma, fibrosis y cáncer pulmonar (círculos rojos) y las enfermedades infecciosas como influenza, COVID-19, neumonía y tuberculosis (círculos morados), y que se han sugerido como pronóstico de la enfermedad, en la respuesta inmunitaria y como objetivos terapéuticos o biomarcadores para el diagnóstico. EPOC = enfermedad pulmonar obstructiva crónica, GM-CSF = factor estimulante de colonias de granulocitos y macrófagos, PBM = proteína básica de mielina, ETA = ácido 5-cetoeicosatetraenoico, POE = peroxidasa de eosinófilos, PCE = proteína catiónica de eosinófilos, EPO = eritropoyetina, MBP = major basic protein o principal proteína básica, KETE = 5-keto eicosatetraenoic acid o ácido 5-ceto eicosatetranoico, TNF-α = factor de necrosis tumoral, Hsp70 = beat shock protein, proteína de shock térmico de 70 KDa.
Comentarios finales
1. El diagnóstico de las enfermedades pulmonares es un desafío debido a que se requiere de procedimientos invasivos para identificar al agente causal, por lo cual, desde hace ya algunas décadas las investigaciones se han enfocado en evaluar los exosomas para su uso clínico. La diferencia de evaluar los miARNs en exosomas, y no la muestra de sangre completa, plasma o suero, es que aumenta la sensibilidad para detectar las secuencias que provienen del sitio de la enfermedad.
2. Los miARNs exosomales diferencialmente expresados en las enfermedades pulmonares se han propuesto como biomarcadores en el diagnóstico, para monitorear las terapias, e incluso como posibles mecanismos para inmunizar. Aunque aún falta por identificar los miARNs que se expresan de manera diferencial en las enfermedades pulmonares como los casos del miR-21 que incrementa su expresión tanto en asma como en fibrosis, del miR-let-7 que disminuye su expresión en asma y fibrosis, del let-7e-5p que incrementó su expresión tanto en tuberculosis como en cáncer y del miR-126 que incrementa su expresión en neumonía y asma, es posible que estos miARNs estén asociados al proceso inflamatorio y no sean específicos de la enfermedad.
3. Los miARNs exosomales que participan en la patología y fisiología de las enfermedades respiratorias se presentan en la Figura 2. Si bien los resultados son prometedores, aún es de gran importancia realizar la investigación de la fase clínica para su uso.

Figura 2: Exosomas y su papel en las enfermedades pulmonares. Efecto de los miARNs en la fisiología y patología de las enfermedades pulmonares no infecciosas (círculo morado). Efecto de los miARNs en la fisiología y patología de las enfermedades pulmonares infecciosas (círculo rojo). TB = tuberculosis, FPI = fibrosis pulmonar idiopática , MPs = micropartículas , PMN = polimorfonucleares, TNF = por sus siglas en inglés, tumor necrosis factor, ATG7 = autophagy related 7 protein, MEC = matriz extracelular, COX-2 = ciclooxigenasa-2, PGE2 = prostaglandina E2, EPOC = enfermedad pulmonar obstructivo crónica, FP = fibrosis pulmonar, CP = cáncer pulmonar, MPs= micropartículas.











 nueva página del texto (beta)
nueva página del texto (beta)


