INTRODUCCIÓN
El ser humano no es un organismo eucariote independiente, coexiste en equilibrio con varios microbios (por ejemplo, comensales, simbiontes y patobiontes). Los microbios, junto con sus productos biológicos y su carga genética, habitan la superficie y el interior del cuerpo formando un órgano operativo único conocido como microbioma humano que es crucial para nuestra salud y fisiología1-4. Sin embargo, bajo ciertas circunstancias, el equilibrio entre los comensales y los patobiontes puede romperse y, en consecuencia, crear un estado de desequilibrio microbiano conocido como disbiosis que puede ser un factor contribuyente de enfermedad. Además, la diversidad y el equilibrio del microbioma están influenciados por factores como el desarrollo humano, la susceptibilidad genética e incluso la localización geográfica. Por lo tanto, se sabe que algunos individuos en ciertas etapas de la vida o diferentes etnias pueden ser más susceptibles a ciertas enfermedades asociadas con la disbiosis en comparación con otros grupos. Actualmente se sabe que la cavidad oral alberga la segunda microbiota más abundante, después del tracto gastrointestinal5 y se ha demostrado que los taxones bacterianos específicos que colonizan la boca (también llamado microbiota bacteriana o bacterioma) se asocian con diferentes enfermedades locales y sistémicas. Sin embargo, los estudios del microbioma oral humano sólo se han realizado en ciertas poblaciones, por lo que sería importante revisar ciertos factores del huésped, intrínsecos o extrínsecos, con el fin de tener un mejor entendimiento de cómo la variabilidad inter e intraindividual podría influir en el microbioma oral humano en estados de salud o enfermedad en diferentes poblaciones.
VARIABILIDAD DEL MICROBIOMA ORAL
La composición del microbioma oral es relativamente estable a lo largo de la vida, sin embargo, es posible observar cambios en el microbioma oral a lo largo de diferentes etapas en un individuo o entre individuos. La gran variabilidad que presenta el microbioma oral humano, está reportada actualmente en la expanded Human Oral Microbiome Database (eHOMD), que incluye 775 especies microbianas, de las cuales, 57% son oficialmente nombradas, 13% sin nombre pero cultivables y 30% son conocidas sólo como filotipos no cultivables, así como un total de 2,074 genomas oral/nasales, representando 529 taxones (95% de los cultivables) de los cuales las phyla Actinobacterias, Bacteroidetes, Firmicutes, Fusobacterias, Proteobacterias, Spirochaetes y Sinergistetes con sus respectivos géneros y especies son las comunidades bacterianas más representativas de la cavidad oral. Adicionalmente, la eHOMD incluye taxones para genomas de un nuevo filo de nanomicroorganismos conocidos como clase candidata de bacterias o CPR (por sus siglas en inglés, candidate phyla radiation) y recientemente designadas como ultramicrobacterias “UMB” que incluyen una amplia variedad de géneros donde destacan Saccaribacteria (TM7), GN02, SR1, WPPS-2, que se estima puedan incluir desde 26 hasta 50% del total de la diversidad bacteriana6,7. finalmente, dentro de la variabilidad del microbioma oral, pueden presentarse diferentes factores que a continuación se describen:
El factor demográfico
La composición del microbioma oral no sólo está influenciada por la edad e ingesta de medicamentos, sino también por variables adicionales, incluyendo la genética del hospedero, el sesgo de género (dimorfismo sexual), el nivel socioeconómico, la etnia y la geografía, las cuales pueden reflejar variaciones en la dieta, y la convivencia de cada individuo8. Por ejemplo, estudios comparativos entre poblaciones europeas, africanas, asiáticas y norteamericanas mostraron una variación microbiana entre poblaciones, así como un microbioma oral conservado en grupos étnicos específicos con diferencias interindividuales, p. ej., un estudio en poblaciones norteamericanas reportó que las phyla Firmicutes, Proteobacterias, Bacteroidetes y Actinobacterias y géneros Streptococcus spp., Haemphilus spp., Neisseria spp., Prevotella spp., Veillonella spp. y Rothia spp. fueron los más abundantes. Con relación a lo anterior, en conjunto, tanto el nivel educativo, la migración y la adaptación a otra cultura son factores contribuyentes asociados con el cambio y la transición microbiana en la cavidad oral y particularmente se ha reportado que la migración y tiempo de residencia a una localidad más urbanizada se asocia negativamente con la riqueza microbiana oral. Con base en lo anterior, en un grupo de mujeres migrantes mexicoamericanas la presencia de Stresptococcus spp. se ha asociado positivamente con obesidad y disminución de la diversidad bacteriana oral, mientras que la presencia de Prevotella spp. se propuso como un indicador de un mayor tiempo de residencia en su país de origen. De manera interesante, se reportó que las características demográficas, como el nivel educativo, no influenciaba cambios en las diversidades bacterianas en este grupo de mujeres. La diferencia demográfica reportada anteriormente, entre Streptococcus spp. y Prevotella spp., coincide con la hipótesis de una firma microbiana oral entre las mujeres más aculturadas y colectivamente sustentan el potencial de cambio de la microbiota oral en respuesta a la adaptación cultural asociada con la migración9. Adicionalmente, la composición de un microbioma oral saludable también podría estar dirigida por un componente específico del sexo10. Con base en lo anterior, en un grupo nigeriano se observaron taxones exclusivos de acuerdo con el sexo, donde Anaerococcus spp., Anoxybacillus spp., Arthrobacter spp., Brevibacterium spp., Bulleida spp., Actinomyces oris, Actinomyces sp., Arthrobacter albus, Bacteroides coprocola fueron géneros y especies bacterianas exclusivas en mujeres, mientras que Altererythrobacter spp., Blautia spp., Citrobacter spp., Desulfobulbus spp., Kluyvera spp., Actinobacillus delphinicola, Actinomyces meyeri, Blautia faecis fueron exclusivas en hombres11. Estos hallazgos apoyan la hipótesis de la presencia de un microgenderoma oral que define la comunicación entre la microbiota, las hormonas sexuales y el sistema inmunitario a través de la comunicación bidireccional con el sistema endocrino.
Saliva y placa dentobacteriana entre diferentes poblaciones
La lengua, los dientes, la mucosa oral y el paladar albergan una microbiota distintiva para cada nicho. Tal es el caso de Pasteurellaceae spp. y Streptococcus spp. observadas en todas las áreas de la cavidad oral, así como Fusobacterium spp. y/o Prevotella spp. en amígdalas y garganta y Veillonella spp. observada en el paladar duro, amígdalas, garganta y parte posterior de la lengua. Sin embargo, se reconocen cambios basados en la geografía y la edad. Por ejemplo, el microbioma salival en daneses muestra estabilidad a largo plazo y se observan géneros como Streptococcus spp., Haemophilus spp., Prevotella spp., Rothia spp. y Neisseria spp. así como las especies periodontopatógenas Porphyromonas gingivalis, Treponema denticola, Prevotella intermedia, Filifactor alocis, Tannerella forsythia y Parvimona micra. De manera contrastante, en una población española, el microbioma salival reporta la presencia de los géneros Prevotella spp. y Veillonella spp., mientras que en la placa dentobacteriana se han observado marcadas diferencias en la diversidad bacteriana de acuerdo con el sitio anatómico12. En contraste, en una población japonesa con un estado de salud periodontal, se pueden observar cambios de inter e intra individuales, donde se ha observado que las phyla Actinobacterias, Bacteroidetes, Spirochaetes, Firmicutes, Proteobacterias, SR1 y Synergistes varían significativamente a nivel interindividual, pero éstas se mantienen estables a nivel interindividual, con excepción de SR1 y Synergistes spp. Lo anterior muestra que el microbioma oral es significativamente distante entre individuos, incluso entre una misma población, lo que podría apoyar la propuesta del uso del microbioma oral como huella digital13.
El microbioma de caries entre poblaciones adultas
La caries es una enfermedad que en etapas avanzadas puede desencadenar condiciones clínicas muy graves, especialmente infecciones que requieren tratamientos dentales complicados e invasivos. Adicionalmente, no hay que olvidar que las interacciones inter e intermicrobianas son relevantes para el desarrollo de este padecimiento. Tal es el caso de Streptococcus spp. que tiende a formar relaciones estrechas con Rothia spp., Actinomyces spp. y Selenomonas spp. lo que es un factor predictivo de la caries de la dentina y que también está altamente conectada a Tannerella spp. y P. olorum14. Considerando lo anterior, en una población española se reportó que el género Actinomyces spp. fue significativamente abundante en estado de salud oral, mientras que Porphyromonas spp., Fusobacterium spp., Capnocytophaga spp., Tannerella spp. y Leptotrichia spp. eran abundantes en caries15. Adicionalmente, se observó que conforme la caries avanza a través de los tejidos dentales, la diversidad del microbioma oral disminuye y se modifica drásticamente16. Otros hallazgos en daneses reportaron que S. salivarius se observó a menudo en presencia de caries17 (figura 1).
Microbioma en caries entre poblaciones infantiles y adolescentes
El microbioma oral es un factor crucial en la etiopatogenia de la caries, particularmente en edades tempranas donde la evolución de este padecimiento es muy agresiva. En infantes británicos que desarrollan caries o que ya tienen la enfermedad, se ha observado a Streptococcus spp. y S. mutans como el género y especie más abundantes. También se ha observado un aumento de Gemella spp., Porphyromonas spp. y Granulicatella spp.18. Por el contrario, en niños españoles con caries, se ha reportado a Streptococcus spp. y Neisseria spp., pero Prevotella spp. y Veillonella spp. como los géneros predominantes19. Así mismo, en infantes estadounidenses con caries, se ha reportado que la diversidad del microbioma oral disminuye en presencia de caries, al igual que Streptococcus spp., Neisseria spp. y Streptococcus sanguinis como los géneros y especie más abundantes y a medida que la enfermedad progresa se acompaña de una menor abundancia de Streptococcus mitis, Neisseria spp. y S. sanguis y. Así mismo, se ha reportado que la diversidad del microbioma oral mantiene una estabilidad temporal en diferentes grados de caries20. De manera contrastante, en infantes asiáticos se ha vinculado a los géneros Streptococcus spp., Porphyromonas spp. y Actinomyces spp. con caries severa temprana (S-ECC)21. Adicionalmente, también se ha referido que Prevotella spp., Lactobacillus spp. spp., Dialister spp. y Filifactor spp. podrían estar relacionados con la patogénesis y la progresión de la enfermedad22. En contraste a lo anterior, se ha reportado que en infantes canadienses Rothia aeria y Corynebacterium matruchotii se asocian significativamente con la ausencia de caries, mientras que en presencia de S-ECC existen diferencias relevantes caracterizadas por la disminución de Firmicutes, Actinobacterias, Fusobacterium, Streptococcus gordonii y Streptococcus sanguinis y un aumento de TM7, S. mutans, Veillonella spp. y Porphyromonas spp.23 (figura 2). Por otra parte, en etapas de edad como la adolescencia se ha observado que en rumanos con alta prevalencia de caries se presenta una mayor abundancia de Firmicutes, Bacteroidetes y Fusobacterias, Bacteroides spp., Porphyromonas spp., Abiotrophia spp., Filifactor spp., Peptostreptococcaceae spp., Pseudoramibacter spp., Streptococcus spp. y Neisseria spp., así como una menor proporción de Actinobacterias, Proteobacterias, Spirochaetes, TM7, Tenericutes, SR1, Actinomyces spp., Selenomonas spp., Veillonella spp., Campylobacter spp. y TM7 [G-1]. En contraste, adolescentes suecos con baja prevalencia de caries, se han reportado las especies S. mutans, S. sobrinus, S. australis, Alloprevotella spp., Leptotrichia spp., Neisseria spp., Porphyromonas spp., Prevotella spp., Actinomyces spp., Selenomonas spp., Mycoplasma spp. y Capnocytophaga spp. Sin embargo, tanto en rumanos como suecos, se ha reportado que la presencia de Streptococcus mitis y streptococcus sp. HOT070 es relevante cuando la caries está ausente. Además, se ha reportado que ambas poblaciones comparten un microbioma oral representado por L. shahii, P. catoniae y Streptococcus sp. HOT07424.
El microbioma de enfermedad periodontal
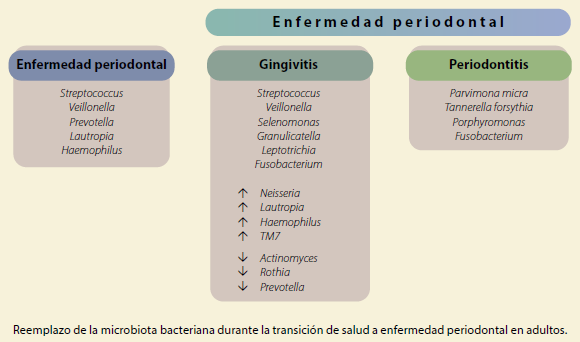
El microbioma oral, en presencia de periodontitis crónica, difiere significativamente según la ubicación geográfica. Se ha observado gran abundancia de Porphyromonas gingivalis en chilenos, brasileños, suecos y estadounidenses, mientras que Treponema denticola varía entre ellos. Por el contrario, se ha reportado que Tannerella forsythensis, no muestra diferencia entre estas poblaciones. Las especies dominantes en chilenos fueron Prevotella melaninogenica y Neisseria mucosa; En brasileños Actinomyces naeslundii y Prevotella intermedia; en suecos, A. Naeslundii, Capnocytophaga gingivalis y Peptostreptococcus micros; en estadounidenses, A. Naeslundii, P. intermedia y C. gingivalis25. De igual manera, en daneses, se asoció la gingivitis con una disminución de Bacteroidetes y Actinobacterias y un aumento de Fusobacterias spp. y TM7. En gingivitis, también se ha observado una mayor abundancia de Streptococcus spp., Neisseria spp., Leptotrichia spp., Actinomyces spp., Prevotella spp., Veillonella spp., Rothia spp., Fusobacterium spp., Lautropia spp., Selenomonas spp., Haemophilus spp., granulicatella spp. Así como Parvimona micra y Tannerella forsythia se asociaron a periodontontitis; mientras que, en salud periodontal se ha asociado una mayor abundancia de Streptococcus spp., Veillonella spp., Prevotella spp., Lautropia spp. y Haemophilus spp.26. Adicionalmente, en presencia de diferentes grados de periodontitis (localizada, agresiva generalizada, crónica generalizada y periimplantitis) se han observado altos niveles de Fusobacterium nucleatum, así como Aggregatibacter actinomycetemcomites en todos los casos de periodontitis agresiva localizada8. En taiwaneses se han observado diferencias bacterianas entre las comunidades asociadas con la salud y la enfermedad periodontal donde Porphyromonas spp. Treponema spp., Tannerella spp., Filifactor spp. y Aggregatibacter spp. eran más abundantes en la enfermedad periodontal; mientras que, en estados de salud periodontal se han observado niveles más altos de Streptococcus spp., Haemophilus spp., Capnocytophaga spp., Gemella spp., Campylobacter spp. y Granulicatella spp.27. Adicionalmente, en una población china se han reportado contrastes significativos en el microbioma de la placa dentobacteriana, pero no en la saliva, tanto de sujetos sanos con de sujetos con gingivitis con diferentes grados de enfermedad periodontal. Por lo tanto, es sugerido que es posible distinguir entre el microbioma oral de la placa dentobacteriana que está asociada a salud gingival de la que está asociada con enfermedad periodontal incluso a diferentes grados de gingivitis y periodontitis28 (figura 3).
El microbioma oral asociado con otras enfermedades
La presencia de las bacterias orales implicadas en el desarrollo de la caries y la enfermedad periodontal se ha asociado con enfermedades sistémicas. A lo largo de toda la vida, un individuo puede estar constantemente expuesto a estas bacterias por medio de bacteriemia, contigüidad e incluso broncoaspiración, que pueden conducir a su translocación sistémica. Es posible que esto ocurra debido a la eliminación del cálculo por procedimientos cotidianos de higiene dental (ya sea en pacientes con enfermedad periodontal o con un periodonto aparentemente saludable), durante procesos de remoción de la caries o por tratamientos del conducto radicular, incluso como consecuencia de complicaciones agudas o crónicas de trastornos endodónticos o periodontales (p. ej., inflamación y necrosis de la pulpa dental, abscesos y angina de Ludwig).
Dado que los géneros Streptococcus, Veillonella, Porphyromonas, Actinomyces, Fusobacterium, Capnocytophaga, Tannerella y Leptotrichia han estado estrechamente relacionados con diferentes trastornos, han llegado a ser contemplados como posibles blancos terapéuticos, así como indicadores de riesgo para la detección temprana de enfermedades orales o sistémicas. Por el contrario, a los géneros y especies de Neisseria spp. y Streptococcus spp. que están asociados con la salud y, por lo tanto, pueden servir como marcadores pronóstico de protección de enfermedad e incluso como parte de terapias alternativas para revertir la disbiosis y restaurar la salud. El microbioma oral no sólo desempeña un papel importante en la enfermedad oral, sino que también los cambios en la microbiota bacteriana salival y la placa dental pueden estar asociados con enfermedades orales de mucosas, que son de interés público, como leucoplasia oral (LO), liquen plano oral (LPO), lupus eritematoso sistémico (LES), cáncer oral (COCE) y complicaciones durante ciertos tratamientos como periimplantis29-32.
Algunas bacterias que se encuentran en la cavidad oral también están relacionadas con la patogénesis de trastornos sistémicos como la artritis reumatoide, infecciones recurrentes en las vías respiratorias, enfermedades pulmonares y cardiovasculares, alteraciones hepáticas y metabólicas como resistencia a la insulina, diabetes mellitus tipo 2 y obesidad32-39, enfermedades inflamatorias intestinales40-42, embarazo prematuro43, diferentes tipos de cáncer44, así como alteraciones nerviosas y cognitivas45-48 (tablas 1a y 1b).
Tabla 1a Microbiota bacteriana oral asociada a algunas alteraciones sistémicas32
| Alteración | Beneficio | Riesgo |
| Inflamación intestinal crónica (ICI)40-42 | Streptococcus, Prevotella, Neisseria, Haemophilus, Veillonella y Gemella, Klebsiella | |
| Cáncer pancreático44 | Leptotrichia | Porphyromonas gingivalis y Aggregatibacter actinomycetemcomitans |
| Síndrome de Alzheimer45,47-48 | Porphyromonas gingivalis, A. actinomycetemcomitans y Tannerella forsythia | |
| Diabetes mellitus tipo II39 | TM7, Aggregatibacter, Neisseria, Gemella, Eikenella, Selenomonas, Actinomyces, Capnocytophaga, Fusobacterium, Veillonella y Streptococcus | |
| Resultados adversos en el embazo43 | Bacteroides forsythus, Campylobacter rectus y F. nucleatum | |
| Artritis reumatoide33-36 | Lactobacillus salivarius | Haemophilus, P. gingivalis y A. actinomycetencomitans |
| Ateroesclerosis37,38,46 | Chryseomonas, Veillonella, Streptococcus y P. gingivalis |
Tabla 1b Microbiota bacteriana oral asociada a algunas alteraciones bucodentales
| Alteración | Bacterias asociadas |
| Periimplantitis | ↑ Bacteroidetes, Fusobacterium nucleatum, Porphyromonas gingivalis, Tannerella forsythia, así como Eubacterium minutum en correlación con Prevotella intermedia |
| Leucoplasia oral (LO) |
↑ Haemophilus y Fusobacteria ↓ Firmicutes Displasia severa: ↑ Leptotrichia spp. y Campylobacter concisus |
| Liquen plano oral (LPO) |
↑ Porphyromonas, Solobacterium, Fretibacterium, Prevotella nigrescens y Selenomonas ↓ Haemophilus, Corynebacterium, Cellulosimicrobium y Campylobacter LPO erosive: ↓ Streptococcus spp. |
| Carcinoma oral de células escamosas (COCE) |
↓ Riqueza y diversidad a nivel de especie ↑ Bacteriodetes y fusobacterias ↑ Capnocytophaga, Pseudomonas, Atopobium, Streptococcus, Rothia, Gemella, Lactobacillus, Micromonas, Porphyromonas, Fusobacterium, Campylobacter concisus, Prevotella salivae, Prevotella loeschii y Fusobacterium oral taxon 204 Abundancia significativamente diferente entre lesiones epiteliales precursoras y COCE: Bacillus, Enterococcus, Parvimonas, Peptostreptococcus y Slackia |
Conclusiones
El microbioma oral posee patrones característicos de la salud y la enfermedad, éste difiere entre individuos de distintas poblaciones, pero en individuos sanos de una misma población se comparte una composición bacteriana oral específica, siempre y cuando existan factores extrínsecos similares, por lo que determinados taxones bacterianos orales podrían sugerir un factor predictivo de salud o enfermedad bucodental en una población. Sin embargo, será válido para las poblaciones estudiadas o en condiciones generales de enfermedad. Aun así, debe tenerse en cuenta que la composición de las comunidades microbianas orales puede variar de manera interindividual, y dependiendo del nicho bucodental. Adicionalmente, se ha establecido que el perfil de la microbiota salival sea comparable con otros nichos, como la lengua y la mucosa, pero completamente distinta a la composición de la placa dentobacteriana aun entre individuos de una misma población. Lo anterior es relevante por el valor representativo del uso de las muestras de saliva para estudios etiológicos y epidemiológicos de enfermedades bucales ya que muestras salivales suelen no ser representativas de los perfiles microbianos en sitios donde se desarrollan enfermedades bucodentales infecciosas locales, por lo que para una mejor comprensión del proceso de la caries y la enfermedad periodontal, sería relevante que para el estudio de estos padecimientos, las muestras a considerar sean sitioespecíficas o preferente de la placa supragingival, mientras que para el estudio de padecimientos sistémicos el uso de saliva sería una muestra más representativa.











 nueva página del texto (beta)
nueva página del texto (beta)