Introducción
El ion Ca2+ es un segundo mensajero que transmite señales para la regulación de procesos celulares como proliferación, muerte celular, transcripción y metabolismo de células malignas.1-3 Las ATPasas de Ca2+ del retículo sarcoplasmático (SERCA) son de las proteínas más importantes en el mantenimiento de la homeostasis y señalización por Ca2+, ya que bombean Ca2+ citosólico hacia el lumen del retículo endoplásmico para restaurar el estado de reposo. Los genes ATP2A1, 2, y 3 codifican para diferentes isoformas de las enzimas SERCA1, 2 y 3, respectivamente.4 La expresión de estas bombas de Ca2+ es regulada por el tipo de tejido y estadio del desarrollo.5 Las alteraciones en la señalización por Ca2+ están involucradas en el desarrollo de patologías cardiacas, neurológicas y cáncer.6
El cáncer de mama (CaMa) es la neoplasia más frecuente en mujeres, tanto en México como a nivel mundial,7,8 y pese a las múltiples estrategias de tamizaje, su incidencia continúa en aumento.9 La primera evidencia de la alteración de la expresión de las bombas de Ca2+ SERCA en CaMa fue dada por el grupo de Brouland, que analizó la expresión de SERCA3 mediante inmunohistoquímica en muestras de pacientes y observó que su expresión correlacionaba inversamente con el grado histológico del tumor y que era abundante en tejido normal.10 Estos hallazgos llevaron a proponer a SERCA3 como marcador de diferenciación, ya que su expresión disminuye incluso en lesiones precancerosas.10
Sin embargo, el papel de las SERCA aún no está claro, ya que la expresión de SERCA3 se ha visto disminuida en ciertos linajes de cáncer, como el gástrico, de colon, mama, hígado y pulmón,10-14 pero también se ha observado que la sobreexpresión de SERCA2 promueve la proliferación en cáncer de colon y próstata.15,16
En este trabajo cuantificamos y comparamos la expresión de los genes ATP2A2 y ATP2A3 en las líneas celulares de CaMa MCF-7 y MDA-MB-231, que representan los subtipos luminal A y triple negativo, respectivamente, y reflejan distintos grados de diferenciación; MCF-7 es representativo de cáncer primario y MDA-MB-231, de cáncer metastásico. Adicionalmente, evaluamos el efecto de resveratrol (RSV), fitoestrógeno que modula la expresión de SERCA3 en la proliferación y muerte celular, procesos celulares cruciales del cáncer.2
Métodos
Cultivos celulares y tratamientos farmacológicos
Las líneas celulares del CaMa MCF-7 y MDA-MB-231 fueron adquiridas en ATCC® y su identidad la verificó el Laboratorio de Diagnóstico Genómico del Instituto Nacional de Medicina Genómica. Las células MCF-7 y MDA-MB-231 se cultivaron en medio Dulbecco's Modified Eagle Medium con glucosa, 10 % de suero fetal bovino, 0.5 % de penicilina/estreptomicina y 70 mg/L de kanamicina. Se incubaron para cultivo a 37 °C, con 95 % de aire y 5 % de CO2. Las células no malignas MCF-10A fueron cultivadas con medio DMEM/F12, con 10 % de suero fetal bovino, insulina, hidrocortisona y factor de crecimiento epidérmico humano. Las células en fase de crecimiento exponencial se tripsinizaron y sembraron en diferentes placas de cultivo. Al alcanzar 60 % de confluencia, se agregaron concentraciones crecientes de RSV (0-200 μM) durante 72 horas y el medio de cultivo se reemplazó cada 24 horas.
Ensayos de proliferación celular
A las células listas para realizar el experimento se les retiró el medio de cultivo y se lavaron con PBS, se lisaron con amortiguador de lisis adicionado con el colorante CyQUANT en una relación 1:80 y se procedió a la medición de la fluorescencia (480 nm de excitación y 520 nm de emisión), directamente proporcional a la cantidad de ADN.
Ensayos de apoptosis
La muerte celular se determinó mediante un ensayo basado en la hidrólisis del péptido sustrato Ac-DEVD-pNA por diversas caspasas y en el que se libera la pNA. Los resultados se presentan como el índice de activación de caspasas comparado con el del control; las células control tratadas únicamente con el vehículo (etanol) fueron consideradas con un índice de activación de 1.
Extracción de ARN y PCR en tiempo real
La concentración de ARN se determinó mediante espectrofotometría y su integridad se evaluó mediante electroforesis en gel de agarosa con formaldehído a 1 %. La reacción de transcripción reversa se realizó con 1 mg de ARN total con la transcriptasa reversa M-MLV. Las reacciones de PCR en tiempo real (qPCR) se realizaron por triplicado con 25 ng de ADN complementario como molde, el cual fue obtenido en el paso anterior. La expresión de b-actina se utilizó para normalizar los resultados.
Análisis de datos públicos de secuenciación de ARN de una sola célula
Para analizar la expresión de los genes ATP2A2 y ATP2A3 en diferentes subtipos de CaMa, se utilizó la base datos con número de acceso GSE75688, la cual proviene de un estudio donde se analizó el perfil de la expresión génica de 515 células derivadas de 11 pacientes con CaMa.17 Los resultados de la expresión de ATP2A2 y ATP2A3 fueron analizados y visualizados en el Single Cell Expression Atlas y para fines de este estudio, las 515 células de CaMa se dividieron en cuatro subtipos: ER+ y HER2+; HER2+; ER+; y PR–, ER– y HER2–.18
Análisis estadístico
Los datos fueron analizados en el programa GraphPad Prism. Los resultados están expresados como la media de tres experimentos independientes ± desviación estándar. Las comparaciones entre grupos experimentales se realizaron mediante el método de Dunnett (Anova de un factor). Las diferencias fueron consideradas como significativas con un valor de p < 0.05.
Resultados
Las células MCF-7 y MDA-MB-231 expresan niveles distintos de los genes ATP2A2 y ATP2A3
Mediante PCR cuantitativo en tiempo real, evaluamos la abundancia de los genes ATP2A2 y ATP2A3 en las líneas celulares CaMa MCF-7, MDA-MB-231 y MCF-10A, esta última utilizada como modelo de epitelio mamario normal. En la Tabla 1 se observa que el valor del ciclo umbral para la amplificación del ARNm para ambos genes es similar entre las tres líneas celulares para el gen ATP2A2, por lo que se concluye que se expresa a niveles similares entre estos linajes. Sin embargo, el ciclo umbral del gen ATP2A3 es menor (Ct1 6.43) en las células MCF-7 que en las células MDA-MB-231 (Ct 22.89), lo cual indica una menor expresión de este gen en las células MDA-MB-231 (Tabla 1). Sorprendentemente, la línea celular MCF-10A mostró un ciclo umbral para ATP2A3 menor que las células MDA-MB-231 (Ct 27.25). La tabla también muestra que la expresión relativa de ATP2A2 con respecto a ß-actina fue similar en las tres líneas celulares. Sin embargo, la expresión relativa de ATP2A3 fue diferente en las tres líneas celulares, siendo más baja en la línea MCF-10A, seguida de la línea triple negativa MDA-MB-231 y la más alta en la línea celular MCF-7.
Tabla 1 Expresión de los genes ATP2A2 y ATP2A3 en las líneas celulares MCF-10A, MCF-7 y MDA-MB-231
| Línea celular | Ciclo umbral ATP2A2 | Expresión ATP2A2 normalizada | Ciclo umbral ATP2A3 | Expresión ATP2A3 normalizada | Ciclo umbral b-actina |
|---|---|---|---|---|---|
| MCF-10A | 14.79 ± 0.23 | 0.68 ± 0.11 | 27.25 ± 0.44 | 0.0013 ± 0.0001 | 8.26 ± 0.05 |
| MCF-7 | 14.64 ± 0.64 | 1.15 ± 0.21 | 16.43 ± 0.78 | 1.16 ± 0.23 | 9.03 ± 0.36 |
| MDA-MB-231 | 13.46 ± 0.14 | 1.06 ± 0.0005 | 22.89 ± 0.12 | 0.0015 ± 0.0003 | 7.68 ± 0.11 |
Se muestran los valores del ciclo umbral obtenidos por qRT-PCR para cada gen. También se muestra cuantitativamente, en las columnas resaltadas en verde, la comparación de la expresión de ATP2A2 y ATP2A3 entre las tres líneas celulares normalizada con la expresión de β-actina y tomando como base la abundancia en la expresión de estos genes en las células MCF-7. Los datos son expresados como el promedio de tres experimentos independientes (n = 3) ± desviación estándar.
RSV afecta la morfología y el número de células en las líneas MCF-7 y MDA-MB-231
Con la finalidad de analizar el efecto del RSV sobre las distintas líneas celulares, se adicionaron diferentes concentraciones (10, 50, 100 y 200 μM) y se realizaron cultivos con 12, 24, 48 y 72 horas de tratamiento. En las Figuras 1A y 1B se puede observar que el etanol (vehículo) no tiene efecto alguno sobre las células MCF-7 y MDA-MB-231, respectivamente. Las células MCF-7 muestran disminución significativa en su número dependiendo de la dosis-tiempo de exposición, especialmente a altas concentraciones (> 50 μM RSV) a las 72 horas (Figura 1A). Las células MDA-MB-231 mostraron ser más resistentes que las células MCF-7 (Figura 1B). En el caso de la línea MCF-10A, se acentúa más con concentraciones altas de RSV (200 μM), incluso a tiempos cortos (24 horas), y muestra signos de senescencia o estrés19 (Figura 1C). Concentraciones bajas de RSV (entre 10 y 50 μM) parecen no impedir significativamente la proliferación, ya que se comportan en forma similar al control.
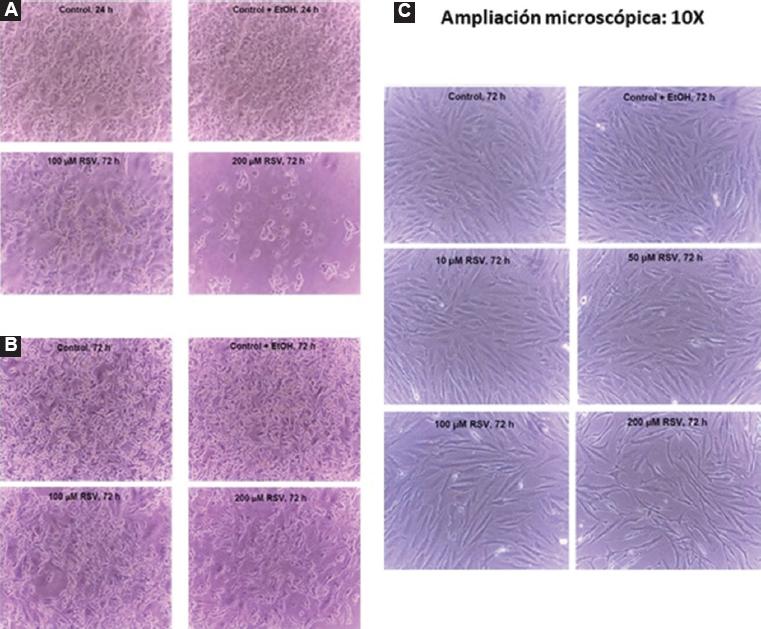
Figura 1 Efecto del RSV sobre las células MCF-7 (panel A), MDA-MB-231 (panel B) y MCF-10 (panel C). Al alcanzar 60 a 70 % de confluencia, las células se expusieron a concentraciones crecientes de RSV (10, 50, 100 y 200 μM) por 12, 24, 48 y 72 horas de tratamiento, con cambio de medio cada 24 horas. Las microfotografías fueron tomadas con una ampliación de 10×.
RSV disminuye la proliferación celular e induce apoptosis en las células malignas MCF-7 y MDA-MB-231, sin afectaciones en las células no malignas MCF-10A
Para cuantificar la proliferación celular utilizamos un colorante (CyQUANT), que al unirse al ADN incrementa su fluorescencia, permitiendo con ello cuantificar el ADN, medida directa del número de células y de su proliferación. En la Figura 2A se puede observar que el RSV disminuye la proliferación de las células MCF-7 y MDA-MB-231 después de 72 horas de forma dosis-dependiente. Las células MDA-MB-231 son más resistentes a bajas dosis, pero al incrementar la concentración de RSV se comportan en forma similar a las células MCF-7. Se puede observar también que las células MCF-10A no muestran disminución, pero su tasa de proliferación es lenta. El etanol no tuvo efecto alguno en la proliferación de los tres linajes.
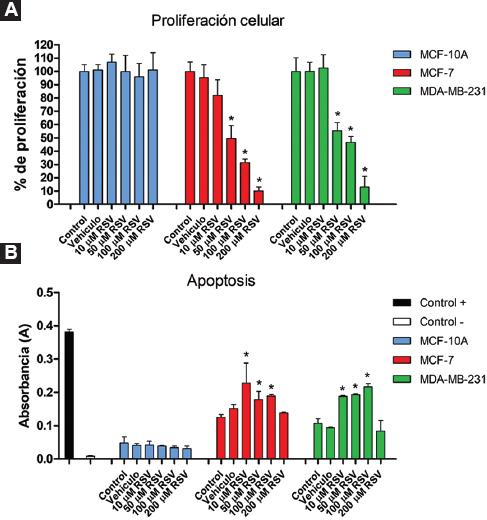
Figura 2 Disminución de la proliferación y aumento de apoptosis en células MCF-7 y MDA-MB-231 tratadas con RSV. Las células fueron tratadas por 72 horas con concentraciones crecientes de RSV (0 a 200 μM), posteriormente fueron lisadas y se procedió a la cuantificación de ADN total para la determinación de la proliferación celular (panel A) y a la utilización de extractos totales para ser incubados con un sustrato hidrolizable por varias caspasas como indicativo de apoptosis (panel B). Los datos son expresados como el promedio de tres experimentos independientes (n = 3) ± desviación estándar. Cada línea celular fue comparada contra su respectivo control: células en condiciones normales de cultivo. En el panel B se graficaron directamente los valores de absorbancia (A).
También se determinó la muerte celular por los cambios en la absorbancia del producto pNA. Se observó que, mientras que en las células no malignas MCF-10A no hay cambios significativos en la absorbancia de pNA, en las células malignas MCF-7 y MDA-MB-231 se detectan incrementos en la absorbancia desde las concentraciones más bajas, lo que indica que el RSV afecta selectivamente a las células malignas (Figura 2B).
La expresión de ATP2A2 y ATP2A3 en los subtipos de cáncer de mama
En la Figura 3A se muestra la distribución de las células acorde a los cuatro subtipos: ER+ HER2+ (amarillo), ER+ (azul claro), HER2+ (azul) y PR−, ER− y HER2− (rojo). Los datos de secuenciación de ARN de una sola célula indican que la expresión de ATP2A2 (azul) es homogénea en los cuatro subtipos de CaMa (Figura 3B), mientras que la expresión de ATP2A3 es heterogénea (azul), lo que muestra los mayores niveles de expresión en las células ubicadas en el subtipo ER+ HER2+, seguido del subtipo PR−, ER− y HER2− y, finalmente, los menores niveles se encontraron en los fenotipos ER+ y HER2+ (Figura 3C).
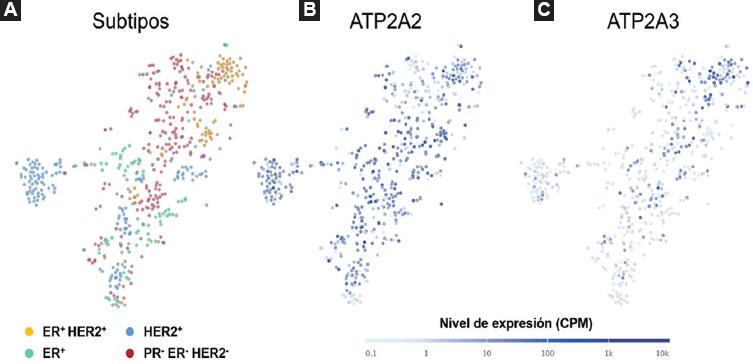
Figura 3 Expresión de ATP2A2 y ATP2A3 en cuatro subtipos de cáncer de mama analizados por secuenciación de ARN de una sola célula. En el panel A se muestra la distribución de las células acorde a los cuatro subtipos: ER+ HER2+ (amarillo), ER+ (verde), HER2+ (azul) y PR-, ER-, HER2- (rojo). Los paneles B y C muestran los resultados de los niveles de expresión (gradiente de azul) de ATP2A2 y ATP2A3, respectivamente. La expresión de ATP2A2 y ATP2A3 se analizó y visualizó en el Single Cell Expression Atlas (EMBL-EBI).
Discusión
La expresión o función de SERCA se encuentra alterada en patologías como cáncer,6 por lo que estudiar su rol en la carcinogénesis y mantenimiento del fenotipo tumoral es importante para entender su etiología y el posible desarrollo de nuevas estrategias terapéuticas y pronósticas. Papp y Brouland reportaron que lesiones precancerosas y cancerosas derivadas de pacientes con CaMa muestran niveles de expresión casi inexistentes de SERCA3 comparadascon tejido no neoplásico,10 lo que sugiere que la pérdida en la expresión de SERCA3 es un evento temprano dentro del proceso carcinogénico. Estos y otros datos obtenidos en cáncer gástrico, de colon y pulmón11,12,20 han llevado a proponer a SERCA3 como un marcador de diferenciación, puesto que su expresión correlaciona proporcionalmente en forma inversa con el grado histológico del tumor.
En este trabajo utilizamos dos líneas celulares que representan distintos subtipos de CaMa, para cuantificar la expresión de ARNm de los genes ATP2A2 y ATP2A3. Por un lado, la línea MCF-7 es representativa de CaMa tipo luminal, el cual conserva algunas características epiteliales, mientras las células MDA-MB-231 representan un tipo triple negativo, muy agresivo y menos diferenciado.21-23
La señalización proliferativa sostenida y la resistencia a la apoptosis son características distintivas del cáncer,2 por lo cual se evaluó el efecto del RSV (que modula selectivamente la expresión de ATP2A3 sin alterar la expresión de ATP2A2)24 en estos dos procesos celulares. Se observó cómo existe una disminución selectiva en el número de células en ambas líneas celulares malignas en forma dosis-tiempo dependiente (Figura 1). La Figura 2 muestra cuantitativamente que el RSV detiene la proliferación celular e induce apoptosis.
En el laboratorio hemos identificado mecanismos transcripcionales y epigenéticos que controlan la expresión de SERCA3 en cáncer. Por ejemplo, en cáncer gástrico y de colon, los factores de transcripción Sp1, Sp3 y KL4 son importantes para la transcripción basal del gen ATP2A3;12 además, su expresión también se controla a nivel epigenético, puesto que la metilación del promotor disminuye conforme las células se diferencian por confluencia, lo que promueve cambios en la estructura de la cromatina a través de marcas de histonas.20 Además, en líneas celulares de CaMa observamos que el RSV induce la expresión de ATP2A3 a través de la remodelación de la estructura de cromatina25 y que el silenciamiento de ATP2A3 mediante ARNi previene parcialmente la muerte celular inducida por RSV,24 por lo que SERCA3 podría ser un buen blanco para tratar tumores proliferativos con gran resistencia a la apoptosis.
Los resultados obtenidos en nuestro laboratorio concuerdan con la evidencia disponible, ya que las células MDA-MB-231 mostraron una expresión de SERCA3 menor que las células MCF-7 (Tabla 1). Interesantemente, al observar la línea celular MCF-10A, la cuantificación de ARNm mostró que expresaba menos SERCA3 que las células MDA-MB-231. Sin embargo, las células MCF-10A son de tipo basal y se caracterizan por la ausencia en la expresión de los receptores ERα−, PR− y HER2−.23 Lo anterior cuestiona la verdadera utilidad de las células MCF-10A como modelo de tejido epitelial mamario normal.26,27 Interesantemente, encontramos que la expresión de ATP2A3 varía en los diferentes subtipos de CaMa (Figura 3C) y que sus mayores niveles de expresión se presentan en el subtipo ER+ HER2+, relacionado con las células MCF-7, seguido por el subtipo triple negativo (PR−, ER− y HER2−), relacionado con las células MDA-MB-231 y que los niveles más bajos se encontraron en el subtipo HER2+, el cual, junto con el triple negativo representan los subtipos de CaMa con peor pronóstico.
Todo esto es importante porque si bien se han mejorado las estrategias de tamizaje para detectar CaMa en etapas más tempranas, aún muchos casos son detectados en etapas avanzadas en las cuales la terapéutica es limitada y menos eficiente, desalentando el pronóstico de las pacientes.











 nueva página del texto (beta)
nueva página del texto (beta)


