La aparición del nuevo virus
A fines de 2019 emergió un nuevo virus en Wuhan, China. Su reporte a la Organización Mundial de la Salud se reflejó en una alerta sanitaria para poner sobre aviso a la comunidad internacional.1 De acuerdo con los estudios genéticos, este nuevo virus pertenece a la familia Coronaviridae, en la que se encuentran los virus del síndrome respiratorio agudo grave o severo (SARS-CoV) y del síndrome respiratorio del Medio Oriente (MERS-CoV). Estos dos virus son causantes de infección respiratoria grave en los humanos y han causado epidemias que han afectado a distintos países.2,3 El nuevo virus fue conocido inicialmente como nuevo coronavirus 2019, novel coronavirus 2019 (2019-nCoV). El Comité Internacional de Taxonomía de Virus denominó a este nuevo virus como coronavirus 2 del síndrome respiratorio agudo grave o SARS-CoV-2.4 La infección por este virus se denominó oficialmente como COVID-19 (coronavirus disease 2019).5
Características de SARS-CoV-2
Los coronavirus son agentes infecciosos frecuentes en animales y humanos. En humanos, los coronavirus pueden causar desde resfriados comunes (HCoV-NL63, HCoV-229E, Beta-CoV1 y HCoV-HKU1), hasta las enfermedades graves mencionadas (SARS y MERS).6 Los coronavirus tienen un genoma de ARN monocatenario no segmentado de sentido positivo de aproximadamente 30 mil nucleótidos. Por microscopia electrónica se observa que su forma general es redondeada con espículas dispuestas en una estructura superficial que recuerda una corona solar (Figura 1), de la cual deriva su nombre.7
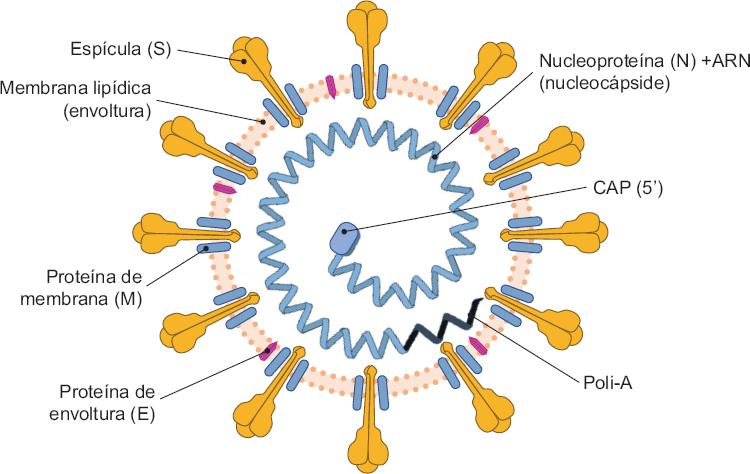
Figura 1 Diagrama esquemático del virión de SARS-CoV-2. Creado con información de Fehr et al.7 y Cui et al.27
El genoma viral contiene una estructura cap en el extremo 5 y una cola poli-A en el extremo 3, por lo que funciona como ARN mensajero. Este ARN es traducido para generar proteínas no estructurales (nsp) que tienen funciones en la replicación y maduración de las proteínas. No obstante, el ARN genómico da origen a otros ARN mensajeros, denominados subgenómicos, que generarán otras proteínas virales. Esta serie de mensajeros tienen en común el extremo 3, por lo que se dice que están anidados, de donde proviene el nombre del orden al que pertenecen los coronavirus: Nidovirales.8
El genoma viral contiene al menos 13 marcos de lectura abiertos, sin embargo, el procesamiento de algunos de los polipéptidos puede originar múltiples proteínas maduras. El virus tiene cuatro proteínas estructurales: la espícula (S, spike), la de envoltura (E), la de membrana (M) y la nucleoproteína (N), mientras que las proteínas no estructurales son al menos 16 y son conocidas como nsp1 a nsp16.9
Las proteínas estructurales forman la partícula viral; la proteína S es la más antigénica y externa, además de que es la responsable, en gran parte, de la forma de corona al virión. En el caso de SARS-CoV-2, es la proteína que reconoce la proteína celular ECA2 (enzima convertidora de angiotensina 2) como receptor para fijarse a la célula hospedera. La proteína N proporciona protección al genoma e interviene en la síntesis del ARN viral, mientras que la M da estructura y estabilidad al virión; la E es un canal iónico con funciones durante el ensamble y la salida de los viriones de la célula hospedera.7,10 Entre las regiones codificantes de las proteínas estructurales se encuentran otros marcos de lectura (ORF, open reading frame) que codifican una serie de proteínas denominadas accesorias y que se nombran como ORF3a, ORF6, ORF7a, ORF7b, ORF8, ORF9b y posiblemente una más denominada ORF14.9 Estas proteínas se consideran no indispensables para el ciclo replicativo de los coronavirus, sin embargo, tienen diferentes funciones al interaccionar con proteínas del hospedero en diferentes vías de señalización relacionadas con la respuesta antiviral. La eliminación de alguno de estos marcos de lectura puede llevar a la atenuación del coronavirus de la hepatitis murina; también se ha propuesto que estas proteínas contribuyen a la patogénesis de SARS-CoV, además de que representan una ventaja para la adaptación de los virus a su hospedero.11 Por otra parte, entre las 16 proteínas no estructurales se encuentran helicasa, trifosfatasa, metiltransferasa y nucleasa. Destaca especialmente la nsp5, la principal de dos proteasas que tiene el virus, conocida como Mpro (main protease) o 3CLpro (3C like protease), proteasa del tipo de la quimiotripsina que interviene en la maduración de 12 proteínas, ya que actúa sobre 11 sitios de corte entre los productos de traducción del genoma viral.7,9
Ciclo replicativo de los coronavirus
El virión de SARS-CoV-2 se une a la célula hospedera mediante la interacción de la proteína S con su receptor, la proteína ECA2, la cual es abundante en el tejido respiratorio, desde el superior, como el de la cavidad nasal y bucal, hasta el inferior, principalmente en los alveolos pulmonares.3
Se ha definido que una proteína del hospedero, la serina proteasa transmembranal 2 (TMPRSS2), también tiene un papel en la entrada del virus a las células, ya que activa la proteína S.12 El virus ingresa a la célula mediante endocitosis. Después de la interacción con el receptor, hay un cambio conformacional en la proteína S, que promueve la fusión de las membranas viral y endosómica en un proceso que requiere acidez del medio. Posterior a la liberación de la nucleocápside en el citoplasma, el ARN genómico viral se traduce directamente en el ribosoma y se produce la poliproteína pp1a. Por un desplazamiento del marco de lectura (ribosomal frameshift) al final del código de la proteína pp1a, el marco de lectura se alarga para generar la poliproteína pp1ab. Estas poliproteínas son digeridas por las proteasas virales PLpro y 3CLpro para generar 16 proteínas no estructurales con distintas funciones enzimáticas y que intervienen en la síntesis del ARN viral y en el procesamiento proteolítico, entre otras actividades. Algunas de estas proteínas forman un complejo que se encarga de la síntesis del genoma viral y de los segmentos de ARN subgenómico que darán origen a las proteínas estructurales que son sintetizadas en ribosomas asociados con el retículo endoplásmico. Posteriormente, los productos virales se ensamblan en el compartimiento intermediario del retículo endoplásmico y el aparato de Golgi, donde los genomas interaccionan con la nucleoproteína para formar la nucleocápside y con los otros componentes estructurales. Finalmente, la partícula viral se forma como una vesícula en la membrana plasmática, después de lo cual los nuevos viriones salen de la célula por exocitosis.10 Los principales pasos de este proceso se observan en la Figura 2.
Probables fármacos para el tratamiento de COVID-19
Es muy relevante la investigación de tratamientos farmacológicos para COVID-19. Hasta el momento no se ha autorizado ningún fármaco para el tratamiento específico. No obstante, diversos estudios muestran que existen moléculas con probable actividad específica contra algún constituyente del virus o que pueden disminuir el proceso inflamatorio en los pacientes. En las primeras semanas de la epidemia en China se reportó el tratamiento con varios fármacos probados con éxito para otros virus, por ejemplo, interferón alfa (citocina antiviral), ribavirina (análogo de guanosina) y la combinación de lopinavir y ritonavir (inhibidores de proteasa retroviral);13-15 sin embargo, no se han obtenido resultados concluyentes que permitan definir que son útiles para el tratamiento de COVID-19.
Algunos otros fármacos se han probado en ensayos in vitro y son capaces, en diferente medida, de inhibir la infección por SARS-CoV, entre ellos nelfinavir, glicirricina y baicaleína,16 por lo que se propone que pudieran tener actividad contra SARS-CoV-2. Otros fármacos que parecen buenas opciones son los anticuerpos que bloquean el receptor para interleucina 6 (por ejemplo, sarilumab y tocilizumab), lo cual puede atenuar la acción de citocinas proinflamatorias característica en los pacientes con COVID-19 grave. Estos anticuerpos se utilizan para el tratamiento de algunas enfermedades inflamatorias como la artritis reumatoide.17,18 Otras moléculas que han probado su actividad in vitro son el mesilato de nafamostat, un anticoagulante que inhibe la enzima TMPRSS2, importante para la entrada del virus a la célula,19 y el lipopéptido EK1C4, que al interactuar con la proteína S viral es capaz de inhibir la fusión de las membranas del virus y del hospedero, lo cual impide que el virus deposite su material genético en el citoplasma.20 También se ha propuesto favipiravir, un análogo de pirazina que ha demostrado su efecto para inhibir la polimerasa de los virus de la influenza y otros virus; sin embargo, en un artículo reciente se describió que tuvo poco efecto sobre la infección por SARS-CoV-2 en un modelo experimental de hámster.21 Algunos de estos fármacos son señalados en su sitio de acción en la Figura 2.
Hay pocos estudios clínicos con estos fármacos que muestren algún efecto de mejoría en los pacientes. En un ensayo clínico con 1063 pacientes, 538 recibieron remdesivir y 521, placebo; los del grupo de remdesivir se recuperaron en promedio cuatro días antes y aunque también se observó disminución en la mortalidad, no fue estadísticamente significativa respecto a la del grupo control.22 Por otra parte, en un ensayo clínico con tocilizumab que incluyó a 21 pacientes graves en China, los síntomas, la hipoxemia y la opacidad en las imágenes de la tomografía computarizada mejoraron con el tratamiento en la mayoría de los pacientes.23 A mediados de junio, el ensayo clínico RECOVERY comunicó que la dexametasona, un antiinflamatorio esteroideo de uso frecuente en el paciente con enfermedad respiratoria crítica, disminuyó la mortalidad en los pacientes hospitalizados por COVID-19 grave. Al comparar dos mil pacientes tratados contra cuatro mil controles, la dexametasona evitó uno de cada ocho muertes en pacientes con ventilación mecánica y una de cada 25 muertes en pacientes que requirieron oxígeno; no tuvo efecto sobre la infección moderada que no requirió oxígeno.24
Otro recurso explorado es el uso de plasma de personas convalecientes. En un estudio clínico realizado en China con 103 pacientes, 52 se trataron con plasma de individuos convalecientes y 51 constituyeron el grupo control; 52 % de los pacientes que recibieron plasma experimentaron mejoría clínica y 43 % del grupo control, si bien las diferencias no fueron significativas estadísticamente. Lo mismo ocurrió con la mortalidad, en la que se registró una tendencia a ser menor en el grupo que recibió plasma de pacientes convalecientes.25 No obstante estos avances, no hay evidencia suficiente para afirmar que existe un tratamiento farmacológico efectivo para los pacientes con COVID-19.
Origen de SARS-CoV-2
Como se ha documentado en otros coronavirus que infectan a humanos, desde el principio se propuso el origen zoonótico de SARS-CoV-2, esencialmente por su gran cercanía filogenética con un coronavirus previamente hallado en un murciélago, con cuyo genoma tiene más de 96 % de identidad.3 Previamente circulaban en humanos dos coronavirus responsables de infección respiratoria leve: 229E y OC43. El virus del SARS, causante de neumonía grave, fue descubierto en 2003. Entre 2004 y 2005 se detectaron en humanos otros dos coronavirus, denominados NL63 y HKU1; ambos ocasionan infecciones generalmente leves. Con estudios filogenéticos se ha evidenciado que NL63 y 229E se originaron en murciélagos, mientras que OC43 y HKU1 provienen de roedores.26 Para ingresar en la población humana, estos virus tuvieron un hospedero intermediario con amplio contacto con humanos, que en el caso de 229E fue la alpaca y en OC43, el ganado bovino. Respecto a los virus NL63 y HKU1, aún se desconoce el hospedero intermediario, mientras que en SARS-CoV y MERS-CoV hay evidencia de que fueron la civeta y el dromedario, respectivamente.26-28
Con base en los estudios filogenéticos se propone que SARS-CoV-2 se originó del linaje de un virus de murciélago denominado Bat-CoV RaTG13, con el que tiene 96 % de identidad. Hasta el momento no se ha definido la especie que pudo haber funcionado como intermediaria para que SARS-CoV-2 pasara de los murciélagos a los humanos. Una de las diferencias principales entre SARS-CoV-2 y Bat-CoV RaTG13 se encuentra en el dominio de unión al receptor de la proteína S; sin embargo, esta pequeña secuencia es muy similar a la encontrada en los coronavirus del pangolín malayo (Manis javanica), aunque la identidad global entre los genomas de los coronavirus del pangolín y SARS-CoV-2 es aproximadamente de 90 %. La presencia en SARS-CoV-2 de esa pequeña secuencia parecida al coronavirus del pangolín sugiere que hubo eventos de recombinación entre un virus de pangolín y uno de murciélago, y que el pangolín podría haber funcionado como intermediario para que el coronavirus de murciélago llegara al humano. No obstante, SARS-CoV-2 tiene otra secuencia en la misma proteína S, rica en aminoácidos básicos, susceptible de digestión por proteasas e importante para el ciclo replicativo que no está presente en los coronavirus de murciélago ni pangolín.29-31 Por ello, la hipótesis del pangolín como hospedero intermediario se encuentra aún en discusión. El comercio ilegal del pangolín es común en China, particularmente por el uso de sus escamas en la medicina tradicional, así como por su carne, considerada de alto valor nutricional,32 de ahí que no es difícil que el contacto humano con estos animales silvestres esté involucrado en la emergencia de SARS-CoV-2.
Comentarios finales
Es difícil predecir el surgimiento de un virus nuevo o desconocido. En el siglo XXI se han presentado algunas epidemias de mayor o menor impacto causadas por diferentes virus con los que no convivimos habitualmente, entre los que podemos mencionar los virus de la influenza A H1N1 (2009), de Zika, chikungunya, Ébola, SARS y MERS. No obstante, si se hace una revisión sobre virus con potencial pandémico, la lista crece en forma importante. La pregunta es si a pesar de no saber cuál o cuáles virus pueden emerger como amenazas a la salud global, las naciones pueden estar mejor preparadas para enfrentarlas.
Con estas crisis sanitarias, en esencia zoonóticas, se volvió primordial la identificación de virus en animales de vida silvestre. De hecho, desde ese entonces se ha reportado una variedad de virus con probabilidades de causar brotes en humanos y con potencial relativo de causar pandemias. No obstante, no deja de ser una actividad científica que para muchos es solamente una curiosidad, sin reparar en la relevancia que puede tener en la salud pública y el bienestar mundial, lo que se refleja esencialmente en la falta de recursos para la investigación científica y tecnológica.33
La mayoría de los virus emergentes es causada por zoonosis, lo cual se debe, entre otros factores, a que los humanos invaden cada vez más zonas no exploradas donde hay especies silvestres con las que antes raramente tenían contacto. Algunos de estos animales son atrapados con propósitos comerciales, por ejemplo, para su uso como alimento, como parte de remedios de la medicina tradicional o como mascotas. La invasión del hábitat de estas especies lleva al contacto con su microbiota y a que se generen emergencias sanitarias de vez en cuando.34 Por ello, las actividades mencionadas, ya reglamentadas en muchos países, deben ser fuertemente vigiladas y, en su caso, sancionadas.
La pandemia de COVID-19 no será la última emergencia sanitaria que enfrente la humanidad. Poco a poco hemos detectado las áreas en las que tenemos que estar preparados y los gobiernos y entidades supranacionales deben asumir la responsabilidad de coordinar este esfuerzo para evitar los daños que en este momento se viven por la emergencia de SARS-CoV-2.











 nueva página del texto (beta)
nueva página del texto (beta)



