Introducción
La sangre de cordón umbilical (SCU) es una fuente de células progenitoras hematopoyéticas que se utilizan como tratamiento de enfermedades oncológicas y no oncológicas; estas células se caracterizan por ser pluripotentes y autorrenovables.1,2
La Fundación Internacional NetCord y la Fundación para la Acreditación de la Terapia Celular (FACT) establecen procedimientos estándarizados para la terapia celular y los bancos de SCU. Las unidades de sangre de cordón umbilical (USCU) deben cubrir los siguientes criterios en las respectivas fases de evaluación:
Antes de la criopreservación: ≥ 5.0 × 108 células nucleares totales, ≥ 85 % de viabilidad celular, ≥ 1.25 × 106 células CD34+, ≥ 85 % células CD34+ viables, tipificación de HLA (human leukocyte antigens), identificación de grupo sanguíneo y factor Rh, cultivos microbiológicos negativos a bacterias aerobias, anaerobias y hongos.
Posterior a la criopreservación y antes de la liberación para trasplante (segmentos): ≥ 70 % células CD34+ viables, ≥ 40 % células CD45+ viables, crecimiento de UFC como control pretrasplante de pluripotencia de las células progenitoras, verificación de HLA.3
Adicionalmente se realiza tamizaje de VIH1, VIH2, VHB, VHC, citomegalovirus, Treponema pallidum (sífilis) y Trypanosoma cruzi (enfermedad de Chagas) como control de calidad. Es posible realizar tamizaje de otros agentes infecciosos transmisibles según la historia del donador, características de las células donadas o región geográfica donde habita el donador.3
El virus del papiloma humano (VPH) es un agente infeccioso que puede transmitirse verticalmente (madre-hijo); el término transmisión transplacentaria suele utilizarse cuando la madre y el recién nacido presentan el mismo genotipo del VPH. A la fecha, se desconoce el mecanismo de transmisión del VPH entre la madre y el recien nacido;4-7 sin embargo, el ADN del VPH se ha identificado en SCU, placenta, fluido amniótico y mucosa oral al momento del nacimiento.5,8 Específicamente, en SCU la frecuencia del VPH es de 2.2 a 6.1 % y en placenta, 4.0 a 4.2 %; los genotipos de alto riesgo VPH16, VPH18, VPH39, VPH52 y VPH83 son los más frecuentes y los genotipos de bajo riesgo VPH6 y VPH11 en población de Finlandia y Brasil.5,6,9 Además, en los recién nacidos positivos a la presencia del genoma del VPH, específicamente en SCU o placenta, se incrementa el riesgo de infección persistente del VPH en mucosa oral. 6,10,11
De acuerdo con las evidencias mencionadas, el VPH se ha detectado en SCU o en placenta; por lo tanto, el objetivo del presente estudio fue optimizar la muestra biológica de las unidades formadoras de colonicas (UFC) de los cultivos clonogénicos para extraer ADN y generar un biobanco de ADN que permita resolver ambigüedades de HLA o implementar el análisis de otros biomarcadores, así como detectar el genoma del VPH en las USCU del biobanco del Centro Nacional de la Transfusión Sanguínea.
Métodos
Se incluyeron 141 muestras biológicas del biobanco del Centro Nacional de la Transfusión Sanguínea: 90 muestras fueron de SCU, que se utilizan en la tipificación del HLA antes de criopreservar las USCU; 41 se trataba de segmentos de USCU criopreservados para verificar el HLA y 10 fueron de UFC de cultivos clonogénicos, como control de pluripotencia. Cada USCU contiene una bolsa de 25 mL y tres segmentos de 50 a 200 mL cada uno, los cuales se utilizan para control pretrasplante (cuantificación de células, verificación de HLA y cultivos clonogénicos). Los criterios de inclusión fueron disponer del consentimiento informado de la madre, historia familiar negativa de enfermedades hereditarias y transmisibles, ≥ 34 semanas de gestación y enfermedades congénitas negativas. Los criterios de control de calidad fueron los siguientes: volumen de SCU ≥ 74 mL, cuantificación de glóbulos blancos totales ≥ 8 × 108, cuantificación de células CD34+ ≥ 2.0 × 106, cultivos microbiológicos negativos y serología no reactiva a VIH, VHB, VHC, Treponema pallidum, Trypanosoma cruzi, citomegalovirus o toxoplasma.
Los cultivos clonogénicos se realizan rutinariamente en el Centro Nacional de la Transfusión Sanguínea cuando se reservan USCU para trasplante; el protocolo ha sido reportado previamente.12 Las UFC se aislaron de 10 cultivos clonogénicos de USCU, de acuerdo con el protocolo descrito por StemCell Technologies Inc.13
La extracción de ADN de las muestras de SCU, segmentos de USCU y UFC se realizó con el kit QIAamp DNA Blood Mini® (Qiagen, California, Estados Unidos), de acuerdo con las especificaciones del proveedor. Los ADN se cuantificaron y almacenaron a 70 °C hasta su uso. Se evaluó la integridad del ADN extraído de las UFC en gel de agarosa a 1 %. Se utilizó el ADN de tres muestras de UFC para la tipificación del HLA con el kit Micro SSP Allele Specific HLA Class I (HLA-A , HLA-B) and Class II (HLA-DRB1, HLA-DQB1) (One Lamda, California, Estados Unidos), de acuerdo con las especificaciones del proveedor.
La detección del genoma del VPH se realizó con 500 ng de ADN genómico de las muestras de SCU, segmentos de USCU o UFC, utilizando los oligonucléotidos MY09/MY1114 y GP5+/GP6+15 (Tabla 1). El ADN de las líneas celulares Hela (VPH18) y SiHa (VPH16) fue utilizado como control positivo. Un fragmento del gen GAPDH se amplificó con 100 ng de ADN de todas las muestras, como control de calidad del ADN extraído (Tabla 1). Además, como control negativo en cada PCR (polymerase chan reaction) se incluyó una reacción sin ADN. Los productos de PCR se analizaron en gel de agarosa a 2 %.
Tabla 1 Secuencias de oligonucleótidos
| Oligonucleótido | Secuencia | Temperatura de alineamiento | Tamaño del fragmento |
|---|---|---|---|
| GAPDH F GAPDH R |
5´-ACC ACA GTC CAT GCC ATC AC-3´ 5´-TCC ACC ACC CTG TTG CTG TA-3´ |
57 ºC | 450 pb |
| MY09 MY11 |
5´- CGT CCM ARR GCA WAC TGA TC -3´ 5´- GCM CAG GGW CAT AAY AAT GG -3´ |
57 ºC | 450 pb |
| GP5+ GP6+ |
5´-TTT GTT ACT GTG GTA GAT ACT AC-3´ 5´-GAA AAA TAA ACT GTA AAT CAT ATT C-3´ |
40 ºC | 150 pb |
El análisis estadístico se realizó con el programa GraphPad Prix versión 7 (GraphPad Software, California, Estados Unido). Se comparó la concentración de ADN y la absorbancia 260/280 de las diferentes muestras biológicas con la prueba no paramétrica U de Mann-Whitney; los resultados se presentan como media con desviación estándar. Los valores de p < 0.05 fueron estadísticamente significativos.
Resultados
Las características de las USCU incluidas en el estudio se describen en la Tabla 2. El rango del porcentaje de glóbulos blancos totales fue de 60 a > 90 %, mientras que el rango inicial de glóbulos blancos totales fue de 8.0 a > 18.0 × 108 y el rango final de células CD34+ fue de 2.0 a > 5.0 × 106; estos parámetros cumplen los criterios establecidos por NetCord-FACT para que las USCU se utilicen con fines terapéuticos (Tabla 2).
Tabla 2 Características de las unidades de sangre de cordón umbilical
| Características | (%) |
|---|---|
| Tipo de muestra | |
| SCU | 63.83 |
| Segmento | 29.08 |
| UFC | 7.09 |
| Edad de la madre (años) | |
| ≤ 20 | 11.34 |
| 21-30 | 48.23 |
| 31-40 | 40.43 |
| Volumen de la USCU (mL) | |
| ≤ 90 | 24.82 |
| > 90-110 | 31.91 |
| > 110-130 | 30.50 |
| > 130 | 12.77 |
| Recuperación (%) | |
| 60-70 | 29.09 |
| > 70-80 | 26.24 |
| > 80-90 | 31.91 |
| > 90 | 12.76 |
| Glóbulos blancos totales x 108, medición inicial | |
| 8.0-10.0 | 19.86 |
| > 10.0-12.0 | 19.15 |
| > 12.0-14.0 | 20.57 |
| > 14.0-16.0 | 17.02 |
| > 16.0-18.0 | 7.80 |
| > 18.0 | 15.60 |
| Células CD34+ × 106 final | |
| 2.0-3.0 | 41.84 |
| > 3.0-4.0 | 25.53 |
| > 4.0-5.0 | 10.64 |
| > 5.0 | 21.99 |
| Grupo sanguíneo | |
| A | 29.08 |
| B | 4.26 |
| AB | 0.70 |
| O | 65.96 |
| Factor RH | |
| Positivo | 97.87 |
| Negativo | 2.13 |
SCU = sangre de cordón umbilical, USCU = unidades de sangre de cordón umbilical, UFC = unidades formadoras de colonias.
Para optimizar las USCU criopreservadas en el biobanco, se realizó la extracción de ADN de UFC obtenidas de cultivos clonogénicos (Figura 1A). La Figura 1B es una imagen representativa de un gel de agarosa con muestras de ADN purificado de las UFC; en la parte superior de cada carril se aprecia una banda que permite confirmar la integridad del ADN sin degradación. En la Figura 1C se muestra la tipificación de HLA mediante técnica sequence specific priming (SSP); se utilizó como templado ADN purificado de una muestra de UFC; la banda superior corresponde a la amplificación del gen endógeno b-globina, que confirma la integridad de la reacción; las bandas inferiores corresponden a la amplificación de los alelos específicos de HLA de la muestra. Los resultados confirman la tipificación de HLA de las tres muestras de ADN seleccionadas de las UFC, debido a que las USCU ya se habían tipificado con SCU antes de su criopreservación o del segmento descongelado, obteniendo los mismos resultados de HLA. Los resultados muestran las potenciales aplicaciones del uso de las UFC para obtener ADN, ARN o proteínas para realizar pruebas adicionales.

Figura 1 Imágenes representativas de UFC de cultivos clonogénicos. A) UFC de cultivos clonogénicos. BFU-E, burst-forming-unit-erythroid; CFU-GM, colony-formed-unit-granulocyte-macrophage; CFU-GEMM, granulocyte-erythrocyte-monocyte-megakaryocyte. B) Electroforesis de ADN extraído de UFC en gel de agarosa a 1 %. C) Tipificación de HLA de una muestra de ADN de UFC mediante la técnica sequence-specific priming. Carril 1, marcador de peso molecular; carril 2, panel superior izquierdo, control de reacción sin ADN; carriles 2 a 9, reacciones con ADN de UFC (banda superior control interno, bandas inferiores alelos de HLA específicos de cada muestra). Los alelos detectados se muestran en recuadro y se interpretan conforme a su tamaño. UFC = unidades formadoras de colonias.
Para la detección del VPH a través de PCR se realizó extracción de ADN. En la Figura 2A se observa la distribución de todas las muestras incluidas en el estudio respecto a la concentración total de ADN en un rango de 1.51 a 96.2 mg y la relación de absorbancia 260/280 en un rango de 1.40 a 2.05. Posteriormente, se comparó la concentración de ADN total en los diferentes tipos de muestras biológicas; la media en SCU fue de 25.81 mg, estadísticamente significativa en comparación con la media en segmento, que fue de 11.19 mg (p < 0.0001). La media en UFC fue de 36.56 mg, estadísticamente significativa en comparación con la media en SCU (p = 0.0041) y la media en segmento (p < 0.0001) (Figura 2B). Sin embargo, cuando se comparó la absorbancia 260/280 de los diferentes tipos de muestras no se identificaron diferencias estadísticamente significativas (Figura 2C).

Figura 2 ADN purificado de SCU, segmento y UFC. A) Distribución de las muestras de ADN purificado. B) Concentración de ADN total. C) Relación de absorbancia 260/280. Los resultados se graficaron en medias y desviación estándar. SCU = sangre de cordon umbilical, UFC = unidades formadoras de colonias.
Una vez cuantificadas las muestras de ADN (en SCU, segmento y UFC) se realizó la detección del genoma del VPH en todas las muestras. La detección se realizó a través de la amplificación por PCR de un fragmento de 450 pb del gen L1 del VPH con los oligonucleótidos MY09/MY11 (Figura 3A) y un fragmento de 150 pb del mismo gen con los oligonucleótidos GP5+/GP6+ (Figura 3B); no detectamos ninguna muestra VPH positiva con ninguno de los oligonucleótidos utilizados. Además, para verificar la integridad del ADN se amplificó un fragmento de 450 pb del gen GAPDH como control endógeno, con el que se observó amplificación de todas las muestras de ADN incluidas en el estudio (Figura 2C).
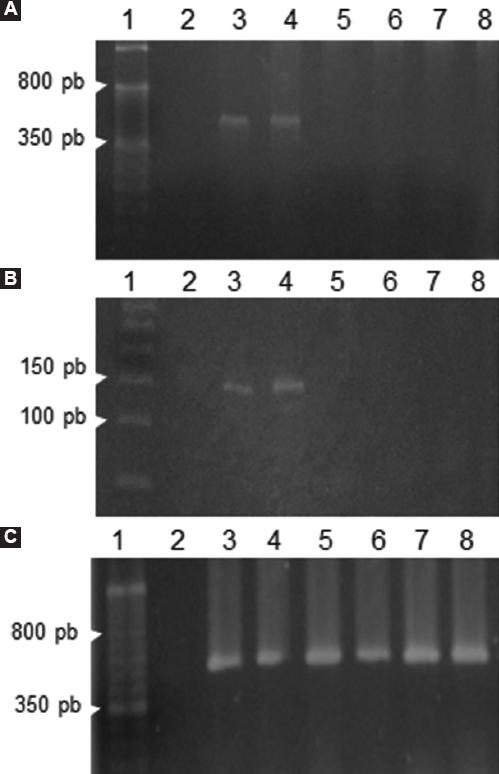
Figura 3 Imágenes representativas de la detección del VPH en las USCU. A) PCR del gen L1 con los oligonucleótidos MY09/MY11, que amplifican un fragmento de 450 pb. B) PCR del gen L1 con los oligonucleótidos GP5+/GP6+, que amplifican un fragmento de 150 pb. C) PCR del gen GAPDH, que amplifica un fragmento de 450 pb. Carril 1, marcador de peso molecular; carril 2, control negativo sin ADN; carril 3, ADN de línea celular HeLa; carril 4, ADN de línea celular SiHa; carril 5-8, muestras de ADN de las USCU. VPH = virus del papiloma humano, USCU = unidad de sangre de cordón umbilical, PCR = polymerase chain reaction.
Discusión
Las USCU criopreservadas en el biobanco del Centro Nacional de la Transfusión Sanguínea fueron VPH negativas, parámetro de control de calidad adicional para que las USCU sean trasplantadas. La optimización de las UFC de cultivos clonogénicos permitirá generar un biobanco de ADN con pureza y concentración adecuada para resolver ambigüedades de HLA o implementar el análisis de otros biomarcadores.
En el presente estudio incluimos muestras de ADN purificado de UFC de cultivos clonogénicos. Aun cuando existe un protocolo de StemCell Technologies Inc. para extraer ácidos nucleicos, no hay evidencia respecto a su uso en técnicas de biología molecular para diagnostico in vitro. Observamos una media de concentración de ADN mayor en este tipo de muestra respecto a la concentración de ADN extraída de SCU antes de su criopreservación o del segmento descongelado. Es necesario mencionar que la concentración del ADN depende del número de UFC y del tamaño de estas en los cultivos clonogénicos; sin embargo, cuando se realizó una correlación entre la concentración de ADN y el número de UFC total no se encontró una correlación positiva (datos no mostrados), debido a que también influye el tamaño de las UFC.
En las muestras de segmentos descongelados de SCU se obtuvo una media menor de concentración de ADN, resultado esperado debido a que cada segmento de SCU contiene un volumen de 50 a 200 mL; además, los segmentos estuvieron criopreservados por meses o años, lo cual, como es conocido en los procesos de criopreservación y descongelación, afecta la viabilidad celular y la función de las células progenitoras hematopoyéticas.16-18 Se ha reportado que el tiempo de almacenamiento de las muestras puede afectar la concentración y pureza del ADN debido al proceso de degradación natural de los ácidos nucleicos.19-21
Otra variable importante del ADN es la pureza. Obtuvimos una relación 260/280 en un rango de 1.64 a 2.05, sin diferencias estadísticamente significativas entre los diferentes grupos analizados. Entre las especificaciones para la tipificación de HLA mediante las técnicas SSP y sequence-specific oligonucleotide se requiere una relación de absorbancia de 1.65 a 1.80, la cual se cumple con los valores de pureza obtenidos. Además, la relación óptima del ADN cuantificado en las longitudes 260/280 debe ser mayor de 1.80; una relación > 2.1 indica presencia de ARN en la muestra y una relación < 1.60 indica presencia de proteínas o contaminantes.22
Respecto a la detección del genoma del VPH no encontramos ninguna USCU positiva, lo cual representa un control de calidad adicional en la detección de un agente infeccioso para que estas unidades sean trasplantadas. Una desventaja del estudio fue el tamaño de la muestra (n = 141), debido a que el VPH se ha observado en SCU con una frecuencia de 2.2 a 6.1 %,5,6,9 por lo tanto, sería conveniente incrementar el tamaño de la muestra. Entre los genotipos del VPH frecuentemente identificados en SCU se encuentran los VPH de alto riesgo que se asocian con alteración de la respuesta inmune, lesiones premalignas y cáncer.5,6,9,10,11 Por lo tanto es pertinente analizar la presencia del VPH en una muestra de mayor tamaño de USCU.
En conclusión, las USCU analizadas fueron VPH negativas; además, la extracción de ADN de UFC de cultivos clonogénicos permitirá generar un biobanco de ADN para implementar en el futuro otros biomarcadores, resolver ambigüedades de HLA o implementar protocolos de investigación.











 nueva página del texto (beta)
nueva página del texto (beta)


