Introducción
Pneumocystis jirovecii es un hongo oportunista cuyo cultivo in vitro no se ha logrado. Causa infección pulmonar en personas inmunocomprometidas, particularmente en las infectadas con el virus de la inmunodefiencia humana (VIH), que han desarrollado síndrome de inmunodeficiencia adquirida (SIDA), desnutridas o sometidas a trasplantantes.1 Pneumocystis jirovecii es adquirido por vía respiratoria en etapas tempranas de la vida y frecuentemente se encuentra como comensal en personas inmunocompetentes. Durante la inmunosupresión, el microorganismo se multiplica en los alveolos induciendo un proceso inflamatorio.2 La neumonía por Pneumocystis jirovecii (PCP) se manifiesta por síntomas inespecíficos, si bien la insuficiencia respiratoria es la complicación más frecuente y se asocia con alta mortalidad.3
Para detectar Pneumocystis jirovecii comúnmente se utilizan tinciones (Gomori-Grocott y Giemsa) y procedimientos moleculares.4,5 La reacción en cadena de la polimerasa (PCR) ha sido dirigida hacia la región del espaciador transcrito interno (inter transcribed spacer, ITS) del ADN ribosómico.2,6 Esta región ITS es el “código de barras” para la identificación de hongos. La comparación de secuencias de la región ITS es ampliamente usada en taxonomía y filogenia molecular porque es muy fácil de amplificar, aún a partir de cantidades pequeñas de ADN y tiene un alto grado de variación, incluso entre especies estrechamente cercanas. Este marcador existe en múltiples copias en la mayoría de las células fúngicas y es recuperable con oligonucléotidos relativamente robustos con un alto grado de confiabilidad.7,8
Para cada gen blanco se han reportado secuencias tipo y las diferencias detectadas en estudios similares han determinado numerosos genotipos.2,9,10 Para la región ITS1 se conocen más de 30 genotipos y para ITS2 más de 40.11
En el mundo, la PCP representa la primera o segunda infección oportunista más común en los pacientes con infección por VIH.12 En México ha sido reportada hasta en 51 y 55 % de los casos de infección por VIH y el diagnóstico se ha basado en tinciones.13,14 El objetivo de esta investigación fue determinar la frecuencia de detección de Pneumocystis jirovecii y sus genotipos en muestras de pacientes infectados por VIH/SIDA o con alguna otra patología inmunosupresora, mediante PCR anidada de la región ITS seguida de secuenciación.
Método
Estudio prospectivo, transversal y prolectivo. Se solicitaron especímenes respiratorios de pacientes hospitalizados con síntomas pulmonares en el Instituto Nacional de Enfermedades Respiratorias y el Hospital Regional “General Ignacio Zaragoza” del Instituto de Seguridad y Servicios Sociales de los Trabajadores del Estado, Ciudad de México.
Se incluyeron pacientes hospitalizados, de cualquier edad, de uno u otro sexo, con síntomas respiratorios, sin tratamiento anti-Pneumocystis. No se solicitó el consentimiento informado debido a que el estudio se realizó con parte de la muestra obtenida para otros análisis. El control negativo incluyó líquido de lavado orofaríngeo de personas sanas, previa solicitud de consentimiento informado.
Para la extracción del ADN fueron consideradas las condiciones descritas por Lu et al.15 y se utilizó el kit High Pure PCR Template Preparation® (Roche, Alemania). El ADN fue almacenado a –20 °C y después utilizado para la PCR.
PCR anidada
– Primera ronda: se utilizaron 20 µL de ADN en un volumen final de 50 µL que contenía 1x de PCR-buffer, 1.5 mM de MgCl2, 1.0 mM de desoxirribonucleótidos trifosfato, 0.2 µM de oligonucleótido 1724F (AAG TTG GTC AAA TTT GGT C), 0.2 µM de oligonucleótido ITS2R (CTC GGA CGA GGA TCC TCG CC), 1.25 U de Taq DNA polimerasa (Invitrogen, Carlsbad, California, Estados Unidos). Temperatura: 94 °C, 5 minutos; 35 ciclos de 94 °C por un minuto, 47 °C por un minuto y 72 °C por dos minutos; finalmente, 72 °C por cinco minutos. El tamaño del producto esperado fue de 693 pares de bases (pb).15 Como control negativo de PCR se emplearon 20 µL de agua miliQ estéril y como control positivo, 3 µL de DNA de una muestra notablemente positiva para Pneumocystis jirovecii por tinción de Gomori-Grocott y por PCR.
– Segunda ronda: se utilizaron 2 µL del producto de amplificación de la primera ronda, en un volumen final de 50 µL. Los oligonucleótidos fueron ITS1F (CGT AGG TGA ACC TGC GGA AGG ATC) e ITS2R1 (GTT CAG CGG GTG ATC CTG CCT G). Las concentraciones de la mezcla de reacción fueron similares a las de la primera ronda. Temperaturas: 94 °C, 10 minutos; 35 ciclos de 94 °C por un minuto, de 58 °C por un minuto, de 72 °C por dos minutos. La extensión final a 72 °C por 10 minutos. El tamaño esperado del producto fue de 550 pb.15
Los amplicones fueron corridos en un gel de agarosa a 1.5 %, teñidos con bromuro de etidio, purificados y enviados a secuenciar al Instituto de Biología de la Universidad Nacional Autónoma de México, Ciudad de México, con el método de inhibición de terminación de cadena.16 Todas las secuencias fueron realizadas por duplicado en sentidos 5’-3’ y 3’-5’.
Análisis de secuencias
Las secuencias sense y antisense fueron analizadas y editadas con el programa ChromasPro y comparadas con tres bases de datos: Basic Local Alignment Search Tool del GenBank (https://www.ncbi.nlm.nih.gov/), The European Molecular Biology Laboratory (http://www.embl.de/) y Kyoto Encyclopedia of Genes and Genomes (http://www.genome.jp/kegg/).
Las secuencias fueron también analizadas con el programa MEGA versión 6.0, para construir un árbol filogenético con el método de neighbour-joining, utilizando el modelo Kimura de dos parámetros con un bootstrap de 1000 repeticiones.17 El alineamiento múltiple se hizo utilizando ClustalW. Se construyó una matriz de distancias genéticas.
Determinación de genotipos
Se tomaron como referencia las secuencias reportadas por Lee et al.,9 quien consideró 15 genotipos para la región ITS1 (A-O) y 14 genotipos para ITS2 (a-n).
Resultados
Del Instituto Nacional de Enfermedades Respiratorias se recibieron 48 muestras de 30 pacientes (23 hombres y 7 mujeres). La edad osciló de cinco a 76 años, con promedio de 35.7. La mayoría de las muestras (30) fueron líquido bronquioalveolar y tejido (18). En 26 pacientes (86.6 %), el principal factor de oportunismo fue la infección por VIH/SIDA, la mayoría de los casos confirmados (Tabla 1).
Tabla 1 Datos generales de pacientes hospitalizados con síntomas pulmonares en el Instituto Nacional de Enfermedades Respiratorias
| Número de muestra | Sexo | Edad (años) | Espécimen | Factor de riesgo | PCR Pneumocystis jirovecii |
|---|---|---|---|---|---|
| 1 | M | — | LBA | VIH/SIDA | Positiva |
| 2a | F | 28 | LBA | VIH | — |
| 2b | F | 28 | BxP | — | — |
| 3a | M | 25 | LBA | VIH | — |
| 3b | M | 25 | LBA | VIH, TbP | — |
| 4a | M | 43 | LBA | VIH/SIDA | — |
| 4b | M | 43 | BxP | VIH/SIDA | — |
| 5 | F | 59 | LBA | VIH | Positiva |
| 6a | M | 32 | LBA | VIH | Positiva |
| 6b | M | 32 | BxP | VIH | — |
| 7 | M | 25 | LBA | VIH/SIDA, TbP | — |
| 8a | M | 26 | LBA | VIH/CM | — |
| 8b | M | 26 | LBA | VIH/CM | — |
| 9a | M | 33 | LBA | VIH | Positiva |
| 9b | M | 33 | BxP | VIH | — |
| 10a | M | 26 | BxP | VIH/PCP | Positiva |
| 10b | M | 26 | LBA | VIH/PCP | Positiva |
| 11a | M | 28 | LBA | VIH/PCP | Positiva |
| 11b | M | 28 | BxP | VIH/PCP | Positiva |
| 12a | M | 25 | BxP | VIH/SIDA | Cryptococcus |
| 12b | M | 25 | LBA | VIH/SIDA | — |
| 13a | F | 33 | BxP | VIH/PCP | — |
| 13b | F | 33 | LBA | VIH/PCP | Positiva |
| 14 | F | 57 | LBA | VIH | — |
| 15 | M | 76 | LBA | VIH | — |
| 16 | M | 53 | LBA | Neumonía | — |
| 17 | F | 5 | BxP | Desconocido | — |
| 18a | M | 44 | BxG | VIH/SK | — |
| 18b | M | 44 | LBA | VIH/SK | Positiva |
| 19 | M | 31 | BxP | VIH | — |
| 20a | M | 28 | LBA | Pb VIH/PCP | — |
| 20b | M | 28 | BxP | Pb VIH/PCP | — |
| 21a | M | 44 | LBA | VIH/neumonía | Positiva |
| 21b | M | 44 | LBA | VIH/neumonía | — |
| 22a | F | 31 | LBA | VIH/PCP | — |
| 22b | F | 31 | BxP | VIH/PCP | — |
| 23a | M | 20 | LBA | Pb VIH | — |
| 23b | M | 20 | BxP | Pb VIH | — |
| 24a | M | 32 | BxP | VIH, TbG | — |
| 24b | M | 32 | LBA | VIH, TbG | — |
| 25 | M | 24 | LBA | VIH/PCP | Positiva |
| 26 | M | 37 | LBA | VIH/PCP | Positiva |
| 27 | M | 31 | BxP | VIH/Pb TbP | — |
| 28 | F | 25 | LBA | PCP | — |
| 29 | M | 74 | LBA | Pb FPI | — |
| 30a | M | 42 | LBA | VIH/TbP | — |
| 30b | M | 42 | BxG | VIH | — |
| 30c | M | 42 | BxP | VIH/TbP | — |
M = masculino, F = femenino, LBA = líquido bronquioalveolar, BxP = biopsia pulmonar, BxG = biopsia ganglionar, Pb = probable, VIH = virus de inmunodeficiencia humana, SIDA = síndrome de inmunodeficiencia adquirida, TbP = tuberculosis pulmonar, CM = complejo Mycobacterium, PCP = neumonía por Pneumocystis, TbG = tuberculosis ganglionar, FPI = fibrosis pulmonar idiopática, SK = sarcoma de Kaposi.
Del Hospital Regional “General Ignacio Zaragoza” se recibieron 30 muestras, correspondientes a 23 pacientes (15 hombres y ocho mujeres). La edad osciló de uno a 86 años, con promedio de 50.6. La mayoría de las muestras (25) fueron de esputo. Los pacientes presentaron diversos factores de oportunismo, con predominio de tuberculosis pulmonar (siete, 29.16 %) probable o confirmada en el momento de la recepción de la muestra (Tabla 2).
Tabla 2 Datos generales de pacientes con síntomas pulmonares del Hospital Regional “General Ignacio Zaragoza”
| Número de muestra | Sexo | Edad (años) | Espécimen | Factor de riesgo |
|---|---|---|---|---|
| 1 | M | 50 | LBA | Desconocido |
| 2 | F | 64 | LBA | Desconocido |
| 3a | M | 38 | Esputo | Tuberculosis pulmonar |
| 3b | M | 38 | Esputo | Tuberculosis pulmonar |
| 3c | M | 38 | Esputo | Tuberculosis pulmonar |
| 3d | M | 38 | Esputo | Tuberculosis pulmonar |
| 4 | F | 52 | Esputo | Desconocido |
| 5 | M | 57 | Esputo | Probable TbP |
| 6a | M | 37 | Esputo | Probable TbP |
| 6b | M | 37 | Esputo | Probable TbP |
| 7a | F | 45 | Esputo | Tuberculosis pulmonar |
| 7b | F | 45 | Esputo | Tuberculosis pulmonar |
| 8 | M | 21 | Esputo | VIH, probable TbP |
| 9a | M | 63 | Esputo | Neoplasia pulmonar |
| 9b | M | 63 | Esputo | Hemoptisis en estudio |
| 10 | M | 43 | Esputo | Tuberculosis pulmonar |
| 11 | M | 31 | Esputo | VIH |
| 12 | M | 67 | Esputo | Desconocido |
| 13 | F | 50 | Líquido pleural | Probable TbP |
| 14 | M | 80 | Líquido pleural | Neoplasia pulmonar |
| 15 | F | 23 | LBA | Epilepsia |
| 16a | M | 57 | LBA | Neumonía |
| 16b | M | 57 | Esputo | Choque séptico |
| 17 | F | 37 | Esputo | Choque séptico |
| 18 | M | 76 | Esputo | Insuficiencia respiratoria |
| 19 | F | 62 | Esputo | Insuficiencia respiratoria |
| 20 | M | 86 | Esputo | EPOC, neumonía |
| 21 | F | 64 | Esputo | Neumonía |
| 22 | M | 1 | Esputo | Sepsis, neumonía |
| 23 | M | 61 | Esputo | Choque séptico |
M = masculino, F = femenino, LBA = líquido bronquioalveolar, TbP = tuberculosis pulmonar, VIH = virus de inmunodeficiencia humana, EPOC = enfermedad pulmonar obstructiva crónica.
El grupo control negativo consistió en 11 muestras de líquido de lavado orofaríngeo de personas adultas, sanas, cuya edad promedio fue de 34.2 años, sin antecedente de tabaquismo o síntomas respiratorios en el momento del muestreo.
Detección de Pneumocystis jirovecii
La Figura 1 muestra las estructuras parasitarias teñidas con Gomori-Grocott y el amplicon correspondiente a Pneumocystis jirovecii obtenido de líquido bronquioalveolar, el cual fue usado como control positivo de PCR. El fragmento fue purificado y secuenciado para corroborar su identidad con Pneumocystis jirovecii.

Figura 1 Estudio de la muestra utilizada como control positivo (muestra 1 de los pacientes del Instituto Nacional de Enfermedades Respiratorias). A: Tinción de Gomori-Grocott con la que se observan las formas quísticas típicas de Pneumocystis jirovecii (tamaño de escala, 5 µm). B: Producto de PCR anidada corrido en un gel de agarosa a 1.5 %, teñido con bromuro de etidio. El amplicón de 550 pb corresponde a la muestra de la Figura 1A. M = marcador de peso molecular de 100 pb.
De los 30 pacientes del Instituto Nacional de Enfermedades Respiratorias, 11 fueron positivos a Pneumocystis jirovecii (Figuras 1B y 2A), todos ellos tenían infección por VIH como factor de oportunismo, nueve eran hombres y dos mujeres. En cinco pacientes previamente se había diagnosticado PCP por tinción de Gomori-Grocott, confirmados por PCR. Los pacientes positivos a Pneumocystis jirovecii representaron 36.6 % de los casos de esta institución.

Figura 2 Amplicones de la región ITS de Pneumocystis jirovecii. A: Muestras que amplificaron un fragmento del tamaño esperado (550 pb). B: Muestra que amplificó un fragmento de mayor tamaño al esperado (620 pb). M = marcador de peso molecular de 100 pb. Los números en la parte superior indican el número de la muestra.
En la muestra 12a, correspondiente a un paciente del Instituto Nacional de Enfermedades Respiratorias, se obtuvo un amplicón de 620 pb, mayor al tamaño esperado (Figura 2B). Este fragmento fue secuenciado y analizado.
Todas las muestras del Hospital Regional “General Ignacio Zaragoza” y del grupo control negativo (individuos sanos) fueron negativas para Pneumocystis jirovecii.
Análisis de similitud de secuencias
La identidad con Pneumocystis jirovecii osciló entre 90 y 100 %. El análisis en las tres bases de datos fue similar. Todos los fragmentos amplificados correspondieron a la región ITS de Pneumocystis jirovecii. En la muestra 12a, el electroferograma fue muy confiable y el análisis comparativo indicó 87 % de identidad con Cryptococcus sp.
Con los datos de la secuenciación se construyó una matriz de distancias genéticas (datos no mostrados) y con el análisis del origen de las muestras (Figura 3) se exploró su correlación. La muestra 12a se mantuvo en una rama separada de las otras. Las muestras 11a y 11b correspondieron al mismo paciente, cuyo valor de distancia genética fue 0.000; ambas agruparon en la misma rama, lo cual indica que es la misma cepa de Pneumocystis jirovecii. Las muestras 10a y 10b también pertenecían al mismo paciente, con una distancia genética de 0.152, y se agruparon en diferentes ramas filogenéticas, lo cual sugirió que había diferencia entre las cepas de Pneumocystis jirovecii.
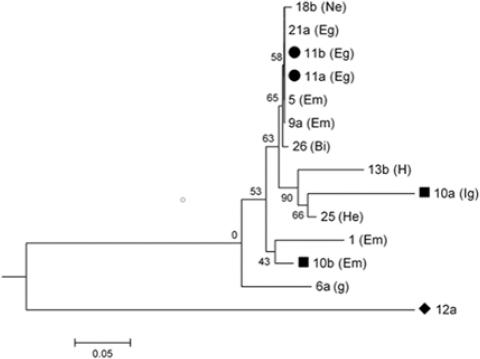
Figura 3 Árbol filogenético construido con el método de neighbour-joining. Los números corresponden a cada muestra procesada y entre paréntesis se indica su haplotipo. Las muestras 10a y 10b y 11a y 11b corresponden al mismo paciente respectivamente. Cryptococcus neoformans fue identificado en la muestra 12a y fue usado como control externo.
Determinación de genotipos
De las 14 muestras positivas a Pneumocystis jirovecii, en 11 se determinó el genotipo en ambas direcciones. En tres secuencias no fue posible determinar el genotipo debido al gran número de inserciones y deleciones. En estas muestras, el procedimiento fue repetido dos veces más, desde la extracción de ADN hasta el análisis de las secuencias y los resultados fueron similares. Para ITS1 fue predominante el genotipo E (siete muestras) y para ITS2, el genotipo g (cinco muestras). Los haplotipos predominantes fueron Em (cuatro casos) y Eg (tres casos). En las muestras 10a y 10b, que correspondieron al mismo paciente, los genotipos fueron diferentes. En las muestras 11a y 11b, que también correspondieron al mismo paciente, se encontró el mismo genotipo.
Discusión
Debido a la imposibilidad para recuperar Pneumocystis jirovecii en cultivos, los procedimientos moleculares han sido particularmente útiles para detectar este organismo en muestras de pacientes en quienes se sospecha PCP. La frecuencia de detección de Pneumocystis jirovecii reportada por otros autores es variable, dependiendo de diversos factores como el tipo de muestra, la carga fúngica, el procedimiento molecular utilizado y el gen seleccionado. La PCR anidada y la PCR en tiempo real que utilizan fragmentos de ADN mitocondrial, ITS1 e ITS2 como genes blanco han mostrado altos valores de sensibilidad y especificidad.2,18
La PCR ha revelado valores de sensibilidad y especificidad para líquido bronquioalveolar que oscilan entre 84 y 100 % y 58 y 100 %, respectivamente,19,20 por ello ha sido el espécimen más usado y confiable. Gupta et al.21 estudiaron 180 muestras de pacientes inmunosuprimidos con sospecha clínica de PCP; por PCR anidada dirigida a la región ITS encontraron 24.69 % de positividad en 81 muestras de líquido bronquioalveolar y en 12 % de 50 muestras de esputo. En otros países, como Brasil, por PCR anidada se ha identificado 44.8 % de positividad a Pneumocystis jirovecii en pacientes VIH positivos.22 En este estudio, de 30 muestras de líquido bronquioalveolar procesados, 11 (36.6 %) fueron positivos.
Los 25 esputos que correspondieron a pacientes del Hospital Regional “General Ignacio Zaragoza” fueron negativos a Pneumocystis jirovecii. En otras investigaciones, los resultados negativos han sido atribuidos a la presencia de inhibidores de la PCR.23 Para el grupo de pacientes de ese hospital es posible que otros factores hayan influido en los resultados negativos, como el menor grado de inmunosupresión causado por la enfermedad subyacente, que fue principalmente tuberculosis pulmonar, ocasionalmente reportada en coinfección con Pneumocystis jirovecii.24 Los dos pacientes VIH positivos del Hospital Regional “General Ignacio Zaragoza” no mostraron evidencia de Pneumocystis, probablemente por una etapa moderada de inmunosupresión, como los casos de tuberculosis pulmonar.
Medrano et al.25 detectaron ADN de Pneumocystis jirovecii en 12 (20 %) de 50 muestras de líquido de lavado orofaríngeo de individuos sanos y Vargas et al.26 en 32 % de 74 niños sanos. En este trabajo, la ausencia de Pneumocystis jirovecii en individuos sanos posiblemente se debió a que las personas no eran portadoras de Pneumocystis jirovecii, a que la carga fúngica era muy baja o a que el procedimiento de lavado orofaringeo fue inadecuado para detectar al microorganismo.
En los últimos años, las bases de datos genéticos han crecido exponencialmente y en la actualidad hay más información sobre la identidad de los microorganismos. El análisis por la herramienta de búsqueda de alineación local básica (BLASTn) de los oligonucleótidos aquí utilizados indica que amplifican para un fragmento de ADN de Pneumocystis jirovecii y Cryptococcus sp. La amplificación de un fragmento correspondiente a Cryptococcus se puede explicar porque los oligonucleótidos descritos para la región ITS son universales para la identificación molecular de especies fúngicas.7,8 Además, no es sorprendente encontrar a Cryptococcus colonizando o infectando a pacientes con infección por VIH, ya que la levadura (principalmente Cryptococcus neoformans) es adquirida por vía respiratoria, inicialmente infecta el pulmón y después invade el sistema nervioso central y otros tejidos.27
La mezcla de genotipos de Pneumocystis jirovecii (como en las muestras 10a y 10b) es una condición frecuente.28 Es posible que la alta variabilidad genética y la dificultad para determinar genotipos en algunas muestras de este estudio se debiera al contacto de los pacientes con diferentes fuentes de infección y cepas.29 Respecto a los genotipos detectados, el haplotipo Em (cuatro muestras) no ha sido reportado como predominante, diferente al haplotipo Eg (tres muestras) encontrado como el más común en otro reporte.30 Esta investigación no es suficiente para afirmar que la población mexicana sea portadora de un genotipo distinto a otras poblaciones analizadas.
Aunque en la mayoría de los estudios se utiliza esputo como espécimen, es posible que la forma de obtenerlo sea un factor decisivo para detectar al parásito fúngico. Casanova et al.13 encontraron Pneumocystis carinii (Pneumocystis jirovecii) en 51 % de 128 pacientes después de obtener el esputo por expectoración inducida y con tinción de Giemsa.
La población infectada con VIH sigue estando en alto riesgo de desarrollar PCP debido a su bajo nivel de linfocitos CD4.31
La PCP es una patología que ocupa un lugar preponderante en la población sometida a algún factor de inmunosupresión. Se estima que en el mundo, la prevalencia anual de neumonía por Pneumocystis jirovecii es de 500 000 casos.32 Para aplicar medidas de control de las enfermedades infecciosas, en particular las infecciones oportunistas como la PCP, es necesario conocer su estado epidemiológico en cada país.
En este trabajo se identificó alta positividad de Pneumocystis jirovecii y sus genotipos en pacientes mexicanos VIH-positivos a través de la PCR anidada dirigida el gen de la región ITS. Es necesario vigilar la población mexicana en riesgo de desarrollar PCP, aplicando procedimientos moleculares para determinar con certeza la carga fúngica y, si es posible, detectar cepas resistentes a los medicamentos de uso terapéutico, para iniciar el tratamiento específico.











 nueva página del texto (beta)
nueva página del texto (beta)


