Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Gaceta médica de México
On-line version ISSN 2696-1288Print version ISSN 0016-3813
Gac. Méd. Méx vol.142 n.3 Ciudad de México May./Jun. 2006
Artículo original
Influencia de alelos y haplotipos del complejo principal de histocompatibilidad en la susceptibilidad a lupus eritematoso generalizado en la población mexicana
Influence of alleles and haplotypes of the main histocompability complex on the susceptibility to sytemic lupus erythematosus in the Mexican population
Julio Granados,a* Joaquín Zúñiga,b Víctor Acuña–Alonzo,c Florencia Rosettid y Gilberto Vargas–Alarcóne
a Departamento de Inmunología y Reumatología, Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán, México D. F.
b Centro de Investigación en Enfermedades Infecciosas para el Paciente Inmunocomprometido, Instituto Nacional de Enfermedades Respiratorias, México D. F.
c Laboratorio de Genética Molecular, Escuela Nacional de Antropología e Historia, México D. F.
d Departamento de Fisiología, Instituto Nacional de Cardiología Ignacio Chávez, México D. F., México
Recibido en su versión modificada: 14 de diciembre de 2005
Aceptado: 17 de febrero de 2006
*Correspondencia y solicitud de sobretiros:
Julio Granados,
Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán.
Vasco de Quiroga 15, Delegación Tlalpan
C. P. 14000, México D.F., México.
Teléfono: 5485 0080.
Correo electrónico: julgrate@yahoo.com
Resumen
A pesar de que se han asociado algunos alelos del complejo principal de histocompatibilidad (MHC) con la susceptibilidad a lupus eritematoso generalizado (LEG) en diferentes grupos étnicos, aún se desconoce la contribución de otros genes como los HSP70 y TNF–α en la susceptibilidad a esta enfermedad. Además, las asociaciones genéticas varían con base en el grupo étnico estudiado, lo que sugiere que la etnicidad tiene un papel importante en la susceptibilidad. En este estudio se analizaron las frecuencias de los genes HLA–DRB1, DQA1, DQB1, HSP70–2 y polimorfismos del promotor de TNF–α en 81 pacientes mexicanos con LEG. Como grupo control se estudiaron 99 sujetos mexicanos mestizos sanos. El haplotipo DRB 1*0301–DQA1*0501–DQB1 *0201 se asoció con LEG. El alelo DRB 1 *1501 se encontró con mayor frecuencia en los pacientes con LEG en comparación con los grupos control. Además se observó menor frecuencia de marcadores autóctonos como el alelo DRB 1 *0802 en los pacientes con LEG. Los alelos del MHC asociados con LEG son poco frecuentes en las poblaciones indígenas mexicanas. Esto podría sugerir que los marcadores de susceptibilidad a LEG se incorporaron a la población mestiza mexicana por flujo genético de poblaciones no indígenas.
Palabras clave: LEG, MHC, HLA, susceptibilidad, TNF–α, polimorfismo
Summary
HLA alelles with susceptibility to systemic lupus erythematosus (SLE) have been found in many ethnic groups. In addition, some neighboring genes such as TNF–α and HSP70, that may contribute to this disease have also been described. Interestingly some of the genetic associations differ among several ethnic groups, which might suggest that ethnicity plays an important role in the predisposition to SLE. In this study, we analyze gene frequencies of HLA–DRB1, DQA1, DQB1, HSP70–2 alelles and the polymorphism of TNF–α promoter region among 81 mexican mestizo SLE patients. A control group of 99 healthy mexican mestizos was included. We found that the HLA–DRB1*0301–DQA1*0501–DQB1*0201 haplotype was significantly increased in SLE patients compared to healthy controls (p=0.01, OR=2.97, IC 95%=1.18–7.68). The DRB 1*1501 allele was more frequent among patients than among controls. A significantly decreased frequency of the HLA–DRB1 *0802 alelle in SLE patients was also observed. Since the HLA alelles associated with SLE are uncommon in mexican ethnic groups, we performed admixture estimates analysis and found that the incorporation of SLE susceptibility markers in mexican mestizo groups might have come from genetic admixture with Caucasian populations.
Key words: SLE, HLA, susceptibility, TNF–α, polymorphism
Introducción
El lupus eritematoso generalizado (LEG) es una enfermedad autoinmune caracterizada por diferentes manifestaciones clínicas e inmunológicas. 1,2 Existe evidencia de que los factores genéticos tienen un papel importante en la susceptibilidad a LEG.3,4 Se ha puesto particular atención a los genes del complejo principal de histocompatibilidad (MHC) para la búsqueda de marcadores de susceptibilidad a LEG, estos genes están localizados en el brazo corto del cromosoma 6.5,6 Estudios previos demuestran una asociación alélica de HLA–B*0801, HLA–DRB1 *0301, *0302, *1501, *15037 con el LEG en poblaciones caucásicas y africanas. En estos grupos étnicos se han observado deleciones en factores del complemento como los alelos nulos del C4 "C4AQ0" y del gen C2 como un papel importante en la susceptibilidad a la enfermedad. El desequilibrio genético del HLA–DR2 y DR3 con alelos específicos de los loci HLA–DQA1, DQB1, TNF–α ha dificultado la asignación de asociaciones con la susceptibilidad a LEG.8–11
En estudios previos de la población mexicana se encontró asociación del alelo HLA–DR3 con el LEG, mientras que se observó una frecuencia significativamente menor de alelos considerados como autóctonos (HLA–B8).12 Sin embargo, se desconoce la posible relevancia en la susceptibilidad a LEG de otros genes como el TNF–α, HSP70, receptor de células T, IL–10.13–16
Poco se sabe de los mecanismos que podrían determinar la susceptibilidad a LEG en relación con estos loci pero es posible que los diferentes patrones de expresión de estos genes influyan significativamente en la regulación inmunológica. La hipótesis de los genes de susceptibilidad desconocidos que se encuentran en desequilibrio genético con estos loci se ha fortalecido en los últimos años, principalmente, por el hecho de que la asociación genética con LEG varía de acuerdo a las diferencias étnicas, lo que sugiere que la etnicidad es crucial en la predisposición a LEG.
Objetivo
Analizar la posible asociación de los genes y haplotipos de la región HLA, así como los genes de proteínas de choque térmico (HSP70–2) y los polimorfismos –238 y –308 del promotor de TNF–α con LEG en la población mexicana. Además se analizó la influencia de la etnicidad con base en los patrones de mezcla genética de pacientes con LEG y sujetos sanos.
Material y métodos
Pacientes
Se estudiaron 81 pacientes con diagnóstico de LEG que cumplieran con los criterios del Colegio Americano de Reumatología. Los pacientes fueron reclutados de la consulta externa del Departamento de Inmunología y Reumatología del Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán.
Controles mestizos mexicanos sanos
Se incluyeron 99 sujetos sanos mestizos mexicanos, todos ellos se analizaron desde los puntos de vista clínico, serológico, psicológico y nutricional para establecer la historia de padecimientos autoinmunes o infecciosos. Mediante cuestionario directo se obtuvieron datos para establecer la historia familiar de enfermedades autoinmunes del tejido conjuntivo. Desde el punto de vista etnológico se consideraron mestizos aquellos mexicanos originarios con dos o más generaciones nacidas en México.7 En un trabajo anterior se caracterizó a la población mestiza de la ciudad de México al calcular la contribución relativa de cada componente ancestral según un modelo trihíbrido de la siguiente forma: 56% de genes indígenas, 41% de genes europeos y 3% de genes africanos.17
Obtención del material genético
Se aisló DNA genómico a partir de células mononucleares de sangre periférica utilizando la metodología descrita por Davis et al.18
Tipificación de genes HLA
La tipificación genética de HLA–DRB1 se realizó por el método de reacción en cadena de la polimerasa e hibridación con sondas específicas de secuencia (PCR–SSO). La amplificación se realizó por el método de PCR con 0.125 U/ul de Taq DNA polimerasa (Promega, Madison, WI). La tipificación de alta resolución por el método de hibridación con sondas específicas de secuencia (PCR–SSOP).
Los oligonucleótidos utilizados para la amplificación fueron DRBAMP–B para la región 3' del exón 2 en todos los casos, y DRBAMP–1, DR–BAMP–2, DRBAMP–3, DRBAMP–4, DRBAMP–B5 y DRBAMP–52 para la región 5' del exón 2 para cada grupo específico de amplificación. Los oligonucleótidos DQAAMP–A, DQAAMP–B, DQBAMP–A y DQBAMP–B fueron usados para la amplificación del gen DQ. Cada membrana donde se depositó la alícuota de DNA amplificado, se hibridó con sondas específicas para los distintos alelos de los loci HLA–DR, DQA y DQB. En el caso del locus HLA–DR se usó un total de 120 sondas y para el locus HLA–DQA se utilizaron 70 sondas y un número similar para el locus DQB. La información de los oligonucleótidos y de las sondas utilizadas fue tomada de las especificidades para DRB1, DRB3, DRB4, DRB5, DQA1 y DQB1 publicadas en el 12° Taller Internacional de Histocompatibilidad.
Genotipificación HSP70–2
Se caracterizó el polimorfismo en la posición 1267 del gen de la proteína de choque térmico (HSP70–2) mediante amplificación y corte con la endonucleasa Pst I. Para la amplificación de la región que contiene el sitio 1267 de este gen se utilizaron los siguientes oligonucleótidos; sentido (nucleótidos 1083–1102 dentro de la región codificante) 5'–CATCGACTTCTACA CGTCCA–3' y antisentido (nucleótidos 2180–2199 dentro de la región 3' no traducida para evitar la homología con HSP70–1) 5'–CAAAGTCCTTGAGTCCCAAC–3'. El DNA genómico fue amplificado por PCR. Los productos de la PCR fueron cortados por la endonucleasa de restricción Pst I (Pharmacia, Uppsala, Sweden) y posteriormente los fragmentos o patrón de restricción fueron analizados en electroforesis en geles de agarosa a 2% teñidos con bromuro de etidio.
Genotipificación TNF –238 y –308
La genotipificación para el polimorfismo –238G/A del TNF se hizo con fragmentos amplificados de PCR usando oligonucleótidos modificados (primer iniciador 5'–AAACAGACCA CAGACCTGGTC–3' y primer inverso 5'–CTCACACT CCCCATCCTCCCGGATC–3') que incluye un sitio de restricción para la enzima BamHI. El polimorfismo del TNF –308 se analizó con los iniciadores 5'–GAGCAATAGGTTTTGAG–CGCCAT–3'y5'–GGGACACACAAGCATCAAG–3' para crear un sitio de restricción para la enzima Ncol. Los fragmentos obtenidos fueron analizados por fototipificación en geles de agarosa a 2%, teñidos con bromuro de etidio.
Análisis estadístico
Se obtuvieron por conteo directo las frecuencias génicas y genotípicas del HLA–DRB1, DQA1, DQB1, TNF –238, TNF –308 y HSP70 y su equilibrio se demostró mediante el análisis de Hardy–Weinberg, excepto para el polimorfismo HSP70–2. Las diferencias entre pacientes y controles sanos se analizaron por el método de Mantel–Haenszel, por la prueba CX basada en tablas de contingencia de 2 x 2 con el programa estadístico EPIINFO. Los valores de p se corrigieron por el método de Bonferroni. Se consideraron valores estadísticamente significativos aquellos con una p igual o menor a 0.05. El intervalo de confianza de 95% (IC95%) se estimó como el índice de probabilidad (RM) según el método de Woolf.
El desequilibrio genético (D = valor de desequilibrio; D'= valor de desequilibrio normalizado) se calculó por medio del programa estadístico ARLEQUIN v 1.1.
Las contribuciones relativas de los componentes ancestrales africano, europeo e indígena, se calcularon con frecuencias del HLA–DRB1 para el grupo de pacientes con LES y para el grupo control utilizando como datos de referencia las frecuencias de población subsahariana19 de Murcia, España y de los indígenas mazatecos utilizando el método de Robert & Hiorns con el programa Leadmix.20
Resultado
En el cuadro I se muestran las frecuencias génicas de los alelos del locus HLA–DRB1 en pacientes con LEG y en controles sanos. Se observó incremento en la frecuencia del alelo DRB1*0301 en los pacientes con LEG al compararse con los controles sanos (p=0.03, RM=2.63, IC 95%=1.08–6.53). En contraposición, la frecuencia del alelo DRB1*0802 fue significativamente menor en el grupo de pacientes con LEG (p=0.03, RM=0.45, IC95%=0.21–0.95).

El cuadro II muestra los alelos HLA– DQA1 y DQB1 más frecuentes, encontrando mayor frecuencia del alelo DQB1*0501 en pacientes con LEG (p=0.004, RM=4.02, IC 95%=1.46–11.64).

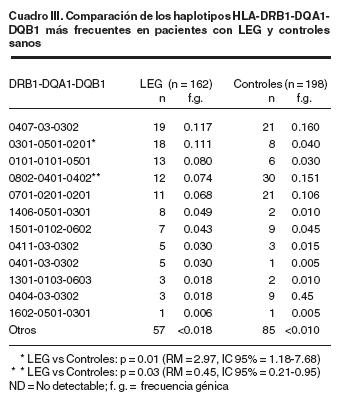
El cuadro III muestra, en orden de frecuencia, los haplotipos clase II del MHC en pacientes con LEG y controles sanos. Como muestra, los haplotipos DRB1*0407–DQA1*03–DQB1*0302, DRB1*0301–DQA1*0501–DQB1*0201, DRB1*1501–DQA1*0102–DQB1*0602 y DRB1*01 01–DQA1 *01 01 –DQB1*0501 fueron más frecuentes en los pacientes con LEG, mientras que en el grupo control los haplotipos más frecuentes fueron DRB1 *0802–DQA1 *0401 –DQB1 *402 y DRB1 *0701 –DQA1 *0201 –DQB1 *0201 con una frecuencia de 0.127 y 0.106 respectivamente. El análisis de los genes de clase III mostró que el genotipo heterocigoto HSP70–2 A/B fue el más común en las dos poblaciones (83% en LEG, 72% en controles) El genotipo homocigoto HSP70–2 B/B no se detectó en los pacientes con LEG y se observó con mayor frecuencia el genotipo B/B en los controles (5%) (Cuadro IV). El análisis del polimorfismo del promotor de TNF–α en las posiciones –238 y –308 mostró incremento en la frecuencia del genotipo TNF –238 G/A en los pacientes con LEG en comparación con los controles (p = 0.03; RM = 4.77; IC 95% = 1.1–23.2). Por otro lado, se encontró aumento significativo en la frecuencia génica del alelo TNF–α –238 en los pacientes con LEG al comparar con controles (p = 0.02; RM = 3.62; IC 95% = 1.1–11.9) (Cuadro V). Mediante el cálculo de delta (D) se detectaron ciertos patrones de desequilibrio genético entre alelos del promotor de TNF–α y alelos HLA–DR en el grupo de pacientes: TNF–α–DRB1*1401 (D = 0.84; D'= 1.0; p = 0.015); TNF–α DRB1*0301 (D = 1.38; D'= 0.41; p = 0.042); TNF2– DRB1*1106 (D = 0.83; D'= 0.45; p = 0.02); TNF2– DRB1*1406 (D = 0.83; D'= 0.45; p = 0.02). TNF2– DRB1*0301 (D = 1.3; D'= 0.4; p = 0.042). Por otro lado, en el grupo control se observó desequilibrio genético entre TNF2–DRB1*0802 (D = 1.4; D'= 0.61; p = 0.004); TNF2–DRB1*1302(D = 0.81; D'=0.30,p = 0.005)TNF–α–DRB1*0404 (D = 0.7; D'= 0.29, p = 0.03).

Además se estimó el grado de mestizaje para conocer la contribución genética europea tanto en pacientes con LEG como en los controles, encontrando que los pacientes cuentan con una aportación génica aproximada: africana 0%, europea 65% e indígena 35%, y los controles: africana 3%, europea 52% e indígena 45%.
Discusión
Los resultados de este estudio confirman el papel del DRB1*0301 y del haplotipo DRB1*0301–DQA1*0501–DQB1*0201 en europeos y parcialmente en africanos.19 Por otra parte, también confirman el papel de TNF y HSP70 en la susceptibilidad genética a LEG en europeos.
Es interesante que los pacientes mexicanos con LEG tengan contribución europea pero no africana pues la prevalencia e incidencia de LEG en africanos es ocho veces mayor que en caucásicos.21 Mas aún, la incidencia de LEG en mexicanos mestizos es considerablemente superior que en caucásicos.22 Esta aparente paradoja sugiere que el mestizaje participa directamente en la susceptibilidad genética a LEG, por lo que no sorprende que el haplotipo más característico de la población amerindia (DRB1*0407–DQA1*0301–DQB1*0302) esté disminuido de forma significativa en el grupo de pacientes con respecto a los sanos.
En conclusión, este estudio confirma que los genes del MHC ubicados en el cromosoma 6 contribuyen directamente al desarrollo de LEG; sin embargo para entender mejor el papel del mestizaje en la fisiopatogenia de esta enfermedad, es necesario realizar estimaciones del mestizaje con marcadores de otros cromosomas, como el uso de Short Tandem Repeats (STR) y Single Nucleotide Tandem Polimorphisms (SNP) en los diversos grupos étnicos que permitan estudiar el mestizaje, por ejemplo en afroamericanos y orientales.
Agradecimientos
Al apoyo otorgado por el Consejo Nacional de Ciencia y Tecnología (CONACyT) No. 45129.
Referencias
1. Tan EM, Cohen AS, Fries JF, Masi AT, McShane DJ, Rothfield NF, et al. The 1982 revised criteria for the classification of systemic lupus erythematosus. Arthritis Rheum 1982; 25:1271–1277. [ Links ]
2. Hochberg MC. Updating the American College of Rheumatology revised criteria for the classification of systemic lupus erythematosus. Arthritis Rheum 1997; 40:1725. [ Links ]
3. Schur PH. Genetics of systemic lupus erythematosus. Lupus. 1995; 4:425–437. [ Links ]
4. Harley JB, Moser KL, Gaffney PM, Behrens TW. The genetics of human systemic lupus erythematosus. Curr Opin Immunol 1998; 10:690–696. [ Links ]
5. Hartung K, Baur MP, Coldewey R, Fricke M, Kalden JR, Lakomek HJ, et al. Major histocompatibility complex haplotypes and complement C4 alleles in systemic lupus erythematosus. Results of a multicenter study. J Clin Invest 1992; 90:1346–1351. [ Links ]
6. Yao Z, Kimura A, Hartung K, Haas PJ, Volgger A, Brunnler G, et al. Polymorphism of the DQA1 promoter region (QAP) and DRB1, QAP, DQA1, DQB1 haplotypes in systemic lupus erythematosus. SLE Study Group members. Immunogenetics 1993; 38:421–429. [ Links ]
7. Burgos–Vargas R, Granados J. Ankylosing spondylitis and related disease in the Mexican mestizo. Spine 1990; 4:161–167. [ Links ]
8. Rood MJ, van Krugten MV, Zanelli E, van der Linden MW, Keijsers V, Schreuder GM, et al. TNF–308A and HLA–DR3 alleles contribute independently to susceptibility to systemic lupus erythematosus. Arthritis Rheum 2000; 43129–134. [ Links ]
9. Howard PF, Hochberg MC, Bias WB, Arnett FC Jr, McLean RH. Relationship between C4 null genes, HLA–D region antigens, and genetic susceptibility to systemic lupus erythematosus in Caucasian and black Americans Am J Med 1986; 81:187–193. [ Links ]
10. Fielder AH, Walport MJ, Batchelor JR, Rynes RI, Black CM, Dodi IA, et al. Family study of the major histocompatibility complex in patients with systemic lupus erythematosus: importance of null alleles of C4A and C4B in determining disease susceptibility. Br Med J 1983; 286:425–428. [ Links ]
11. Dunckley H, Gatenby PA, Hawkins B, Naito S, Serjeantson SW. Deficiency of C4A is a genetic determinant of systemic lupus erythematosus in three ethnic groups. J Immunogenet 1987; 14:209–218. [ Links ]
12. Granados J, Vargas–Alarcon G, Andrade F, Melin–Aldana H, Alcocer–Varela J, Alarcon–Segovia D. The role of HLA–DR alleles and complotypes through the ethnic barrier in systemic lupus erythematosus in Mexicans. Lupus 1996; 5:184–189. [ Links ]
13. Rudwaleit M, Tikly M, Khamashta M, Gibson K, Klinke J, Hughes G, et al. Interethnic differences in the association of tumor necrosis factor promoter polymorphisms with systemic lupus erythematosus. J Rheumatol. 1996; 23:1725–1728. [ Links ]
14. Jarjour W, Reed AM, Gauthier J, Hunt S 3rd, Winfield JB. The 8.5–kb PstI allele of the stress protein gene, Hsp70–2: an independent risk factor for systemic lupus erythematosus in African Americans? Hum Immunol 1996; 45:59–63. [ Links ]
15. Theofilopoulos AN. The basis of autoimmunity. Genetic predisposition. Immunol Today 1995; 16:150–159. [ Links ]
16. Brennan P, Hajeer A, Ong KR, Worthington J, John S, Thomson W, et al. Allelic markers close to prolactin are associated with HLA–DRB1 susceptibility alleles among women with rheumatoid arthritis and systemic lupus erythematosus. Arthritis Rheum 1997; 40:1383–1386. [ Links ]
17. Lisker R, Perez–Briceno R, Granados J, Babinsky V. Gene frequencies and admixture estimates in the state of Puebla, Mexico. Am J Phys Anthropol 1988; 76:331–335. [ Links ]
18. Davis RW, Thomas M, Cameron J, St John TP, Scherer S, Padgett RA. Rapid DNA isolations for enzymatic and hybridization analysis. Methods Enzymol. 1980; 65:404–411. [ Links ]
19. http://www.ncbi.nlm.nih.gov/mhc [ Links ]
20. Wang J. Maximum likelihood estimation of admixture proportions from genetic data. Genetics 2003; 64:747–765. [ Links ]
21. Molokhia M, Hoggart C, Patrick AL, McKeigue P. Risk for rheumatic disease in relation to ethnicity and admixture. Arthritis Res 2002 ;(2):115–125. [ Links ]
22. Alarcón–Segovia D, Alarcón–Riquelme ME, Cardiel MH, Caeiro F, Massardo L, Villa AR, et al. Grupo Latinoamericano de Estudio del Lupus Eritematoso (GLADEL). Familial aggregation of systemic lupus erythematosus, rehumatoid arthritis, and other autoimmune diseases in 1177 lupus patients from the GLADEL cohort. Arthritis Rheum 2005; 52:1138.1147. [ Links ]














