Services on Demand
Journal
Article
Indicators
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Gaceta médica de México
On-line version ISSN 2696-1288Print version ISSN 0016-3813
Gac. Méd. Méx vol.141 n.1 Ciudad de México Jan./Feb. 2005
Artículos de revisión
La vía de CD1 y la activación de células T NK hacia los antígenos glicolipídicos de Mycobacterium tuberculosis
CD1 pathway and NK T Cell activation to glycolipid antigens from Mycobacterium
Isabel Sada–Ovalle,* Luis Torre–Bouscoulet,* María del Carmen Jiménez–Martínez,* Salvador Martínez–Cairo,** Edgar Zenteno,*** Ricardo Lascurain***
* Departamento de Bioquímica. Unidad de Investigación. Instituto Nacional de Enfermedades Respiratorias. Secretaría de Salud. México, D.F.
** Coordinación de Investigación en Salud. Centro Médico Nacional Siglo XXI, IMSS
*** Departamento de Bioquímica, Fac. Medicina, UNAM.
Correspondencia y solicitud de sobretiros:
Isabel Sada–Ovalle.
Departamento de Bioquímica. Unidad de Investigación. Instituto Nacional de Enfermedades Respiratorias, Calzada de Tlalpan 4502,
14080, México D. F.
Fax: (55) 56–65–46–23 Tel. (55) 56–65–43–79.
e–mail: i_sadamx@yahoo.com.mx
Recepción versión modificada: 16 de diciembre de 2004
Aceptación: 08 de enero de 2004
Resumen
El objetivo de esta revisión es analizar el estado actual de nuestro conocimiento sobre las moléculas de superficie celular involucradas en la presentación de antígenos glicolipídicos, denominadas familia CD1. Estas proteínas constituyen la tercera clase de moléculas presentadoras de antígeno. Las proteínas CD 1 controlan diversas funciones inmunes importantes en la defensa del hospedero contra las infecciones microbianas. En años recientes estas proteínas han sido involucradas en la generación de una respuesta inmune celular contra Mycobacterium tuberculosis. Aquí, nosotros analizaremos aspectos relevantes acerca de las proteínas CD 1 y las células T específicas para antígenos glicolipídicos.
Palabras clave: CD1, linfocitos T CD8+, linfocitos T NK, Mycobacterium tuberculosis
Summary
The aim of this review is to analyze the current state of our knowledge about cell surface molecules involved in glycolipid antigen presentation, named CD1 family. These proteins constitute a third class of antigen–presenting molecules. CD 1 molecules develop diverse important immune functions in host defenses against microbial infections. In recent years these proteins have been involved in the generation of cell–mediated immune response against Mycobacterium tuberculosis. Here, we analyze relevant roles of CD1 proteins and glycolipid antigen–specific T cells.
Key words: CD1, CD8+ T cells, NK T cells, Mycobacterium tuberculosis
Introducción
La tuberculosis pulmonar es causada por Mycobacterium tuberculosis (Mtb), un patógeno intracelular que infecta a un tercio de la población mundial.1 La inmunidad mediada por células es una respuesta que aporta beneficio al hospedero, sin embargo, los mecanismos a través de los cuales algunas subpoblaciones de linfocitos T participan en el control de esta enfermedad no han sido claramente establecidos. Informes recientes en la literatura muestran un papel importante de los linfocitos T en la inmunidad celular, en especial aquellos linfocitos T CD8+ que reconocen y matan macrófagos alveolares infectados.2 Los linfocitos T CD8+ reconocen antígenos peptídicos presentados por moléculas clásicas de clase I pertenecientes al sistema HLA (Human Leucocyte Antigen por sus siglas en inglés); aunque también ha sido descrito que pueden reconocer antígenos de naturaleza no proteica como la lipoarabinomanana (LAM) y los manósidos de fosfatidilinositol;3 los antígenos derivados de Mtb. Interesantemente estos linfocitos T CD8+ con especificidad por glicolípidos micobacterianos pueden lisar macrófagos infectados con Mtb, incluso se ha descrito que algunos de sus efectos bactericidas son mediados por granulisina.3
Presentación de antígenos proteicos y la familia de moléculas CD1
Se han descrito dos mecanismos para la presentación y reconocimiento de antígenos proteicos por las dos subpoblaciones principales de linfocitos T; los linfocitos T CD4+ (cooperadores) y los T CD8+ (citotóxicos). Los linfocitos T CD4+ reconocen antígenos extracelulares provenientes de proteínas propias o extrañas que son fagocitadas y presentadas en la superficie celular, en complejo con moléculas de clase II del sistema HLA. Las moléculas HLA de clase II son sintetizadas en el retículo endoplásmico (RE) y transferidas a endosomas, los cuales se fusionan con los fagolisosomas donde se encuentra el antígeno proteico. En estos compartimientos, diversos procesos degradan al antígeno proteico en pequeños péptidos permitiendo su adecuada asociación con las moléculas HLA de clase II.4–7
Los linfocitos T CD8+ reconocen también antígenos proteicos pero de origen endógeno, como proteínas celulares defectuosas, proteínas virales, neoantígenos tumorales, etc. Estas proteínas son procesadas en el complejo proteasoma y los productos peptídicos son transferidos al RE en donde se unen a moléculas HLA de clase I, posteriormente son transportados en endosomas y presentados en la superficie celular.6,7 Sin embargo, la acumulación de evidencia experimental de que los lípidos y/o glicolípidos (propios o derivados de microorganismos) son reconocidos por algunas subpoblaciones linfocitarias, originó las incógnitas sobre la vía de procesamiento y cómo es que estas subpoblaciones de linfocitos dirigen una respuesta inmune esencial en diversas patologías.
Recientemente, se ha estudiado y descrito una vía diferente para el procesamiento y presentación de lípidos y glicolípidos de diferentes orígenes, destacando aquellos antígenos derivados de la pared celular de micobacterias entre los que se incluye a: monomicolato de glucosa, ácidos micólicos y lipoarabinomanana (LAM).8,9 Esta vía de procesamiento de antígenos glicolipídicos, denominada vía de CD1, está compuesta por un grupo heterogéneo de moléculas que son glicoproteínas transmembranales unidas a la β–2 microglobulina ((3–2m) y por proteínas adaptadoras heterotetraméricas denominadas AP1, AP2, AP3 y AP4.10 La familia de proteínas CD1, está codificada por genes distantes de los pertenecientes al sistema HLA, se localizan en el cromosoma 1 y comprende 5 genes (isotipos) conservados en diferentes especies de mamíferos y son clasificados en 2 grupos en base a su secuencia homóloga.11,12 El grupo 1 está integrado por CD1 a, CD1 b, CD1 c y CD1 e, y el grupo 2 sólo incluye a CD1 d, la cual se encuentra en todas las especies estudiadas actualmente (Figura 1). Estos dos grupos no se encuentran estrechamente relacionados, lo que sugiere que diferentes isoformas pueden tener diferentes funciones. Las proteínas CD1 tienen un tallo citoplásmico (no ha sido identificado en CD1 a) que contiene la secuencia YXXZ, donde Y es tirosina, X corresponde a cualquier aminoácido y Z es un grupo heterogéneo de aminoácidos hidrofóbicos.13,14 Esta secuencia en las moléculas CD1 facilita su localización en los diferentes compartimentos endosomales (Figura 2).15–17


Estructura de las proteínas CD1
Los antígenos presentados por CD1 son moléculas glicolipídicas anfipáticas, esto es, consisten de una cabeza hidrofílica con grupos polares y una cola hidrofóbica compuesta por una o dos cadenas hidrocarbonadas.18 La presentación de antígenos glicolipídicos por moléculas CD1 a linfocitos T (también llamada restricción antigénica) muestra semejanzas con el reconocimiento antigénico de los complejos péptido/HLA. Las moléculas CD1, al igual que las moléculas clase I del sistema HLA, son heterodímeros constituidos por una cadena a asociada a la (β2m, asimismo, estas moléculas se sintetizan a nivel del RE.14 Las cinco isoformas de CD1 (humano) se asocian de manera no covalente a la β2m aunque con diferentes afinidades; la interacción más fuerte se ha identificado en la molécula CD1 b–β2m, la cual es resistente a la disolución ocasionada con pH de 3.0.17 La cadena α se pliega en tres dominios (α1 , α2 y α3), los cuales son conocidos como cadenas pesadas por su parecido con las cadenas pesadas de las moléculas HLA clase I. Los dominios α1, α2 y α3 se extienden hacia el lumen del RE, mientras que una pequeña porción de aproximadamente 10 aminoácidos se orienta a través de la membrana del RE hacia el citoplasma formando el tallo citoplásmico.10 El plegamiento y la asociación de la molécula CD1 con la β2m más un glicolípido endógeno (habitualmente fosfatidilinositol, residente del RE) es dirigida por las chaperonas conocidas como Calnexina y Calreticulina. El surco de unión para el antígeno se constituye por la proximidad de los residuos de aminoácidos hidrofóbicos de los dominios α1 y α2 formando 4 canales hidrofóbicos no polares denominados A', F' T' y C'.19 Sin embargo, estesurco de unión para el antígeno en la proteína CD1 es más profundo y estrecho que el descrito en las moléculas HLA. Este surco de unión antigénico está compuesto por láminas p plegadas en la base y estructuras a hélices en los bordes, semejante en su aspecto físico al surco de unión antigénico encontrado en las moléculas clase I y II del sistema HLA.20
Se ha propuesto un modelo que predice el anclaje de los glicolípidos dentro del surco de unión en las moléculas CD1, de modo que los lípidos con cadenas alkil cortas ocupan los canales C y A' dejando libres a los T' y F' y la cabeza polar queda localizada fuera de la abertura del surco de unión, en donde interactúa con el receptor del linfocito T (TCR).17 Análisis realizados en líneas celulares humanas CD1b+ portando mutaciones puntuales permitieron conocer que el TCR interacciona con las α–hélices de la molécula CD1 y con el propio antígeno glicolipídico.18,21
Importancia del CD1 en macrófagos infectados por micobacterias
Durante la infección por Mtb, el macrófago ( ) es la célula hospedera de la micobacteria, localizándose especialmente en el compartimiento fagosomal temprano, el cual no es acidificado debido a que la micobacteria inactiva la bomba de protones. Esto retarda el proceso de maduración fagosomal permitiendo a la micobacteria residir indefinidamente dentro del
) es la célula hospedera de la micobacteria, localizándose especialmente en el compartimiento fagosomal temprano, el cual no es acidificado debido a que la micobacteria inactiva la bomba de protones. Esto retarda el proceso de maduración fagosomal permitiendo a la micobacteria residir indefinidamente dentro del  .22,23 En otras condiciones el material fagocitado por el macrófago, es transportado a través de una serie de eventos de fusión y fisión del fagosoma con la vía endosomal/lisosomal en donde este material es degradado.24,25 Sin embargo, en el caso de Mtb, observaciones experimentales sugieren que los sulfátidos micobacterianos tienen la capacidad de inhibir la fusión fagolisosomal.22,23 Por lo tanto, el impedimento en la maduración del fagosoma es un proceso activo desarrollado por la micobacteria como un mecanismo de evasión de la función bactericida del
.22,23 En otras condiciones el material fagocitado por el macrófago, es transportado a través de una serie de eventos de fusión y fisión del fagosoma con la vía endosomal/lisosomal en donde este material es degradado.24,25 Sin embargo, en el caso de Mtb, observaciones experimentales sugieren que los sulfátidos micobacterianos tienen la capacidad de inhibir la fusión fagolisosomal.22,23 Por lo tanto, el impedimento en la maduración del fagosoma es un proceso activo desarrollado por la micobacteria como un mecanismo de evasión de la función bactericida del  que correlaciona con la sobrevivencia del bacilo.26 Asimismo, la residencia de Mtb en el fagosoma induce en la membrana plasmática de este organelo, la expresión de moléculas características de los endosomas tempranos y tardíos como transferrina, rab5 y el receptor de transferrina, los cuales son utilizados por la Mtb para la obtención de nutrimentos del citosol.27–30
que correlaciona con la sobrevivencia del bacilo.26 Asimismo, la residencia de Mtb en el fagosoma induce en la membrana plasmática de este organelo, la expresión de moléculas características de los endosomas tempranos y tardíos como transferrina, rab5 y el receptor de transferrina, los cuales son utilizados por la Mtb para la obtención de nutrimentos del citosol.27–30
La envoltura celular de la micobacteria está compuesta por dos estructuras principales: una membrana plasmática y una pared celular a su vez constituida por peptidoglicanos y arabinogalactanos unidos por enlaces covalentes.31 Interesantemente, algunos glicolípidos de la pared celular de la micobacteria son exportados desde el fagosoma inmaduro, movilizándose en el interior de las células infectadas para así ser transportados a las células vecinas por un proceso de exocitosis. De esta forma, los antígenos glicolipídicos micobacterianos entran en contacto con las moléculas CD1.32–34 Algunas de las moléculas CD1, principalmente CD1 b han sido localizadas en compartimientos endosomales del complejo multivesicular denominado MIIC (MHC class II peptide–loading compartment por sus siglas en inglés), el cual está formado por extensas invaginaciones membranales donde se encuentran moléculas clase II del sistema HLA.35,36 Es posible que la asociación de los antígenos glicolipídicos micobacterianos con moléculas CD1 en los MIIC se realice en un sitio especial para el procesamiento y carga de antígenos.37–39
Distribución celular de las moléculas CD1
Las isoformas de CD1 se localizan diferencialmente en los compartimentos endosomales:
CD1 a. Se expresa en células dendríticas derivadas de monocitos así como en células de Langerhans. La molécula es expresada en la superficie celular, sin embargo, algunos estudios de localización intracelular han mostrado que CD1a no se encuentra en los endosomas tardíos, aunque se ha encontrado en vesículas que contienen clatrina, lo cual sugiere que su entrada a los compartimentos intracelulares es mediante clatrina. La distribución intracelular de CD1 a se debe a que no posee una secuencia basada en tirosina en su tallo citoplásmico que le permite tener diferentes localizaciones endosomales.40–43
CD1b. Las células dendríticas derivadas de monocitos son la principal subpoblación celular que la expresa en la membrana celular. La molécula CD1b después de ser sintetizada en el retículo endoplásmico es inicialmente transportada a la membrana celular debido a la unión con las proteínas AP2 y AP3 y posteriormente internalizada por un mecanismo dependiente de clatrina, así que puede ser encontrada en los endosomas tardíos y en los MIIC.14,44–46
CD1c. Es expresada en la membrana de células dendríticas y puede ser localizada en compartimentos de la red endosomal junto con receptores de transferrina. Se ha mostrado que la proteína CD1 c es capaz de activar a linfocitos T autorreactivos así como facilitar la presentación de fosfolípidos micobacterianos a linfocitos T.47
CD1d. Esta molécula es también internalizada por un mecanismo dependiente de clatrina e interactúa con la proteína AP3. Puede localizarse en endosomas tardíos y en los MIIC, se ha descrito que CD1d puede presentar otra vía de tráfico intracelular asociada a la cadena Ii en el ratón o a la molécula clase II en el humano, a través de la cual se integra como parte de la red endosomal.48–51
CD1e. Ha sido identificada solamente como una proteína expresada a nivel intracelular y no ha sido asociada a presentación o activación de linfocitos T.52
Procesamiento de antígenos glicolipídicos
Las células dendríticas que expresan CD1 pueden también albergara Mtb, sin embargo, son los  principal sitio donde esta bacteria reside. Cabe mencionar que los
principal sitio donde esta bacteria reside. Cabe mencionar que los  prácticamente no expresan moléculas CD1, más aún, la expresión de CD1b es regulada negativamente por Mtb.31,53,54 Por lo tanto, la exocitosis de vesículas que contienen antígenos micobacterianos y su captura por células presentadoras de antígeno (CPA) vecinas, quizá permite la inducción de una respuesta inmune aun cuando la capacidad de las CPA–CD1+ infectadas por Mtb sea ineficiente. La captura de LAM por las CPA para su presentación a linfocitos T, es un proceso mediado por el receptor de manosa, sin embargo, este receptor no participa en el procesamiento intracelular de los antígenos micobacterianos.35 Se ha sugerido que la captura de los antígenos glicolipídicos micobacterianos por las CPA–CD1 + y su distribución en los diferentes compartimientos endosomales mediante un muestreo, es un requisito para hacer más eficiente la presentación antigénica. En muchos de los casos estudiados, la presentación de antígenos glicolipídicos por las moléculas CD1b humana y CD1d murina es inhibida por substancias como cloroquina y concanamicina A cuya función es evitar la acidificación endosomal sugiriendo que hay una asociación entre los compartimentos ácidos y el procesamiento de antígenos glicolipídicos.14 Esta hipótesis se fundamenta en la evidencia de que las moléculas CD1 humanas han sido localizadas en diferentes compartimientos de la vía endosomal. Un aspecto importante sería conocer cuáles son las características moleculares de estos glicolípidos que determinan su entrada a la vía endosomal.
prácticamente no expresan moléculas CD1, más aún, la expresión de CD1b es regulada negativamente por Mtb.31,53,54 Por lo tanto, la exocitosis de vesículas que contienen antígenos micobacterianos y su captura por células presentadoras de antígeno (CPA) vecinas, quizá permite la inducción de una respuesta inmune aun cuando la capacidad de las CPA–CD1+ infectadas por Mtb sea ineficiente. La captura de LAM por las CPA para su presentación a linfocitos T, es un proceso mediado por el receptor de manosa, sin embargo, este receptor no participa en el procesamiento intracelular de los antígenos micobacterianos.35 Se ha sugerido que la captura de los antígenos glicolipídicos micobacterianos por las CPA–CD1 + y su distribución en los diferentes compartimientos endosomales mediante un muestreo, es un requisito para hacer más eficiente la presentación antigénica. En muchos de los casos estudiados, la presentación de antígenos glicolipídicos por las moléculas CD1b humana y CD1d murina es inhibida por substancias como cloroquina y concanamicina A cuya función es evitar la acidificación endosomal sugiriendo que hay una asociación entre los compartimentos ácidos y el procesamiento de antígenos glicolipídicos.14 Esta hipótesis se fundamenta en la evidencia de que las moléculas CD1 humanas han sido localizadas en diferentes compartimientos de la vía endosomal. Un aspecto importante sería conocer cuáles son las características moleculares de estos glicolípidos que determinan su entrada a la vía endosomal.
A la fecha se sabe que los antígenos de origen glicolipídico, tanto exógenos como endógenos, pueden ser presentados por moléculas CD1.14,53 Para el procesamiento de antígeno, inicialmente fueron descritos dos mecanismos: uno de ellos postula que los antígenos glicolipídicos con grandes cadenas alkil (~ 80 carbonos) requieren la ruptura enzimática de enlaces covalentes liberando pequeños fragmentos antigénicos que pueden ser reconocidos por las células T;18 en el otro mecanismo se propone que el antígeno sufre algunas modificaciones estructurales dependientes de pH.21 Un tercer mecanismo recientemente descrito, sugiere que las cadenas alkil de algunos antígenos glicolipídicos pueden plegarse dentro del surco de unión en la molécula CD1.54–57
Para la presentación de antígenos glicolipídicos, los estudios sobre la vía de presentación se han realizado con la molécula CD1 b, la cual recién sintetizada en el RE se asocia a un glicolípido endógeno y entonces viaja a la superficie celular. Posteriormente la molécula CD1 b se internaliza dentro de compartimentos endosomales en donde los lípidos endógenos son removidos y un glicolípido exógeno es colocado. CD1 es regresada a la superficie celular y ahora potencialmente presentará glicolípidos extraños (Figura 3).44–47

Reconocimiento de antígenos micobacterianos por linfocitos T CD8+ restringidos a CD1
La subpoblación de linfocitos T que más ha sido estudiada por su asociación a las moléculas CD1 son los linfocitos T tipo NK; fenotípicamente estos linfocitos pueden ser CD4+, CD8+, CD4–CD8– (dobles negativos) y TCRγδ+.58 La primera descripción que se hizo de los linfocitos T tipo NK fue la de Fowlkes B.J. en 1987, al estudiar timocitos en un modelo murino.59 Esta población de linfocitos puede ser identificada por la expresión de moléculas de superficie celular características de las células NK (NK1.1 y Ly–49 en el ratón y CD57 en el humano entre otros), y por su escasa capacidad proliferativa.60 Otra característica importante es su repertorio inmunológico restringido, también llamado canónico, en donde la mayoría de los linfocitos T tipo NK humanos expresan una cadena a que no varía (Vα24–JαQ humana ó Vα14–Jα281 murina).Este TCR reconoce una familia conservada de glicolípidos, entre los que se encuentra el glicosilfosfatidilinositol (GPI) el cual puede ser parte de ciertos antígenos bacterianos, aunque la naturaleza de estos glicolípidos no ha sido claramente establecida.61 Estudios más recientes sobre esta subpoblación celular, han establecido que prácticamente todos los linfocitos T tipo NK (humanos y murinos) reconocen la α–galactosilceramida, un glicolípido que fue obtenido de las esponjas marinas durante la búsqueda de sustancias que pudieran prevenir metástasis tumorales.62–64 Entre los glicolípidos derivados de la pared celular de Mtb, CD1 b presenta ácidos micólicos, monomicolato de glucosa, lipoarabinomanana (LAM) y gangliósidos;54,57CD1c presentaf osfoisoprenoides; CD1d presenta glicofosfolípidos (manósido de fosfatidilinositol) y glicoesfingolípidos(gangliósidoGM1 y α–galactosilceramida) y CD1 a presenta algunos sulfátidos.14
La respuesta inmune ante Mtb es dependiente de células T y comprende no solamente a los linfocitos T CD4+ y CD8+ convencionales, sino que incluye de manera especial a aquellos linfocitos que reconocen glicolípidos presentados por CD1.65–67 Estos linfocitos T que pueden reconocer glicolípidos presentados por moléculas CD1, producen citocinas como: IFN–γ, IL–4 y TNF–α, además de presentar importante actividad citolítica.68–70 El IFN–γ muestra un efecto sinérgico con el TNF–α para activar a los MO, lo cual lleva a la muerte de las mico bacterias. La activación de los linfocitos T tipo NK induce la activación de células del sistema inmune innato como las células "natural killer" (NK) y las células dendríticas, así como la expresión de moléculas co–estimuladoras. Por otra parte, esta subpoblación de linfocitos puede dirigir la inmunidad mediada por células contra bacterias intracelulares que requieren la lisis de células infectadas así como la muerte del patógeno.71 Los linfocitos T tipo NK activados pueden por sí mismos lisar a sus células blanco por tres vías principales: primero, citotoxicidad por exocitosis de gránulos que contienen perforina y granzima; segundo, citotoxicidad mediada a través del sistema Fas/FasL y tercero, actividad microbicida directa, liberando gránulos que contienen granulisina.72,73 Además se ha propuesto que esta subpoblación celular puede contribuir a la inmunidad antimicobacteriana gracias a su capacidad para activar a las células NK y la producción de citocinas. En la figura 1 podemos observar un macrófago alveolar infectado por Mtb en interacción con un linfocito T CD8+ tipo NK (que coexpresa la molécula CD57, marcador característico de células NK). Los linfocitos T CD8+ tipo NK tienen la capacidad de reconocer antígenos lipídicos presentados por moléculas CD1 y una vez que son activados puede ejercer acciones bactericidas por diferentes mecanismos como citotoxicidad mediada por gránulos (perforina, granzimas y granulisina) o citotoxicidad directa a través del sistema Fas–FasL (Figura 4).
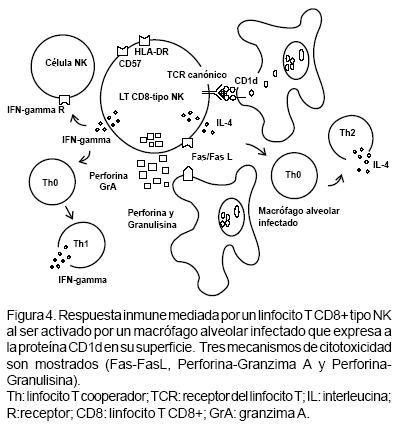
Conclusiones
La capacidad de Mtb para persistir en los compartimentos endosomales y detener su maduración en estadios tempranos, es quizá, una de las cualidades más relevantes del patógeno, porque facilita su supervivencia dentro del M(|). El punto central para la resolución de la enfermedad es generar una respuesta inmune capaz de activar al mayor número de macrófagos por medio de citocinas, junto con la eficiente actividad citolítica de los linfocitos T CD8+. Estas acciones son realizadas en conjunto por las principales poblaciones implicadas: linfocitos T CD4+, linfocitos TCD8+, linfocitos Ty8y las células T restringidas a CD1. Aunque Mtb aparentemente reside dentro del fagosoma, se ha propuesto que tiene la capacidad de formar poros en la membrana fagosomal que le permiten tener contacto directo con las moléculas clase I y con las proteínas CD1. Sin embargo, Mtb también es un potente inductor de apoptosis para el M^ que lo hospeda, de tal modo que los antígenos micobacterianos pueden ser capturados por otros macrófagos que ya se encuentren activados y presentados por las células CD1+ a los linfocitos T CD8+. El papel de los linfocitos T tipo NK restringidos a CD1 en tuberculosis aún no está completamente esclarecido, sin embargo, se puede asumir que participan de manera activa en la inmunidad protectora contra Mtb por dos mecanismos principales: el primero es su capacidad para producir citocinas como IFN–y mientras que el segundo es su actividad microbicida directa. Aún falta por determinar qué tan importante puede ser la contribución de estos linfocitos T CD8+ tipo NK restringidos a CD1 en la respuesta inmune contra patógenos como Mtb, por lo que es necesario realizar futuras investigaciones en este campo.
Agradecimiento: CONACYT 34814–M
Referencias
1. World Health Organization. The world health report 1999. Making a difference. Geneva: World Health Organization (1999). [ Links ]
2. Stenger S, Mazzccaro RJ, Uyemura K, Cho S, Barnes PF, Rosat JP, et al. Differential effects of cytolytic cells subsets on intracellular infection. Science 1997;276:1684–87. [ Links ]
3. Rosat JP, Grant EP, Beckman EM, Dascher CC, Sieling PA, Frederique D, et al. CD1–restricted microbial lipid antigen–specific recognition found in the CD8+ alpha beta T cell pool. J Immunol 1999;162:366–71. [ Links ]
4. Chapman HA. Endosomal proteolisis and class II MHC function. Curr Opin Immunol 1998;10:93–102. [ Links ]
5. Klein J, Sato A. The HLA system. First of two parts. N Engl J Med 2000;343:702–9. [ Links ]
6. Klein J, Sato A. The HLA system. Second of two parts. N Engl J Med 2000;343:782–6. [ Links ]
7. Pamer E, Creswell P. Mechanisms of MHC class I–restricted antigen processing. Annu Rev Immunol 1998;16:323–58. [ Links ]
8. Schaible UE, Kaufmann SH. CD1 molecules and CD1–dependent T cells in bacterial infections: a link from innate to acquired immunity?. Semin Immunol 2000;12:527–35. [ Links ]
9. Shamishiev A, Donda A, Carena I, Mori L, Kappos L, De Libero G. Self glycolipids as T–cell autoantigens. Eur J Immunol 1999;29:1667–75. [ Links ]
10. Moody DB, Porcelli SA. Intracellular pathways of CD1 antigen presentation. Nat Rev Immunol 2003;3:11–22. [ Links ]
11. Yu CY, Milstein C. A physical map linking the five CD1 human thymocyte differentiation antigen genes. EMBO J 1989;8:3727–32. [ Links ]
12. Albertson DG, Fishpool R, Sherrington P, Nacheva E, Milstein C. Sensitive and high resolution in situ hybridization to human chromosomes using biotin labelled probes: assignment of the human thymocyte Cd1 antigen genes to chromosome 1. EMBO J 1988;7:2801–5. [ Links ]
13. Park SH, Bendelac A. CD1–restricted T cell responses and microbial infection. Nature 2000;406:788–92. [ Links ]
14. Porcelli SA, Modlin RL. The CD1 system: antigen–presenting molecules for T cell recognition of lipids and glycolipids. Annu Rev immunol 1999;17:297–329. [ Links ]
15. Boehm M, Bonofacino JS. Genetic analyses of adaptin function from yeast to mammals. Gene 2002;286:175–86. [ Links ]
16. Sugita M, Cao X, Watts GF, Rogers RA, Bonifacino JS, Brenner MB. Failure of trafficking and antigen presentation by CD1 in AP–3–deficient cells. Immunity 2002;16:697–706. [ Links ]
17. Martin LH, Calabi F, Milstein C. Isolation of CD1 genes: a family of major histocompatibility complex–related differentiation antigens. Proc Natl Acad Sci USA 1986;83:9154–8. [ Links ]
18. Moody DB, Briken V, Cheng TY, Roura–Mir C, Guy MR, Geho D et al. Lipid length controls antigen entry into endosomal and nonendosomal pathways for Dc1b presentation. Nature Immunol 2002;3:435–42. [ Links ]
19. Zeng Z, Castano AR, Segelke BW, Stura EA, Peterson PA, Wilson IA. Crystal structure of mouse CD1: An MHC–like fold with a large hydrophobic binding groove. Science 1997;277:339–45. [ Links ]
20. Gadola SD, Zaccai NR, Harlos K, Sheperd D, Castro–Palomino JC, Ritter G, et al. Structure of human CD1b with bound ligands at 2.3 A, a maze for alkyl chains. Nat Immunol 2002;3:721–6. [ Links ]
21. Beckman EM, Porcelli SA, Morita CT, Behar SM, Furlong ST, Brenner MB. Recognition of a lipid antigen by CD1–restricted alpha beta+ T cells. Nature 1994;372:691–4. [ Links ]
22. Kaufmann SH. How con immunology contribute to the control of tuberculosis? Nat Rev Immunol 2001;1:20–30. [ Links ]
23. Clemens DL, Horwitz MA. Characterization of the Mycobacterium tuberculosis phagosome and evidence that phagosomal maturation is inhibited. J Exp Med 1995; 181:257–70. [ Links ]
24. Desjardins M, Huber LA, Parton RG, Griffiths G. Biogenesis of phagolysosomes proceeds through a sequential series of interactions with the endocytic apparatus. J Cell Biol 1994;124:677–88. [ Links ]
25. Russell DG, Dant J, Sturgill–Koszycki S. Mycobacterium–avium and Mycobacterium tuberculosis–containing vacuoles are dynamic, fusion–competent vesicles that are accessible to glycosphingolipids from the host cell plasmalemma. J Immunol 1996; 156:4764–73. [ Links ]
26. Armstrong JA, Hart PD. Phagosome–lysosome interactions in cultured macrophages infected with virulent tubercle bacilli. Reversal of the usual nonfusion pattern and observations on bacterial survival. J Exp Med 1975;142:1–16. [ Links ]
27. Xu S, Cooper A, Sturgill–Koszycki S, van Heyningen T, Chatterjee D, Orme I, et al. Intracellular trafficking in Mycobacterium tuberculosis and Mycobacterium avium–infected macrophages. J Immunol 1994;153:2568–78. [ Links ]
28. Ferrari G, Langen H, Naito M, Pieters J. A coat protein on phagosomes envolved in the intracellular survival of mycobacteria. Cell 1999;97:435–47. [ Links ]
29. Clemens DL, Horwitz MA. The Mycobacterium tuberculosis phagosome interacts with early endosomes and is accessible to exogenously administered transferrin. J Exp Med 1996;184:1349–55. [ Links ]
30. Sturgill–Koszycki S, Schaible UE, Russell DG. Mycobacterium–containing phagosomes are accessible to early endosomes and reflect a transitional state in normal phagosome biogenesis. EMBO J 1996;15:6960–8. [ Links ]
31. Ortalo–Magne A, Lemassu A, Laneelle MA, Bardou F, Silve G, Gounon P, et al. Identification of the surface–exposed lipids on the cell envelops of Mycobacterium tuberculosis and other mycobacterial species. J Bacteriol 1996;178:456–61. [ Links ]
32. Draper P. The outer parts of the mycobacterial envelope as permeability barriers. Frontiers in Bioscience 1998;3:d1253. [ Links ]
33. Asselineau J, Lanéelle G. Mycobacterial lipids a historical perspective. Frontiers in Bioscience 1998;3:e164–74. [ Links ]
34. Beatty WL, Rhoades ER; Ullrich HJ, Chatterjee D, Heuser JE, Russell DG. Trafficking and release of mycobacterial lipids from infected macrophages. Traffic 2000;1:235–47. [ Links ]
35. Prigozy TI, Sieling PA, Clemens D, Stewart PL, Behar SM, Percelli SA, et al. The mannose receptor delivers lipoglycan antigens to endosomes for presentation to T cells by CD1b molecules. Immunity 1997;6:187–97. [ Links ]
36. Dutronc Y, Porcelli SA. The CD1 family and T cell recognition of lipid antigens. Tissue antigens 2002;60:337–53. [ Links ]
37. Moody DB, Besra GS. Glycolipid targets of CD1–mediated T cell responses. Immunology 2001;104(3):243–51. [ Links ]
38. Peters PJ, Neefjs JJ, Oorschot V, Ploegh HL, Geuze HJ. Segregation of MHC molecules from MHC class I molecules in the Golgi complex for Transport to lysosomal compartment. Nature 1991;349:669–76. [ Links ]
39. Sugita M, Jackman RM, van Donselaar E, Behar SM, Rogers RA, Peters PJ, et al. Cytoplasmic tail–dependent localization of CD1b antigen–presenting molecules to MIICs. Science 1996;273:349–52. [ Links ]
40. Briken V, Moody DB, Porcelli SA. Diversification of CD1 proteins: sampling the lipid content of different cellular compartments. Semin Immunol 2000;12:517–25. [ Links ]
41. Shamishiev A, Gober HJ, Donda A, Mazorra Z, Mori L, De Libero G. Presentation of the same glycolipid by different Cd1 molecules. J Exp Med 2002;195:1013–21. [ Links ]
42. Schaible UE, Hagens K, Fischer K, Collins HL, Kaufmann SH. Intersection of group I CD1 molecules and mycobacteria in different intracellular compartments of dendritic cells. J Immunol 2000; 164:4843–52. [ Links ]
43. Longley J, Graus J, Alonso M, Edelson R. Molecular cloning of CD1a (T6), a human epidermal dendritic cell marker related to class I MHC molecules. J Invest Dermatol 1989;92:628–31. [ Links ]
44. Porcelli SA, Morita CT, Brenner MB. CD1b restricts the response of human CD4–CD8– T lymphocytes to a microbial antigen. Nature 1992;360:593–97. [ Links ]
45. Ernst WA, Maher J, Cho S, Niazi KR, Chatterjee D, Moody DB, et al. Molecular interaction of CD1b with lipoglycan antigens. Immunity 1998;8:331– 40. [ Links ]
46. Melian A, Watts GF, Shamishev A, De Libero G, Caltworthy A, Vincent M, et al. Molecular recognition of human CD1b antigen complexes: evidence 64. for a common pattern of interaction with alpha beta TCRs. J Immunol 2000; 165:4494–504. [ Links ]
47. Moody DB, Ulrichs T, Muhlecker W, Young DC, Gurcha SS, Grant E, et al. CD1c–mediated T cell recognition of isoprenoid glycolipids in Mycobacterium tuberculosis infection. Nature 2000;404:884–8. [ Links ]
48. Roberts TJ, Sriram V, Spence PM, Gui M, Hayakawa K, Bacik I, et al. Recycling CD1d1 molecules present endogenous antigens processed in an endocytic compartment tto NKT cells. J Immunol 2002;168:5409–14. [ Links ]
49. Joyce S, Woods AS, Yewdell JW, Bennink JR, De Silva AD, Boesteanu A, et al. Natural ligand of mouse CD1d: cellular glycosylphosphatidylinositol. Science 1998;279:1541–4. [ Links ]
50. Spada FM, Koezuka Y, Porcelli SA. CD1d–restricted recognition of synthetic glycolipid antigens by human natural killer T cells. J Exp Med 1998;188:1529–34. [ Links ]
51. Kang SJ, Cresswell P. Regulation of intracellular trafficking of human CD1d by association with MHC class II molecules. EMBO J 2002;21:1650–60. [ Links ]
52. Angenieux C, Salamero J, Fricker D, Cazenave JP, Gound B, Hanau D, et al. Characterization of CD1e, a third type of CD1 molecule expressed in dendritic cells. J Biol Chem 2000;275:37757–64. [ Links ]
53. Shinkai K, Locksley RM. CD1, tuberculosis, and the evolution of major histocompatibility complex molecules. J Exp Med 2000;191:907–14. [ Links ]
54. Beckman EM, Melian A, Behar SM, Sieling PA, Chatterjee D, Furlong ST, et al. CD1c restricts responses of mycobacteria–specific T cells. Evidence for antigen presentation by a second member of the human CD1 family. J Immunol 1996;157:2795–803. [ Links ]
55. Stenger S, Niazi KR, Modlin RL. Down–regulation of CD1 on antigen–presenting cells by infection with Mycobacterium tuberculosis. J Immunol 1998;161:3582–8. [ Links ]
56. Moody DB, Besra GS, Wilson IA, Porcelli SA. The molecular basis of CD1–mediated presentation of lipid antigens. Immunol Rev 1999;172:285–96. [ Links ]
57. Sugita M, Grant EP, van Donselaar E, Hsu VW, Rogers RA, Peters PJ, et al. Separate pathways for antigen presentation by CD1 molecules. Immunity 1999;11:743–52. [ Links ]
58. Vicari AP, Zlotnik A. Mouse NK1.1+ T cells: a new family of T cells. Immunol today 1996;17:71–75. [ Links ]
59. Fowlkes BJ, Kruisbeek AM, Ton–That H, Weston MA, Coligan JE, Schwarts RH, et al. A novel polulation of T cell receptor alpha beta–bearing thymocytes which predominantly xpresses a single V beta gene family. Nature 1987;329:251–55. [ Links ]
60. Kronenberg M, Gapin L. The unconventional life style of NKT cells. Nat Rev Immunol 2002;2:557–68. [ Links ]
61. Bendelac A, Rivera MN, Park SH, Roark JH. Mouse CD1–specific NK1 T cells: development, specificity and function. Annu Rev Immunol 1997;15:535–62. [ Links ]
62. Singh N, Hong S, Scherer DC, Serizawa I, Burdin N, Kronenberg M, et al. Cutting Edge: Activation of NK T cells by CD1d and á–Galactosylceramide directs conventional T cells to the acquisition of a Th2 phenotype. J Immunol 1999;163:2373–77. [ Links ]
63. Hong S, et al. The natural killer T cell ligand á–galactosylceramide prevents autoimmune diabetes in non–obese diabetic mice. Nature Med 2001;7:1052– 56. [ Links ]
64. van der Vliet HJ, Nishi N, Koezuka Y, von Blomberg BM, van den Esrtwegh AJ, Porcelli SA, et al. Potent expansion of human natural killer T cells using alpha–galctosylceramide (KRN7000)–loaded monocyte–derived dendritic cells, cultured in the presence of IL–7 and IL–15. J Immunol Methods 2001; 247:61–72. [ Links ]
65. Hiromatsu K, Dascher CC, LeClair KP, Sugita M, Furlong ST, Brenner MB, et al. Induction of CD1–restricted immune responses in guinea pigs by immunization with mycobacterial lipid antigens. J Immunol 2002;169:330–9. [ Links ]
66. Kaufmann SH. Protection against tuberculosis: cytokines, T cells, and macrophages. Ann Rheum Dis 2002;61(suppl II):ii54–58. [ Links ]
67. Russell DG. Mycobacterium tuberculosis: here today, and here tomorrow. Nat Rev Mol Cell Biol 2001;2:569–77. [ Links ]
68. Romero JF, Eberi G, MacDonald HR, Corradin G. CD1d–restricted NKT cells are dispensable for specific antibody responses and protective immunity against liver–stage malaria infection. Parasite Immunol 2000;164:5005–9. [ Links ]
69. Nishimura T, Kitamura H, Iwakabe K, Yahata T, Ohta A, Sato M, et al. The interface between innate and acquired immunity: glycolipid antigen presentation by CD1d–expressing dendritic cells to NKT cells induces the differentiation of antigen–specific cytotoxic T lymphocytes. Int Immunol 2000;12:987–94. [ Links ]
70. Burdin N, Brossay L, Kronenberg M. Immunization with alpha–galactosylceramide polarizes CD1–reactive NK T cells towards Th2 cytokine synthesis. Eur J Immunol 1999;29:2014–25. [ Links ]
71. Carnaud C, Lee D, Donnars O, Park SH, Beavis A, Koezuka Y, Bendelac A. Cutting edge: Cross–talk between cells of the innate immune system: NKT cells rapidly activate NK cells. J Immunol 1999;163:4647–50. [ Links ]
72. Lazarevic V, Flynn J. CD8+ T cells in tuberculosis. Am J Respir Crit Care Med 2002;1666:1116–21. [ Links ]
73. Frassanito MA, Silvestris F, Cafforio P, Dammacco F. CD8+/CD57 cells and apoptosis suppress T cell functions in multiple myeloma. Br J Haematol 1998;100:469–77. [ Links ]














