Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Chapingo. Serie horticultura
versión On-line ISSN 2007-4034versión impresa ISSN 1027-152X
Rev. Chapingo Ser.Hortic vol.16 no.2 Chapingo may./ago. 2010
Evaluación y caracterización de Phaseolus spp. como fuente de resistencia a Fusarium oxysporum f sp. phaseoli (Fop)
Evaluation and characterization of Phaseolus spp. as a source of resistance to Fusarium oxysporum f sp. phaseoli (Fop)
J. C. Jiménez–Galindo1; E. Valadez–Moctezuma2*; N. Marbán–Mendoza3
1 Instituto Nacional de Investigaciones Forestales Agrícolas y Pecuarias, Campo Experimental Sierra de Chihuahua, Avenida Hidalgo Núm. 1213. Colonia Centro, Cd. Cuauhtémoc, Chihuahua. C. P. 31500. MÉXICO.
2 Posgrado en Biotecnología Agrícola, Departamento de Fitotecnia, Universidad Autónoma Chapingo, km 38.5 Carretera México–Texcoco, Chapingo, Estado de México. C. P. 56230. MÉXICO. Correo–e: evaladez@correo.chapingo.mx (*Autor responsable).
3 Posgrado en Protección Vegetal, Departamento de Parasitología Agrícola, Universidad Autónoma Chapingo, km 38.5 Carretera México–Texcoco, Chapingo, Estado de México. C.P. 56230. MÉXICO.
Recibido: 7 de Julio, 2009.
Aceptado: 12 de Abril, 2010.
Resumen
Los objetivos del presente estudio fueron evaluar con marcadores de DNA, plantas de frijol Tepary (Phaseolus acutifolius) y frijol común (Phaseolus vulgaris L.) con distinto nivel de resistencia a Fusarium oxysporum f sp. phaseoli (Fop) y rastrear la herencia de los marcadores de DNA asociados. Se realizaron cruzas de Tepary Pinto x Pinto Americano y Pinto Americano x Bayo Blanco con niveles contrastantes de resistencia al hongo para obtener progenies segregantes F1 y F2. Para la búsqueda de los marcadores asociados se inocularon plántulas progenitoras y F2 con una suspensión de 2.5 x 105 conidios·mL–1 del hongo. DNA de plantas susceptibles y resistentes seleccionadas con base en la presencia de síntomas de marchitamiento u hojas cloróticas y plantas sin síntomas de marchitez, respectivamente, se utilizaron para rastrear los marcadores de DNA asociados a los fenotipos contrastantes de resistencia y susceptibilidad. El análisis molecular se hizo con la técnica DAF–PCR con los iniciadores aleatorios A–01, A–04 y A–13. Se observaron polimorfismos de DNA en las poblaciones F1, lo que indicó la conformación de híbridos entre P. acutifolius y P. vulgaris. En F2 se detectaron marcadores dominantes de P. acutifolius asociados con la resistencia a Fop.
Palabras clave: Cruzas inter e intraespecíficas, frijol Tepary, frijol común, pudrición de raíz y marchitamiento, perfiles genómicos.
Abstract
The objectives of this study were to evaluate Tepary bean (Phaseolus acutifolius) and common bean (Phaseolus vulgaris L.) with different levels of resistance to Fusarium oxysporum f. sp. phaseoli (Fop), using DNA markers, and to trace the inheritance of DNA associated markers. Crosses were made between Tepary Pinto x Pinto Americano and Pinto Americano x Bayo Blanco contrasting levels of resistance to the fungus to obtain F1 and F2 segregating progenies. To search for the DNA associated markers, seedlings from parents and F2 were inoculated with a suspension of 2.5 x 105 fungus conidia·mL–1. DNA from selected susceptible and resistant plants based on the presence of symptoms of wilting or chlorotic leaves and plants without wilting symptoms, respectively, were used to track DNA markers associated with contrasting phenotypes of resistance and susceptibility. Molecular analysis was done with DAF–PCR technique using the random primers A–01, A–04 and A–13. DNA polymorphisms were observed in F1 populations indicating the formation of hybrids between P. acutifolius and P. vulgaris. In F2 dominant markers were detected in P. acutifolius associated with resistance to Fop.
Key words: Inter– and intra–specific crosses, Tepary bean, common bean, root–rot and wilting, genomic profiles.
INTRODUCCIÓN
El frijol (Phaseolus vulgaris L.) es de las leguminosas de grano más importantes del mundo; su cultivo está extendido en los cinco continentes y es uno de los alimentos básicos en África, América Latina y el Caribe (Lépiz, 2000). Para México es un cultivo estratégico, ya que ocupa el segundo lugar en superficie a escala nacional, con un promedio de 1.688 millones de hectáreas. Su producción es de casi un millón de toneladas con un valor de 6.94 mil millones de pesos (SIAP, 2009). La gama de frijoles bayos y pintos ocupan el segundo y tercer lugar de consumo en México, aunque se muestra una tendencia al alza debido al precio, sabor y calidad culinaria (Plan Rector Sistema Nacional Frijol, 2009).
Durante el periodo de cultivo, el frijol puede ser afectado por múltiples factores adversos que reducen el rendimiento; algunos son las enfermedades causadas por patógenos de la raíz y foliares que lo afectan hasta un 50 %; esto ha obligado a investigadores a desarrollar nuevas estrategias que permitan estabilizar la producción y contribuir a la competitividad del frijol con otros cultivos (Van Bruggen et al., 1986).
En los programas de mejoramiento genético, el investigador considera genotipos contrastantes respecto al carácter de interés; sin embargo, la segregación de genes puede reflejarse en la variedad mejorada, ya que no todos los individuos presentan el carácter deseado absoluto. Esto dependerá de la base genética y del nivel de recombinación (Staub et al., 1996).
La resistencia genética mediante la incorporación de genes que ayuden a contrarrestar los efectos causados por los patógenos, ha sido una herramienta importante no sólo para la incorporación de caracteres de calidad, sino también para el manejo de enfermedades en muchos cultivos, entre ellos el frijol. Las técnicas tradicionales y moleculares se han utilizado en los programas de mejoramiento para transferir a los genotipos mejorados, genes de resistencia procedentes de variedades silvestres o de especies cultivadas no comerciales (Geffroy et al., 1997). También se utilizan métodos para la identificación de marcadores potenciales de DNA asociados a resistencia o susceptibilidad a patógenos, para posteriormente realizar selecciones apropiadas durante el proceso de obtención de nuevas variedades comerciales (Tarlan et al. , 2001).
Para la búsqueda de marcadores de DNA se utilizan los RFLP (polimorfismo de fragmentos de restricción), la electroforesis de campos pulsantes o PFGE y el complejo de amplicones múltiples aleatorios (MAAP) (Tenover et al., 1997). De estas herramientas, la más utilizada es la caracterización con MAAP (terminología propuesta por Caetano et al. , 1992), que comprende tres técnicas basadas en PCR: el polimorfismo de DNA amplificado al azar (RAPD), la amplificación de huellas de DNA (DAF) y la reacción en cadena de la polimerasa con el uso de iniciadores arbitrarios (AP–PCR).
La PCR es una técnica que se utiliza en variados campos científicos, y su éxito se debe a la capacidad para producir literalmente millones de copias de fragmentos de DNA con alta fidelidad (Tenover et al., 1997). Particularmente la técnica DAF presenta ventajas respecto a otras técnicas de PCR: por ejemplo, la obtención de alto número de fragmentos discriminativos; el diseño de iniciadores no requiere de información genética previa; las cantidades del DNA molde son mínimas; la reproducibilidad de resultados es mayor que RAPD y los costos unitarios por ensayo son bajos; pero también tienen el inconveniente de seguir una herencia dominante y la falta de conocimiento de la identidad de los productos amplificados que se obtienen (Struelens, 1998). Hoy en día, esta técnica y RAPD son consideradas las más adecuadas para realizar una genotipificación comparativa rápida (Williams et al., 1990; Welsh et al., 1990). Con base en la utilidad que ofrecen las técnicas de PCR, en el presente estudio se utilizaron marcadores de DNA de plantas de frijol Tepary (Phaseolus acutifolius) y frijol común (Phaseolus vulgaris L.) con distinto nivel de resistencia a Fusarium oxysporum f sp. phaseoli (Fop), y se rastreó la herencia de los marcadores de DNA asociados.
MATERIALES Y MÉTODOS
Variedades evaluadas
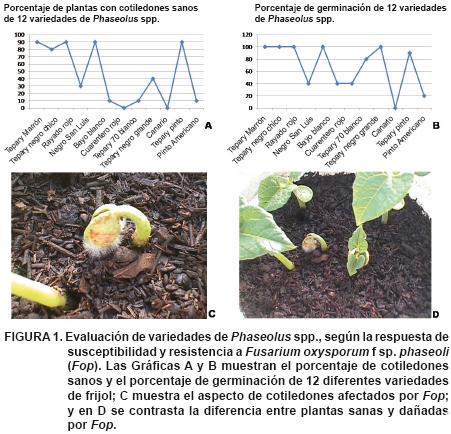
Se utilizaron seis materiales genéticos de Phaseolus acutifolius (Tepary Marrón, Tepary Negro Chico, Tepary Negro Grande, Tepary 60 Blanco, Tepary 70 Blanco y Tepary Pinto) y seis variedades comerciales de Phaseolus vulgaris L. (Rayado Rojo, Negro San Luis, Bayo Blanco, Cuarentero Rojo, Canario y Pinto Americano). Se inocularon 20 semillas de cada variedad con una suspensión de 2.5 x 105 conidios·mL–1 de Fusarium oxysporum f sp. phaseoli (Fop) para conocer sus niveles intrínsecos de resistencia y susceptibilidad al hongo fitopatógeno. Antes de la inoculación, las semillas se desinfestaron con una solución de Extrán 10 % durante 10 minutos, se enjuagaron varias veces con agua destilada estéril y se sumergieron en la suspensión de conidios durante cinco minutos. En seguida se sembraron en macetas con suelo de monte esterilizado con bromuro de metilo a cuatro cm de profundidad y se evaluaron los porcentajes de germinación y de cotiledones sanos (Figura 1). Se seleccionaron las variedades Tepary Pinto, Pinto Americano y Bayo Blanco por presentar porcentajes contrastantes de resistencia y susceptibilidad a Fop.
CRUZAS INTRA E INTERESPECÍFICAS
Con la finalidad de rastrear las huellas de DNA asociadas a resistencia y susceptibilidad a Fop, se realizaron dos cruzamientos, uno de Tepary Pinto (T) x Pinto Americano (P) y otro de Pinto Americano (P) x Bayo Blanco (B). Las variedades Tepary Pinto y Bayo Blanco se consideraron como resistentes y Pinto Americano como susceptible, con base en las respuestas que mostraron en la evaluación indicada en la Figura 1. En los progenitores y las progenies F2 de las cruzas, se analizó la herencia del marcador de DNA asociado al nivel de resistencia. Las cruzas se representan en el esquema siguiente:
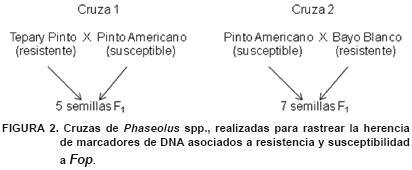
Las semillas de la progenie F1 se sembraron en invernadero en macetas de 10 kg de sustrato de tierra de monte desinfestada con bromuro de metilo para la obtención de la generación F2.
Inoculación con Fop de progenitores y progenies segregantes F2
Para detectar los marcadores moleculares asociados a resistencia y/o susceptibilidad a Fop en los progenitores y en las progenies segregantes F2 de frijol, se inocularon 10 semillas de cada progenitor y 10 semillas de las progenies F2 por inmersión durante cinco minutos en una suspensión de 2.5 x 105 conidios·mL–1 de Fop. Después se sembraron en macetas de un kilogramo bajo condiciones de invernadero con temperatura de 20–23 °C. La selección de muestras de plantas susceptibles y resistentes se realizó 25 días después de la inoculación, con base en la presencia de síntomas de marchitamiento u hojas simples cloróticas y plantas sin síntomas de marchitez, respectivamente; su DNA se analizó de acuerdo a su polimorfismo mediante la técnica DAF–PCR.
Estimación de la variabilidad genética y detección de marcadores DAF
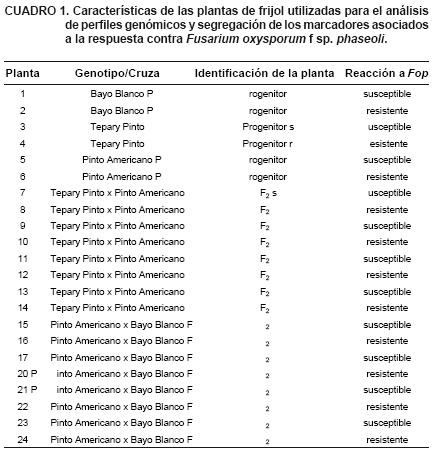
Para la estimación inicial de la variabilidad genética de los progenitores, el DNA de las 10 plantas utilizadas se caracterizó finalmente con los iniciadores aleatorios A–01 (5'–CAGGCCCTTC–3') y A–04 (5'–AATCGGGCTG–3') de Carl Roth Gbmh (Germany) (Figura 3). Para detectar la presencia de DNA de los progenitores en la progenie F1, se conformaron muestras compuestas de hojas de todas las plantas resultantes (cinco plantas en la cruza 1 y siete plantas en la cruza 2), y el análisis del DNA conjunto se hizo con los iniciadores A–01 y A–04, además del iniciador A–13 (5'–CAGCACCCAC–3') (Figura 4). Los marcadores de DNA asociados a la resistencia y susceptibilidad en F2 fueron rastreados en plántulas de los tres progenitores y de la descendencia F2 inoculadas, utilizando los mismos tres iniciadores aleatorios A–01, A–04 y A–13 (Figura 5). Las plantas susceptibles seleccionadas, mostraron síntomas severos o muerte post emergente a los 25 días después de la inoculación. Para la búsqueda de los marcadores de DNA se consideraron una planta con fenotipo susceptible y otra con fenotipo resistente de cada progenitor (dada la variabilidad intrínseca de respuesta frente a Fop) y ocho plantas F2 de cada cruzamiento, cuatro susceptibles y cuatro resistentes para cada cruza, conformando dos repeticiones con plantas diferentes en cada caso (TxP a; TxP b; PxB a y PxB b) (Figura 5, Cuadro 1).


Extracción de DNA
Para la extracción de DNA se utilizó el protocolo descrito por Dellaporta et al. (1983) utilizando hojas jóvenes de cada uno de los progenitores segregantes F1 y F2. Para el caso de F1 se consideraron muestras compuestas por hojas de toda la progenie y se utilizaron 1.4 g de hojas que se molieron con nitrógeno líquido en un mortero. El tejido pulverizado se transfirió a un tubo que contenía 15 mL de amortiguador de extracción precalentado a 65 °C (100 mM Tris–HCl, pH 8.0; 50 mM EDTA, pH 8.0; 500 mM NaCl2, 10 mM β–mercaptoetanol y SDS 1.3 %), se mezcló el contenido y se incubó a 65 °C en baño de agua por 10 min; luego se añadieron a la mezcla 5 mL de acetato de potasio 5 M y posteriormente se dejó enfriar durante 30 min. Después, los tubos de las muestras se centrifugaron a 4,000 rpm durante 20 min a 4 °C, y el sobrenadante se transfirió a otro que contenía un volumen de isopropanol frío; el contenido se mezcló lentamente por inversión y los tubos se mantuvieron por 30 min a –20 °C. Posteriormente, el DNA (en forma de hilos) se transfirió a un nuevo tubo que contenía 700 μL de amortiguador TE (50 mM Tris–HCl, pH 8.0 y 10 mM de EDTA, pH 8.0); se agregaron 4 μL de RNAsa (10 mgmL–1) para eliminar el RNA mediante incubación a 37 °C por una hora. En seguida se adicionaron 50 μL de acetato de sodio 3M, pH 5.2 y 500 μL de isopropanol frío para precipitar el DNA; los tubos se mezclaron nuevamente y se mantuvieron a –20 °C por 2 h. Para recuperar el DNA, los muestras se centrifugaron a 15,000 rpm por 5 min; se eliminó el sobrenadante y la pastilla resultante se lavó con etanol 70 % y posteriormente se disolvió en 100 μL de amortiguador TE.
Técnica DAF–PCR
Antes de la amplificación con PCR, el DNA de las plantas inoculadas F1 y F2 fue digerido con la enzima de restricción Eco RI. La mezcla de reacción para PCR contenía: 200 μM de dNTPs, 1X de amortiguador Taq, 2 mM de MgCl2, 20 pmol del iniciador respectivo (A–01, A–04, A–13), 1 U de enzima Taq DNA polimerasa y 25 ng del DNA digerido correspondiente. El programa de amplificación fue: 1 ciclo a 94 °C, 1 min; 38 ciclos [94 °C, 20 s; 40 °C, 15 s; 72 °C, 1 min] y 1 ciclo de extensión final a 72 °C por 2 min.
Separación de productos amplificados DAF–PCR
Para el análisis de la variabilidad de los progenitores, los productos de PCR se separaron por electroforesis en geles de agarosa 1.2 %, utilizando el amortiguador TAE 1X (40 mM Tris–HCl, pH 8.0; 20 mM acetato de sodio y 2 mM de EDTA). Para esto se colocaron 10 μL del producto de PCR mezclado con 0.1 volumen del amortiguador de carga 10X (0.1 % azul de bromofenol p/v, 0.1 % de xileno cianol p/v, 40 % de glicerol y 60 mM de EDTA, pH 8.0). El DNA se corrió a 80 V durante 1.5 h; posteriormente se tiñeron con bromuro de etidio y las bandas se documentaron con el sistema Kodak Digital Science 1D v. 2.0.
Los productos de PCR para la F1 y plantas resistentes y susceptibles a Fop de progenitores y progenies F2, fueron separados en geles de acrilamida 5 % (acrilamida–bisacrilamida 29:1, agua deionizada, TBE 5X, persulfato de amonio 10%, TEMED 0.5 %) de acuerdo a Sambrook et al. (1989). Se cargaron 10 μL de las muestras amplificadas de DNA con 0.1 de volumen del amortiguador de carga en cada uno de los pozos del gel y se corrieron a 70 V para la electroforesis respectiva. Los geles fueron teñidos con bromuro de etidio y se documentó con el sistema Kodak. En cada corrida se incluyó el marcador de peso molecular de 1 kb (Gibco–BRL).
RESULTADOS Y DISCUSIÓN
Estimación de la variabilidad de perfiles genómicos de los progenitores
Los perfiles de DNA observados en las plantas representantes de los progenitores Pinto Americano y Bayo Blanco a través de las huellas DAF, indicaron que el grado de variación intrapoblacional fue bastante estable en comparación con el progenitor Tepary Pinto, que presentó perfiles heterogéneos entre las plantas analizadas. Este comportamiento en las variedades probablemente se debe, en primer lugar, al alto nivel de endogamia característica del género Phaseolus, y en segundo lugar, lo que era de esperarse, a que son variedades comerciales cuyo origen se remonta a una sola planta obtenida después de muchas generaciones mediante autofecundación (líneas puras). El frijol Tepary a diferencia de las variedades comerciales, en México se considera como silvestre, por lo que es muy variable. En la Figura 3 se muestran perfiles DAF obtenidos con los iniciadores utilizados A–01 y el A–04. Nótese que las variedades Bayo Blanco y Pinto Americano presentan bandas de DNA comunes con ambos iniciadores (elipses), mientras que los representantes de Tepary Pinto exhiben mayor variación en los perfiles (flechas).
Análisis de perfiles genéticos en la progenie F1
Los perfiles genómicos tipo DAF demostraron que las progenies F1 contenían material genético de los progenitores utilizados en las cruzas respectivas, tal como se observa en la Figura 4. Para la evaluación de las progenies se utilizó la mezcla de DNA de todas las plantas F1 obtenidas en cada cruzamiento. En la cruza de Tepary Pinto x Pinto Americano (panel A, carril 5) se aprecia un fragmento de DNA de alrededor de 1,010 bp señalado con una flecha, que posiblemente se deba a una nueva recombinación genética ocurrida en la cruza. En los carriles 6 y 7 del panel A correspondientes a la cruza Pinto Americano x Bayo Blanco, existe una banda de aproximadamente 1,220 bp perfectamente definida (señalada con una flecha) heredada del progenitor Bayo Blanco (carril 2). En el panel B no se observaron fragmentos nuevos heredados a la progenie F1. Con el iniciador A–13 (panel C) se observaron dos bandas de alrededor de 490 bp y 520 bp en los carriles 4 y 5 (señalados con flechas) en la cruza cruza T x P claramente heredadas del progenitor Tepary.
Análisis de perfiles genómicos en la progenie F2
Al analizar los perfiles DAF de las plantas inoculadas con el patógeno (progenitores y progenie F2) mostrados en la Figura 5, el iniciador A–01 detectó un polimorfismo de alrededor de 1,020 bp en el carril 4 (indicado con una flecha), el cual está presente en el progenitor resistente Tepary Pinto. En los carriles 15, 16, 17 y 18, que corresponden a plantas resistentes y susceptibles de la cruza de Pinto Americano x Bayo Blanco, se detectaron dos fragmentos polimórficos, en los carriles 16 y 18, de alrededor de 2,500 pb y 500 pb (marcados con flechas) que no están presentes en los progenitores y que están asociados a plantas resistentes. En los carriles 15 y 17, que corresponden a plantas susceptibles, se detectó un polimorfismo de alrededor de 1,300 pb asociado a susceptibilidad que también está presente en los progenitores Pinto y Bayo. Fue posible apreciar que las plantas que mostraron fragmentos de DNA asociados a este carácter procedentes de la variedad Pinto Americano, desarrollaron síntomas de pudrición de raíz y marchitez. Al realizar cortes longitudinales en el tallo de estas plantas, se observó oscurecimiento del sistema vascular y coloración marrón rojiza, característica de las infecciones obstructivas causadas por Fop (datos no mostrados).
Con relación al iniciador A–04, no se detectaron datos informativos; de hecho, se obtuvieron pocos fragmentos de DNA. El iniciador A–13 logró detectar un polimorfismo de alrededor de 520 pb (indicado con una flecha) en el progenitor Tepary Pinto del carril 4 que corresponde a una planta resistente a Fop. Este marcador también está presente en las plantas de los carriles 10 y 14, que corresponden a plantas resistentes al hongo en las cruzas de TxP. En el carril 6, que corresponde a una planta resistente del progenitor Pinto Americano, se aprecia una banda de DNA de alrededor de 1,200 pb (señalada con flecha) también presente en las muestras de los carriles 16 y 18 correspondientes a plantas resistentes de la cruza PxB; lo que sugiere que este marcador posiblemente también esté asociado al carácter de resistencia al hongo.
Además de la detección de fragmentos de DNA asociados a resistencia o susceptibilidad en los progenitores y progenie F1, fue posible detectar polimorfismos nuevos en la progenie F1 de la cruza Tepary Pinto x Pinto Americano que no estaban presentes en los padres, y polimorfismos heredados de la variedad Bayo Blanco a la progenie de Pinto Americano x Bayo Blanco; lo que sugiere la transferencia de información genética a través de las cruzas inter e intraespecíficas. Estos resultados coinciden con lo encontrado por Michelmore et al. (1991), quienes señalan que una población F2 provee mayor amplitud genética de la región a ser detectada, que una población derivada de una retrocruza, y que todos los ligamientos así detectados deberían ser confirmados por el análisis de poblaciones segregantes.
Lépiz (2000) señala al frijol como un cultivo atractivo para marcar genes de resistencia, porque existen polimorfismos que permiten asociar caracteres genotípicos con respuestas fenotípicas que no están influidas por el ambiente. En la actualidad, la identificación de marcadores asociados a un carácter fenotípico se considera una herramienta fundamental para el desarrollo de la selección asistida por marcadores (MAS), debido a que pueden ser secuencias de un gen o de una porción de DNA cuya herencia es factible de detección en la progenie. Dependiendo de la técnica empleada para su detección, los marcadores pueden ser dominantes o codominantes (Staub y Serquen, 1996); asimismo, además de carecer de efectos pleiotrópi–cos, son estables y no están sujetos al ambiente en donde se desarrollan las plantas. Estas propiedades hacen que estos marcadores sean extremadamente útiles comparados con los marcadores morfológicos o bioquímicos (Valadez y Kahl, 2000).
Al analizar estadísticamente los datos genómicos con los programas NTSYS–pc: Numerical Taxonomy and multivariate analysis system, version 2.0, se obtuvo el dendrograma de la Figura 6, donde se aprecian dos grupos principales; el primero comprende a todas las plantas relacionadas con las variedades comerciales Pinto, Bayo y cruzas respectivas, mientras que el segundo contiene a las relacionadas con el frijol Tepary y cruzas respectivas. Al interior de ambos grupos se conformaron subgrupos que representan a plantas que se comportaron como resistentes o susceptibles a Fop; esto sugiere que los polimorfismos asociados a estos fenotipos fueron detectados y heredados a las progenies evaluadas.

CONCLUSIONES
Con base en los resultados obtenidos, se concluye que existe la posibilidad de transferir caracteres genéticos asociados a caracteres de resistencia de Phaseolus acutifolius a Phaseolus vulgaris contra la infección de Fusarium oxysporum f. sp. phaseoli, agente causal de la pudrición de raíz y marchitamiento del frijol. Con el análisis de perfiles genómicos de los progenitores y progenie segregante F1, se apreció la recombinación genética ocurrida a través de las cruzas realizadas. La presencia de bandas de DNA específicas de los progenitores y progenie F2 de plantas resistentes al hongo patógeno, refiere polimorfismos asociados a ambos caracteres respecto a Fop que fueron heredados por los progenitores. Las plantas resistentes al patógeno en la progenie F2 exhibieron polimorfismos nuevos como producto de la recombinación genética ocurrida. La incorporación de caracteres de resistencia a plantas susceptibles mediante el esquema desarrollado en el presente trabajo, es aplicable no sólo a la búsqueda de marcadores asociados a resistencia de fitopatógenos, sino que puede ser de utilidad para rastrear marcadores relacionados, por ejemplo, a condiciones de estrés en plantas o cultivos.
AGRADECIMIENTOS
Los autores agradecen al M en C. Jaime B. Díaz de la Cruz y al Ing. Juan Aguilar Moreno por sus valiosas sugerencias en el escrito.
El primer autor expresa su agradecimiento al Consejo Nacional de Ciencia y Tecnología (CONACYT–México) por financiar sus estudios de maestría en la especialidad de Protección Vegetal en la Universidad Autónoma Chapingo.
LITERATURA CITADA
CAETANO A. G.; BASSAM, B. J.; GRESSHOFF, P. M. 1992. DNA fingerprinting: MAAPing out a RAPD redefinition. BioTechnology 10:937. [ Links ]
DELLAPORTA, S. L.; WOOD, J.; HICKS, J. B. 1983. A plant DNA minipreparation version II. Plant. Mol. Bio. Rep. 1:19–21. [ Links ]
GEFFROY, V.; CREUSOT, F.; FALQUET, J.; and SEVIGANAC, M. 1997. A family of LRR sequences in the vicinity of the Co–2 locus for anthracnose resistance in Phaseolus vulgaris and its potential use in marker–assisted selection. Theoretical and Applied Genetics. 96: 3–4, 494–502. [ Links ]
LÉPIZ I. R. 2000. Simposio: Contribución de la fitopatología al mejoramiento de los cultivos agrícolas. El caso del frijol. Rev. Mex. Fitopatol. 17: 54–72. [ Links ]
MICHELMORE, R. W.; PARAN, I.; KESSEL, R. V. 1991. Identification of marker linked to disease genes by bulked segregant analysis: a rapid method to detect markers in specific genomic regions using segregating populations. Proc. Natl. Acad. Sci. USA. 88:9828–9832. [ Links ]
PLAN RECTOR SISTEMA NACIONAL FRIJOL. 2009. Segunda fase: diagnóstico inicial, base de referencia, estructura estratégica. SAGARPA, Tecnológico de Monterrey, INCA Rural. [ Links ]
SAMBROOK, J.; FRITCH, E. F.; MANIATIS, T. 1989. Molecular Cloning: a Laboratory Manual. Second edition. Ed. Cold Spring Harbor Laboratory. Cold Spring Harbor, NY, USA. 18.51–18.57 pp. [ Links ]
SIAP. 2009. Servicio de información agroalimentaria y pesquera. Anuario estadístico de la producción agrícola. Producción agrícola del frijol. En: http://reportes.siap.gob.mx/aagricola_siap/icultivo/index.jsp Último acceso disponible: 20–junio–2009. [ Links ]
STAUB, J. E.; SERQUEN, F. C.; GUPTA, M. 1996. Genetic markers, map construction, and their application in plant breeding. HortScience Vol. 31(5):729–741. [ Links ]
STRUELENS, M. 1998. Molecular epidemiologic typing systems of bacterial pathogens: Current issues and perspectives. Mem Inst Oswaldo Cruz 1998;93:581–5. [ Links ]
TARLAN, B.; MICHAELS, T. E.; PAULS, K. P. 2001. Mapping genetic factors affecting the reaction to Xanthomonas axonopodis pv. phaseoli in Phaseolus vulgaris L. under field conditions. Genome 44:1046–56. [ Links ]
TENOVER, F. C.; ROBERT D. A.; GOERING, R. V. 1997. The Molecular Typing Group of the Society for Healthcare Epidemiology of America. How to select and interpret molecular strain typing methods for epidemiological studies of bacterial infections: a review for healthcare epidemiologists. Infection Control and Hospital Epidemiology. Vol. 18 (6): 426–439. [ Links ]
VALADEZ M. E.; KAHL, G. 2000. Huellas de ADN en genomas de plantas (teoría y protocolos de laboratorio). Ed. Mundi–Prensa México, S. A. 147 pp. [ Links ]
VAN BRUGGEN, A. H. C.; WHALEN, C. H.; and ARNESON, P. A. 1986. Emergence, growth, and development of dry bean seedlings in response to temperature, soil moisture, and Rhizoctonia solani. Phytopathology 76:568–572. [ Links ]
WARBURTON, M. L.; CROSSA, J. 2000. Data analysis in the CIMMYT, Applied Biotechnology Center for fingerprint and Genetic Diversity Studies. CIMMYT, México, D.F. 30 pp. [ Links ]
WELSH, J.; MCCLELLAND, M. 1990. Fingerprinting genomes using PCR with arbitrary primers. Nucleic Acids Res. 18: 7213–7218. [ Links ]
WILLIAMS, G. K.; KUBELIK, A. R.; RAFALSKI, J.; TINGEY, V. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucleic Acids Res. 18:6531–6535. [ Links ]














