Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Salud Pública de México
versión impresa ISSN 0036-3634
Salud pública Méx vol.48 no.3 Cuernavaca may./jun. 2006
ARTÍCULO DE REVISIÓN
La influenza, un problema vigente de salud pública
Influenza, an existing public health problem
Juan García-García, MVZ, PhDI; Celso Ramos, QBP, Dr en CII
IInvestigador independiente
IIDepartamento de Arbovirus, Centro de Investigaciones sobre Enfermedades Infecciosas, Instituto Nacional de Salud Pública. Cuernavaca, Morelos, México
RESUMEN
La influenza estacional es una enfermedad respiratoria aguda, recurrente y común que se conoce desde la antigüedad y se presenta sobre todo durante los meses de invierno con un elevado impacto para la salud pública mundial. La enfermedad se manifiesta con altas tasas de morbilidad en individuos de todas las edades y elevadas tasas de mortalidad en niños, individuos mayores de 60 años, pacientes con enfermedades crónicas y mujeres en gestación. Las estrategias de prevención incluyen el uso de vacunas: inactivadas, subunitarias o vacuna con virus genéticamente modificados. Dos subtipos de virus de influenza tipo A y un virus de influenza tipo B causan la enfermedad en humanos. Los virus de influenza A que afectan a los humanos mutan con facilidad, por lo que con frecuencia aparecen nuevas variantes antigénicas de cada subtipo, lo que obliga a incluir dichas variantes en las vacunas anuales para brindar una adecuada protección a la población. La influenza pandémica se refiere a la introducción y posterior diseminación mundial de un nuevo virus de influenza en la población humana, lo que ocurre de manera esporádica, y que debido a que los humanos carecen de inmunidad para el nuevo virus pueden suscitarse epidemias graves con elevadas tasas de morbilidad y mortalidad. Históricamente el origen de las pandemias de influenza se debe a la transmisión de virus de aves al hombre o la transferencia de genes de éstos a los virus de la influenza estacional. En las aves acuáticas silvestres, tanto migratorias como costeras, se mantiene una gran diversidad de subtipos de virus de influenza, los cuales se introducen eventualmente en aves domésticas, donde algunos virus adquieren la capacidad de infectar a mamíferos, incluido el hombre. El proceso de adaptación de los virus aviarios a hospederos mamíferos requiere tiempo, por lo que la presentación de estos casos puede tardar varios años. Desde diciembre de 2003, en varios países del sureste asiático, las aves domésticas han sido afectadas por una epidemia de influenza aviaria (subtipo H5N1) de grandes proporciones. A febrero de 2006 la epidemia ya afectó a países de Europa y Africa, con un fuerte impacto económico para la avicultura comercial por el sacrificio de más de 180 millones de aves. Algunos linajes de este virus adquirieron la capacidad de cruzar la barrera de especie e infectaron de manera directa pero incipiente a la población humana. El virus todavía no adquiere del todo la eficiencia de infección y transmisión de persona a persona, lo que ha limitado su diseminación en humanos. Debido a que la tasa de mortalidad en los individuos afectados con este virus es mayor de 50%, la Organización Mundial de la Salud (OMS) convocó a los países miembros para que establezcan planes de preparación y emergencia ante la amenaza de una posible pandemia causada por el virus H5N1 o por algún virus reasociante con el de la influenza común; estas acciones han de evitar o disminuir el impacto de la amenaza y así impedir catástrofes previas como la de 1918, en la que murieron alrededor de 40 millones de personas en todo el mundo. Los planes de prevención y control previstos consideran, entre otras actividades, la vacunación y el uso de medicamentos antivíricos. Sin embargo, a la fecha no hay vacunas aplicables en caso de emergencia por un virus pandémico de influenza y tal vez los países que fabrican la vacuna anual del tipo endémico carezcan de capacidad para producir la del virus pandémico. Además, se han detectado cepas de los virus resistentes a los agentes antivíricos de uso común. La presente revisión pretende actualizar los conceptos básicos sobre la influenza con el fin de fortalecer la vigilancia epidemiológica de la enfermedad y resaltar las acciones de prevención y control en caso de que acontezca una pandemia.
Palabras clave: Influenza; prevención; control; México
ABSTRACT
Seasonal influenza is an acute and recurring respiratory disease known since ancient times, occuring, in particular, during winter months and having an elevated effect on public health worldwide. The disease has high morbidity rates for people of all ages and particularly high mortality rates for children, adults over 60 years old, patients with chronic illnesses and pregnant women. Prevention control strategies include vaccination using inactivated, subunit or genetically modified virus vaccines. Influenza in humans is caused by two subtypes of influenza virus A and one of influenza virus B. The influenza virus A that affects humans mutates easily, thereby often causing new antigenic variants of each subtype to emerge, requiring the inclusion of such variants in annual vaccines in order to assure proper immunization of the population. The influenza pandemic refers to the introduction and later worldwide spread of a new influenza virus in the human population, which occurs sporadically. Due to the lack of immunity in humans against the new virus, serious epidemics can be provoked resulting in high morbidity and mortality rates. Historically, influenza pandemics are a result of the transmission of the virus from birds to humans, or the transfer of such genes to seasonal influenza. Wild waterfowl –both migratory and shore birds– carry a large diversity of influenza virus subtypes, which are eventually transmitted to domestic birds. Some of those viruses cross the species barrier and infect mammals, including humans. The adaptation process of the avian virus to mammal hosts requires time. Therefore, the presentation of these cases can take several years. Since December 2003, in several Southeast Asian countries a large proportion of domestic birds have been affected by an avian influenza epidemic (subtype H5N1). By Februrary 2006, the epidemic had already affected countries in Europe and Africa, having a significant economic impact on commercial poultry due to the more than 180 million birds that were sacrificed. Some strains of this avian influenza virus have directly, although incipiently, infected the human population. The virus has not yet acquired with complete efficiency person-to-person infection and transmission, which has limited its spread among humans. Since the mortality rate in infected individuals is greater than 50%, the World Health Organization (WHO) called on their member countries to establish preparation and emergency plans against the threat of a possible pandemic associated with H5N1 virus, or another virus related to common influenza. These actions are intended to prevent or reduce the impact of the threat, as experienced in previous pandemics, such as in 1918 when roughly 40 million people died worldwide. The prevention and control plans include, among other strategies, vaccination and antiviral medications. Nevertheless, to date there are no vaccines to be administered to the population in the case of a new influenza pandemic emergency and it is possible that countries that produce the annual seasonal influenza vaccine lack the capacity to produce the pandemic virus vaccine in a short period of time. In addition, recent studies have identified the existence of influenza virus strains resistant to common antiviral agents. The purpose of this review is to update the basic concepts of influenza in order to strengthen epidemiological surveillance of the disease and reinitiate prevention and control actions in the event of a pandemic.
Key words: Influenza; prevention; control; Mexico
La influenza es una enfermedad común de origen vírico, de tipo respiratorio, aguda y muy contagiosa. La infección se trasmite de persona a persona, principalmente por medio de aerosoles que contienen virus y que se generan por la tos o el estornudo de individuos enfermos, e infectan a las células superficiales del epitelio respiratorio del receptor.1 A pesar del uso de vacunas para su control, las epidemias invernales continúan exhibiendo un alto impacto en la población mundial.2,3 Los virus de influenza evolucionan de forma constante por acumulación de mutaciones puntuales en cada uno de sus ocho segmentos de RNA, lo que da lugar a la aparición de nuevas variantes antigénicas de manera regular que pueden causar epidemias de impacto global.4-6 La aparición de nuevos virus que se transmiten de especies animales domésticas a humanos, y para los cuales no existe inmunidad, también puede originar pandemias con resultados catastróficos.7-11 El análisis de las características del virus, de la influenza y de la enfermedad desde perspectivas históricas y contemporáneas es fundamental para definir estrategias adecuadas para su prevención y control, ya que por sus características de distribución y variación genética la influenza no representa una enfermedad erradicable.
Aspectos históricos
La primera descripción de un padecimiento identificable como influenza la realizó Hipócrates en el año 492 A.C.12,13 Más tarde, durante la Edad Media, se describieron numerosos episodios relacionables con este mal.12 En América, la primera descripción de un problema respiratorio severo de este tipo se documentó en Texcoco, en 1552, y se le denominó "pestilencia catarral".14,15 No obstante, la primera pandemia reconocida ocurrió en 1580,15 que los italianos describieron como la "influencia planetaria", por la asociación que hicieron con una serie de fenómenos astrales observados antes del brote, de donde se derivó el nombre actual de influenza.13 El origen de esta pandemia se localizó en Asia, de donde luego se extendió a Europa, África y el continente americano.16 En España se documentó una elevada morbilidad y mortalidad indicativa de la introducción de un virus muy agresivo con una transmisibilidad muy eficiente.15
La pandemia de 1918-1919 causó la muerte mundial de 20 a 40 millones de personas, lo que representó de 2.5 a 5% de la población global, y se calcula que al menos 20% de la población mundial se infectó con el virus H1N1.1,4 Otras pandemias registradas en 1957, 1968 y 197716-18 con diferentes virus de influenza resultaron menos impactantes debido a cierta inmunidad que los individuos exhibieron al nuevo virus y a las estrategias de control que se utilizaron, como la oportuna preparación y aplicación de vacunas.1,17 A partir de 1997 un nuevo virus de influenza aviar de alta patogenicidad (H5N1) adquirió la capacidad de causar infección en humanos en Hong Kong.19 Un virus semejante se identificó en Corea del Sur en 2003, se extendió a varios países de Asia, y para febrero de 2006 se difundió a Medio Oriente, Europa y África. El virus causa una mortalidad mayor de 50% de los individuos infectados y si la enfermedad se ha limitado a un número reducido de personas se debe a que el virus todavía no adquiere eficiencia en su transmisión entre humanos.20-22
Otros virus de influenza de origen aviario que afectaron a humanos se describieron en China (H9N2),23 Holanda (H7N7)24,25 y Canadá (H7N3),26,27 donde los signos más comunes fueron desde inaparentes hasta trastornos respiratorios o conjuntivitis, con un caso fatal en Holanda.24 En Italia, un análisis retrospectivo demostró seroconversión en individuos que estuvieron en contacto con aves infectadas por virus H7N1 y H7N2 de 1999 a 2003, y la infección se presentó incluso en personas que estuvieron en contacto con aves infectadas por virus de baja patogenicidad.28,29 Hace poco en Japón se describió seroconversión en trabajadores de granjas afectadas con el virus H5N2 de baja patogenicidad.30
Clasificación taxonómica del virus
Los virus de la influenza pertenecen a la familia Orthomyxoviridae y se distribuyen en tres géneros: Influenzavirus A, Influenzavirus B e Influenzavirus C,31 que corresponden a los virus de influenza tipo A, B y C, respectivamente,32 que aún utilizan algunos autores. La diferencia principal entre los géneros radica en las variaciones antigénicas en la proteína de la matriz y de la nucleoproteína que se utilizan para la caracterización del virus y que son específicas para cada género.31
Los virus de influenza A se describieron por primera vez en 1933; afectan a animales y al hombre; y son factor causal de epidemias y pandemias.31,33 Los virus de influenza B se identificaron en 1940; afectan a los humanos causando epidemias moderadas,34 aunque tiene poco tiempo que también se encontraron en focas.35 Los virus de influenza C se describieron en 1950,36 infectan de manera limitada a humanos y existen evidencias serológicas de infección en cerdos y perros.37-39
Por su importancia en salud pública, derivada de las epidemias anuales y de las pandemias identificadas hasta la fecha, esta revisión se limitará a la descripción de los virus de influenza A.
Este tipo de virus se divide, a su vez, en subtipos de acuerdo con las características antigénicas de la hemaglutinina (HA) y la neuraminidasa (NA), que son glicoproteínas en forma de espículas localizadas en la envoltura del virus.31 La denominación internacional de los virus de influenza debe indicar: a) el género (tipo) del virus; b) la especie animal (en inglés) de la que se aisló –excepto cuando procede de humanos–; c) el lugar del aislamiento; d) el número de caso del laboratorio; e) el año del aislamiento; y f) el subtipo de HA y NA del virus entre paréntesis.40 De modo que, por ejemplo, un virus aislado de humanos en Hong Kong en 1997 se describe como "A/Hong Kong/156/97 (H5N1)" y un virus aislado de pollos en el mismo lugar y año se identifica como "A/chicken/Hong Kong/Y280/97 (H5N1)".41 Los virus que tienen con mezclas de genes de diferentes virus de influenza A se denominan genotipos42 y las variaciones genéticas dentro de un mismo subtipo de virus se denominan linajes.43
Ecología de los virus de influenza A
Las aves silvestres acuáticas migratorias, tanto de lagos como costeras, son los reservorios naturales de toda la constelación genética de los virus de influenza,1-4,8,31 en particular las especies de los órdenes Anseriforme (como patos, por ejemplo), Passeriforme (por ejemplo, gansos), ambas aves silvestres migratorias de lagos, y las del orden Charadriforme (gaviotas y golondrinas marinas, entre otras), que son aves acuáticas migratorias de las costas.44 En todas estas aves silvestres, los virus han alcanzado un estado evolutivo estable y por lo general no causan trastornos clínicos en ellas.14,45 Todos los subtipos de HA, incluidos los identificados en los últimos 16 años (HAs 14,46 15,47 y 1648) se aislaron de patos y gaviotas, lo que demuestra que ocurrieron mayores cambios genéticos virales en estas aves.14,44 El análisis filogenético del mismo subtipo de virus encontrados en aves migratorias muestra linajes diferentes entre aquellos que circulan en Europa y Asia y los del continente americano, lo que pone en evidencia que las migraciones longitudinales son más importantes que las migraciones intercontinentales en la difusión global de los virus.1,49 En los patos silvestres los virus se replican en primer lugar en las células del tracto intestinal y se excretan por las heces en altas concentraciones (hasta 108.7 dosis infectante en embrión de pollo (DIEP)50%/g4). Los virus que se eliminan en heces frescas contaminan el suelo y agua de los lagos o canales, en donde las aves domésticas, en especial aquellas que se crían en espacios abiertos, buscan alimento y consumen agua, lo que constituye una eficiente ruta de transmisión.14,44,49 Las aves domésticas se consideran especies aberrantes,49 lo que origina mutaciones en los virus como un mecanismo de adaptación para una óptima replicación y transmisión en la nueva especie.42,49 Los patos y gansos domésticos, así como los pavos, aparentemente son las aves que se infectan primero. Los virus se difunden con cierta facilidad hasta alcanzar gallináceas que se crían en traspatio y de ahí llegan a las explotaciones comerciales de pollos de engorda y gallinas de postura donde la difusión es muy rápida debido a los sistemas de manejo, que movilizan al virus entre granjas.42,50 A su vez, los virus de influenza de las aves acuáticas silvestres llegan a diferentes especies de mamíferos de manera directa o a través de aves domésticas y se han descrito infecciones en mamíferos marinos como focas y ballenas,51,52 mamíferos terrestres como cerdos,53 caballos,54 visones,4 tigres y leopardos,55 y en fecha reciente perros.56
El hombre desempeña un papel importante como transmisor directo o indirecto del virus, ya sea por transporte mecánico a través de la ropa, equipos o vehículos contaminados, o bien por el contrabando de aves silvestres, de ornato, de pelea y comerciales.2,14,42
También se reconoce el papel del cerdo como hospedador intermediario, adaptador y amplificador de ciertos subtipos virales para infecciones en humanos.53 Los estudios genéticos de los virus de influenza clásica del cerdo y en humanos de los subtipos H1 y H3 indican que tienen un ancestro común de origen aviar y una relación filogenética cercana entre ellos.57,58
Características del virus de influenza A
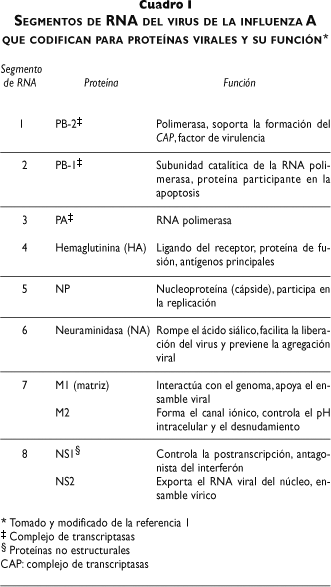
La partícula vírica tiene un tamaño de 80 a 120 nm de diámetro, forma estructuras pleomórficas o esféricas31 y contiene un genoma de ocho segmentos de RNA de cadena sencilla y de polaridad negativa13 (figura 1). Cinco de los segmentos son monocistrónicos y codifican para una proteína, mientras que los otros tres contienen marcos abiertos de lectura (orf) para dos proteínas de cada uno de los tres segmentos.31,59 En el cuadro I se describen las proteínas que se expresan por cada uno de los segmentos del RNA viral y las funciones que realizan. La nucleoproteína (NP) está unida a las cadenas de RNA que en el microscopio electrónico se observan como estructuras helicoidales.1 La envoltura viral es lipídica, se deriva de la membrana plasmática de la célula infectada y contiene dos glicoproteínas a manera de espículas (HA y NA). En los virus de influenza A se describen 16 diferentes subtipos antigénicos de HA (H)48 y nueve diferentes subtipos de NA (N).31 Cada virus de influenza contiene en cualquier combinación sólo un subtipo de HA (H1-H16) y un solo subtipo de NA (N1-N9), lo que puede generar un número extenso de posibles combinaciones o subtipos, de los cuales casi todos se encuentran en las aves silvestres acuáticas.4,13-31

La HA es una glicoproteína integral de superficie que forma espículas y desempeña tres funciones importantes: adherencia a la célula susceptible, fusión de membranas y servir para el reconocimiento antigénico que induce la respuesta inmune.4
La NA es un tetrámero glicoproteínico en forma de hongo que se encuentra en la envoltura del virus; tiene como función principal catalizar el rompimiento del ácido siálico (AS) para liberar los viriones de la célula infectada; también permite el transporte del virus a través de la capa de mucina del tracto respiratorio.60
La proteína M2 es una proteína integral de la envoltura viral que forma un poro o canal iónico encargado de controlar el pH intracelular; esto lo logra al permitir la entrada de iones al virión durante la fase de desnudamiento en el ciclo de replicación.1,4,13,61 Esta proteína es el blanco de agentes antivirales como la amantadina y la rimantadina, y las mutaciones que ocurren en el gen que codifica esta proteína determinan la resistencia a los fármacos antivirales.62
Ciclo de replicación viral
En la figura 2 se muestra de manera esquemática la replicación de los virus de influenza.

Mutaciones
Los virus de influenza experimentan un proceso de evolución continua mediado por mutaciones que les permite adaptarse al hospedero y a las condiciones del ambiente, lo cual se conoce como presión selectiva o evolución obligada.4,6,7 Las mutaciones se presentan como sustituciones de nucleótidos, inserciones y deleciones de tripletes que son causados principalmente por errores de la enzima polimerasa de RNA durante la trascripción31 y ocurren en cada uno de los ocho segmentos genómicos del virus. Las mutaciones se van acumulando en cada ciclo de trascripción y generan variantes de estructura o función de las proteínas que expresan.63,64 Hay cambios mayores como los que se observan al comparar las 16 diferentes hemaglutininas, que muestran diferencias de al menos 30% entre ellas, lo que las hace diferentes desde el punto de vista antigénico.4,42,65 Los cambios menores pueden ser irrelevantes, como los que ocurren en los virus encontrados en las aves silvestres acuáticas, que muestran que la mayoría de las sustituciones de nucleótidos no origina cambios en los aminoácidos de las proteínas, lo que indica una restricción en la evolución del virus en estas aves, mientras que las mutaciones que se acumulan en los virus en aves domésticas y mamíferos originan cambios en los aminoácidos y generan alteraciones importantes en su comportamiento biológico.45-49 Esto es lo que les permite infectar a especies diferentes, seleccionar poblaciones que no sean reconocidas por la respuesta inmune (escape viral), generar familias resistentes a agentes antivirales o cambiar su tropismo celular, con lo que modifican su patogenia.64-68
Variación antigénica
Las mutaciones en los determinantes antigénicos de las glicoproteínas de la envoltura viral dan sustento a las variaciones antigénicas, que se conocen como "drift antigénico".1-4,6,8,13,31,63,64 Éste ocurre por la acumulación de mutaciones puntuales de nucleótidos, en particular en el segmento que codifica para la HA y que resulta en sustituciones de aminoácidos en los determinantes antigénicos; de ello se deriva la imposibilidad para que puedan unirse los anticuerpos generados por infecciones o inmunizaciones previas.1-2,64
Variación genética
La variación genética es otro tipo de mutación que ocurre cuando una célula es infectada por dos virus diferentes de influenza cuyos segmentos genómicos se reasocian durante el ensamble en una sola partícula viral.1,4,6,8,13,42,63,64 Algunas de estas reasociaciones le pueden conferir al virus ventajas para infectar a otras especies, y en general sustituyen a alguno de los virus que le dieron origen,21 como ocurrió en las pandemias de 1957 y 1968; a este proceso se le denomina variación genética o shift.13, 31,64,68
Receptores celulares para los virus de influenza
La replicación viral se inicia mediante la adherencia del virus a la célula, que tiene lugar por la interacción de una región de la HA (ligando) de la porción globular con el receptor de la célula. El receptor celular para los virus de influenza es el ácido siálico (AS), que se encuentra en la porción distal de los oligosacáridos y glicolípidos que forman el glicocáliz de las células.69-73 Las diferencias en la forma de unión entre el AS con las dos moléculas adyacentes de azúcares determinan dos tipos de cadenas: AS-a2-3-Gal-b-1-3-N-acetilglucosamina (AS-a2,3-Gal) y AS-a2-6-Gal-b-1-4-N-acetilglucosamina (AS-a2,6-Gal), que se encuentran distribuidas de manera diferente en las especies animales.70-73 Las cadenas AS-a2,3-Gal se encuentran en las aves y caballos, y la AS-a2,6-Gal, en humanos.66 Ambas cadenas del sitio receptor están presentes en el glucocáliz de las células de los cerdos.71,73
Los aminoácidos de la HA de los subtipos H1, H3, H5 y H9 de los virus de las aves que se unen al receptor SA-a2,3-Gal son: tirosina 205 (Tir-205), glutamina 226 (Gln-226) y serina 228 (Ser-228); de manera similar, los aminoácidos serina 205 (Ser-205), leucina 226 (Leu-226) y glicina 228 (Gli-228) determinan la especificidad de adherencia al receptor SA-a2,6-Gal en humanos.72,76,77 Las mutaciones que ocurren en la región de unión de la HA parecen determinar la capacidad para que los virus de influenza crucen la barrera de especie de aves a los mamíferos.70 La presencia de ambos tipos de receptores en los cerdos sugiere que éstos pueden infectarse con virus de aves y de humanos, mezclando y amplificando a los virus con potencial zoonótico.73-77
Respuesta inmune contra la infección
La resistencia natural a la infección por virus de influenza, que no depende de una previa exposición al virus, se debe a la inducción de IFNa (interferón alfa), que tiene la propiedad de interferir con la replicación viral.78,79 Durante la fase aguda de la infección el IFNa se detecta en las secreciones respiratorias y en el suero de los pacientes.78 Hace poco tiempo se describió el mecanismo por el cual la proteína NS1 del virus interfiere con la producción de IFN1.80
La respuesta inmune que se genera después de una infección conduce a la formación de anticuerpos que se localizan en las mucosas y en el suero, los cuales desempeñan dos funciones principales: neutralización de la infección e interferencia en la liberación de nuevas partículas virales de la membrana de las células infectadas.81,82 A pesar de que en una infección se producen anticuerpos para contrarrestar las proteínas virales, los más importantes para limitar la infección se dirigen contra la HA y la NA.81,82 En las secreciones respiratorias y en el suero se detectan los anticuerpos IgA e IgG, respectivamente.80,81 La concentración de anticuerpos contra la HA en el suero se correlaciona con la protección.81 Es importante hacer notar que los patos desarrollan una pobre o inexistente respuesta de anticuerpos, lo que tiene implicaciones en la vigilancia serológica de la infección en las aves silvestres.82
Los estudios de la respuesta celular en infecciones por el virus de influenza han sido limitados; algunos trabajos sugieren la participación de las células T citotóxicas CD8+, que aparecen de tres a cuatro días después de la infección y se dirigen contra las proteínas HA, NP, PB2 y M2.83
Influenza en aves domésticas
Los virus de influenza que infectan a las aves domésticas pueden dividirse en dos grupos según su capacidad para causar enfermedad en los pollos.84 En el primer grupo se encuentran todos los subtipos de virus de influenza que han sido identificados en las aves, y su replicación se limita a células de los tractos respiratorio y digestivo; causan leves manifestaciones clínicas o su presencia transcurre de forma imperceptible y se denominan virus de baja patogenicidad. Las infecciones con este grupo de virus pueden ser agravadas por infecciones secundarias con otros agentes patógenos e incrementar con ello la morbilidad relacionada con signos respiratorios, retraso en el crecimiento o disminución en la postura, así como aumentar la mortalidad.46 El segundo grupo se genera por mutaciones en los virus de los subtipos H5 y H7, que adquieren la capacidad de causar infecciones sistémicas; ocasionan tasas de mortalidad en las parvadas afectadas hasta de 100% y se denominan virus de alta patogenicidad.4,49,84
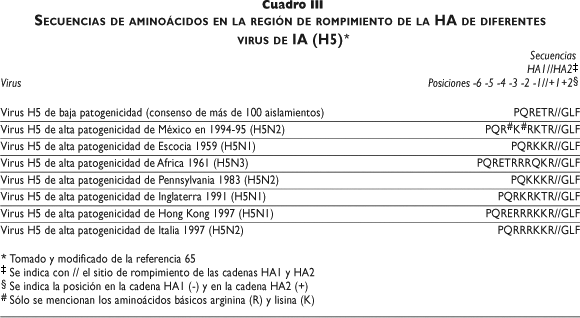
La influenza aviar se describió por primera vez en aves domésticas en 187885 en Italia, en un brote que afectó a gallináceas con una elevada mortalidad; le denominaron "peste aviar", y fue causada por un virus de alta patogenicidad. De 1959 a la fecha se han notificado a escala mundial diversos brotes causados por virus de alta patogenicidad de los subtipos H5 y H7 en las aves domésticas65,86 (cuadro II), y su efecto en el hombre sólo se demostró en años recientes.19,22,23,42 La patogenicidad de estos dos subtipos de virus se mide por su capacidad para causar la muerte en un grupo de ocho aves susceptibles inoculadas por vía intravenosa, en un periodo no mayor a 10 días después de la inoculación. Aquellos que causan una mortalidad igual o mayor a 75% se consideran de alta patogenicidad y los demás se consideran virus de baja patogenicidad.84 Las bases moleculares que determinan el cambio en la patogenicidad se ubican en la HA,87 la cual se sintetiza en un precursor denominado HA0 de 562 a 566 aminoácidos. A causa de un proceso postraduccional se produce un corte enzimático que divide a la HA0 en dos moléculas, HA1 y HA2, que permanecen unidas (figura 3).4,31,66 El corte o rompimiento de la HA produce cambios estructurales en la región del ligando que exponen los aminoácidos de unión al receptor, lo que permite que el virus se adhiera con mayor facilidad a la célula.31 Los virus de baja patogenicidad contienen al menos dos aminoácidos básicos del tipo lisina (K) y arginina (R) en la región terminal de la HA1, adyacente al sitio de rompimiento.87 Enzimas semejantes a la tripsina que realizan el corte y que se encuentran en los tractos respiratorio y digestivo de las aves reconocen esta secuencia.49,64,66 Por mutaciones puntuales se pueden generar sustituciones e inserciones de múltiples aminoácidos básicos (K y R) en la región terminal de la HA1 que reconoce enzimas intracelulares del tipo de la furina, la cual es una proteasa intracelular que realiza el corte en la secuencia de aminoácidos básicos de HA1 que convierte a los virus en agentes de alta patogenicidad.64,66,87 En el cuadro III se presentan ejemplos de secuencias determinadas en el sitio de rompimiento de la HA de algunos virus de baja y alta patogenicidad. Por lo general, los virus de influenza H5 y H7 de las aves silvestres acuáticas infectan a las aves domésticas como virus de baja patogenicidad y las mutaciones que ocurren en la HA1 los convierten en virus de alta patogenicidad al cabo de un tiempo, que puede variar de días a años.87,88 El virus que causó el brote en Pensilvania en 1983 contenía al principio aminoácidos básicos en el sitio de rompimiento de la HA compatible con virus de alta patogenicidad, pero no causaba la muerte de las aves al inocularse por vía intravenosa, debido a que presentaba, un sitio de glicosilación cercano al sitio de rompimiento que actuaba como una barrera física que impedía la acción enzimática. La mutación que causó la pérdida de ese sitio de glicosilación determinó que el virus se convirtiera en una variedad de alta patogenicidad.89 En Asia continúa sin identificarse el precursor de baja patogenicidad del virus H5N1.90


Historia natural de la influenza humana
La introducción de nuevas cepas virales a la población humana se ha relacionado con un incremento sustancial en la morbilidad y mortalidad, por lo que se vuelve necesario efectuar una descripción de los acontecimientos en que los virus ocasionaron pandemias en la población.
Pandemia de 1918
Durante los primeros meses de 1918 Estados Unidos de América (EUA) decidió reforzar el frente armado en Europa para concluir la Primera Guerra Mundial, por lo que inició un reclutamiento de individuos en los fuertes militares. El caso índice de la pandemia con el virus del subtipo H1N1 se identificó durante el mes de mayo en el fuerte Funston, en Kansas, donde se notificaron enfermos con una mortalidad moderada.91 El brote se difundió a otros centros de reclutamiento militar durante ese mes, quizá por la movilización de soldados infectados; el virus causó un cuadro respiratorio agudo y cianosis. Las instalaciones de los servicios médicos fueron insuficientes para atender a los enfermos, por lo que se habilitaron salones de actos y gimnasios para poder brindar la atención médica debida.92 Durante los meses de junio y julio los soldados norteamericanos fueron trasladados a Francia. A la llegada del ejército estadounidense el virus empezó a difundirse en una segunda oleada, más agresiva que la primera, en que los individuos enfermaban en la mañana, se agravaban por la tarde y morían en la noche.91 El brote se extendió a los países de Europa, África, Medio Oriente, Asia y regresó a América a través de buques españoles que llegaron a EUA, Argentina, Brasil y México. Debido a esta situación y al nombre de la estrategia militar a través de España (que en realidad llegó a Francia), se denominó a esta enfermedad como "influenza española".1,91 La pandemia tuvo tres episodios que concluyeron en octubre de 1919, con la estimación de alrededor de 40 millones de fatalidades en todo el mundo. La cifra es imprecisa debido al desconocimiento del impacto que tuvo la enfermedad en poblaciones del continente africano, India, países del Medio Oriente, sureste asiático y Sudamérica.11,69 En México se documentó el efecto que tuvo en el estado de Sinaloa, donde se notificaron 20 000 muertes de una población total de 350 000 habitantes.15. La alta virulencia del virus, la falta de inmunidad a este nuevo agente infeccioso, el contacto cercano entre los pacientes y la falta de control de infecciones secundarias contribuyeron de forma notable a incrementar los efectos de la pandemia.11 Las investigaciones recientes revelaron que el virus causal fue de origen aviar y que probablemente estuvo evolucionando por un periodo de 5 a 10 años hasta que adquirió la capacidad de transmitirse de manera eficiente entre humanos.4,92 Los casos de influenza de los cerdos, identificados de manera simultánea durante la pandemia y con el mismo subtipo de virus H1N1, sugieren la posibilidad de que este animal haya desempeñado un papel importante en la adaptación del virus para infectar a humanos.31,66 El origen del virus que causó esta pandemia se ubicó en Asia y se sugiere que llegó a América traído por aves migratorias a través del Círculo Polar Artico.14
Influenza asiática de 1957
El virus de influenza H1N1 circulaba desde 1918 en la población humana como único virus de influenza. La pandemia de 1957 se inició cuando un virus de origen aviar del subtipo H2N2 aportó, mediante un proceso de reasociación, tres genes al virus H1N1 circulante (variación genética o shift). Los genes donados fueron los de la HA, NA y PB1.93-95 El virus resultante, con nuevas proteínas HA y NA, con las cuales la población humana no había tenido contacto, se difundió con celeridad en dos oleadas que causaron una elevada morbilidad y una mortalidad que, aunque no fue semejante a la de 1918, se calcula que alcanzó de uno a cuatro millones de personas. Como el origen se identificó en Singapur, se le denominó "influenza asiática".1,4,8,61 La alerta temprana de un problema respiratorio con elevada morbilidad en Hong Kong, la pronta identificación y caracterización del nuevo virus de influenza, y el rápido desarrollo y distribución de la vacuna específica contribuyeron a minimizar los efectos de la pandemia.1,96 A partir de 1957 el nuevo virus H2N2 reemplazó al virus H1N1 en todo el orbe.
Pandemia de 1968
Una nueva reasociación ocurrió entre el virus H2N2, circulante en humanos, con un virus aviar del subtipo H3, sin que hasta la fecha se haya identificado el subtipo de NA. Este virus de las aves aportó dos genes al virus circulante (HA y PB1).93-95 El nuevo virus se originó en Hong Kong y produjo una elevada morbilidad, pero limitada mortalidad. La segunda oleada se presentó 12 meses después del pico de la primera8,61 y el total de fallecimientos global se estimó en cuatro millones de personas debido a que, en contraste con la detección temprana del virus de la pandemia anterior, en 1968 la detección tuvo lugar de manera tardía y no se produjo vacuna con oportunidad.1,96 La dominancia del nuevo virus H3N2 en humanos marcó la desaparición mundial del virus H2N2 precedente.
Pandemia de 1977
También se conoce como "la influenza rusa", en la que reapareció el virus del subtipo H1N1. El origen del virus es incierto ya que permaneció oculto por al menos 20 años ya que los estudios de secuenciación indicaron su similitud con el virus H1N1 que circulaba en 1957 y que pudo haber sido liberado por algún laboratorio de manera accidental.1,96,97 El brote se inició en la región sur de Rusia en octubre y para febrero del siguiente año el virus se había difundido a todo el mundo. Debido a que la población mayor de 20 años había tenido contacto previo con el virus el impacto fue mayor en individuos menores de esta edad, en quienes causó una elevada tasa de morbilidad y mortalidad.
Emergencia de la cepa A/Fujian/411/2002 (H3N2)
En la temporada invernal de 2003-2004 los casos de influenza en humanos se incrementaron sustancialmente en el mundo a pesar de la vacunación. La elevada morbilidad en escolares y ausentismo en individuos económicamente activos por enfermedad respiratoria fueron los indicadores iniciales de la pandemia.98-100 La mortalidad fue mayor en sujetos mayores de 60 años y en niños en cuidados maternos. Los países desarrollados, que cuentan con una red centinela de médicos para la notificación de casos con problemas respiratorios, fueron los que alertaron a la comunidad médica mundial sobre el hecho poco usual, y esa voz resultó fundamental para prevenir el impacto de una pandemia.98,100 El virus prevalente en esa temporada se identificó como uno similar a la cepa A/Fujian/411/2002 (H3N2), que no estaba incluida en la fórmula de la vacuna utilizada durante ese periodo. El análisis genético demostró una mutación discreta en dos aminoácidos localizados en el extremo distal de la HA, adyacente al ligando, y que fue la determinante del cambio antigénico (drift) de la cepa A/Panamá/99 (H3N2), recomendada para producir la vacuna que se aplicó durante ese periodo.101 El comité de expertos de la OMS aconsejó la inclusión de esta nueva cepa viral para la fabricación de la vacuna destinada a la temporada invernal 2004-2005.98 Pero durante el invierno de 1998-99 se había presentado otro acontecimiento pandémico en que se identificó la cepa A/Sydney/05/97 (H3N2) como el agente causal.102 Desafortunadamente, las variantes antigénicas que causan estos eventos se identifican en especial durante la temporada invernal, una vez que el programa de vacunación ya se inició, y como no resulta posible elaborar la nueva vacuna de manera inmediata, hay que esperar hasta el siguiente año para incluir la cepa variante o una similar en la nueva vacuna y proteger a la población.1,100,101 En este punto conviene plantear una consideración importante: cuando una variante antigénica emerge en la población, suele ser la cepa dominante, pero las cepas anteriores persisten circulando en combinación con la nueva, por lo que pueden suscitarse múltiples reasociaciones a lo largo del tiempo y generarse diversos linajes dentro de un mismo subtipo de virus.103
Influenza en Asia 1997-2003
A mediados de mayo de 1997 se recibió en un hospital de Hong Kong a un niño de tres años de edad con problemas respiratorios agudos. A pesar del tratamiento médico el niño murió y el 21 del mismo mes, a partir de muestras de la necropsia, se aisló un virus de influenza que no pudo clasificarse con los reactivos comunes que proporciona la OMS. El virus fue enviado para su subtipificación a Holanda y al hospital pediátrico de San Judas, en Memphis, Tennessee, EUA. En agosto del mismo año, el virus fue oficialmente caracterizado como del subtipo H5N1 de origen aviar.104-107 Cuatro meses después se identificaron en Hong Kong 17 individuos más con problemas respiratorios, de quienes se aisló un virus idéntico al que causó la muerte del niño en mayo. Del total de 18 personas infectadas murieron seis (33% de mortalidad).104-105 Al realizar un análisis retrospectivo en la población avícola se encontró que durante los meses de marzo y abril del mismo año se habían notificado casos de influenza aviar (IA) de alta patogenicidad en granjas de Hong Kong y se reconoció el contacto del niño y de los demás pacientes con aves enfermas.108 Los estudios moleculares indicaron que el virus aviar y el virus humano eran genéticamente el mismo.107 En diciembre de 1997 se identificaron tres casos aviares en mercados de aves vivas y uno en granjas. Ante esta situación y con el propósito de eliminar al virus causal, el gobierno tomó la decisión de sacrificar a todas las aves comerciales de la isla (aproximadamente un millón y medio), tanto de los mercados de aves vivas como de granjas, junto con un programa de repoblación de granjas con aves procedentes de zonas libres de IA y la prohibición de criar y mantener juntas aves acuáticas domésticas con gallináceas. De este modo se logró la eliminación del virus potencialmente infeccioso para los humanos.109 El estudio molecular sobre su origen indica que se debió a la reasociación de tres virus que se encontraban circulando en aves silvestres y domésticas (figura 4).

En 1999 se aisló un virus H5N1 a partir de excretas de gansos domésticos, genéticamente diferente al virus de 1997 pero similar a un virus aislado de gansos silvestres en 1996 [A/Goose/Guandong/1/96 (H5N1)].51 Luego se aislaron virus similares en codornices y aves silvestres y, en el año 2000 exclusivamente de aves acuáticas. Los estudios moleculares de los aislamientos de 2001 identificaron seis diferentes genotipos (A, B, C, D, E y X0) generados por la reasociación entre el virus de Guandong con otros virus de aves silvestres y de aves domésticas de diferentes subtipos identificados en los mercados. Las reasociaciones y la presión selectiva originó que en 2002 se identificaran seis nuevos genotipos (Z, Z+, Y, B, W y X0-X3). En 2003 la prevalencia se limitó a tres genotipos (Z, Z+ y V), con lo cual se confirma que la dominancia de un virus reduce la circulación de los otros.21,90 En 2004 sólo se identificó el genotipo Z. Todos los genotipos, con excepción del virus de Guandong y el X0-X3, mostraron una deleción de cinco aminoácidos (posición 80-84) en la proteína NS1. De manera adicional los virus aislados desde 2002, excepto los genotipos B, W y Z+, tuvieron una deleción de 20 aminoácidos en la NA (posición 49-68).21 En febrero de 2003 en Hong Kong se reconoció que el virus del genotipo Z+ causó la muerte de una persona que había viajado al sur de China y que su hija se enfermó del mismo padecimiento aunque se recuperó. Durante el primer trimestre de 2003, China notificó la muerte de un infante con la sospecha de haber sido infectada por el virus H5N1.41 Aparentemente, para 2004 y 2005 sólo el virus del genotipo Z continuaba circulando en humanos90 y está estrechamente relacionado con los virus aviares identificados en la región.41
Epidemia de influenza aviar en Asia 2003-febrero de 2006
El brote en Asia se inició en agosto de 2003 y se considera sin precedente alguno por su fácil diseminación y la extensión geográfica que alcanzó hasta la fecha.110-111 La falta de métodos de diagnóstico apropiados y el comercio insalubre de aves domésticas permitió la difusión del virus con rapidez a diferentes países. En diciembre de 2003 se notificó a la Organización Mundial de Salud Animal (OIE) el caso índice de esta epidemia, que se ubicó en Corea del Sur y que causó una elevada mortalidad en aves comerciales.112 En enero de 2004 se identificó en Vietnam el primer caso de IA. La enfermedad se extendió en tres semanas, con 400 casos reconocidos, aunque afectó a más de tres millones de aves.113 El brote de IA en Vietnam, causado por el virus H5N1, se identificó como la causa de muerte de al menos tres personas en Hanoi, por lo que atrajo la atención de los expertos de la OMS y de los medios de comunicación, que denominaron a la enfermedad "la gripe del pollo".114 En enero de 2004 Japón notificó a la OIE el primer caso de IA causado por virus del subtipo H5N1.115 El 23 de ese mes Tailandia notificó al mismo organismo internacional un brote de influenza en las aves en una granja con 70 000 gallinas. Este mismo país también informó a la OMS de los dos primeros casos de influenza en humanos transmitidos por aves infectadas.113-114 El brote aviar se extendió con rapidez afectando a 32 provincias con una población de 11 millones de gallinas, pollos y patos, que se convirtió en la principal fuente de infección para los humanos. Después, en el mismo mes, se notificaron casos de IA en Camboya, Hong Kong, República Democrática Popular de Laos y China.113-114,116 En este último país el brote afectó a más de 60% de sus 13 mil millones de pollos y gallinas, cuya mayoría se cría en traspatio, en pequeñas granjas rurales al aire libre y que combinan con la crianza de patos. En febrero de 2004, la OIE recibió la notificación de la presencia del mismo virus en Indonesia,113 en donde el efecto a la avicultura fue importante debido a la escasez de recursos para contener el brote. Los virus de IA en Asia infectaron no sólo a humanos, sino también a otras especies como leopardos y tigres,55 otras aves silvestres que no son acuáticas como palomas, cuervos y águilas, han ocasionado mortalidad en aves acuáticas silvestres que no se notificaba desde 1959.90
Estrategias de control de la IA en Asia
Tanto Japón como la República de Corea establecieron de inmediato un programa de sacrificio de las parvadas infectadas, lo que condujo a la eliminación del virus de su avicultura comercial, sin presentación de casos en humanos.117Por su parte, China decidió implementar el sacrificio y un programa nacional de vacunación en las aves; empero, la falta de medidas de bioseguridad y la limitación en el uso de vacunas ha dado como resultado el mantenimiento de la circulación del virus en su avicultura, que terminó por causar infecciones en humanos.114,118 Tailandia optó por el sacrificio de las parvadas afectadas; no obstante, la deficiente notificación de casos volvió difícil el control de la enfermedad, lo que dio como resultado la afección en humanos. Otros países como Camboya, Laos y Vietnam fueron incapaces, por razones económicas, de establecer medidas de control eficientes y surgieron casos de infecciones en humanos en Camboya y Vietnam.119,122 Por el número creciente de casos en Vietnam, en particular en los meses de mayo a agosto de 2005, las autoridades de ese país iniciaron un programa piloto de vacunación de aves en dos provincias que limitó la infección en humanos.123 En Indonesia, los productores comerciales establecieron un programa de sacrificio y vacunación en una campaña gubernamental desde julio de 2004, pero el ocultamiento de la mortalidad, así como la falta de vacunación en pollos de engorda y de medidas de bioseguridad en las granjas, fueron factores que limitaron el control de la enfermedad. El primer caso fatal humano en ese país se notificó durante el mes de agosto de 2005, para luego incrementarse a 25 en seis meses con 18 muertes.114 Omitir la notificación respectiva y la falta de diagnóstico apropiado están contribuyendo a que el virus pueda difundirse con facilidad en los humanos, lo que constituye un factor de riesgo para el incremento de casos e inicio de la pandemia.
El futuro del control de esta enfermedad en la región es incierto.123 El brote aviar se empezó a extender al norte de Asia, Medio Oriente, Europa y Africa, en donde el virus se identificó en primer lugar en aves silvestres, algunos casos en avicultura comercial y de manera eventual ha causado infecciones en humanos,113-114 como lo demuestran las notificaciones que se presentan en el cuadro IV.

Influenza aviar en Holanda, Bélgica y Alemania
En marzo de 2003 se identificó el primer caso de IA en una granja de gallinas de postura en Holanda, donde el virus causal fue del subtipo de alta patogenicidad H7N7.24,25,124 Holanda inició de inmediato un programa de vigilancia tomando muestras de todas las granjas comerciales alrededor del caso índice y el sacrificio de más de 20 millones de aves.25 El brote se extendió a Bélgica y Alemania,125 países que tomaron la misma decisión. Más de 80 personas que estuvieron en contacto con las aves enfermas se infectaron y los síntomas observados variaron de conjuntivitis a problemas respiratorios compatibles con influenza. Un veterinario especialista en aves, que había visitado una de las granjas afectadas en Holanda, murió a causa de bronconeumonía intersticial bilateral aguda y a partir de muestras de necropsia se aisló un virus similar al que causó la epidemia en las aves.24
Influenza aviar en Canadá
En febrero de 2004 un brote de IA por virus del subtipo H7N3 de baja patogenicidad fue reconocido en una granja en el estado de Columbia Británica, en la costa oeste de Canadá.26,27,126 En un periodo de dos semanas la granja vecina notificó una elevada mortalidad y se aisló el virus de alta patogenicidad H7N3. A pesar de la cuarentena establecida, del sacrificio de las aves afectadas y de las medidas de bioseguridad implementadas, el brote se extendió a 42 granjas comerciales en 92 días.126 Las autoridades gubernamentales decidieron el sacrificio y eliminación de las aves de estas granjas y el procesamiento de las aves de 410 granjas localizadas en la zona. Debido a la preocupación sobre la transmisión de virus aviares a humanos el Centro de Control de Enfermedades del estado de Columbia Británica estableció una vigilancia del personal expuesto a las aves afectadas. Los resultaron indicaron que 57 personas cumplieron con los criterios de definición de caso sospechoso de influenza, ya que presentaron conjuntivitis y/o síntomas parecidos a influenza. Sólo en dos de los individuos sospechosos se aisló el virus y no ha habido confirmación serológica de la infección. No se identificaron casos secundarios, lo que sugiere la ausencia de transmisión de persona a persona.26
Influenza aviar en México
El virus de la IA se identificó por primera vez en México en mayo de 1994 y correspondió al subtipo H5N2 de baja patogenicidad. Sin embargo, desde octubre de 1993 se habían observado problemas respiratorios de etiología desconocida en aves comerciales.127 La falta de diagnóstico apropiado por tratarse de una enfermedad exótica permitió que la infección se difundiera con celeridad en las zonas avícolas del país.127-129 A finales de junio del mismo año los estudios epidemiológicos indicaron que las principales zonas productoras de aves en el centro del país se encontraban infectadas, ya que se aisló el virus H5N2 en aves domésticas en los estados de Aguascalientes, Distrito Federal, Guanajuato, Guerrero, Hidalgo, Jalisco, México, Morelos, Puebla, Querétaro, Sonora y Veracruz.126 El gobierno federal estableció un programa novedoso de eliminación de la enfermedad que consistió en que las aves de granjas infectadas se debían procesar en rastros autorizados al final de su ciclo de producción, con lo que se evitó la venta de aves vivas;130 sin embargo, la infección se extendió a otros estados.
En diciembre de 1994 y enero del año siguiente, se identificaron casos de IA de alta patogenicidad en los estados de Puebla y Querétaro.128,130 En respuesta, las autoridades activaron el Dispositivo Nacional de Emergencia en Salud Animal para el control de la IA, que incluyó, entre otras medidas, el sacrificio de las parvadas infectadas y un programa de vacunación para reducir el riesgo en los estados con casos de infección.130 El programa dio como resultado que en mayo de 1995 se eliminara el virus de alta patogenicidad; sin embargo, el virus de baja patogenicidad se ha mantenido en la avicultura comercial.131 El análisis filogenético del virus H5N2 aislado al principio en México muestra un gran parecido con un virus identificado en EUA en aves costeras silvestres, que también se había detectado en avestruces y emúes en los estados de la Florida y Texas.132 Lo anterior genera incertidumbre con respecto a la llegada del virus a México, ya que pudo ser causada por aves silvestres acuáticas migratorias o costeras, o tal vez por el contrabando de aves infectadas. En noviembre de 1994 el virus de baja patogenicidad mutó, se convirtió en virus de alta patogenicidad y circuló hasta junio de 2005. Por su parte, el virus de baja patogenicidad se mantuvo en circulación en algunas regiones avícolas del país133 y se extendió a Guatemala y El Salvador en el año 2000.134-135 Los estudios filogenéticos indican que los virus encontrados en el periodo de 1994 a 1997 se dividen en dos grupos que se denominaron Jalisco y Puebla, pero de 1998 a 2000 fueron sustituidos por dos nuevos linajes denominados A y B. El linaje A agrupa a virus de todo el país y el linaje B a virus de Puebla, Chiapas, Guatemala y El Salvador.136
En México no se ha identificado la transmisión del virus aviar a humanos, por lo que la infección se limita a la población avícola, sin ningún riesgo aparente para la salud humana. El número de casos de IA en este país tiene una variación anual y en 2005 se detectó un incremento de casos en estados considerados libres de la enfermedad (Villareal C, comunicación personal). El control se ha establecido mediante el sacrificio de las parvadas afectadas una vez que concluye su ciclo de producción, es decir, cuando alcanzan el peso para su venta o al final del ciclo de producción de huevo, en establecimientos aprobados por la Secretaría de Agricultura, Ganadería, Pesca y Alimentación (SAGARPA).128,131 Las vacunas contra la IA en México son inactivadas y recombinantes y se requiere autorización de la SAGARPA para su aplicación; la vacunación se realiza en las zonas identificadas bajo control, donde se ha detectado la infección.137
Vacuna de influenza humana
El impacto sobre la morbilidad y la mortalidad humanas ocasionadas por la infección con virus de influenza endémica estacional se puede prevenir con el uso de vacunas.138-140 Desde el brote de influenza de 1946-47 con el subtipo H1N1, que afectó severamente al ejército estadounidense, el Instituto de Investigaciones Médicas Walter Reed, de Washington D. C., inició estudios para proponer estrategias de prevención y control de la siguiente pandemia de influenza.1,4,31 Con el descubrimiento de que la antigenicidad del virus del brote era diferente a la del virus vacunal, se reconoció la variación antigénica, demostrable mediante la apropiada y constante vigilancia epidemiológica por serología y aislamiento viral.138
En la actualidad la OMS cuenta con un sistema global de vigilancia de influenza que le permite identificar variantes antigénicas y virus con potencial pandémico, para lo cual dispone de una red internacional de médicos que informan a lo largo del año sobre el desarrollo de padecimientos respiratorios compatibles con la enfermedad y envían muestras de pacientes a laboratorios y centros internacionales dedicados a aislar y caracterizar los virus.141 La información se concentra en el Centro de Vigilancia de Enfermedades de la OMS, localizado en los CDC de Atlanta, Georgia. Después de analizar los informes anuales, un grupo de expertos de la OMS recomienda las cepas virales que deben incluirse en la elaboración de la vacuna para la siguiente temporada invernal en los hemisferios norte y sur.114 El sistema permite detectar con prontitud variaciones en las tasas de morbilidad y mortalidad, así como variaciones antigénicas de los virus endémicos de influenza. Las mutaciones puntuales que se acumulan en la región antigénica de la HA de los virus de los subtipos H1 y H3 se reconocen y se evalúa su distribución mundial año con año para recomendar las cepas virales capaces de inducir la inmunidad contra los virus circulantes. La OMS emite las recomendaciones durante el primer trimestre del año.114 En el cuadro V se enlistan las cepas virales recomendadas para las temporadas invernales de 1999-2000 a 2004-2005 en el hemisferio norte. Las pruebas de eficacia e inocuidad del producto concluyen para el hemisferio norte aproximadamente en agosto de cada año e inician su distribución en septiembre para que la vacuna se aplique en octubre. La campaña termina en enero del siguiente año, debido a que se recomiendan dos aplicaciones, con lo que se espera que la población se encuentre protegida adecuadamente durante el invierno.142

De acuerdo con la OMS los grupos de mayor riesgo son los infantes de seis a 23 meses, adultos mayores de 65 años o personas de 60 años con enfermedades complicadas.140
En la actualidad sólo nueve países albergan las 14 compañías que producen vacunas contra la influenza humana,142-144 por lo que su disponibilidad es limitada. En países industrializados la vacuna se aplica a todos los individuos que la solicitan, además de la población de alto riesgo. En la mayoría de los países en desarrollo la vacuna no está disponible para toda la población.
Tipos de vacuna contra la influenza endémica estacional
Vacuna inactivada
Es la más utilizada y es trivalente, ya que la constituyen antígenos o virus completos inactivados de los subtipos H1N1, H3N2 y de influenza B.114 Los resultados indican que ofrece una efectividad de 60 a 90% en niños y adultos mayores.1,144 De manera habitual la vacuna se produce en embriones de pollo, de cuyo fluido alantoideo se rescatan los virus que luego se purifican. Este proceso consiste en eliminar los residuos del embrión con el propósito de disminuir las reacciones adversas de hipersensibilidad que en ocasiones se observan en algunos sujetos vacunados.140 El requerimiento internacional establece que la vacuna debe contener 15 µg de HA de cada uno de los virus incorporados y se recomiendan dos aplicaciones para garantizar una mejor respuesta inmune protectora.114,142,144 Desafortunadamente, hasta la fecha no se utilizan adyuvantes en la formulación, a pesar de que los resultados en ensayos piloto indican una mejoría en la producción de anticuerpos aun con la mitad de antígeno de cada uno de los virus de la fórmula.145 Otros estudios han demostrado la factibilidad de reemplazar los embriones de pollo por el cultivo de células para la producción de las vacunas.1
Vacuna de subunidades
En particular, estas vacunas contienen HA y NA y su propósito es concentrar y mejorar la pureza de los antígenos para reducir los efectos secundarios de las vacunas inactivadas. Sin embargo, estas vacunas no son muy eficientes, ya que los estudios realizados muestran un baja inducción de anticuerpos, en especial en infantes de 6.5 meses a tres años de edad. En general se requieren dos o más aplicaciones para alcanzar una adecuada inmunidad y por su pobre reactividad en el sitio de inoculación se recomienda para niños de tres a 12 años de edad.1, 145
Vacuna adaptada al frío
Acaba de registrarse en varios países, incluido México, una vacuna de virus vivo, atenuada y adaptada al frío,2,146-147 que se fabrica por reasociación in vitro a partir de una semilla maestra de virus, la cual contiene seis de los segmentos genómicos con alteraciones estables que limitan su replicación, y a la que se le insertan los genes de HA y NA de los virus recomendados por la OMS para la temporada invernal. Las alteraciones en el virus de la semilla maestra permiten su replicación sólo a 20°C, por lo que se les denomina "virus adaptados al frío".146,148 El producto se administra por vía intranasal en humanos y la infección se limita al tracto respiratorio alto.
En la actualidad el uso de este producto suscita argumentos a favor y en contra.149-150 Por un lado, los argumentos favorables destacan su seguridad, ya que no contiene antígenos de microorganismos aviares presentes en el embrión ni proteínas de aves que en algunos casos inducen sensibilización alergénica. También, subrayan la simplicidad de su aplicación en niños. Las consideraciones en contra señalan que el virus vacunal activo se puede reasociar con virus circulantes y dar origen a nuevos virus con potencial pandémico, además de que el virus de campo podría adquirir genes del vacunal con efectos inciertos en la población. Además, se indica la necesidad de evaluar su utilidad en sujetos inmunodeprimidos, para sopesar su eficacia y posibles efectos adversos. Otra situación contemplada consiste en determinar la eficiencia de la vacuna en sujetos que antes recibieron el mismo producto, a fin de evaluar el efecto de la respuesta inmune generada por vacunaciones previas. Por último, se señala el requerimiento de mantener la cadena fría durante el almacenamiento y transporte del producto para evitar su inactivación.148-150
Vacunas en estudio
Se encuentran en desarrollo otros tipos de vacunas dirigidas a prevenir la influenza endémica. La vacuna de DNA se ha ensayado por más de una década151-152 y en ella se utilizan plásmidos con genes clonados del virus de la influenza (como HA, NA, y NP). En estudios experimentales con ratones vacunados que se desafían con virus homólogo virulento se observa una protección satisfactoria.153 Estas vacunas están en proceso de evaluación en humanos y su aceptación por los comités internacionales de expertos, ya que el uso de DNA puede tener algún riesgo para la población.
Vacuna contra la influenza pandémica
A raíz de los hechos mundiales conocidos en que virus de aves pueden resultar pandémicos, la OMS puso a disposición de los investigadores los distintos aislamientos existentes para desarrollar una vacuna eficiente que prevenga y controle la siguiente pandemia de influenza. Se han publicado trabajos donde se abordan otras estrategias tecnológicas para la generación de vacunas contra la influenza.154-155 Sin embargo, la dificultad estriba en que no se puede predecir con seguridad cuál será el virus causal de la próxima pandemia. Los candidatos posibles son virus de los subtipos H5 o H9, que circulan en Asia y Medio Oriente, o el H7, que afecta a países de Europa y a Canadá.144 También existe el riesgo de que se reintroduzca un virus conocido, como el que causó la alarma mundial en marzo de 2005. Esta se debió al descuido y desconocimiento del riesgo potencial para la salud pública de una empresa norteamericana de certificación de control de calidad, que distribuyó a 18 países en América, Europa, Medio Oriente y Asia un estuche con virus activo del subtipo H2N2, inexistente desde 1968 en la población humana.156
A lo anterior hay que agregar que hoy por hoy existe un limitado número de laboratorios fabricantes de la vacuna, que serían insuficientes para proveer a la población en riesgo, e incluso para cubrir las necesidades propias de los países productores. A ello se suma el tiempo requerido para su desarrollo, que en el mejor de los casos sería de al menos siete meses, sin considerar los posibles problemas técnicos que puedan surgir durante su proceso de escalamiento y producción.141,142,144 También habría que definir los grupos en riesgo para recibir la vacuna y los costos de producción y compra del producto. Esta situación puede originar una presión de la opinión pública y pánico por el desabasto.
Los planes de emergencia elaborados en estos países prescriben la aplicación de la vacuna endémica estacional, que poca o ninguna protección brindaría en caso de una pandemia con un nuevo virus. Las previsiones respecto al uso de una vacuna pandémica se limitan a la población de niños y adultos mayores por considerarlos de manera errónea como de mayor riesgo, ya que en caso de una pandemia por un nuevo virus todos los individuos deben considerarse en riesgo, según lo dicta la experiencia de pandemias anteriores.1,2,31,45,96 En marzo de 2006 se publicó un trabajo que describe el desarrollo y las pruebas clínicas realizadas con una vacuna inactivada contra el virus H5N1.157 Esta vacuna puede ser la más adecuada para la prevención de la ya tan anunciada pandemia.157-158 La vigilancia de la evolución de los virus de influenza, tanto en humanos como en animales, debe constituir una actividad primordial para detectar con prontitud variaciones antigénicas o de transferencia entre especies y así definir las acciones a seguir. Los hechos biológicos no son constantes, sino que, por el contrario, representan situaciones impredecibles por el comportamiento biológico tan variable de los microrganismos, aun cuando se trate de individuos del mismo género y especie.159 El concepto de pandemia no debe ser alarmista sino más bien orientador de intereses para estructurar de manera científica y logística las acciones que se deben emprender para evitarla.
Agentes antivirales
El uso de medicamentos antivirales contra la influenza humana es una alternativa a considerar en el control de la enfermedad. Hasta el momento existen cuatro medicamentos antivirales que han sido utilizados en el control de la infección.160-161 Se trata de la amantadina y la rimantadina, que bloquean al canal iónico que forma la proteína M2 en la envoltura viral, de manera que evita el cambio de pH requerido para separar la nucleoproteína de los segmentos genómicos durante el proceso de desnudamiento y la fusión de la envoltura vírica con la membrana endosómica.32,62 Los inconvenientes para su uso provienen de la rápida resistencia que se produce a la acción de ambos medicamentos y de la producción de efectos colaterales, que en el caso de la amantadina se verifican en el sistema nervioso central y el hígado, y en el de la rimantadina, en los riñones.161 La rimantadina puede utilizarse en una dosis diaria en lugar de dos con amantadina. Además, la rimantadina expone a menos reacciones adversas. La dosis recomendada en el adulto es de 200 mg diarios para ambos medicamentos y de 5 mg por kg de peso sin exceder 150 mg de dosis total en niños; también puede administrarse la misma dosis como medida profiláctica en sujetos de alto riesgo.162
Los estudios realizados para demostrar la efectividad de estos medicamentos en infecciones por virus del subtipo H5N1 de Asia mostraron una sensibilidad variable y se detectaron cepas resistentes a ambos medicamentos.163-168
En fecha reciente se desarrollaron y registraron nuevos medicamentos para el control de la influenza164-166 que se basan en el bloqueo de la neuraminidasa. Las moléculas son inhibidores competitivos análogos del AS que compiten con éste para prevenir la liberación de la partícula viral y su posterior distribución en el tracto respiratorio. Se han fabricado dos sustancias denominadas oseltamivir y zanamivir que se administran por vía oral y en aerosol intranasal, respectivamente, y ambas han demostrado eficiencia para el control de infecciones por virus de los subtipos H1 y H3, así como efecto variable con el virus H5N1.163 El oseltamivir se administra a dosis de 75 mg en individuos de trece años en adelante o mayores de 40 kg de peso, para fines profilácticos y terapéuticos.160 El zanamivir se recomienda en disparos nasales a dosis de 10 mg diarios en una sola aplicación, cuando es profiláctico, o en dos aplicaciones de 5 mg cada una en pacientes infectados. Este medicamento mostró resultados variables en personas infectadas por el virus H5N1, aunque resultó efectivo en animales con infección experimental.162
Los costos estimados por paciente con el uso de antivirales oscilan de 30 a 70 dólares americanos diarios. El tratamiento completo se extiende en promedio de 10 a 15 días.
Periodos de incubación y clínico en infecciones humanas por virus H5N1
El periodo de incubación en humanos infectados por el virus H5N1 suele ser más largo que el habitual por virus de influenza endémicos y en promedio dura entre dos y cuatro días, con un rango de hasta ocho días.19,122
La mayoría de los pacientes presenta fiebre (mayor de 38°C) y de manera habitual no desarrolla conjuntivitis. La diarrea acuosa, vómito, dolor abdominal y pleural, así como epistaxis y gingivorragia son manifestaciones comunes en los estados iniciales de la enfermedad.19,163 Algunos pacientes pueden exhibir compromiso del sistema nervioso central. La disnea se presenta en un periodo de 1 a 16 días, con una media de cinco, con inspiraciones dolorosas. La presencia de esputo es variable y algunas veces acusa un aspecto sanguinolento. Casi todos los pacientes desarrollan neumonía con cambios radiográficos que muestran infiltrados multifocales o difusos y consolidación lobular; estos cambios son ostensibles en pacientes de 3 a 17 días de curso infeccioso, con una media de siete días. La insuficiencia respiratoria sobreviene a consecuencia de infiltrados bilaterales difusos. La afectación renal y cardiaca, así como la neumonía, hemorragia pulmonar, neumotórax y el síndrome séptico forman parte del cuadro clínico que acompaña a las fases finales de la infección.162
Diagnóstico de laboratorio
Los resultados de laboratorio muestran leucopenia, en particular linfopenia con ligera o moderada trombocitopenia, una ligera o moderada elevación de aminotransferasas, marcada hiperglicemia, tal vez secundaria al uso de corticoesteroides, y creatinina elevada. Son de pronóstico grave la leucopenia, linfopenia y trombocitopenia al ingreso del paciente.19,162
Se pueden realizar pruebas rápidas específicas para la identificación del virus, conocidas como Directigen, pero el diagnóstico confirmatorio depende del aislamiento del virus o de la identificación de ácidos nucléicos por pruebas de RT-PCR.169-173 Las muestras más convenientes son las de faringe, comparadas con las nasales y traqueales. También ha sido posible realizar el aislamiento a partir de muestras de heces. Asimismo el virus ha sido aislado de sangre, suero, heces y líquido cefalorraquídeo.162 El virus se puede demostrar en las muestras faríngeas mediante pruebas de inmunofluorescencia o inmunoperoxidasa con anticuerpos monoclonales para el tipo y subtipo de virus (A/H5N1).19
Cuidados del paciente
En el mismo momento de su ingreso los pacientes sospechosos deben ser aislados. Mientras se realizan las pruebas confirmatorias de la infección se inicia el tratamiento con antivirales y una terapia de apoyo con oxígeno o ventilación. A este respecto, se contraindica el uso de nebulizadores para evitar los aerosoles que contribuyen a diseminar la infección.169-170 El empleo de antibióticos es recomendable para prevenir infecciones secundarias, mientras que los corticoesteroides han demostrado ejercer efectos negativos en el tratamiento. Los familiares en contacto previo con el paciente deben ser instruidos sobre la enfermedad para que asistan a la unidad médica en caso de presentar síntomas de diarrea, dificultad para respirar y dolor abdominal o pleural.162
Planes de preparación y respuesta ante una pandemia
Después del incidente de Hong Kong en 1997, la OMS promovió con mayor esfuerzo que todos los países establecieran procedimientos a seguir en caso de presentarse una pandemia.173 La propuesta del plan consiste en que los países constituyan el marco jurídico y ético necesario para implementar las medidas de control que se determinen para cada una de las fases. Las fases determinadas por la OMS se describen en el cuadro VI. El plan entraña definir la coordinación y los niveles de actuación de las diferentes entidades en las distintas fases, y debe centrarse en la vigilancia epidemiológica para la identificación oportuna de potencial pandémico en animales y humanos; el logro de lo anterior requiere el fortalecimiento de la infraestructura diagnóstica en el país y la detección en puertos y fronteras. Este debe estar conectado a la red internacional de información sobre influenza para evaluar los riesgos del ingreso de cualquier virus con potencial pandémico identificado en cualquier lugar del mundo. Los países deben conformar y ubicar las unidades médicas que han de servir para el aislamiento de pacientes sospechosos, con el fin de reducir la posibilidad de transmisión de la enfermedad. Además, deben asegurar una reserva estratégica de vacunas y medicamentos antivirales para el momento que se requieran y han de establecer un plan de actualización médica para concientizar a los trabajadores de la salud sobre la enfermedad, así como a los diversos sectores gubernamentales para que actúen en las diferentes fases. Además, deben contar con sistemas de comunicación para informar a la población con objetividad durante todas las fases determinadas.174 En relación con todo ello, México ha establecido un plan para enfrentar la pandemia en cuanto se reconozcan los casos.175
Conclusiones
La influenza común estacional es una infección que se puede prevenir mediante el uso adecuado de vacunas, sobre todo, en las poblaciones en riesgo. Los virus de influenza endémicos mutan con facilidad, por lo que es necesario adecuar anualmente las vacunas con los virus circulantes para su prevención.
La influenza pandémica se origina por la transmisión eficiente de un virus nuevo en un hombre que carece de inmunidad para controlarla. Supone una emergencia de salud pública con implicaciones políticas, sociales y económicas, por lo que todos los sectores de la población deben participar en los planes de preparación y cualquier decisión al respecto se debe tomar sobre la base del conocimiento científico.
La introducción de virus de aves acuáticas silvestres a los humanos requiere un proceso complicado de adaptación viral en hospederos intermediarios y de evolución genética para lograr la eficiente transmisión entre humanos.
El virus H5N1 que circula en la población de aves domésticas en los países asiáticos ha afectado a 170 personas con 92 casos fatales al 20 de febrero de 2006; la OMS ubica la situación en la fase interpandémica con tendencia al periodo 3, que se caracteriza por la transmisión del virus entre humanos. La infección en Asia ha tenido tres etapas y en la última la distribución alcanzó a algunos países de Europa y África.
Otros virus de influenza de los subtipos H5, H7 y H9 han ocasionado infecciones de menor impacto en humanos.
Al momento no existe una vacuna para ser aplicada en caso de una pandemia con virus de influenza. Es conveniente, por tanto, fortalecer y apoyar la investigación básica y aplicada y la infraestructura para el estudio de la influenza en México, en particular la formación de recursos humanos y el entrenamiento técnico que coadyuve a apoyar las acciones de prevención y control, así como a motivar el desarrollo de vacunas para humanos a través de la colaboración entre las instituciones mexicanas.
Referencias
1. Hilleman MR. Realities and enigmas of human viral influenza: pathogenesis, epidemiology and control. Vaccine 2002;20:3068-3087. [ Links ]
2. Cox NJ, Subbarao K. Influenza. Lancet 1999;354:1277-1282. [ Links ]
3. Simonsen L. The global impact of influenza on morbidity and mortality. Vaccine 1999;17:S3-S10. [ Links ]
4. Webster RG, Bean WJ, Goren OT, Chambers TM, Kawaoka Y. Evolution and ecology of influenza A viruses. Microbiol Rev 1992;56:53-179. [ Links ]
5. Webby RJ, Webster RG. Emergence of influenza A viruses. Phil Trans R Soc Lond B 2001;356:1817-1828. [ Links ]
6. Webster RG, Laver WG, Air GM. Antigenic variation among influenza type A influenza viruses. En: Palese P, Kingsbury DW, ed.: Genetics of influenza viruses. New York: Springer, 1983:127-168 [ Links ]
7. Hampson AW, Cox NJ. Global surveillance for pandemic influenza: are we prepared. En: Brown LE, Hampson AW, Webster RD, ed.: Options for the control of influenza. Amsterdam: Elsevier, 1996:50-59. [ Links ]
8. Hoy AJ, Gregory V, Douglas AR, Lin YP. The evolution of human influenza viruses. Philos Trans R Soc Lond B 2001;356:1861-1870. [ Links ]
9. Claas EC, Osterhaus ADME, van Beek R, DeJong JC, Rimelzwaan GF, Senne DA et al. Human influenza A H5N1 virus related to a highly pathogenic avian influenza virus. Lancet 1996;351:472-477. [ Links ]
10. Cox NJ, Subbarao K. Global epidemiology of influenza: past and present. Annu Rev Med 2000;51:407-421. [ Links ]
11. Webster RG. Spanish influenza: the secrets remain. Proc Natl Acad Sci USA 1999;96:1164-1166. [ Links ]
12. Kolata G. Flu: the story of the great influenza pandemic of 1918 and the search for the virus that caused it. New York: Farrar, Straus and Giroux, 1999:330. [ Links ]
13. Murphy BR, Webster RG. Orthomyxoviruses. En: Knipe DM, Howley PM, ed. Fields virology vol. 1. 3a. ed. Philadelphia: Lippincott-Raven, 1996:1397-1445. [ Links ]
14. Viesca TC. Medicina prehispánica de México. México, Panorama Editorial,1996;49-50. [ Links ]
15. Valdez AR. Pandemia de gripe. Sinaloa, 1918-1919. Elementos 2002;47:37-43. [ Links ]
16. Hinshaw VS, Webster RG. The natural history of influenza A viruses. En: AS Beare ed.: Basic and applied influenza research. Boca Raton, Fla: CRC Press, Inc, 1982;79-104. [ Links ]
17. Hilleman MR. Asian influenza: initial identification of Asiatic virus and antibody response in volunteers to vaccination. En: US Department of Health, Education and Welfare. Proceedings of the Special Conference on Influenza. Washington, DC: USDHEW, 1957;17-24. [ Links ]
18. Lindstrom SE, Cox NJ, Klimov A. Genetic analysis of human H2N2 and early H3N2 influenza viruses, 1957–1972: Evidence for genetic divergence and multiple reassortment events. Virology 2004;328:101-119. [ Links ]
19. Yuen KY, Chan PK, Peiris M, Tsang DN, Que TL, Shortridge KF et al. Clinical features and rapid viral diagnosis of human disease associated with avian influenza A H5N1 virus. Lancet 1998;351:467-471. [ Links ]
20. Claas EC, Osterhaus AD, Van Beek R, De Jong JC, Rimmelzwaan GF, Senne DA et al. Human influenza A H5N1 virus related to a highly pathogenic avian influenza virus. Lancet 1998;351:472-477. [ Links ]
21. Li KS, Guan Y, Wang J, Smith GJ Xu KM et al. Genesis of a highly pathogenic and potentially pandemic H5Nl influenza virus in eastern Asia. Nature 2004;430:209-213. [ Links ]
22. Shortridge KF, Peiris JSM, Guan Y. The next influenza pandemic: lessons from Hong Kong. J Appl Microbiol 2003;94;70S-79S. [ Links ]
23. Peiris M, Yuen KY, Leung CW, Chan KH, Ip PL, Lai RWM et al. Human infection with influenza H9N2. Lancet 1999;354:916-917. [ Links ]
24. Kemink SA, Fouchier RA, Rozendaal FW, Broekman JM, Koopmans M, Osterhaus AD et al. A fatal infection due to avian influenza-A (H7N7) virus and adjustment of the preventive measures. Ned Tijdschr Geneeskd 2004;148:2190-2194. [ Links ]
25. Stegeman A, Bouma A, Elbers AR, de Jong MC, Nodelijk G, de Klerk F et al. Avian influenza A virus (H7N7) epidemic in The Netherlands in 2003: course of the epidemic and effectiveness of control measures. J Infect Dis 2004;190:2088-2095. [ Links ]
26. Tweed SA, Danuta MS, Samara TD, Andrew L, Petric M, Wayne L et al. Human illness from avian influenza H7N3, British Columbia S. Emerg Infect Dis 2004;10:2196-2199. [ Links ]
27. Hirst M, Astell CR, Griffith M, Coughlin SM, Moksa M, Zeng T et al. Novel avian influenza H7N3 strain outbreak, British Columbia. Emerg Infect Dis 2004;10:2192-2195. [ Links ]
28. Campitelli L, Mogavero E, De Marco MA, Delogu M, Puzelli S et al. Interspecies transmission of an H7N3 influenza virus from wild birds to intensively reared domestic poultry in Italy. Virology 2004;323:24-36. [ Links ]
29. Puzelli S, Di Trani L, Fabiani C, Campitelli L, De Marco MAl, Capua I et al. Serological analysis of serum samples from humans exposed to avian H7 influenza viruses in Italy between 1999 and 2003. J Infect Dis 2005;192:1318-1322. [ Links ]
30. Ministry of Health, Labour and Welfare of Japan disponible en: <http://www.mhlw.go.jp/houdou/2006/01/h0110-4.html>
31. Wright PF, Webster RG. Orthomyxoviruses. En: Knipe DM, Howley PM, ed Fields virology. vol. 1. 4a. ed. Philadelphia: Lippincott Williams and Wilkins, 2001:1533-1579. [ Links ]
32. Lamb RA. Genes and proteins of the influenza viruses. En: Krug RM, Fraenkel-Conrat H, Wagner RR, ed. The influenza viruses. New York, Plenum Press, 1989:1-88. [ Links ]
33. Smith W, Andrewes CH, Laidlaw PP. A virus obtained from influenza patients. Lancet 1933;2:66-68. [ Links ]
34. Francis Jr T. A new type of virus from epidemic influenza. Science 1940;92:405-408. [ Links ]
35. Osterhaus AD, Rimmelzwaan GF, Martina BE, Bestebroer TM, Fouchier RA. Influenza B virus in seals. Science 2000;288:1051-1053. [ Links ]
36. Francis Jr T, Quilligan Jr JJ, Minuse E. Identification of another epidemic respiratory disease. Science 1950;112:495-497. [ Links ]
37. Kimura H, Abico C, Peng G, Muraki Y, Sugawara K, Hong S et al. Interspecies transmission of influenza C virus between humans and pigs. Virus Res 1997;48:71-79. [ Links ]
38. Yamaoka M, Hotta H, Itoh M, Homma M. Prevalence of antibody to influenza C virus among pigs in Hyogo Prefecture, Japan. J Gen Virol 1991;72:711-714. [ Links ]
39. Ohwada K, Kitame F, Sugawara K, Nishimura H, Homma M, Nakamura K. Distribution of antibody to influenza C virus in dogs and pigs in Yamagata prefecture, Japan. Microbiol Immunol 1978;31:1173-1180. [ Links ]
40. WHO Memorandum. A revised system of nomenclature for influenza viruses. Bull WHO 1980;58:585-591. [ Links ]
41. Guan Y, Peiris JS, Lipatov AS, Ellis TM, Dyrting KC et al. Emergence of multiple genotypes of H5N1 avian influenza viruses in Hong Kong SAR. Proc Natl Acad Sci USA 2002;99:8950-8955. [ Links ]
42. Webster RG, Peiris M, Chen H, Guan Y. H5N1 outbreaks and enzootic influenza. Emerg Infect Dis 2006;12:3-8. [ Links ]
43. Spackman E, Senne DA, Davison S, Suarez DL. Sequence analysis of recent H7 avian influenza viruses associated with three different outbreaks in commercial poultry in the United States. J Virol 2003;77:13399-13402. [ Links ]
44. Dennis JA. A review of avian influenza in different bird species. Vet Microbiol 2000;74:3-13. [ Links ]
45. Taubenberger JK, Morens DM. 1918 influenza: the mother of all pandemics. Emerg Infect Dis 2006;12:15-22. [ Links ]
46. Kawaoka Y, Yamnikova S, Chambers TM, Lvov DK, Webster RG. Molecular characterization of a new hemaglutinin subtype H14 of influenza virus A. Virology 1990;179:759-767. [ Links ]
47. Röhm C, Zhou N, Suss J, Mackenzie J, Webster RG. Characterization of a novel influenza hemagglutinin, H15. Criteria for determination of influenza subtypes. Virology 1996;217:508-516. [ Links ]
48. Fouchier RA, Munster V, Wallensten A, Bestebroer TM, Herfst S, Smith DR et al. Characterization of a novel influenza A virus hemagglutinin subtype (H16) obtained from black-headed gulls. J Virol 2005;79:2814-2822 . [ Links ]
49. Suarez DL. Evolution of avian influenza viruses. Vet Microbiol 2000;74:15-27. [ Links ]
50. Kennedy FS, Gao P, Guan Y, Ito T, Kawaoka Y, Markwell D et al. Interspecies transmission of influenza viruses: H5N1 virus and Hong Kong SAR perspective. Vet Microbiol 2000;74:141-147. [ Links ]
51. Geraci JR, St Aubin DJ, Barker IK, Webster RG, Hinshaw VS, Bean WJ et al. Mass mortality of harbor seals: pneumonia associated with influenza A virus. Science. 1982;215:1129-1131. [ Links ]
52. Hinshaw VS, Bean WJ, Geraci JR, Fiorelli P Early G et al. Characterization of two influenza A viruses from a pilot whale. J Virol 1986;58:655-656. [ Links ]
53. Gaydos JC, Hodder RA, Top FH. Swine influenza A at Fort Dix, New Jersey (January-February 1976). IIl transmission and morbidity in units with cases. J Infect Dis 1977;136:363-368. [ Links ]
54. Sovinova O, Tumova B, Pouska F, Nemec J. Isolation of a virus causing respiratory disease in horses. Acta Virol 1958;2:52-61. [ Links ]
55. Keawcharoen J, Oraveerakul K, Kuiken T, Fouchier RAM, Amonsin A, Payungporn S et al. Avian influenza H5N1 in tigers and leopards. Emerg Infect Dis 2004;10:2189-2191. [ Links ]
56. Crawford PC, Dubovi EJ, Castlemen WL, Stephenson I, Gibbs EP, Chen L et al. Transmission of equine influenza virus to dogs. Science 2005;310:482-485. [ Links ]
57. Olsen CW, Brammer L, Easterday BC, Arden N, Belay E, Baker I et al. Serological evidence of H1 swine influenza virus infection in swine farm residents and employees. Emerg Infect Dis 2002;8:814-819. [ Links ]
58. Brown IH. The epidemiology and evolution of influenza viruses in pigs. Vet Microbiol 2000;74:29-46. [ Links ]
59. Chen W, Calvo PA, Malide D, Gibbs J, Shubert U, Bacik I et al. A novel influenza A virus mitochondrial protein that induces cell death. Nat Med 2001;7:1306-1312. [ Links ]
60. Colman PM. Neuraminidase; enzyme and antigen. En: Krugh RM, ed. The influenza viruses. New York: Penum Press, 1989:175-218. [ Links ]
61. Zebedee FL, dhi RA, Influenza A virus M2 protein: monoclonal antibody restriction of virus growth and detection of M2 in virions. J Virol 1988;62:2762-2772. [ Links ]
62. Horimoto T, Kawaoka Y. Influenza lessons from past pandemics, warnings from current incidents. Nat Rev Microbiol 2005;3:591-600. [ Links ]
63. Webster RG. Antigenic variation in influenza viruses. En: Domingo E, Webster R, Holland J, ed. Origin and evolution of viruses. San Diego, CA: Academic Press, 1999:377-390. [ Links ]
64. Webster RG, Hulse DJ. Microbial adaptation and change: avian influenza. Rev Sci Tech 2004;23:453-465. [ Links ]
65. Alexander DJ. A review of avian influenza in different bird species. Vet Microbiol 2000;74:3-13. [ Links ]
66. Webster RG. Influenza viruses. En: Webster RG, Granoff A, ed. Encyclopedia of virology. New York: Academic Press, 1994;2:709-715. [ Links ]
67. Holland J, Spindler K, Horodyski F, Grabau E, Nichol E, VandePol S. Rapid evolution of RNA genomes. Science 1982;215:1577-1585. [ Links ]
68 Webby RJ, Webster RG. Emergence of influenza A viruses. Philos Trans R Soc Lond B Biol Sci 2001;356:1816-1828. [ Links ]
69. Kawaoka J, Krauss YS, Webster RG. Avian-to-human transmission of the PB1 gene of influenza A viruses in the 1957 and 1968 pandemics. J Virol 1989;63:4603-4608. [ Links ]
70. Weis W, Brown JH, Cusack S, Paulson JC, Skehel JJ, Wiley DC. Structure of the influenza virus haemagglutinin complexed with its receptor, sialic acid. Nature 1988;333:426-431. [ Links ]
71. Rogers GN, Paulson JC. Receptor binding properties of human and animal H1 influenza virus isolates. Virology 1989;173:317-322. [ Links ]
72. Ha Y, Stevens DJ, Skehel JJ, Wiley DC. X-ray structures of H5 and H9 swine influenza virus hemagglutinins bound to avian and human receptor analogs. Proc Nat Acad Sci USA 2001;98:11181-11186. [ Links ]
73. Rogers GN, Pritchet TJ, Lane JL, Paulson JC. Differential sensitivity of human, avian and equine influenza A viruses to a glycoprotein inhibitor of infection: selection of receptor specific variants. Virology 1983;131:394-408. [ Links ]
74. Castrucci MR, Donatelli I, Sidoli L, Barigazzi, Kawaoka Y, Webster RG. Genetic reassortment between avian and human influenza A viruses in Italian pigs. Virology 1993;193:503-506. [ Links ]
75. Connor RJ, Kawaoka Y, Webster RG, Paulson J. Receptor specificity in human, avian and equine H2 and H3 influenza viruses isolates. Virology 1994;205:17-23. [ Links ]
76. Sizuki Y. Sialobiology of influenza molecular mechanisms of host range variation of influenza viruses. Biol Pharm Bull 2005;28:399-408. [ Links ]
77. Ito T, Kawaoka Y. Host range barrier of influenza A viruses. Vet Microbiol 2000;74:71-75. [ Links ]
78. Conn CA, McClellan Jl, Maasab HF, Smitka CW, Majde JA, Kluger MJ. Cytokines and the acute phase response to influenza virus in mice. Am J Physiol 1995;268:78-84. [ Links ]
79. Bielefeldt-Ohmann H. Role of cytokines in the pathogenesis and treatment of respiratory diseases. En: Myers MJ, Murtaugh MP, ed. Cytokines in animal health and disease. New York: Marcel Dekker, 1995:291-332. [ Links ]
80. Falcon AM, Fernandez-Sesma, Nakaya Y, Moran TM, Ortiz J et al. Attenuation and immunogenicity in mice of temperature-sensitivity influenza viruses expressing truncated NS1 proteins. J Gen Virol 2005;86:2817-2821. [ Links ]
81. Taylor HP, Dimmock NJ. Mechanism of neutralization of influenza virus by secretory IgA is different from that of monomeric IgA or IgG. J Exp Med 1985;161:198-209. [ Links ]
82. Suarez DL, Schultz-Cherry S. Immunology of avian influenza virus: a review. Dev Comp Immunol 2000;24:269-283. [ Links ]
83. Karson DT. Cytotoxic T cells in influenza immunity. Semin Virol 1996;6:906-908. [ Links ]
84. Alexander DJ. Highly pathogenic avian influenza. En: OIE. Manual of standars for diagnostic tests and vaccines. 4a. edición. París: OIE, 2004:212-220 [ Links ]
85. Perroncito E. Epizoozia tifoide nei gallinnacei. Annali Accademia Agriculture Torino 1878;21:87-126. [ Links ]
86. Capua I, Alexander DJ. Avian influenza: recent developments. Avian Pathol 2004;33:392-404. [ Links ]
87. Kawaoka Y, Webster RG. Sequence requirements for cleavage activation of influenza virus hemagglutinin expressed in mammalian cells. Proc Natl Acad Sci USA 1988;85:324-328. [ Links ]
88. Perdue ML, Suarez DL. Structural features of avian influenza virus hemagglutinin that influence virulence. Vet Microbiol 2000;74:77-86. [ Links ]
89. Bean WJ, Kawaoka Y, Wood JM, Pearson JE, Webster RG. Characterization of virulent and avirulent A/chiken/Pennsylvania/83 influenza A viruses: potential role of defective interfering RNAs in nature. J Virol 1985;54:151-160. [ Links ]
90. Guan Y, Poon LLM, Cheung CY, Ellis TM, Lim W et al. H5N1 influenza: a protean pandemic threat. Proc Natl Acad Sci USA 2004;101:8156-8161. [ Links ]
91. Crosby AW. America´s forgotten pandemic: the influenza of 1918. Cambridge: Cambridge University Press, 1989:329. [ Links ]
92. Taubenberger JK, Reid AH, Janczewsky TA, Fanning TG. Integrating historical clinical and molecular genetic data in order to explain the origin and virulence of the 1918 Spanish influenza virus. Phil Trans R Soc London B Biol Sci 2001;356:1829-1839. [ Links ]
93. Belshe RB. Origins of pandemic influenza lessons from 1918 virus. N Engl J Med 2005;353:2209-2211. [ Links ]
94. Scholtissek C, Rohde W, von Hoyningen V, Rott R. On the origin of the human influenza virus subtype H2N2 and H3N2. Virology 1978;87:13-20. [ Links ]
95. Kawaoka Y, Krauss S, Webster RG. Avian-to-human transmission of the PB1 gene of influenza A viruses in the 1957 and 1968 pandemics. J Virol 1989;63:4603-4608. [ Links ]
96 Hileman MR. Personal historical chronicle of six decades of basic and applied research in virology, immunology, and vaccinology. Immunol Rev 1999;170:7-27. [ Links ]
97. Nakajima K, Desselberger U, Palese P. Recent human influenza A viruses are closely related genetically to strains isolated in 1950. Nature 1978;274:334-339. [ Links ]
98. World Health Organization. Wkly. Epidemiol Rec 2003;9:57-64. [ Links ]
99. Lu B, Zhou H, Ye D, Kemble G, Jin H. Improvement of influenza A/Fujian/411/02 (H3N2) virus growth in embryonated chicken eggs by balancing the hemagglutinin and neuroaminidase activities, using reverse genetics. J Virol 2005;79: 6763-6771. [ Links ]
100. Holmes EC, Ghedin E, Lipman DJ, Taubenberger JK. Whole-genome analysis of human influenza A virus reveals multiple persistent lineages and reassortment among recent H3N2 viruses. PLoS Biology 2005;3:1579-1589. [ Links ]
101. Jin H, Zhou H, Liu H, Chan W, Adhikary L et al. Two residues in the hemagglutinin of A/Fujian/411/02-like influenza viruses are responsible for antigenic drift from A/Panama/2007/99. Virology 2005;336:113-119. [ Links ]
102. Rao BL, Kadam SS. Isolation of recent variant influenza types A (H3N2) & B strains in Pune, India, during 1998. Indian J Med Res 2000;111:3-5. [ Links ]
103. Lindstrom SE, Cox NJ, Klimov A. Genetic analysis of human H2N2 and early H3N2 influenza viruses, 1957-1972: evidence for genetic divergence and multiple reassortment events. Virology 2004; 328:101-109. [ Links ]
104. CDC. Isolation of avian influenza A (H5N1) viruses from humans; Hong Kong, May-December 1997. MMWR 1997;46:1204-1207. [ Links ]
105. Subbarao K, Klimov A, Katz J, Regnery H, Lim W et al. Characterization of an avian influenza A (H5N1) virus isolated from a child with fatal respiratory illness. Science 1998;279:393-396. [ Links ]
106. Claas EC, Osterhaus AD, Van Beek R, De Jong JC; Rimeelzwaan GF, et al. Human influenza A H5N1 virus related to highly pathogenic avian influenza virus. Lancet 1998;351:472-477. [ Links ]
107. Suarez DL, Perdue ML, Cox N, Rowe T, Bender C et al. Comparisons of highly virulent H5N1 influenza A viruses isolated from humans and chickens from Hong Kong. J Virol 1998;72:6678-6688. [ Links ]
108. Shortridge KF, Zhpu NN, Guan Y, Gao P, Ito T et al. Characterization of avian H5N1 influenza viruses from poultry in Hong Kong. Virology 1998;252:331-342. [ Links ]
109. Webster RG. Influenza: An emerging disease. Emerg Infect Dis 1998;4:436-441. [ Links ]
110. Martin V, Sims L, Lubroth J, Pfeiffer D, Slingenbergh J et al. Epidemiology and ecology of highly pathogenic avian influenza with particular emphasis on South East Asia. Dev Biol 2006:124:23-36. [ Links ]
111. Webster RG, Guan Y, Poon L, Krauss S, Webby R et al. The spread of H5N1 bird flu epidemic in Asia in 2004. Arch Virol 2005;19:117-129. [ Links ]
112. World Organization for Animal Health (OIE). Highly pathogenic avian influenza in the Republic of Korea. Suspected outbreak. Disease information 2003;16: 270. Disponible en: ftp://ftp.oie.int/infos–san–archives/eng/2003/en–031212v16n50.pdf [ Links ]
113. World Organization for Animal Health (OIE). Update on avian influenza in animals (type H5). Disponible en: http://www.oie.int/downld/AVIAN%20INFLUENZA/A_AI-Asia.htm [ Links ]
114. World Health Organization Epidemia and pandemic alert and response (EPR) Avian Influenza. Disponible en: http://www.who.int/csr/disease/avian_influenza/en/index.html [ Links ]
115. Mase M, Tsukamoto K, Imada T, Imai K, Tanimura N et al. Characterization of H5N1 influenza A viruses isolated during the 2003-2004 influenza outbreaks in Japan. Virology 2005;5:167-176. [ Links ]
116. Desvaux S, Sorn S, Holl D, Chavernac D. Goutard F et al. HPAI surveillance programme in Cambodia: results and perspectives. Dev Biol 2006;124:211-224. [ Links ]
117. Mase M, Kim JH, Lee YJ, Tsukamoto K, Imada T et al. Genetic comparison of H5N1 influenza A viruses isolated from chickens in Japan and Korea. Microbiol Immunol 2005;49:871-874. [ Links ]
118. Alexander DJ. Avian influenza viruses and human health. Desv Biol 2006;124:77-84. [ Links ]
119. Snacken R, Kendal AP, Haaheim LR, Wood JM. The next influenza pandemic: lessons from Hong Kong, 1997. Emerg Infect Dis 1999;5:195-203. [ Links ]
120. World Health Organization Global Influenza Program Surveillance Network. Evolution of H5N1 avian influenza viruses in Asia. Emerg Infect Dis 2005;11:1515-1521. [ Links ]
121. Thorson A, Petzold M, Nguyen TK, Ekdahl K. Is exposure to sick or dead poultry associated with flulike illness?: a population-based study from rural area in Vietnam with outbreaks of highly pathogenic avian influenza. Arch Intern Med 2006;166:119-123. [ Links ]
122. Hien TT, Liem NT, Dung NT, San LT, Mai PP et al. Avian influenza A (H5N) in 10 patients in Vietnam. New Engl J Med 2004;350:1179-1188. [ Links ]
123. Morris RS, Jackson R. Epidemiology of H5N1 avian influenza in Asia and implications for region control. FAO report 2004. [ Links ]
124. Koopmaus M, Fouchier R, Wilbrink B, Meijer A, Natrop G, et al. Update of human infections with highly pathogenic avian influenza virus A/H7N7 during an outbreak in poultry in The Netherlands. Euro Surveill 2003;7:1-5. [ Links ]
125. Werner O, Starick E, Ground CH. Isolation and characterization of low pathogenic H7N7 influenza virus from a turkey in a small mixed free-range poultry flock in Germany. En: Proceedings of the fifth international Symposium on Avian Influenza, Athens, april 14-17. Avian Dis 2002;47:1104-1106. [ Links ]
126. Bowes VA. An overview of the 2004 outbreak of highly pathogenic avian influenza in British Columbia. En: Proceedings of the fifty-fourth Western Poultry Disease Conference. Vancouver, Canada: 25-27 2005;10-13. [ Links ]
127. García GJ, Méndez OM. "Estudios sobre algunas características de los virus de influenza Aviar de baja patogenicidad aislados en México, de enero a septiembre de 1994. En: Memorias de la XXI Convención Anual ANECA y 45th Western Poultry Disease Conference. Mayo 1995 Cancún, Q. Roo. 1995;295-296. [ Links ]
128. García-García J, Medina PA. La influenza aviar en México. En: Memorias del Simposio Internacional de Influenza Aviar. Sociedad Venezolana de Veterinarios Especialistas en Aves. Maracay, Venezuela: 4 y 5 noviembre 1999;32-42. [ Links ]
129. García-García J, Medina PA. Influenza aviar. En: Memorias del Simposio Internacional de influenza aviar. Sociedad Venezolana de Veterinarios Especialistas en Aves. Maracay, Venezuela: 4 y 5 noviembre 1999;1-11. [ Links ]
130. Villareal CL, Flores AO. The Mexican avian influenza (H5N2) outbreak. En: Proceedings of the Fourth International Symposium on Avian Influenza. Atenas: 14-17 de abril 1997;18-22. [ Links ]
131. García-García J. Avian influenza update from American countries. En: Procedings of IDEXX Poultry Show Conference. Atlanta, GA: 25 de enero 2006;1-10. [ Links ]
132. Garcia M, Crawford JM, Latimer JW, Rivera-Cruz E, Perdue ML. Heterogenecity in the hemagglutinin gene and emergence of highly pathogenic phenotype among recent H5N2 avian influenza viruses from Mexico. J Gen Virol 1996;77: 1493-1504. [ Links ]
133. Horimoto T, Rivera E, Pearson J, Senne D, Krauss S et al. Origin and molecular changes associated with emergence of highly pathogenic H5N2 influenza virus in Mexico. Virology 1995; 213: 223-230. [ Links ]
134. Swyne DE, Suarez DL. Avian influenza in Europe, Asia and Central America during 2001. En: Proceedings of the 104th Annual Meeting of the US Animal Health Association. Richmond, VA: noviembre 2001;465-470. [ Links ]
135. Capua I, Alexander DJ. Avian influenza: recent developments. Avian Path 2004;33:393-404. [ Links ]
136. Lee CW, Denne DA, Suarez DL. Effect of vaccine use in the evolution of Mexican lineage H5N2 avian influenza virus. J Virol 2004;78:8372-8381. [ Links ]
137. García-García J, Rodríguez VH, Hernández MA, Cerón HM. Vacunas contra influenza aviar. En: Curso de Bioseguridad para el Control y Prevención de la Influenza Aviar. México, D.F.: AN4ECA, 20 de octubre 1995;27-35. [ Links ]
138. Hirst GK. Comparison of influenza virus strains from three epidemics. J Exp Med 1947;86:367-381. [ Links ]
139. Francis Jr T. Vaccination against influenza. Bull Who 1953;8:725-741. [ Links ]
140. World Health Organization. Influenza vaccines. Wkly Epidemiol Rec 2002;77:230-239. [ Links ]
141. World Health Organization. WHO global influenza programme: survey on capacities of national influenza centres, January-June 2002. Wkly Epidemiol Rec 2002;77:350-356. [ Links ]
142. World Health Organization. Vaccines for pandemic influenza. Informal meeting of WHO, influenza vaccine manufacturers, national licensing agencies, and government representatives on influenza pandemic vaccines. Summary report. Ginebra Suiza: noviembre 2004. Disponible en: http://www.who.int/csr/disease/influenza/vaccinerecommendations1/en/ [ Links ]
143. World Health Organization. Influenza vaccines. Wkly Epidemiol Rec 2002;75:281-288. [ Links ]
144. Liebert MA. Profile, interview with David S. Fedson MD. Biosecur Bioterror 2005;3:9-15. [ Links ]
145. Barry DW, Mayner RE, Staton E, Dunlap R, Rastogy JE et al. Comparative trial of influenza vaccines in man. Am J Epidem 1976;104:34-46. [ Links ]
146. Kendal AP. Cold-adapted live attenuated influenza vaccines developed in Russia: Can they contribute to meeting the needs for influenza control in other countries. Eur J Epidemiol 1997;13:591-609. [ Links ]
147. Edwards KM, Dupont WD, Westrich MK, Plummer Jr WD, Palmer PS. A randomized controlled trial of cold-adapted and inactivated vaccines for prevention of influenza A disease. J Infect Dis 1994;169:68-76. [ Links ]
148. Mendelmen PM, Cordova J, Cho I. Safety and effectiveness of influenza virus vaccine, trivalent, types A and B, types A and B, live, cold-adapted (CAIV-T) in healthy children and healthy adults. Vaccine 2001;19:2221-2226. [ Links ]
149. Pfleiderer M, Löwer J, Kurth R. Cold-attenuated live influenza vaccine, a risk-benefit assessment. Vaccine 2002;20:886-894. [ Links ]
150. Youngner JS, Treanor JJ, Betts RF, Whitaker-Dowling P. Effect of simultaneous administration of cold-adapted and wild-type influenza infection in humans. J Clin Microbiol 1994;32:750-754. [ Links ]
151. Webster RG, Robinson HL. DNA vaccines: a review of developments. Biol Drugs 1997;8:273-292. [ Links ]
152. Donnelly JJ, Ulmer JB, Shiver JW, Liu MA. DNA vaccines. Annu Rev Immunol 1997;15:617-648. [ Links ]
153. Ulmer JB, Donnelly JJ, Parker SE, Rhodes GH, Felgner PL et al. Heterologous protection against influenza by injection of DNA encoding a viral protein. Science 1993;259:1745-1749. [ Links ]
154. Fedson DS. Pandemic influenza and the global vaccine supply. Clin Infect Dis 2003;36:1552-1561. [ Links ]
155. Fedson DS. Vaccination for pandemic influenza and severe acute respiratory syndrome: common issues and concerns. Vaccine 2003;36:1562-1563. [ Links ]
156. World Health Organization. WHO update: international response to the inadvertent distribution of H2N2 influenza virus: destruction of virus panels proceedings. WHO 2005; disponible en: http://www.who.int/csr/disease/influenza/h2n2_2005_04_15/en/ [ Links ]
157. Ford SM. Grabenstein JD. Pandemics, avian influenza A (H5N1), and a strategy for pharmacists. Pharmacotherapy 2006;26:312-322. [ Links ]
158. Lu JH, Long JX, Shao WX, Wei DP, Liw XF. Generation of attenuated H5N1 and H5N2 subtypes of influenza virus recombinants by reverse genetics system. Wei Sheng Wu Xue Bao 2005;45:53-57. [ Links ]
159. Poland GA, Rottinghaus ST, Jacobson RM. Influenza vaccines: a review and rationale for use in developed and underdeveloped countries. Vaccine 2001;19:2216-2220. [ Links ]
160. Anónimo. Antiviral drugs for prophylaxis and treatment of influenza. Obstet Gynecol 2005;105:427-429. [ Links ]
161. Stephenson I, Nicholson KG. Influenza: vaccination and treatment. Eur Respir J 2001;17:1282-1293. [ Links ]
162. The writing committee of the World Health Organization (WHO) Consultation on avian influenza A/H5 current concepts avian influenza A (H5N1) infection in humans. N Engl J Med 2005;353:1374-1385. [ Links ]
163. Yuen KY, Wong SSY. Human infection by avian influenza A H5N1. Hong Kong Med J 2005;11:189-199. [ Links ]
164 Govorkova EA, Leneva IA, Goloubeva OG, Bush K, Webster RG. Comparison of efficacies of RWJ-270201, zanamivir, and oseltamivir against H5N1, H9N2, and other avian influenza viruses. Antimicrob Agents Chemother 2001;45:2723-2732. [ Links ]
165. Ward P, Small I, Smith J, Suter P, Dutkowski R. Oseltamivir (Tamiflu) and its potential for use in the event of an influenza pandemic. Antimicrob Agents Chemother 2005;55:15S-21S. [ Links ]
166. Nicholson KG, Aoki FY, Osterhaus AD, Trottier S, Carewicz O, et al. Efficacy and safety of oseltamivir in treatment of acute influenza: a randomised controlled trial. Neuraminidase Inhibitor Flu Treatment Investigator Group. Lancet 2000;355:1845-1850. [ Links ]
167. McClellan K, Perry CM. Oseltamivir: a review of its use in influenza. Drugs 2001;61:263-283. [ Links ]
168. Hayden FG, Gubareva LV, Monto AS, Klein TC, Elliott MJ et al. Inhaled zanamivir for the prevention of influenza in families. Zanamivir Family Study Group. N Engl J Med 2000;343:1282-1289. [ Links ]
169. Chotpitayasunondh T, Ungchhusak K, Hanshoworakui W, Chunsuthiwat S, Sawanpanyalert P et al. Human disease from influenza A (H5N1), Thailand 2004. Emerg Infect Dis 2005;11:201-209. [ Links ]
170. Ungchhusak K, Auwarakul P, Dowell SF, Kitphati R, Auanit W et al. Probable person-to person transmission of avian influenza A/(H5N1). N Engl J Med 2005;352:333-340. [ Links ]
171. Enders KO, Cheng PKC, Ng AYY, Hoan GTL, Lim WL. Influenza A H5N1 Detection. Emerg Infect Dis 2005;11:1303-1305. [ Links ]
172. Padhi S, Panigrahi PK, Mahapatra A, Mahapatra S. Avian influenza A (H5N1): a preliminary review. Indian J Med Microbiol 2004;22:143-146. [ Links ]
173. World Health Organization. WHO global Influenza preparedness plan: the role of WHO and recommendations for national measures before and during pandemics. Ginebra, Suiza: World Health Organization, 2005. Disponible en: http://www.who.int/csr/resources/publications/influenza /GIP_2005_5Eweb.pdf [ Links ]
174. WHO consultation on priority public health interventions before and during an influenza pandemic. Ginebra, Suiza: World Health Organization, 2004. (Document WHO/CDS/CSR/RMD/2004.9.). Disponible en: http://www.who.int/csr/disease/avian_influenza/consultation/en/ [ Links ]
175. Secretaría de Salud. Plan de preparación y respuesta ante una pandemia de influenza. Secretaría de Salud 2005. Disponible en; http://www.dgepi.salud.gob.mx/pandemia/ppi.pdf [ Links ]
Fecha de aprobado: 16 de marzo de 2006
Solicitud de sobretiros: Juan García-García. Salvador Gómez M2, L6B, Col. Ampliación Santa Martha Acatitla, 09510, México, DF, México. Correo electrónico: juangarciagarcia@yahoo.com.mx














