Introducción
México es el centro de origen y diversidad del maíz (Zea mays L.), lo que ha dado lugar a un mayor número de razas de maíz endémicas, variedades y genotipos silvestres Centro de Estudios para el Cambio en el Campo Mexicano (Ceecam, 2012). En América Latina se han descrito cerca de 220 razas de maíz (Goodman y Bird, 1977), de las cuales 64 se han identificado y descrito en su mayoría para México (Anderson, 1946; Hernández y Alanís, 1970; Sánchez et al., 2000).
Las razas de maíz de México se han agrupado, con base en caracteres morfológicos, de adaptación y genéticos (isoenzimas) en siete grupos o complejos raciales (Goodman y Bird, 1977; Sánchez et al., 2000). El INIFAP y sus antecesores desde 1942 han desarrollado y liberado más de 250 variedades e híbridos de maíz, que en algunos casos no han sido suficientes y debe reconocerse que en general la investigación, desde sus inicios, ha privilegiado la agricultura de mayor potencial productivo (Espinosa et al., 2009). El cultivo de maíz ocupó el primer lugar por área sembrada en México con 7.6 millones de has, y una producción total de 19 504 050 t (SIAP, 2015).
Todas las entidades del país presentan algún nivel de producción de este cultivo; sin embargo, siete concentran 64.5% del volumen, siendo el estado de Sinaloa el principal productor con 20.7% en una superficie cosechada de 359, 400 ha (SIAP, 2015). Entre las enfermedades de mayor importancia económica se encuentra las causadas por el género Fusarium sp. (Hernández et al., 2016) ya que es un microorganísmo con una gran diversidad de especies, formas especiales y razas, asociadas con enfermedades de plantas (Leslie y Summerell, 2006). Las especies fitopatógenas de este género constituyen un grupo de hongos filamentosos que están ampliamente distribuidas en el suelo y que colonizan las partes aéreas y subterráneas de una amplia gama de especies de plantas, generando síntomas de amarillez, marchitez, pudrición radicular, chancros y finalmente la muerte de muchos cultivos (Leslie y Summerell, 2006).
En diversos países, se han encontrado una gran diversidad de especies de Fusarium infectando plantas (Mesterház et al., 2012). En México se ha documentado la diversidad de especies de Fusarium que generan pudriciones en poblaciones comerciales y nativas de maíz (Morales et al., 2007; García y Martínez, 2010; Leyva et al., 2014; Briones et al., 2015).
Además de su comportamiento fitopatógeno, especies del género Fusarium, en su proceso de infección, sintetizan metabolitos secundarios denominados micotoxinas, dentro de las cuales se han descrito las del grupo zearalenona, tricotecenos y fumonisinas (Eckard et al., 2011), que son de alta toxicidad y que generan al ser humano y animales disturbios en la salud.
Se tienen reportes que F. solani (Martius) Apple & Wollenweber emend. Snyder & Hansen (teleomorfo= Nectria haematococca (Berkeley & Broome) Samuel & Niberberg), F. oxysporum Schlechten emend. Snyder & Hansen (sin reproducción sexual) y F. verticilloides (Saccardo) Nirenberg (anteriormente llamado F. moniliforme (teleomorfo= Gibberella moniliformis Wineland) son las especies que ocasionan daños al ser humano (Tezcan et al., 2009: de Souza et al., 2014); sin embargo, recientemente se diagnosticó a F. subglutinans Wollenber & Reiking Nelson, Toussoun & Marasas (teleomorfo= Gibberella subglutinans Nelson, Toussoun & Marasas (Campos et al., 2013) y F. napiforme Marasas, Nelson & Rabie (sin reproducción sexual) que causan micosis en humano (de Souza et al., 2014) y dañan a gramíneas (Leslie y Sumerell, 2006).
Fusarium proliferatum (Matsushima) Nirenberg (teleomorfo= Gibberella intermedia (Kuhlman) Samuels, Nirenberg & Seifert), F. subglutinans y F. verticillioides son las especies más conocidas como agentes causales del daño de la raíz y mazorca del maíz a nivel mundial (Bertechini et al., 2012; Kauret al., 2014), en México, F. verticillioides y F. subglutinans (Morales et al., 2007; Figueroa et al., 2010; López et al., 2014) han sido los más reportados en esta enfermedad.
El género Fusarium también es conocido por sus dificultades taxonómicas en la definición e identificación de especies a nivel morfológico, de ahí la pertinencia de integrar otros elementos como la caracterización patogénica y molecular que además han demostrado ser fiable en la evaluación de la diversidad genética dentro de este organismo (Bacon et al., 1994). Actualmente las técnicas de biología molecular basadas en la PCR con el uso de iniciadores específicos y marcadores moleculares han sido utilizadas para la identificación de especies de Fusarium. Se ha reportado el uso del gen parcial de la calmodulina para la identificación de F. verticillioides, F. proliferatum y F. subglutinans (Mulé et al., 2004).
Otra alternativa es el gen del factor de elongación 1 alfa (EF-1α o TEF), el cual codifica para una proteína esencial de la maquinaria de traducción. Su utilidad filogenética reside en que su secuencia es altamente conservada a nivel de género Fusarium, por lo cual se han diseñado iniciadores para una mejor oportunidad de separar especies, cuyos productos amplificados generan un fragmento de ~700 pb flanqueando 3 intrones (O’Donell et al., 1998; Geiser et al., 2004). Por otro lado, los ITS (Lin et al., 2014), los RAPDs (Kauret al., 2014) y los RFLPs (Hsuan et al., 2010) también se utilizan como herramientas moleculares para la identificación de especies de Fusarium. El objetivo del presente estudio fue conocer las especies de Fusaria que causan enfermedades en el cultivo del maíz, en siembras de riego del ciclo otoño-invierno en el estado de Sinaloa, México.
Materiales y métodos
El trabajo se realizó durante los ciclos agrícolas primavera-verano, otoño-invierno 2012-2013 y 2013-2014, colectando plántulas, plantas adultas y mazorcas de maíz en diferentes sitios agrícolas en los Municipios de Ahome, Culiacán y Elota en el estado de Sinaloa (Figura 1). La investigación de laboratorio se llevó a cabo en la Unidad de Biotecnología del Campo Experimental Valle de Culiacán del Instituto Nacional de Investigaciones Forestales, Agrícolas y Pecuarias (INIFAP), 24° 37’ 59.3” latitud norte, 107° 26’ 31.0” longitud oeste a 54 msnm, en Culiacán, Sinaloa.
Tejidos de tallo de plántulas y plantas de maíz colectadas se cortaron en trozos longitudinales de 3 mm además de granos de mazorcas, los cuales fueron desinfestados superficialmente con hipoclorito de sodio al 2% por 2 min y etanol al 70% por 2 min, seguido de tres lavados consecutivos con agua destilada estéril. Para el crecimiento y desarrollo del hongo, el tejido vegetal fue colocado en cajas de Petri con medio de cultivo papa-dextrosa-agar (PDA-Difco) suplementado con 1.5 mL L-1 de PCNB (pentacloronitrobenceno) y cefuroxima (200 mg L-1), incubándose a una temperatura de 25 °C durante cinco días. La identificación del hongo se realizó en un microscopio compuesto (Olympus Cx31), con base a la morfología del micelio, microconidias y macroconidias propuestas por Leslie y Summerell, (2006).
Para obtener cultivos monospóricos de Fusaria, se cortó un pequeño fragmento del micelio crecido en medio PDA, el cual contenía el fungicida PCNB y cefuroxima, resuspendido en 1 mL de agua destilada estéril y mediante un hematocitómetro se contaron el número de conidias en diluciones seriadas (Hernández y Rangel, 2011), a partir de éstas se obtuvieron alícuotas que fueron distribuidas sobre medio PDA en cajas Petri, a partir de donde se obtuvo el cultivo monospórico. El crecimiento del hongo fue observado al microscopio compuesto, ubicando esporas individuales germinadas, las cuales fueron seleccionada y transferidas individualmente a una nueva caja de Petri con medio PDA suplementado con PCNB y cefuroxima. Estas se incubaron bajo las mismas condiciones antes mencionadas. Un pequeño trozo de medio PDA con el hongo desarrollado fue transferido a tubos de ensaye que contenían arena tamizada estéril (8 x 10 hilos cm-3) con medio líquido Komada (Komada, 1975) y fueron almacenados en un refrigerador a una temperatura de 4°C para su preservación y uso posterior.
Extracción de ADN de cultivos monospóricos
Para la extracción del ADN, se empleó el método previamente descrito (Velarde et al., 2015), para ello el micelio del hongo fue obtenido raspando con una asa bacteriológica estéril a partir del medio sólido, este fue colocado en mortero y pistilo de porcelana, previamente esterilizados durante 5 min en un horno de microondas electrodoméstico (LG, modelo MS-1446SQP/01), funcionando en la frecuencia de las microondas, en torno a los 2.45 GHz (GigaHertz) y finalmente enfriados a -70 ºC. Después se agregó 1 mL de amortiguador de extracción que contenía: NaCl 30 mM, ácido etilendinitrilotetracético 30 mM (EDTA) y Tris Base 250 mM (pH 8.5), con el cual fue macerado. El producto de la maceración se colocó en tubos Eppendorf de 1.5 mL con su respectiva etiquetación. Enseguida se añadió a la muestra 100 L de cetiltrimetilbromuro de amonio al 10% (CTAB) y 250 L de Cloruro de Sodio 5M (NaCl) incubándose a 95 ºC durante 10 min y posteriormente fue centrifugada a 12 000 rpm durante 10 min; transcurrido este tiempo, la solución acuosa (sobrenadante) fue transferida a un tubo nuevo. A la solución acuosa se le añadió un volumen de cloroformo frío (v/v) y se agitó en un equipo vortex durante unos segundos y posteriormente se centrifugó a 10 000 rpm durante 5 min, una vez transcurrido este tiempo, la solución acuosa fue transferida a un tubo nuevo, agregándosele un volumen de isopropanol absoluto frio y agitándose manualmente.
Posteriormente, las muestras fueron almacenadas durante una hora a -20 ºC para permitir la precipitación del ADN. Ocurrido este tiempo, las muestras en tubos fueron centrifugadas a 12 000 rpm durante 10 min para obtener la pastilla de ADN, la cual se dejó secar a temperatura de laboratorio por dos horas, finalmente el ADN obtenido de los diferentes aislamientos se re suspendieron en 50 L de agua libre de nucleasas (Promega) y almacenado a 4 ºC para su conservación.
Análisis mediante PCR
Un grupo de pares de iniciadores fueron empleados para el análisis por la PCR, del ADN de Fusaria, que originalmente se describieron para separar especie-específica y que en nuestro caso los empleamos para generar información de primera mano para estudios epidemiológicos sobre la incidencia de Fusaria: el par FOF1/FOR1 para F. oxysporum (Mishra et al., 2003), VER1/2, PRO1/2 y SUB1/2 para especie-específica tales como Fusarium verticillioides, F. proliferatum y F. subglutinans (Mulé et al., 2004) respectivamente (Cuadro 1). Para la reacción se utilizó un termociclador (Nyx Technik Amplitronyx series 6 A6 (ATC401) Thermal Cycler). La mezcla de reacción final (15 μL) contenía 100 ng de ADN, una mezcla equimolar de dATP, dCTP, dGTP y dTTP, MgCl2, amortiguador de PCR, ADN Taq polimerasa (provisto por Promega® PCR Master Mix, Catalogo No. M7502), 40 pmoles de cada oligonucleótido (Sigma®).
Cuadro 1 Iniciadores específicos, empleados para la amplificación de fragmentos predichos para especies de Fusaria.
| Nombre del iniciador | Secuencias de iniciadores 5’→3’ | Especies específicas |
|---|---|---|
| SUB1 | CTGTCGCTAAACCTCTTTATCCA | F. subglutinansa |
| SUB2 | CAGTATGGACGTTGGTATTATATCTAA | |
| PRO1 | CTTTCCGCCAAGTTTCTTC | F. proliferatuma |
| PRO2 | TGTCAGTAACTCGACGTTGTTG | |
| VER1 | CTTCCTGCGATGTTTCTCC | F. verticillioidesa |
| VER2 | AATTGGCCATTGGTATTATATATCTA | |
| FOF | ACATACCACTTGTTGCCTCG | F. oxysporumb |
| FOR | CGCCAATCAATTTGAGGAACG | |
| EF1 | ATGGGTAAGGA(A/G)GACAAGAC | Fusariac,d |
| EF2 | GGA(G/A)GTACCAGT(G/C)ATCATGTT |
a= Mulé et al. (2004); b= Mishra et al. (2003); c= O’Donnell et al. (1998); d= Geiser et al. (2004).
Las condiciones de amplificación para F. oxysporum fueron: 1 ciclo a 95 ºC, 5 min; 30 ciclos (95 ºC, 1 min; 53 ºC, 1 min; 72 ºC, 1 min) y un ciclo de extensión final a 72 ºC, 10 min y 4 ºC, mientras que para F. verticillioides, F. proliferatum y F. subglutinans consistieron en: 1 ciclo a 95 ºC, 5 min; 30 ciclos (95 ºC, 1 min; 56 ºC, 1 min; 72 ºC, 1 min), un ciclo de extensión final a 72 ºC, 10 min y 4 ºC. Los productos amplificados fueron analizados en geles de agarosa al 1%, teñidos con una solución de Gel Red (Biotium, catálogo núm. 41003).
Con la finalidad de confirmar la identidad de las especies detectadas con el juego de iniciadores antes descritos, se realizó un segundo análisis de PCR en donde fueron seleccionados el par de iniciadores correspondientes a la región del gen TEF, para ello fueron seleccionados ADNs de 33 cepas monospóricas. Los pares de iniciadores EF1 y EF2 (O’Donnell et al., 1998; Geiser et al., 2004) (Cuadro 1) fueron usados con una temperatura de alineamiento de 55 °C.
Secuenciación enzimática
Solamente los productos amplificados de la región del gen TEF fueron escindidos de la agarosa y purificados a través de columnas de sílica (EZ-10 Spin Column DNA Gel Extraction Kit BS354, Bio Basic Inc.). Una vez obtenido el fragmento de PCR purificado, se procedió a enviar estas muestras para su secuenciación a el Laboratorio Nacional de Genómica para la Biodiversidad (LANGEBIO) del Centro de Investigación y Estudios Avanzados (CINVESTAV-IPN), Unidad Irapuato, Guanajuato, México, basada en el método ddNTPs (Sanger et al., 1977), utilizando un secuenciador 3730 XL DNA (Applied Biosystems, Foster City, CA) y el kit Big DyeTerminator 3.1 (Applied Biosystems, Foster City, CA). La búsqueda de similitud entre secuencias de ADN se realizó por medio del programa BLAST, con el cual se compararon las secuencias de nucleótidos en estudio, con las bases de datos del Centro Nacional para la Información de Biotecnología (NCBI) (http://www.ncbi.nlm.nih.gov/BLAST/), identificándose los valores de homología.
Postulado de Koch de Fusarium cf. Bullatum
Las semillas del maíz criollo Aperlado fueron obtenidas del banco de germoplasma del maíz del Campo Experimental Valle de Culiacán-INIFAP. Para la preparación del inoculo del hongo (número de accesión en el GenBank-NCBI, KR612339), este fue crecido en medio de cultivo PDA suplementado con PCNB (1.5 mL L-1) y cefuroxima (200 mg L-1) durante una semana, a 25 ºC. De este medio, se cortaron ocho fragmentos de micelio y fueron depositados en un matraz con medio AMA (arena 450 g, harina de maíz 50 g, agua estéril 50 mL) incubados bajo las mismas condiciones durante 15días. La desinfestación de las semillas de maíz fueron lavadas en una solución de hipoclorito de sodio al 3% durante 3 min y posteriormente enjuagadas en agua destilada estéril y para su germinación estas fueron depositadas en una incubadora a 25 °C durante 48 h.
Germinadas las semillas, estas fueron seleccionadas por uniformidad en tamaño, depositando una plántula por maceta. Para la preparación de la tierra-sustrato, se realizó una mezcla de tierra muerta (1.5 kg), turba (0.5 kg) y medio AMA (0.250 kg) (con inóculo del hongo) y 600 g de esta mezcla fueron depositados en macetas previamente esterilizadas. La cantidad de inoculo para esta especie fue 5 000 ufc g-1 de suelo. El experimento fue realizado con base al protocolo de Trapero y Jiménez (1985) y Navas et al. (2007) en un invernadero bajo condiciones controladas a temperatura de 25 ±3 °C y un fotoperiodo de12 h, durante 60 días. Se realizaron dos repeticiones de macetas incluidos los testigos sin inoculo.
Análisis filogenético
La alineación de las secuencias nucleotídicas del factor de elongación EF-1α de especies de Fusarium se realizó con el método Clustal W y el dendograma se construyó con el método de Máxima Parsimonia usando el algoritmo SPR (Nei y Kumar, 2000) y un bootstrap de 1000 réplicas (Felsenstein, 1985). Todos los análisis se realizaron con el programa MEGA versión 6.0 (Tamura et al., 2013).
Resultados y discusión
Identificación molecular de Fusarium spp mediante PCR
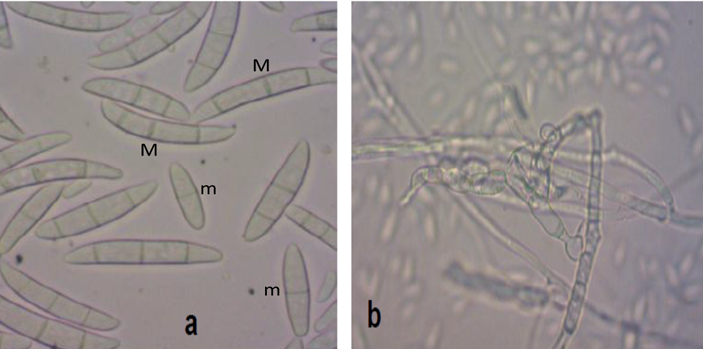
Se obtuvieron 116 cultivos monospóricos, 25 aislados de plántula, 36 de planta adulta y 55 de mazorca, todas ellas con daños de pudrición a los cuales se identificó al género Fusarium mediante la morfología de las microconidias bicelulares, macroconidias septadas y en forma de ‘canoa’ y el micelio septado, de acuerdo a las claves propuestas por Leslie y Summerell (2006) (Figura 2).
A cada aislado monospórico se les extrajo el ADN para la amplificación específica mediante PCR con el empleo de iniciadores descritos para las especies: F. oxysporum, F. verticilloides, F. subglutinansy F. proliferatum (Cuadro 1). De las 116 muestras que morfológicamente fueron identificadas como Fusarium spp., 35 (30.17%) amplificaron el fragmento predicho de 350 pb, para F. oxysporum (Figura 3), especie que se ha mencionado causando daños vasculares en este cultivo en el estado de Puebla, México (García y Martínez, 2010).
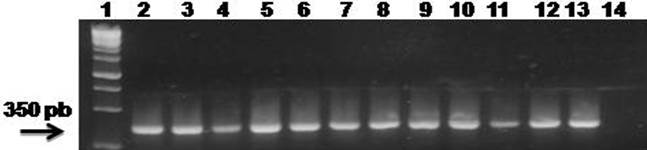
Figura 3 Análisis electroforético en agarosa al 1%. Amplificación del fragmento de 350 pb mediante PCR con iniciadores FOF y FOR específicos para detectar a F. oxysporum. Carriles: 1= Gene Ruler 1Kb DNA Ladder; 2-12= ADN de Fusarium obtenido de maíz enfermo colectado en diferentes localidades del estado de Sinaloa; 13= control positivo (ADN de F. oxysporum) y 14= control negativo (sin ADN).
El ADN extraído del total de cepas monospóricas, 70 muestras amplificaron con los iniciadores para F. verticilloides (60.34%), esta especie que presentó mayor frecuencia de amplificaciones en este estudio, que confirma su prevalencia mundial (Gimeno y Martins, 2011). Estudios recientes indican que F. verticilloides es la especie causante de daños de la pudrición de la raíz y el tallo en el maíz en el estado de Sinaloa (López et al., 2014). En la Figura 4, se observan los fragmentos amplificados con un tamaño predicho de 578 pb descritos para la especie F. verticilloides (Cuadro 1).
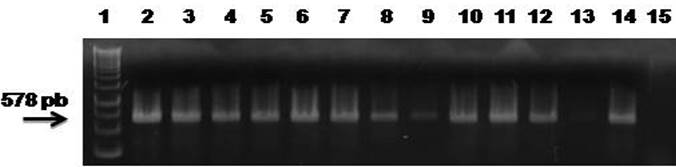
Figura 4 Análisis electroforético en agarosa al 1%. Amplificación del fragmento de 578 pb mediante PCR con iniciadores VER1 y VER2 específicos para detectar a F. verticilloides. 1= Gene Ruler 1Kb DNA Ladder, 2-13= ADN de Fusarium de maíz enfermo colectado en localidades de Sinaloa, 14= control positivo (ADN de F.verticilloides); 15= control negativo (sin ADN).
Sólo tres de las 116 muestras analizadas amplificaron con los iniciadores correspondientes a F. subglutinans (2.59%). En la Figura 5, se observan los fragmentos amplificados con un tamaño predicho de 631pb. A nivel mundial esta especie después de F. verticilloides es el segundo agente causal de la pudrición radicular del maíz (Gimeno y Martins, 2011). En México F. subglutinans, se ha encontrado en el estado de Guanajuato (Figueroa et al., 2010) y en el Estado de México (Morales et al., 2007; Rivas et al., 2011).

Figura 5 Análisis electroforético en agarosa al 1%. Amplificación del fragmento de 631 pb mediante PCR con iniciadores SUB1 y SUB2 específicos para detectar a F. subglutinans . 1= Gene Ruler 1Kb DNA Ladder; 2-6= ADN de Fusarium obtenido de maíz enfermo colectado en localidades de Sinaloa; 6= control positivo (ADN de F. subglutinans); 7= control negativo (sin ADN).
En cuanto a la detección con los iniciadores específicos de F. proliferatum, no se observaron amplificaciones, en las ocho muestras de ADN analizadas, las cuales fueron amplificadas con los iniciadores TEF y que se incluyeron entre las 33 muestras secuenciadas.
Secuenciación enzimática
De los 116 amplificados que en total se obtuvieron, 32 fragmentos fueron secuenciados. La identidad de las secuencias en nuestro estudio, manifestaron una alta similaridad para siete diferentes especies. En el Cuadro 2, se observan los números de accesión en el NCBI, tejido donde fueron aisladas, localización y el porcentaje de homología. Para ello, cada una de nuestras accesiones fue comparada con la base de datos del Centro Nacional para la Información de Biotecnología (NCBI-USA), lo que generó los valores de homología con cada accesión descrita para las diferentes especies, de donde se confirmó la identidad de: F. verticilloides, F. oxysporum , F. subglutinans, F. nygamai Burgess &Trimboli (teleomorfo: Gibberella nygamai Klaasen & Nelson), F. andiyazi Marasas, Rheeder, Lamprecht, Zeller & Leslie (sin reproducción sexual), F. cf. bullatum y F. equiseti (Corda) Saccardo (teleomorfo:Gibberella intricans Wollenweber) cuyos números de accesión se describen en el Cuadro 2.
Cuadro 2 Identidad de especies de Fusarium basado en la secuenciación del gen TEF(EF-1α).
| Cepa | Localización | Tejido | Accesión al GenBank |
Asociación basada en la secuencia |
Identidad (%) |
Accesión comparada del GenBank |
|---|---|---|---|---|---|---|
| 526MC | Ahome | Mazorca | JN806238 | F. verticilloides | 100% | JF740729 |
| 527MC | Ahome | Mazorca | JN806239 | F. verticilloides | 100% | JF740729 |
| 528MC | Ahome | Mazorca | JN806240 | F. verticilloides | 100% | JF740729 |
| 529MC | Ahome | Mazorca | JN806241 | F. verticilloides | 100% | JF740729 |
| 530MC | Ahome | Mazorca | JN806242 | F. verticilloides | 100% | JF740729 |
| 532MC | Ahome | Mazorca | JN806243 | F. verticilloides | 100% | JF740729 |
| 533MC | Ahome | Mazorca | JN806244 | F. verticilloides | 100% | JF740729 |
| 536MC | Ahome | Mazorca | JN806245 | F. verticilloides | 100% | JF740729 |
| 538MC | Ahome | Mazorca | JN806246 | F. verticilloides | 100% | JF740729 |
| 610CS | Culiacán | Plántula | KR905566 | F. verticilloides | 100% | JF740729 |
| 613CUL | Culiacán | Plántula | KF753752 | F. cf. bullatum | 99% | JX268977 |
| 622CS | Culiacán | Plántula | KR905551 | F. verticilloides | 100% | JF740729 |
| 623MS | Ahome | Plántula | KR905567 | F. cf. bullatum | 100% | JX268977 |
| 627CS | Culiacán | Plántula | KR905552 | F. verticilloides | 100% | JF740729 |
| 630CS | Culiacán | Plántula | KR905553 | F. verticilloides | 100% | JF740729 |
| 635CS | Culiacán | Plántula | KR905554 | F. verticilloides | 100% | JF740729 |
| 638ES | Elota | Plántula | KR905555 | F. verticilloides | 100% | JF740729 |
| 646ES | Elota | Plántula | KR905556 | F. verticilloides | 100% | JF740729 |
| 649ES | Elota | Plántula | KR905557 | F. verticilloides | 100% | JF740729 |
| 652MS | Ahome | Plántula | KR905558 | F. nygamai | 100% | JF740790 |
| 655ES | Elota | Planta adulta | KR905559 | F. verticilloides | 100% | JF740729 |
| 663ES | Elota | Planta adulta | KR905560 | F. verticilloides | 100% | JF740729 |
| 676ES | Elota | Planta adulta | KR905562 | F. verticilloides | 100% | JF740729 |
| 677CS | Culiacán | Planta adulta | KR905563 | F. verticilloides | 100% | JF740729 |
| 678CS | Culiacán | Planta adulta | KR612341 | F. nygamai | 100% | JF740790 |
| 680MS | Ahome | Planta adulta | KR706385 | F. equiseti | 91% | KF514661 |
| 681CS | Culiacán | Plántula | KR706384 | F. equiseti | 91% | KF514661 |
| 706ES | Elota | Plántula | KR612339 | F. cf. bullatum | 100% | JX268977 |
| 771CS | Culiacán | Planta adulta | KR706383 | F. subglutinans | 99% | DQ837698 |
| 777ES | Elota | Planta adulta | KR612338 | F. andiyazi | 99% | KM462947 |
| 789ES | Elota | Planta adulta | KR905564 | F. oxysporum | 99% | KM092371 |
| 843CS | Culiacán | Planta adulta | KR612340 | F. oxysporum | 100% | DQ435354 |
La especie F. equiseti se le ha identificado en la zona del Bajío, Guanajuato, México (Figueroa et al., 2010); sin embargo, Leslie y Summerell (2006) afirman que la especie está considerada como saprofito o invasor secundario; no obstante, Madania et al. (2013) lo ha reportado en Siria causando daños en plantas de maíz. En cuanto a F. nygamai ha sido identificado en Sinaloa a nivel mundial causando daños (Leyva et al., 2014), aunado que fue identificado en este estudio.
Con respecto a F. andiyazi ha sido descrito con daños de pudrición de la mazorca en China (Zhang et al., 2014), Siria (Madania et al., 2013) y también en Sinaloa, México (Leyva et al., 2014). Finalmente, para F. cf. bullatum existe un reporte en Irán que informa su aislamiento y daño en el maíz (Rahjoo et al., 2008).
Postulado de Koch de Fusarium cf. Bullatum
Después de 60 días de la inoculación bajo condiciones controladas, se observó que el maíz criollo Aperlado fue dañado generando pudrición radicular por Fusarium cf. bullatum (Figura 6), posteriormente se aislaron cepas monospóricas y se observaron las macroconidias (4-5-septas) (Figura 7a , m), microconidias (1-2-septas) (Figura 7a, m) y fiálides en agar hoja de clavel (CLA) (Figura 7-b). Posteriormente, a una cepa se le asiló el ADN y mediante el gen TEF se identificó molecularmente que es Fusarium cf. bullatum. La secuención enzimática fue registrada en el banco de genes del NCBI (GenBank-NCBI-USA) con número de accesión KX545253.

Figura 6 Postulado de Koch de Fusarium cf. bullatum mostrando daño al maíz del criollo Aperlado bajo condiciones controladas en invernadero.
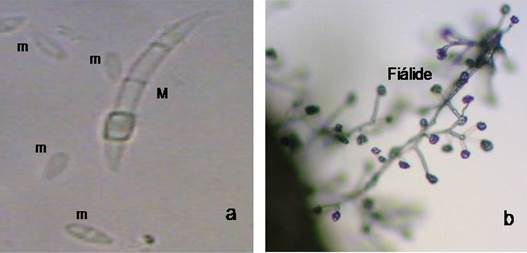
Figura 7 Morfología de Fusarium cf. bullatum: a) microconias (m), macroconidias (M); b) fiálides. Análisis filogenético
La relación filogenética de Máxima Parsimonia del factor de elongación EF-1α de especies de Fusarium se analizó en 32 secuencias con 587 posiciones utilizando el modelo de sustitución nucleotídica SPR (poda y reconexión de un subárbol), que mejor ajustó a EF-1α. El análisis de las secuencias generó 8 árboles de máxima parsimonia, con índices de consistencia de 83.5% y de retención de 92%, ambos índices de amplia confiabilidad en las comparaciones filogenéticas (Nei y Kumar 2000). El consenso de estos árboles retiene un total de 9 clados.
De acuerdo con el Fusarium mating population (Summerell y Leslie, 2006), F. verticillioides y F. andiyazi, sinónimos de Gibberella fujikuroi pertenecen al mating population A, en coincidencia, formaron parte de un mismo grupo en nuestro dendograma (Figura 8). En el cluster de 99% en el que se encuentran F. oxysporum, F. nygamai y F. subglutinans, que previamente se han descrito dentro del mating population G (Summerell y Leslie, 2006); en este análisis, dicha agrupación entre estas especies, también se reflejó en el dendrograma (Figura 8).
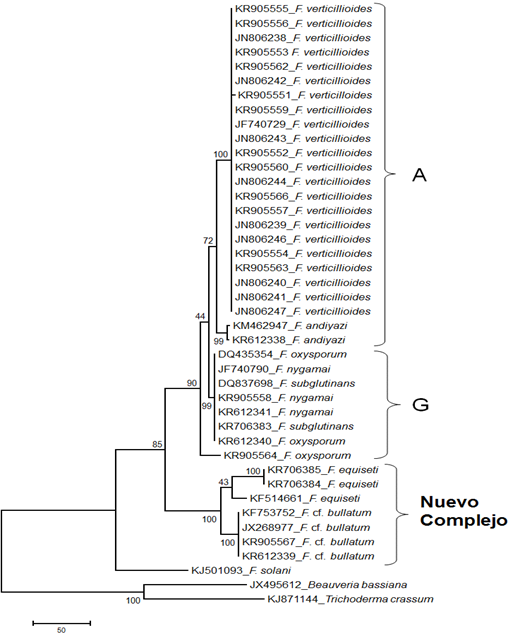
Figura 8 Dendograma de Máxima Parsimonia basado en secuencias del factor de elongación EF-1α de especies de Fusarium. El dendograma se construyó con 32 secuencias nucleotídicas, analizando 587 posiciones y un índice de Felsenstein de 1 000 réplicas.
El cluster donde se agrupan F. equiseti y F. cf. bullatum, está separado del mating population A y G, el cual recientemente fue descrito como un nuevo complejo (Castellá y Cabañes, 2014); no obstante, Summerell y Leslie (2006) mencionan como F. equiseti var bullatum dentro de una misma especie.
Conclusiones
La distinción de especies dentro del género Fusariumutilizando caracteres morfológicos fue poco precisa.
El uso de iniciadores específicos empleados para la identificación de especies de Fusaria con fines de conocer, de primera mano, la incidencia de cada especie detectada arrojó resultados coincidentes con la secuenciación de las especies: F. verticilloides, F. subglutinans y F. oxysporum.
Con el empleo de los iniciadores TEF, fue posible confirmar la identidad y presencia de siete especies de Fusaria aislados en el maíz: F. verticilloides, F. subglutinans, F. oxysporum, F. equiseti, F. nygamai, F. andiyazi y F. cf. bullatum.
F. verticilloides fue la especie predominante con un 60.34%, F. oxysporum fue la especie que ocupó el segundo lugar, con una incidencia de 30.17%, F. subglutinans y F. cf. bullatum en tercer lugar con una incidencia de 2.59%.
De acuerdo con nuestros resultados, la estrecha relación filogenética entre las especies F. verticillioides y F. andiyazi coincidieron con el mating population A descrito por Summerell y Leslie (2006), así como F. oxysporum, F. nygamai y F. subglutinans, dentro del mating population G y para el caso de F. equiseti y F. cf. bullatum que formaron un grupo separado, se asociaron a un nuevo complejo descrito por Castellá y Cabañes, (2014).
Esta investigación reporta por primera vez la presencia en México a Fusarium cf. bullatum causando daños en el maíz.











 texto en
texto en