Introducción
Los flavonoides, el grupo más amplio de polifenoles, han sido ampliamente estudiados por su actividad antioxidante, antimicrobiana, antiinflamatoria, entre otras actividades biológicas (Li et al., 2014; Calabrone et al., 2015). En años recientes se ha estudiado su interacción con proteínas, como las enzimas digestivas para evaluar su posible uso en el control de la obesidad y diabetes (Li et al., 2011; Zeng et al., 2015; Martinez-Gonzalez et al., 2017, 2019). La inhibición de enzimas digestivas está relacionada con la elevada afinidad de los flavonoides de interaccionar con las cadenas polipeptídicas, generando interacciones no covalentes con la enzima. En donde, la estructura de los flavonoides ha sido señalada como una de las responsables de esta interacción enzima-ligando (Gonzales et al., 2015).
Los flavonoides derivados del 2-fenil-benzo-γ-pirano tienen una estructura común de dos anillos aromáticos (de-nominados “A” y “B”), unidos por un anillo heterocíclico central (anillo “C”) (Figura 1) (Harborne, 1964; Ribeiro et al., 2015). Existen aproximadamente 4,000 diferentes compuestos miembros de esta familia (Huang et al., 2009) y sus variaciones dependen principalmente del grado de oxidación del anillo C (Rawel et al., 2002). Se pueden encontrar distintas clasificaciones de los flavonoides. De manera sencilla, se pueden clasificar en dos grandes categorías, dependiendo de la presencia o ausencia del grupo hidroxilo en la posición C3, y se nombran como 3-hidroxiflavonoides y 3-desoxiflavonoides, respectivamente. Estos dos grupos se pueden dividir dependiendo de las sustituciones en otras posiciones de los anillos. Las principales subfamilias de flavonoides son las flavanonas, las flavonas, los flavonoles, y los flavan-3-oles o flavanoles (Erlund, 2004; Gonzales et al., 2015) (Figura 1). De esta forma, las subfamilias de flavonoides presentan diferencias estructurales, en el número y tipo de sustituyentes, en la presencia de dobles enlaces, entre otras (Martinez-Gonzalez et al., 2017b). Los flavonoides pueden tener glicosilaciones (u otras sustituciones como metilaciones) como sustituyentes de los anillos. No obstante, la flexibilidad conformacional del flavonoide depende de la estructura de los anillos B y C (Amat et al., 2008), la estructura plana del anillo C y las sustituciones en los diferentes anillos, son al parecer las principales características señaladas para la interacción con enzimas como las digestivas (Gonzales et al., 2015).

Figura 1 Estructura química de los flavonoides evaluados. Luteolina (LUT), Quercetina (QUE) y
Rutina (RUT) se muestran en una misma estructura (Figura 1a), se diferencian en el sustituyente en el
C3 del anillo C. El grupo R de LUT es un H, para QUE R=OH, y RUT el
grupo R corresponde a un rutinósido. La CAT y HES se muestran en las
estructuras (b) y (c), respectivamente.
Figure 1. Chemical structure of evaluated flavonoids. The
same structure (Figure 1a) was
employed to show Lutein (LUT), Quercetin (QUE) and Rutin (RUT), and
their differences are due to the substitution at C3 position. The
radical group (R) of LUT is a hydrogen, whereas a hydroxyl and a
rutinoside are the R groups for QUE and RUT. CAT and HES are shown, (b)
and (c) respectively.
En la Figura 1 se presentan las estructuras de algunos subgrupos de flavonoides. Se muestran las sustituciones o modificaciones estructurales correspondientes, tales como la carencia del grupo hidroxilo en el carbono 3 (C3) para la flavona luteolina (LUT, Figura 1a), a diferencia del flavonol quercetina (QUE) que cuenta con este grupo hidroxilo; o la presencia de una glicosilación en esta posición para el flavonol glicosilado, rutina (RUT). Mientras que en la Figura 1b se muestra la estructura del flavanol catequina (CAT) que carece del doble enlace entre el C2 y C3 en el anillo C. La flavanona hesperetina (HES) carece también de este doble enlace (Figura 1c), pero a diferencia de los otros flavonoides, presenta en su estructura un grupo metoxi en el C4’ (anillo B). Así, estas estructuras presentan diferencias importantes como la presencia del doble enlace entre el C2 y el C3 del anillo C, la sustitución en el C3 por un grupo hidroxilo o una glicosilación, y la presencia de un grupo metoxi en el C4’, que son características estructurales que han sido señaladas relevantes para su interacción con otras proteínas como las de la leche (Gonzales et al., 2015)2015. Sin embargo, aspectos como la estructura plana del anillo C, otorgada por el doble enlace C2=C3 no ha sido completamente evaluado. La importancia de esta característica ha sido valorada para otras proteínas como las del factor de crecimiento endotelial vascular (Cerezo et al., 2015).
El análisis de la unión de estos flavonoides con enzimas digestivas podría ayudar a elucidar un mecanismo de inhibición. La inhibición reversible de enzimas como lipasa pancreática, podría ser parte de un tratamiento alternativo para enfermedades no transmisibles como la obesidad (Buchholz y Melzig, 2015). La lipasa pancreática es la enzima encargada de hidrolizar alrededor del 70 % de los triglicéridos ingeridos por el ser humano (Birari y Bhutani, 2007). La disminución de su actividad enzimática por inhibidores reversibles, como los polifenoles, es objeto de estudio ya que podrían eliminar los efectos secundarios de un inhibidor no reversible (Sakulnarmrat y Konczak, 2012). Se ha reportado una actividad inhibitoria en valores de IC50 (concentración del ligando a la cual se inhibe la mitad de la actividad enzimática), más eficiente para flavonoides como la quercetina, en comparación con ácidos fenólicos como el ácido caféico (Martinez-Gonzalez et al., 2017a). Extractos vegetales ricos en flavonoides como QUE han sido evaluados por su elevada actividad inhibitoria frente a lipasa (Sakulnarmrat y Konczak, 2012), pero no se les había asociado con su estructura. Comparar a la quercetina con flavonoides de estructura similar, podría permitir elucidar el mecanismo de inhibición de estos flavonoides que cuentan con insaturación C2=C3. Analizar el efecto de la inhibición de la lipasa por parte de los flavonoides por sus interacciones con los sitios hidrofóbicos de unión de la enzima, considerando que el sitio de unión del sustrato es hidrofóbico, permitiría elucidar la relevancia de los aspectos estructurales de los flavonoides importantes para la unión con la enzima y su consecuente inhibición. En este sentido, en el presente estudio se pretende evaluar el efecto de diferentes substituciones en los anillos de los flavonoides, como el doble enlace entre C2 y C3 (anillo C), sobre sus interacciones con lipasa pancreática y como afectan estas interacciones en la actividad enzimática de la lipasa pancreática.
Materiales Y Métodos
Materiales
La lipasa pancreática porcina tipo II, p-nitrofenil laurato (pNPL), Tris, Triton X-100, ácido clorhídrico, acetato de sodio, reactivo de Bradford, albúmina sérica bovina (BSA), ácido 1-anilino-8-naftalenosulfónico (ANS), y los flavonoides o ligandos: Luteolina (LUT), Quercetina (QUE), Catequina (CAT), Hesperetina (HES) y Rutina (RUT) fueron adquiridos de Sigma Aldrich Co. (Control Técnico y Representaciones, CTR, S.A. de C.V., Nuevo León, México). Etanol y metanol se adquirieron de J.T. Baker (CTR, Nuevo León, México). Todos los reactivos fueron de pureza grado reactivo o superior.
Análisis de la actividad enzimática de lipasa pancreática
La inhibición de la actividad de la enzima lipasa pancreática se realizó de acuerdo a Martinez-Gonzalez et al. (2017a). La lipasa se disolvió en una disolución amortiguadora Tris 20 mM (pH = 7.2), a una concentración final de 2.0 mg/mL. La concentración de la proteína se determinó por el método Bradford, empleando una disolución de 1 mg/mL de albúmina de suero bovina (BSA) como disolución estándar, midiendo la absorbancia a 595 nm en un espectrofotómetro UV-Vis (Agilent 8453 HP G1103A; Agilent Technologies, Santa Clara, California, Estados Unidos de América). El sustrato, pNPL, se disolvió (0.08 % de relación masa volumen) en acetato de sodio 5 mM (pH = 5.0, Triton X-100 al 1 %) para tener concentraciones finales de 11.7-124.2 µM. Los flavonoides se disolvieron en metanol a una concentración final de 12.5, 25 y 50 µM. Por problemas de solubilidad, la quercetina se disolvió en etanol a las mismas concentraciones. El ensayo se llevó a cabo en la disolución amortiguadora Tris 200 mM (pH = 8.2), de acuerdo a Martinez-Gonzalez et al. (2017a), y la reacción se midió a 400 nm durante 1000 s a 37 °C en un espectrofotómetro de UV-Visible (Agilent 8453 HP UV-Vis spectrometer G1103, Agilent Technologies, Santa Clara, E.U.A.). Todas las muestras se midieron por triplicado. Además se determinó el porcentaje de inhibición para cada uno de los ligandos a partir de medición realizada al término de la cinética (1000s) (Dalar y Konczak, 2013), así como el valor de IC50 a partir de un gráfico de porcentaje de inhibición frente a concentración de sustrato. A partir de los valores cinéticos (velocidad inicial, v 0) se calcularon los valores de velocidad máxima (v max) y cons-tante de Michaelis-Menten (K M), ajustando los valores de v 0 a las diferentes concentraciones de inhibidor y de sustrato a la ecuación de Michaelis-Menten (análisis no lineal, Ecuación 1), y la ecuación de Lineweaver-Burk (análisis lineal, Ecuación 2) con el programa GraphPad Prism v. 5 (GraphPad Software).
Las constantes de inhibición para una inhibición reversible mixta K i y K i’ correspondientes a la enzima libre y al complejo enzima-sustrato, respectivamente, fueron calculadas mediante las ecuaciones 3 y 4 (Tipton, 1996).
Donde v max’ y K M’ corresponden a los valores obtenidos en presencia de los inhibidores, y la concentración del inhibidor (flavonoide) se representa con [I].
Ensayo de la fluorescencia extrínseca del ANS
La posible unión de los flavonoides en sitios hidrofóbicos de lipasa pancreática, se evaluó de acuerdo a Martinez-Gonzalez et al. (2019) con algunas modificaciones. Se midió la intensidad de la fluorescencia (IF) emitida por una disolución de lipasa pancreática 0.05 mg/mL (Tris 200 mM pH 8.2) con ácido 8-anilino-1-naftalenosulfónico (ANS) 150 µM en un espectrofluorómetro (Shimadzu RF-5301, Shimadzu Corporation, Kioto, Japón). Se monitoreó la IF a λex de 380 nm y λem de 400 a 700 nm a 37 °C durante 900 s, en una celda de cuarzo de 1 cm. Se tituló a diferentes concentraciones de los flavonoides (hasta 300 µM), midiendo la IF en el máximo de emisión (519 nm) y se realizaron las correcciones del cambio de volumen y efectos del filtro interno.
Se calculó el valor de la constante aparente de disociación KD para el complejo enzima-ANS, en presencia de los ligandos, empleando la Ecuación 5.
Donde ΔFI es el cambio en la intensidad de la fluorescencia a 340 nm (F0-F); ΔFmax es el máximo valor de ΔFI; y KD (Ecuación 5) corresponde al valor de la constante aparente de disociación para la unión entre la enzima y el ANS.
Docking molecular
El posible sitio de unión de los flavonoides en la proteína se analizó mediante el software USCF-Chimera v. 4 con AutoDock Vina (Estados Unidos de América), de acuerdo a Martinez-Gonzalez et al. (2019). Se emplearon las estructuras tridimensionales del cristal de lipasa pancreática (Código 1ETH.pdb) y de los ligandos: luteolina, quercetina, catequina, hesperetina y rutina obtenidos de la base de datos de PubChem (Estados Unidos de América) y minimizados con el software PyMOL v. 13 (Estados Unidos de América). Las estructuras de los ligandos se consideraron como flexibles y la estructura de la proteína como fija, para obtener valores de energía de unión (cambio en la entalpía, ΔH y energía libre de Gibbs, ΔG) y determinar el mejor modelo para la posible unión entre la proteína y el ligando.
Resultados Y Discusiones
Estudios de inhibición enzimática de flavonoides frente a lipasa
La actividad enzimática de lipasa pancreática fue evaluada en presencia de distintas concentraciones de los flavonoides. En la Figura 2 se muestra la actividad de la lipasa pancreática en presencia de distintas concentraciones de flavonoides con la enzima (valores de V 0). El control presentó diferencias estadísticas significativas respecto a todos los flavonoides (p < 0.05) es decir, todos los ligandos disminuyeron la actividad enzimática. En esta figura, se observa que el efecto inhibitorio de CAT y HES fue menor que la de los demás flavonoides analizados. Estos compuestos (CAT y HES) comparten similitudes estructurales, como la carencia del doble enlace entre C2 y C3. Este doble enlace al parecer es esencial para el acomodo de los flavonoides en su sitio de unión en la proteína. Resultados similares se observaron con la α-amilasa, en donde LUT y QUE que presentan este doble enlace, presentaron una mejor capacidad inhibitoria (Martinez-Gonzalez et al., 2019). La estructura plana del anillo C, debido a la presencia de este doble enlace, aumenta la capacidad inhibitoria de los flavonoides (Lo Piparo et al., 2008). Estos resultados son similares a los previamente reportados de una mayor inhibición de lipasa pancreática para el flavonoide QUE (Martinez-Gonzalez et al., 2017). Esta mayor inhibición puede explicarse considerando la estructura plana del anillo C del flavonol QUE y de la flavona LUT, mismo que les confiere una mayor capacidad para interaccionar con la cadena polipeptídica. La menor inhibición por parte de CAT y HES se explica por una ausencia de este doble enlace en su estructura y es similar a lo reportado para la inhibición de la α-amilasa (Martinez-Gonzalez et al., 2019). La relevancia de esta característica estructural para su unión con proteínas ha sido previamente reportada (Tadera et al., 2006; Proença et al., 2017; Martinez-Gonzalez et al., 2019).

Figura 2 Efecto de los flavonoides Hesperetina (HES), Luteolina (LUT), Quercetina (QUE),
Catequina (CAT), y Rutina (RUT) (25 µM) sobre la hidrólisis del
pNPL catalizada por lipasa. Los símbolos
representan los valores de velocidad inicial (V
0) expresada como µM s-1 ± desviación
estándar. Las líneas representan el ajuste no lineal de
Michaelis-Menten a los datos experimentales. Los asteriscos
representan diferencias estadísticas entre los tratamientos respecto
al control (Análisis de la diferencia mínima significativa de
Fisher, p < 0.05).
Figure 2. Effect of the flavonoids Hesperetin (HES),
Lutein (LUT), Quercetin (QUE), Catechin (CAT) y Rutin (RUT) (25 μM)
on pNPL hydrolysis catalyzed by pancreatic lipase. Symbols represent
the initial velocity (V0) values expressed as μM s-1 ± standard
deviation. Lines are the Michaelis-Menten curves fitted to the
experimental data. * Statistical difference (Fisher’s Least
Significant Difference, LSD, p < 0.05) between all treatments
respect to control.
La RUT se comportó de manera similar a QUE, probablemente debido a la similitud estructural entre ambas moléculas, ya que RUT es el flavonol QUE glicosilado con el disacárido rutinósido en posición C3. Otros autores tampoco han observado diferencias significativas para la inhibición de lipasa pancreática entre flavonoides glicosilados y sin glicosilación, como la QUE y QUE-arabinósido (Zhang et al., 2015). Se ha señalado que aunque estos compuestos glicosilados, de mayor tamaño que sus respectivas agliconas, no se pudieran acomodar en un sitio de unión, el aumento en el número de grupos hidroxilo por la glicosilación, les permitiría formar un mayor número de puentes de hidrógeno con la lipasa (Li et al., 2011).
A partir del análisis lineal (Lineweaver-Burk; datos no mostrados) y no lineal (Figura 2) se calcularon los parámetros cinéticos para cada uno de los flavonoides (Tabla 1). Del análisis de esta tabla, se observó que su capacidad inhibitoria disminuyó en el siguiente orden: QUE > RUT > LUT > CAT > HES. Los valores calculados de IC50 respaldarían esta aseveración, ya que estos fueron 10.30 ± 2.45, 13.50 ± 0.70, 14.70 ± 0.90, 28.50 ± 1.05, y 30.50 ± 2.10, respectivamente. Estos valores coinciden en que CAT y HES fueron los inhibidores con menor eficiencia frente a lipasa. QUE, RUT y LUT comparten la estructura de catecol en el anillo B (hidroxilaciones en las posiciones C2’ y C3’), que ya había sido señalada para la inhibición de enzimas como la α-amilasa y α-glucosidasa (Gonzales et al., 2015).
Tabla 1 Valores de los parámetros cinéticos, V
max, K
M, K
i y K
i′, así como el tipo de inhibición observada para la
actividad de lipasa sobre el sustrato pNPL en
presencia de los flavonoides.
Table 1. Kinetic parameters, V
max, K
M, K
i y K
i′, and inhibition type determined for pancreatic lipase
activity with pNPL as substrate in presence of the evaluated
flavonoids.
| Ligando | Concentración (μM) | V max (·10-4 mM min-1) | K M (mM) | Tipo de Inhibición | K i and K i′(mM) | |
|---|---|---|---|---|---|---|
| CONTROL | 0.00 | 0.07 ± 0.03a | 2.05 ± 0.80e | Ninguno | 0.00 | 0.00 |
| HES | 12.60 25.00 50.00 |
0.07 ± 0.00a
0.05 ± 0.01a 0.03 ± 0.00b |
3.27 ± 0.35e
3.54 ± 0.22e 9.87 ± 1.14d |
Mixto | 17.82 ± 1.06a | 25.10 ± 3.02a |
| LUT | 12.67 25.35 50.00 |
0.03 ± 0.00b
0.02 ± 0.00c 0.02 ± 0.01bc |
3.23 ± 0.60e
32.64 ± 3.10b 40.54 ± 3.02a |
Mixto | 11.00 ± 1.30b | 19.50 ± 2.90b |
| QUE | 12.51 24.83 49.66 |
0.03 ± 0.00b
0.03 ± 0.00b 0.02 ± 0.01bc |
2.41 ± 0.50e
28.93 ± 1.00b 30.03 ± 1.08b |
Mixto | 8.90 ± 0.65c | 15.70 ± 4.00b |
| CAT | 12.32 24.45 50.00 |
0.05 ± 0.01a
0.03 ± 0.01ab 0.03 ± 0.00b |
1.87 ± 0.05e
1.25 ± 0.00f 1.03 ± 0.01g |
Acompetitivo | n.d. | n.d. |
| RUT | 12.84 25.57 50.00 |
0.04 ± 0.21a
0.02 ± 0.01bc 0.01 ± 0.00c |
10.36 ± 0.71d
11.15 ± 0.09d 14.14 ± 1.35c |
Mixto | 10.63 ± 2.00bc | 16.40 ± 0.80b |
Los datos son representados como media ± desviación estándar de los análisis realizados por triplicado. Las diferentes letras en una columna representan las diferencias significativas (Análisis de la diferencia mínima significativa de Fisher, p < 0.05), n.d. significa no determinado. HES, Hesperetina, LUT, Luteolina, QUE, Quercetina, CAT, Catequina, RUT, Rutina.
Values are mean ± standard deviation of triplicate analysis. Different letters in columns represent significant differences (Fisher, p < 0.05), n.d. not determined.
HES, Hesperetin, LUT, Lutein, QUE, Quercetin, CAT, Catechin, RUT, Rutin.
El tipo de inhibición se determinó por el cálculo de los parámetros cinéticos V max y K M (Tabla 1), y el análisis de Lineweaver-Burk (datos no mostrados). Para la mayoría de los flavonoides, se observó una disminución estadísticamente significativa del valor de V max y un aumento en el valor de la K M, esto indicó que fueron inhibidores reversibles mixtos. Para CAT se observó una inhibición acompetitiva, en donde el flavonoide sólo tendría preferencia por la unión con el complejo enzima-sustrato. Este flavonoide no se uniría como LUT y QUE (menor valor de IC50 que estos). La capacidad inhibitoria de la CAT se puede atribuir a la carencia del grupo oxo en el C4 y el doble enlace (C2 = C3) que disminuye su afinidad por la enzima libre. Se ha descrito que la presencia del grupo oxo en el C4 aumenta las interacciones de los flavonoides (por ejemplo QUE) frente a diferentes proteínas como el factor de crecimiento endotelial vascular (Cerezo et al., 2015).
Los valores de las constantes de inhibición K i y K i’ (Tabla 1), muestran que el componente competitivo de la inhibición mixta fue predominante (K i < K i’). Algunos de estos flavonoides, como QUE y RUT, ya han sido reportados como inhibidores mixtos de la actividad de lipasa (Li et al., 2011; Martinez-Gonzalez et al., 2017a). Estos flavonoides podrían ejercer desde un mismo sitio de unión su efecto competitivo y no competitivo de la inhibición. De acuerdo con cálculos del coeficiente de Hill (ɳ), en ausencia y presencia de los ligandos, que fue de aproximadamente 1.00 (datos no mostrados), se concluye que existe un sólo sitio de unión para el sustrato. Resultados similares de coeficiente de Hill (ɳ igual a 1.00) han sido reportados para flavonoides como LUT y genisteína con proteínas humanas, como quinasas de tirosina (Kuhnert et al., 2011). Esto podría indicar que los ligandos, aunque impidieran el ingreso del sustrato al sitio activo (competitivo), luego permitirían la unión entre ambos y los flavonoides se mantendrían unidos a este complejo (enzima-sustrato; no competitivo).
Análisis de los sitios hidrofóbicos de unión en la enzima
La fluorescencia extrínseca permite evaluar la interacción del compuesto apolar ANS con los sitios hidrofóbicos de la enzima. Una disminución en la IF del ANS se relaciona con un desplazamiento del ANS de estos sitios apolares por parte de los flavonoides (Halim et al., 2017). La Figura 3 muestra el efecto en la IF del ANS en presencia de los flavonoides. Se observa que los flavonoides que disminuyeron la intensidad fueron, en orden decreciente, RUT, QUE y LUT. Asimismo, se puede observar en las Figuras 3-internas, el ajuste no lineal (Ecuación 5) realizado, para calcular la K D app de los flavonoides que mostraron disminución de la IF del ANS.

Figura 3 Efecto de flavonoides en la fluorescencia extrínseca del complejo lipasa-ANS. Se
emplearon diferentes concentraciones (hasta 300 µM) de los
flavonoides, HES Hesperetina (a), LUT Luteolina (b), QUE Quercetina
(c), CAT Catequina (d), y RUT Rutina (e). La Figura interna muestra
la variación en el diferencial de la intensidad de la fluorescencia
corregida (ΔIFc, UA) frente a diferentes concentraciones del flavonoide.
Figure 3. Effect of flavonoids on lipase-ANS complex
extrinsic fluorescence. Flavonoids employed were HES Hesperetin (a),
LUT Lutein (b), QUE Quercetin (c), CAT Catechin (d), and RUT Rutin
(e) at different concentrations (1 – 300 μM). Inset shows the change
in the intensity of the corrected fluorescence (ΔIFc, AU) at
different flavonoid concentrations.
Además, QUE mostró un desplazamiento batocrómico (16 nm), que correspondería a un aumento en la polaridad alrededor del ANS, explicado por el desplazamiento de esta molécula de la región apolar de la enzima hacia el exterior de la proteína más polar (Lee et al., 2014). La disminución en la fluorescencia generada por los flavonoides como QUE, se puede deber a cambios de polaridad del medio que rodea al ANS, por un desplazamiento de este por parte del flavonoide o por cambios estructurales de la proteína por la interacción flavonoide-lipasa (Martinez-Gonzalez et al., 2017). La posible interacción lipasa-QUE tendría el mayor efecto sobre el medio que rodea a los sitios hidrofóbicos en la enzima, debido a que las interacciones hidrofóbicas son las que tienen la mayor contribución a la estabilidad de las proteínas (Pace et al., 2004). Un desplazamiento similar, pero de menor intensidad (p < 0.05) se observó para LUT. En el caso de RUT, esta se comportó similar a QUE, aun cuando la variación de intensidad de fluorescencia (ΔIFc) fue mayor que para QUE, lo que indicaría que este flavonoide glicosilado tiene mayor afinidad por lipasa que QUE, como fue señalado por otros autores por medio de estudios de fluorescencia intrínseca (Li et al., 2011). El mayor tamaño de RUT, por su componente glicosilado, podría impedir la unión del ANS en su sitio de la enzima, el cual podría ser el mismo sitio para el resto de los flavonoides. En un análisis de docking molecular realizado para las interacciones lipasa-ANS (datos no mostrados), se observó que el ANS podría unirse en un sitio cercano a la tríada catalítica de la enzima, y que sería el mismo que para la mayoría de los flavonoides. Esto también explicaría el menor efecto observado para CAT, mismo que coincide con su poca actividad inhibitoria. El desplazamiento hipsocrómico observado por CAT podría estar asociado también con el tipo de inhibición de este flavonoide, que expuso mayores sitios para la unión con el ANS. La competencia por el sitio de unión con el ANS señala el componente competitivo de la inhibición mixta de los flavonoides y coincide con la inhibición acompetitiva observada para el ANS.
Los valores calculados de la K D app para RUT, QUE y LUT fueron 96.70 ± 1.30, 92.41 ± 2.10, y 65.40 ± 7.30, respectivamente. Estos resultados indican que, en presencia de la RUT, el ANS presenta poca afinidad (valor alto de K D app). Es decir, la RUT tiene alta afinidad por un sitio hidrofóbico en la enzima, y desplaza al ANS. Diversas interacciones no covalentes como interacciones hidrofóbicas entre flavonoides y la lipasa pancreática han sido descritas (Li et al., 2011; Hu et al., 2015). Sin embargo, este efecto no ha sido descrito, mediante un análisis de interacciones de sitios hidrofóbicos con ANS. Este análisis es importante debido a que la lipasa cuenta con una región hidrofóbica que favorece la unión del sustrato, específicamente de las cadenas alifáticas del sustrato (Acharya y Madhusudhana, 2003), y es en esta región donde se podrían ubicar sitios de unión para los flavonoides hidrofóbicos por sus anillos aromáticos. Es decir, la unión de LUT, QUE y RUT con lipasa sería principalmente en sitios hidrofóbicos (desplazamiento del ANS). Características estructurales presentes en estos flavonoides, como el grupo oxo en el anillo C, la doble hidroxilación del anillo B (catecol), y el doble enlace C2 = C3 en el anillo C, permitirían estas uniones, en concordancia con los resultados cinéticos descritos previamente.
Por otra parte, el aumento en la intensidad de la fluorescencia del ANS en presencia de HES y CAT indicaría que el ANS tuvo mayor interacción con la proteína (Figura 4h). Probablemente, hubo una mayor exposición de regiones hidrofóbicas de la proteína para su unión con el ANS. Esto se podría deber a que la mayor flexibilidad de los anillos B y C de ambos flavonoides por la carencia del doble enlace C2 = C3, permitiría mayor movimiento sobre la superficie de la proteína, generando cambios en su estructura terciaria (Hawe et al., 2008), mismos que permitirían una mayor unión del ANS. Estos resultados concuerdan con los obtenidos de inhibición acompetitiva de CAT, en donde este flavonoide tendría mayor afinidad por la enzima unida al sustrato, que en este caso sería el ANS (por su sitio de unión cercano al sitio activo). En el caso de la HES, su comportamiento se podría atribuir a dos razones; i) La mayor unión del ANS con la enzima por el movimiento del flavonoide, ii) formación de un complejo enzima-ANS en el cual el ANS emite fluorescencia debido a sus dos grupos auxocromos (amino y sulfonato) unidos a la cadena polipeptídica (Gasymov y Glasgow, 2007), junto con un segundo complejo, ANS-HES, en el que el ANS emita fluorescencia por la interacción de su grupo metoxi con estos grupos auxocromos. Se requieren otros estudios para determinar la presencia de estas interacciones entre grupos metoxi y auxocromos.
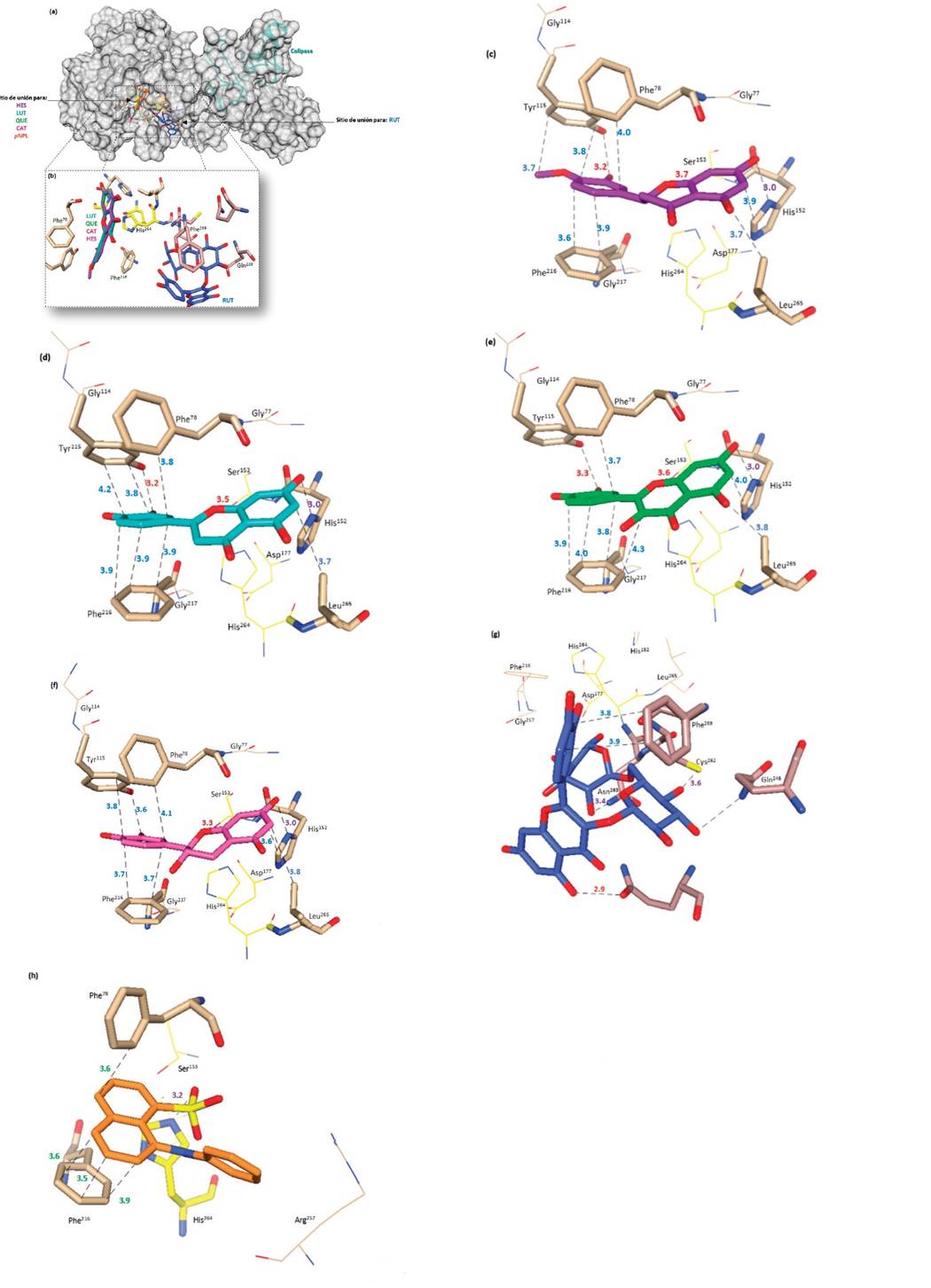
Figura 4 Docking molecular de la lipasa pancreática con los flavonoides Hesperetina (HES),
Luteolina (LUT), Quercetina (QUE), Catequina (CAT), Rutina (RUT), y
el sustrato, pNPL. Representación como superficie
de la estructura tridimensional completa de lipasa (gris oscuro),
indicando los aminoácidos del sitio activo (palos amarillos) y la
unión de los ligandos (a). El sitio de unión para los flavonoides y
los principales residuos (palos) para las interacciones se muestran
(b). Se señalan las interacciones específicas para la HES (c), LUT
(d), QUE (e), CAT (f), RUT (g) y ANS (h). Las distancias están
marcadas en líneas punteadas (negro) y con sus distancias (Å) en
distintos colores para los diferentes tipos de interacciones
evaluados (puentes de hidrógeno, rojo; electrostáticas, morado; e
hidrofóbicas, azul). Estas imágenes se obtuvieron usando USCF Chimera.
Figure 4. Molecular docking of the pancreatic lipase
with the flavonoids Hesperetin (HES), Lutein (LUT), Quercetin (QUE),
Catechin (CAT), Rutin (RUT), and the substrate pNPL. Surface
representation for the full tridimensional structure of lipase (dark
gray), the amino acids of the active site are represented (yellow
sticks) and the binding of the ligands (a). Flavonoids binding sites
and main amino acid residues (sticks) (b), as well as specific
interactions for HES (c), LUT (d), QUE (e), CAT (f ), RUT (g), and
the ANS (h) are shown. The dot lines (black) and distances (Å) with
colors correspond to different interactions such as hydrogen binding
(red), electrostatics (purple), and hydrophobic binding (blue). The
images were obtained with USCF Chimera
Análisis de las interacciones lipasa-flavonoides mediante docking molecular
El análisis de docking predice las interacciones entre lipasa y los flavonoides y los resultados se presentan en la Figura 4. Se muestran dos posibles sitios de unión en la proteína, uno para LUT, QUE, CAT y HES, y otro para RUT (Figura 4a). Ambos sitios se encuentran en cavidades cercanas al sitio activo. El sitio para la mayoría de los flavonoides está más cercano al sitio catalítico, como ha sido descrito para la interacción de lipasa con compuestos polifenólicos como la QUE (Martinez- Gonzalez et al., 2017a). Adicionalmente, los autores señalaron que este sitio es el mismo para sustratos, como el pNPL, e inhibidores de la actividad de lipasa, como el Orlistat. Esto concuerda con el análisis de docking realizado para el pNPL (Figura 4a). Mientras que el sitio para RUT, diferente al del resto de los flavonoides, se encuentra ubicado en una región comprendida entre el sitio activo y la colipasa (Zhang et al., 2018). La inhibición mixta observada para la mayoría de los flavonoides, indica que los flavonoides se unen al enzima antes y después de que el sustrato esté unido (Birari et al., 2011; Li et al., 2011). El componente competitivo de esta inhibición se presenta cuando los flavonoides se unen al residuo de Ser153, que pertenece a la tríada catalítica (Kokotos, 2003). Teniendo en cuenta los resultados cinéticos, que indican que la CAT presenta una inhibición acompetitiva, a partir de los resultados del docking, se propone que la CAT sólo se podría unir al complejo enzima-sustrato, probablemente debido a que la interacción lipasa-CAT no es tan estable como la de QUE. Un comportamiento similar se observa para HES. Ambos flavonoides (HES y CAT) tienen el mismo sitio de unión que LUT y QUE, sin embargo, la falta de estructura plana en su estructura no les permite mantener los enlaces con lipasa. En el caso de la RUT, su mayor tamaño por la presencia del disacárido en posición C3, no le permite ingresar en el mismo sitio que el resto de los flavonoides (Figura 4b). La estructura plana del anillo C de LUT y QUE les permite ingresar e interactuar con los residuos de aminoácidos del sitio activo (Cerezo et al., 2015). En este sentido, la mayoría de los flavonoides interactúan con el subsitio 1 (mayor actividad) de los tres sitios descritos por otros autores, y RUT con el subsitio 3, ambos hidrofóbicos (Zhang et al., 2018). Se señala que el Orlistat se une a los tres subsitios e impide el ingreso del sustrato. De esta manera, se propone que el mecanismo de inhibición de la enzima por los flavonoides está regulado por la entrada del sustrato al sitio activo, siendo esta la principal diferencia con el Orlistat.
En la Tabla 2 se muestran las principales interacciones analizadas en las predicciones para la unión lipasa-flavonoides. En concordancia con resultados previos, todas las interacciones son no covalentes (He et al., 2006; Birari y Bhutani, 2007; Zhang et al., 2014; Martinez-Gonzalez et al., 2017a). Se observaron uniones a través de fuerzas de Van der Waals con la His152 para los flavonoides. Pero, la mayoría de las interacciones son hidrofóbicas debido a que ambos sitios de unión están conformados principalmente por residuos hidrofóbicos, ya que corresponden a una región empleada en la interfase para la unión con el sustrato (Miled et al., 2000). Lo anterior concuerda con los estudios de fluorescencia extrínseca y con los resultados observados para la interacción con el ANS (ΔG = -9.4, Figura 4h).
Tabla 2 Resultados de la energía libre de Gibbs (ΔG, kCal mol-1), los residuos de aminoácidos presentes en la cavidad de lipasa, así como la distancia (Å) para las posibles conformaciones enzima-ligandos.
| Ligandos | Aminoácidos, distancia y energía libre de Gibbs para la interacción enzima-ligando | |||
|---|---|---|---|---|
| Interacción | ||||
| Fuerzas de Van der Waals | Hidrofóbica | Puentes de Hidrógeno (Å) | ΔG | |
| HES | His152(3.0) | Tyr115(3.7, 3.8), Phe78 (4.0), Phe216 (3.6; 3.9; 3.9), His152 (3.9), Leu265(3.7) | Tyr115 (3.2), Ser153(3.7) | -9.9 |
| LUT | His152(3.0) | Tyr115(4.2; 3.8), Phe78(3.8), Phe216(3.9; 3.9; 3.9), Leu265(3.7) | Try62(3.2), Ser153(3.5) | -10.0 |
| QUE | His152(3.0) | Phe78(3.7), Phe216(3.9; 4.0; 3.8; 4.3), His152 (4.0), Leu265 (3.8) | Tyr115(3.3), Ser153(3.6) | -9.8 |
| CAT | His152(3.0) | Tyr115 (3.8; 3.6), Phe78(4.1), Phe216(3.7; 3.7), His152(3.6), Leu265(3.8) | Ser153(3.3) | -9.7 |
| RUT | Asn263(3.4), Gln245(3.8), Cys262(3.6) | Phe259(3.9; 3.8) | Gln239 (2.9) | -9.0 |
Los datos son representados como media ± desviación estándar de los análisis realizados por triplicado. Las diferentes letras en una columna representan las diferencias significativas (Análisis de la diferencia mínima significativa de Fisher, p < 0.05), n.d. significa no determinado. HES, Hesperetina, LUT, Luteolina, QUE, Quercetina, CAT, Catequina, RUT, Rutina.
Values are mean ± standard deviation of triplicate analysis. Different letters in columns represent significant differences (Fisher, p < 0.05), n.d. not determined. HES, Hesperetin, LUT, Lutein, QUE, Quercetin, CAT, Catechin, RUT, Rutin.
En la interacción con flavonoides, se observaron interacciones del tipo pi-stacking con los residuos de Phe en las posiciones 78, 216 para el primer sitio (QUE, CAT, HES, LUT, pNPL), y 259 para el sitio de RUT. La unión con los flavonoides genera un cambio conformacional en la lipasa, abriendo la estructura. La conformación abierta de la lipasa permite la entrada del sustrato (Egloff et al., 1995; van Tilbeurgh et al., 1999), lo que coincide con la descripción para la inhibición mixta.
Sin embargo, en la Tabla 2 se indica que todos los flavonoides se podrían unir a este sitio, y entonces ¿en dónde radica la diferencia del poder inhibitorio de CAT sobre lipasa? La respuesta podría radicar en la movilidad del anillo C, que no le permite mantener interacciones hidrofóbicas entre anillos aromáticos, aunque haya proximidad del flavonoide con el sitio de unión. Se deben considerar aspectos de la estructura de la CAT, que a pesar de contar con anillos A y B similares a los de QUE, exhibió un efecto diferente al de este flavonoide. La carencia del grupo oxo en la posición 4 (anillo central, C) es un componente estructural de la CAT, el cual se podría relacionar con la carencia de interacciones estables. El 4-oxo es un grupo funcional que ha sido señalado como relevante respecto al mayor poder antioxidante de QUE en comparación con CAT (Cook y Samman, 1996; Plumb et al., 1999; Heim et al., 2002). Se requieren otros estudios para determinar si la responsabilidad del efecto inhibitorio de CAT (o HES) es debido a la presencia o ausencia del 4-oxo, aunado a la carencia del doble enlace y la flexibilidad, como análisis de microestados realizados por dinámica molecular (De Vivo et al., 2016).
Conclusiones
Quercetina, Luteolina y Rutina fueron los mejores inhibidores de la actividad de lipasa. Al parecer, el mejor inhibidor debe poseer tres características estructurales para tener la capacidad de interactuar con lipasa: i) Doble enlace entre el C2 y el C3, ii) la estructura de catecol en el anillo B, y iii) la doble hidroxilación en el anillo A. En donde, el doble enlace que le confiere estructura plana y rigidez a la estructura es la característica más relevante, al comparar los resultados con los de Hesperetina y Catequina.











 nueva página del texto (beta)
nueva página del texto (beta)


