Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Salud Pública de México
versión impresa ISSN 0036-3634
Salud pública Méx vol.44 no.5 Cuernavaca sep. 2002
Principales características y diagnóstico de los grupos patógenos de Escherichia coli
Guadalupe Rodríguez-Angeles, M en C.(1)
Rodríguez-Angeles G.
Principales características y diagnóstico de los grupos patógenos de Escherichia coli
Salud Publica Mex 2002;44:464-475.
El texto completo en inglés de este artículo está disponible en: http://www.insp.mx/salud/index.html
Resumen
Escherichia coli coloniza el intestino del hombre pocas horas después del nacimiento y se considera de flora normal, pero hay descritos seis grupos de E. coli productora de diarrea: enterotoxigénica (ETEC), enterohemorrágica (EHEC), enteroinvasiva (EIEC), enteropatógena (EPEC), enteroagregativa (EAEC) y de adherencia difusa (DAEC). La bacteria se puede aislar e identificar tradicionalmente con base en sus características bioquímicas o serológicas, pero también se pueden estudiar sus mecanismos de patogenicidad mediante ensayos en cultivos celulares o modelos animales y, más recientemente, empleando técnicas de biología molecular que evidencian la presencia de genes involucrados en dichos mecanismos. La intención del presente trabajo es resaltar la importancia del estudio y diagnóstico de E. coli como patógeno capaz de causar casos aislados o brotes de diarrea, síndrome urémico hemolítico, colitis hemorrágica y cuadros de disentería, principalmente en niños; por esto es necesario conocer mejor a la bacteria y mantener la vigilancia epidemiológica. El texto completo en inglés de este artículo está disponible en: http://www.insp.mx/salud/index.html
Palabras clave: Escherichia coli; sondas; diarrea; epidemiología
Rodríguez-Angeles G.
Diagnosis and main characteristics of Escherichia coli pathogenic groups.
Salud Publica Mex 2002;44:464-475.
The English version of this paper is available at: http://www.insp.mx/salud/index.html
Abstract
Escherichia coli colonizes the human intestinal tract within hours of birth and is considered a non-pathogenic member of the normal intestinal flora. However, there are six pathogenic groups that may produce diarrhea: enterotoxigenic (ETEC), enterohemorrhagic (EHEC), enteroinvasive (EIEC), enteropathogenic (EPEC), enteroaggregative (EAEC) and diffusely adherent (DAEC) groups. E. coli can be isolated and classified using traditional methods, by identifying its biochemical or serum characteristics. The pathogenic mechanisms may be studied in cell cultures and animal model assays, as well as more up to date molecular biology methods for study and diagnosis. The latter have proven that genes are involved in pathogenesis. The objective of the present work is to draw attention to the importance of E. coli as a pathogenic organism. This microorganism is an etiologic agent of sporadic cases of diarrhea, hemorrhagic colitis, dysentery, and hemolytic uremic syndromes and outbreaks. Diarrheic E. coli manifestations occur mainly among infants, and deep knowledge and understanding of this microorganism are crucial to better epidemiologic surveillance.
Key words: Escherichia coli; probes; diarrhea; epidemiology
Escherichia coli es un bacilo gram negativo, anaerobio facultativo de la familia Enterobacteriaceae, tribu Escherichia, cuyas principales características bioquímicas se indican en el cuadro I.1-3 Esta bacteria coloniza el intestino del hombre pocas horas después del nacimiento y se le considera un microorganismo de flora normal, pero hay cepas que pueden ser patógenas y causar daño produciendo diferentes cuadros clínicos, entre ellos diarrea.

Para determinar el grupo patógeno al que pertenecen Kauffman desarrolló un esquema de serotipificación que continuamente varía y que actualmente tiene 176 antígenos somáticos (O), 112 flagelares (H) y 60 capsulares (K). El antígeno "O" es el responsable del serogrupo; la determinación del antígeno somático y flagelar (O:H) indica el serotipo, el cual en ocasiones se asocia con un cuadro clínico en particular. El cuadro II muestra algunos serotipos más frecuentemente asociados con los grupos patógenos.2-5
La serotipificación de E. coli requiere de gran número de antisueros. Como hay pocos laboratorios que la realizan, se prefiere identificar las cepas mediante sus factores de virulencia empleando ensayos in vitro como por ejemplo ensayos de adherencia en células Hep-2 y ensayos de toxigenicidad en células. También se pueden realizar ensayos in vivo, como el asa ligada o la prueba de Sereny, así como ensayos inmunológicos y pruebas de biología molecular, para poner de manifiesto la presencia de fragmentos de genes involucrados en el mecanismo de patogenicidad y que sirvan de marcadores moleculares. En el cuadro III se muestran algunos métodos de estudio de los diferentes grupos patógenos o patotipos de E. coli.1,2,4
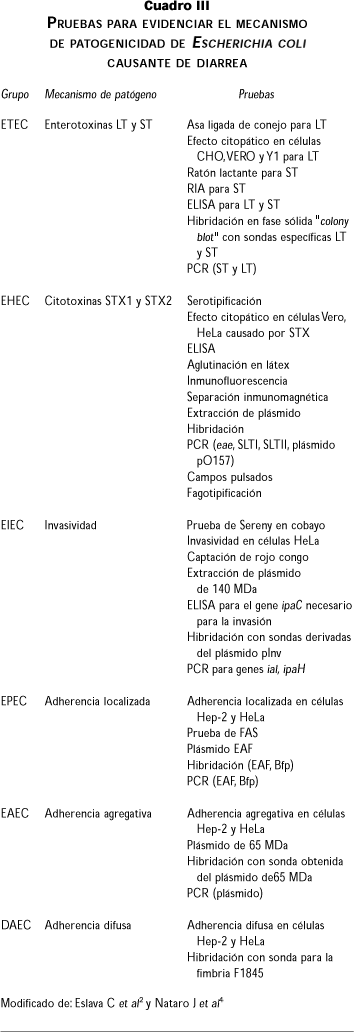
Con base en su mecanismo de patogenicidad y cuadro clínico, las cepas de E. coli causantes de diarrea se clasifican en seis grupos: enterotoxigénica (ETEC), enterohemorrágica también conocidas como productoras de toxina Vero o toxina semejante a Shiga (EHEC o VTEC o STEC), enteroinvasiva (EIEC), enteropatógena (EPEC), enteroagregativa (EAEC) y adherencia difusa (DAEC), cuyas características principales se describirán brevemente y se resumen en el cuadro IV.1,2,4
E.coli enterotoxigénica
Las ETEC colonizan la mucosa del intestino delgado por medio de pilis o fimbrias que tienen diversas formas denominadas CFA (colonization factor antigens), siendo su principal mecanismo de patogenicidad la síntesis de alguna o ambas enterotoxinas llamadas toxina termolábil (LT) y toxina termoestable (ST). Sus genes están en un plásmido que también puede tener información genética para los CFA´s, aunque algunos genes de ST se han encontrado en transposones.6-8 Las toxinas LT y ST aumentan el nivel intracelular de cAMP y cGMP respectivamente, que se encuentran en la membrana de las células intestinales, provocando la salida de agua y iones.9
Las ETEC son importantes en lactantes, principalmente en niños menores de dos años, y en particular durante los primeros seis meses de vida. La frecuencia de aislamiento de este grupo patógeno de E. coli en niños con diarrea es de 10 a 30%. En los niños en edad escolar y en adultos puede ser asintomática y poco frecuente o producir la diarrea del viajero. La enfermedad tiene un periodo de incubación de 14 a 50 h. El cuadro clínico se caracteriza por diarrea aguda, generalmente sin sangre, sin moco, sin pus y en pocos casos se presentan fiebre y vómito. La diarrea producida por ETEC puede ser leve, breve y autolimitada pero también puede ser grave.2,4,10
La contaminación fecal de agua y alimentos es la principal fuente de infección, siendo la dosis infectiva de 108 UFC (unidades formadoras de colonias). El cuadro III indica los métodos de diagnóstico.
E. coli enterohemorrágica
Riley describió y relacionó a EHEC con brotes caracterizados por dolor abdominal, diarrea acuosa con sangre y poco o nada de fiebre, cuadro al que se le llamó colitis hemorrágica (CH) y que era debido a la ingestión de carne cruda o mal cocida.11 La bacteria aislada de todos los casos fue E. coli del serotipo O157:H7. Karmali en 1983,12 la asoció con casos aislados de síndrome urémico hemolítico (SUH) caracterizado por daño renal agudo, trombocitopenia y anemia hemolítica microangiopática, precedida por diarrea con sangre, con la presencia en heces de E. coli productora de una citotoxina con actividad en células Vero, por lo que se le llama verotoxina (VT), y a las cepas capaces de producirla se les denominó E. coli verotoxigénicas (VTEC).13 Además, se observó que la citotoxina se neutralizó con antitoxina obtenida a partir de Shigella dysenteriae tipo 1, por lo que también se le llamó "shiga-like toxin" o toxina semejante a shiga (SLT) o "shiga toxin" (STX), y a las E. coli capaces de producirla se les da el nombre de STEC.4,14
La capacidad toxigénica de las cepas es necesaria para que el paciente desarrolle colitis hemorrágica y diarrea con sangre, ya que la citotoxina STX es el principal mecanismo de patogenicidad de EHEC y su síntesis está relacionada con la presencia del bacteriófago STX, que está insertado en el genoma. La STX actúa a nivel de síntesis de proteínas ya que se une a la subunidad 60S de los ribosomas de las células intestinales o renales del hospedero.15 En las cepas EHEC aisladas, se han encontrado las variantes STX1 y STX2 que son inmunológicamente diferentes, de tal manera que se pueden aislar bacterias que sinteticen alguna de las toxinas o ambas.4,5,16 Además de la toxina, las EHEC tienen otros mecanismos de patogenicidad como el fenómeno de adherencia y esfacelación (A/E), y presentan el gene cromosomal eae que codifica para la proteína de membrana externa (OMP) de 94 kilodaltones (kDa), llamada intimina, cuya expresión es regulada por genes plasmídicos; el gene eae también se encuentra en las cepas EPEC. Otro factor de patogenicidad es el plásmido pO157, de 60 megadaltones (MDa), que codifica para la enterohemolisina.17,18
Actualmente hay al menos dos clasificaciones del grupo EHEC. Una es en función de la presencia de sus factores de patogenicidad: a) cepas típicas cuando tienen el fago, el plásmido de 60 MDa y presentan el fenómeno de A/E, y b) cepas atípicas, cuando no producen lesiones de A/E y pueden presentar o no el plásmido de 60 MDa.
La otra clasificación es en función del serotipo:
a) cepas E. coli O157:H7. Este serotipo no fermenta el D-sorbitol ni la ramnosa y no produce ß-glocuronidasa; esta bacteria puede producir principalmente SUH y CH. E. coli O157:H7 se puede encontrar en bovinos, cabras, borregos y con menos frecuencia en cerdos y pollos; su principal reservorio es el intestino de ganado bovino.5 También se ha logrado recuperar de frutas y vegetales como lechuga, rábanos, alfalfa; además en productos industrializados como mayonesa y jugos de naranja y manzana no pasteurizados, aun cuando estos alimentos tengan un pH de 3.4, condición en la que puede sobrevivir varios días. La transmisión de E. coli O157:H7 puede ser por ingerir carne cruda o mal cocida, leche bronca, agua contaminada; también puede ser de persona a persona o debida a los manipuladores de alimentos.19-21 Hay estudios que sugieren la importancia de la mosca doméstica como vector en la transmisión de E. coli O157:H7,22 y b) cepas no-O157:H7 cuya frecuencia de aislamiento es cuatro veces mayor que las O157:H7.
Estas cepas pueden ser sorbitol positivo, y sus serotipos son diferentes del O157:H7. Actualmente hay más de 200 serotipos, como muestra el cuadro II. El cuadro clínico causado por las cepas no-0157 se caracteriza por diarrea acuosa con dolor abdominal y colitis hemorrágica. Las cepas no-O157 son capaces de causar brotes o casos aislados de diarrea y en ocasiones no se logra establecer la fuente de contaminación, aunque se sabe que se pueden aislar de los mismos alimentos que las cepas de serotipo O157:H7 y también de carne de guajolote, ternera, pescado y mariscos.4,5
El principal mecanismo de patogenicidad de las cepas EHEC no-O157:H7 es la toxina STX; también tienen el fenómeno de A/E y la presencia de pO157.
El periodo de incubación de EHEC es de 1 a 8 días; inicialmente produce diarrea sin sangre, con o sin vómito, dolor abdominal, fiebre, y después de 1 a 2 días la diarrea se torna sanguinolenta y se intensifica el dolor abdominal, de una duración de 4 a 10 días, con heces abundantemente sanguinolentas. Se cura o bien llega hasta SUH.
La identificación de EHEC se puede hacer como indica el cuadro III. Las pruebas moleculares como RFLP, hibridación, campos pulsados, PCR y RAPD-PCR pueden detectar hasta 102 UFC/0.1g de materia fecal, además de que permiten realizar una subtipificación con fines epidemiológicos de este grupo de bacterias.23-25
Es importante el trabajo conjunto del laboratorio y los epidemiólogos para la vigilancia y detección oportuna de E. coli O157:H7 para prevenir posibles brotes en México, principalmente en zonas turísticas y fronterizas.
E. coli enteroinvasiva
El grupo EIEC y Shigella spp están relacionados genética y bioquímicamente ya que son descarboxilasa negativas, no móviles y lactosa negativas. El mecanismo de patogenicidad de EIEC es la invasión del epitelio del colon; para ello el primer paso es la adherencia de la bacteria a las vellosidades de la mucosa requiriendo de mucinasa y adhesinas, para después entrar por endocitosis a la célula, y posterior multiplicación de la EIEC dentro de la célula y diseminación a células sanas adyacentes.4,26 La información genética para este mecanismo está en loci del cromosoma y del plásmido, además de tener la capacidad de elaborar una o más enterotoxinas que pudieran ser importantes en la producción de diarrea. Los genes necesarios para la invasión se encuentran en un plásmido de 140 MDa llamado pInv, que codifica para proteínas, como por ejemplo las Ipa y otras que están involucradas en el proceso de patogénesis.2,27,28
Los síntomas característicos en personas infectadas por EIEC son diarrea acuosa, con sangre y moco, pero algunos casos sólo presentan diarrea, ésta en ocasiones es indiferenciable de la que produce ETEC. Las cepas EIEC se asocian más con brotes que con casos aislados, en los cuales la transmisión puede ser de persona a persona, por ingestión de alimentos y agua contaminada, convirtiéndose en un patógeno importante en niños mayores de seis meses.2,29
El diagnóstico de EIEC se hace por prueba in vivo como la de Sereny, que es la inoculación de un cultivo puro de la bacteria en un ojo de un cobayo en el cual después de 24 a 96 h se produce una ulceración en el ojo, pero también hay métodos inmunológicos y moleculares como se indica en el cuadro III.30-32
E. coli enteropatógena
EPEC fue el primer grupo que se identificó serológicamente y se asoció con casos de diarrea en infantes, siendo la adherencia su principal factor de patogenicidad. El proceso de adherencia íntima entre la bacteria y la membrana de las células del epitelio intestinal, seguida de la destrucción de la microvellosidad, con polimerización de actina, que lleva a la alteración del citoesqueleto en el sitio de la unión de la bacteria, debido al aumento de los niveles de calcio intracelular y de proteína cinasa C, ha sido denominado adherencia y esfacelamiento (A/E).2,33 La adherencia está mediada por pilis o fimbrias rizadas que se llaman Bfp (bundle-forming pilus) cuya información genética está codificada en un plásmido de 50-70MDa denominado EAF (EPEC adherence factor) y de algunos genes cromosomales.34,35 En la adherencia es necesaria la síntesis de una proteína de membrana externa de 94 kDa llamada intimina, codificada por el gene cromosomal eae y que sirve como señal en A/E.2,4
En ensayos in vitro las cepas EPEC se caracterizan por formar microcolonias en el citoplasma de las células Hep-2 y su estudio incluye factores de patogenicidad como el efecto A/E, presencia de plásmidos y fimbrias.2,4,36
Las cepas EPEC se consideran típicas cuando tienen los genes eae para la intimina, que participa en A/E, y el plásmido EAF que codifica para el Bfp; se dice que son atípicas cuando sólo presentan los genes eae pero no el plásmido EAF.4,37
EPEC puede ocasionar brotes o casos aislados de diarrea. Este grupo afecta principalmente a niños menores de seis meses y a los de dos años. También puede aislarse en adultos enfermos y sanos, principalmente cuando hay un factor predisponente como diabetes. La forma de transmisión de la enfermedad es fecal-oral por manos contaminadas de manipuladores de alimentos. Los reservorios de EPEC pueden ser niños y adultos con o sin síntomas.
El cuadro clínico que produce EPEC se manifiesta con diarrea aguda, la cual puede ser leve o grave, con vómito, fiebre baja y mala absorción .
El diagnóstico de EPEC incluye ensayos in vitro en cultivos celulares y métodos moleculares como se observa en el cuadro III.
E. coli enteroagregativa
Scaletsky y Nataro encontraron cepas aisladas de pacientes con diarrea, las cuales por serología no correspondían al grupo EPEC pero si presentaban un patrón característico de adherencia diferente a EPEC y que además eran negativas a la prueba de EAF. En estudios posteriores se encontró el fenotipo de adherencia agregativa, caracterizada por autoaglutinación de las bacterias entre sí y por ser inespecífica, porque las bacterias se adhieren a la superficie de las células Hep-2 y a la superficie del cubreobjetos libre de células Hep-2.2,4,38
La adherencia a células Hep-2 y la hemaglutinación de eritrocitos humanos se debe a la presencia de una fimbria o adhesina flexible llamada fimbria I de adherencia agregativa (AAF/I), codificada por el gene aggA que se encuentra en un plásmido de 60 MDa. También se ha descrito la fimbria AAF/II inmunológicamente diferente a AAF/I y que está codificada por el gene aafA; sin embargo, no todas las EAEC presentan estas fimbrias.39,40
En el mecanismo de patogenicidad de EAEC están implicadas la bacteria y diversas moléculas que ella produce; también se sabe que las cepas EAEC tienen la capacidad de incrementar en la mucosa la producción y secreción de moco que atrapa a las bacterias que se autoaglutinan en una fina película en el epitelio intestinal. La producción de moco puede estar relacionada con la capacidad de EAEC para colonizar persistentemente el intestino y causar diarrea. En el plásmido de 60 MDa de EAEC también se encuentran los genes que codifican para la toxina EASTI.41,42
Eslava y colaboradores identificaron dos proteínas de alto peso molecular de cepas EAEC, aisladas de niños que murieron de diarrea persistente. Estas proteinas fueron probadas en asa ligada de rata observándose vellosidades cortas, hemorragia y necrosis. El gene para una de estas proteínas se encontró en un plásmido de 65 MDa y a la proteína se le dio el nombre de Pet (plasmid-encoded toxin) la cual tiene la capacidad de producir efecto citopático en células Hep-2, caracterizado por arredondamiento y desprendimiento de las células así como contracción del citoesqueleto y pérdida de fibras de actina. En EAEC se ha descrito también la proteína Pic que está codificada en el genoma y que tiene actividad de proteasa.43-45
El sitio blanco de daño de EAEC puede ser la mucosa del intestino grueso y delgado, con un periodo de incubación de menos de ocho horas y puede durar hasta 18 o 20 días. Esta bacteria puede causar brotes o casos aislados de diarrea persistente. En niños puede manifestarse con diarrea líquida, de color verde, con moco, sin sangre, y que en ocasiones puede llegar a ser severa y requerir rehidratación intravenosa. Algunas veces el cuadro clínico se presenta como diarrea con moco con o sin sangre, vómito y sin o con poca fiebre.4,46,47 La prueba de referencia para el diagnóstico de EAEC es la observación de la adherencia agregativa en células Hep-2 de cultivos bacterianos, previamente inoculados en medio Luria e incubados en condiciones estacionarias y a 37 °C. Otras pruebas de diagnóstico se indican en el cuadro III.36,48,49
E. coli de adherencia difusa
Las cepas de E. coli de adherencia difusa, no forman microcolonias cuando se adhieren a células Hep-2 (2). Se sabe poco de su mecanismo de patogenicidad pero se ha caracterizado una fimbria de superficie, conocida como F1845, involucrada en el fenómeno de adherencia difusa.50 Los genes que codifican para esta fimbria se pueden encontrar en el cromosoma o en un plásmido. El fenómeno de adherencia difusa también se ha asociado con una proteína de membrana externa de 100 kDa, en una cepa del serotipo 0126:H27, cuyos genes se han secuenciado pero sólo se han encontrado en una minoría de las cepas aisladas.51 Al realizar ensayos in vitro en células CaCo y Hep-2, las cepas DAEC tienen la capacidad de inducir la formación de estructuras protuberantes, semejantes a dedos, las cuales confieren protección a las bacterias, pero la presencia de dichas estructuras no se ha demostrado in vivo.
El grupo DAEC se puede aislar tanto de personas sanas como en personas con diarrea, siendo más importante en niños de 4 a 5 años. Los principales síntomas que se presentan son diarrea acuosa sin sangre y sin leucocitos. En el cuadro III se indican los métodos de diagnóstico. La hibridación con sondas derivadas de un fragmento del gene daaC, que codifica para la fimbria F-1845, también se ha empleado para el diagnóstico pero puede presentar falsos positivos, y el diagnóstico empleando PCR aún no se ha desarrollado.4,52
Aislamiento, identificación y caracterización de E. coli patógena
Para el aislamiento, la identificación y la caracterización de cepas de E. coli se aplican métodos tradicionales, métodos in vivo e in vitro y de biología molecular; a continuación se mencionarán los métodos tradicionales y los de biología molecular.1,3,4
El método tradicional es el aislamiento de la bacteria, tomada directamente de materia fecal o con hisopo rectal. Después se siembra con la punta del hisopo en la parte superior de una placa de agar Mac Conkey u otro medio selectivo y, con una asa redonda de nicromel, se continúa el aislamiento, sembrando por estría cruzada; después se incuba a 37 °C durante 18-24 h. Posteriormente se seleccionan de 5 a 10 colonias típicas de E. coli lactosa positivas.
En muestras provenientes de casos de diarrea con sangre se deben seleccionar también cepas lactosa negativa que pudieran ser EIEC.
La identificación se hace mediante pruebas bioquímicas en tubo como TSI, LIA, MIO, citrato, sorbitol, mucato, urea, rojo de metilo, Voges Proskauer, malonato y caldo manitol-rojo de fenol. Estas pruebas se interpretan de acuerdo con el cuadro I. Simultáneamente se siembra la cepa en tubos de agar base sangre (BAB), sin sangre para posteriormente hacer la serología.2,4,5
En los laboratorios y hospitales que cuentan con antisuero polivalente A, B o C de EPEC se realiza la prueba de aglutinación en niños con diarrea con moco y sangre, especialmente menores de un año.
Cuando se sospecha la presencia de EHEC, en muestras provenientes de diarrea con sangre, el diagnóstico se hace empleando agar Mac Conkey con 1% de sorbitol (SMAC) en lugar de lactosa, se seleccionan de tres a 10 colonias sorbitol negativo que son incoloras y sospechosas de ser O157:H7. Este agar se debe considerar sólo como un medio de selección y nunca como una forma definitiva de diagnóstico ya que no todas las cepas sorbitol negativo son E. coli O157:H7 y hay cuadros de SUH producidos por cepas no-O157:H7 que son sorbitol positivo.19,53,54
Para aumentar la posibilidad de aislar E. coli O157:H7 se puede efectuar un preenriquecimiento selectivo de 4 h, o de toda la noche, en medio de soya tripticaseína suplementado con 50 ng/ml de cefaxima y 40 µg/ml de vancomicina. El medio SMAC se puede hacer selectivo y diferencial si se adicionan cefaxima y telurito que permiten el crecimiento de E. coli O157:H7 y Shigella sonnei pero inhibe, parcial o totalmente, el crecimiento de otras E. coli. También se puede usar novobiocina para aumentar la selectividad del medio.4,5,21 En el laboratorio se debe tener cuidado con el número de resiembras que se hacen a las cepas después del primoaislamiento, ya que la estabilidad de la capacidad toxigénica de la bacteria varía de un microorganismo a otro y se pierde con un alto número de pases, principalmente en los serotipos O2:H5 y O73:H34 mientras que en las cepas O26:H11 es muy estable.2,4
La identificación del grupo EHEC se puede hacer por serotipificación o bien por poner de manifiesto la producción de la toxina STX también se puede cuantificar la elevación de anticuerpos dirigidos hacia el lipopolisacárido de E. coli O157:H7, así como demostrar la presencia de factores de virulencia como pO157, el fenómeno de A/E o los genes involucrados, además de la fagotipificación.24
La demostración de la producción de las toxina LT y STX de E. coli se puede realizar por un modelo in vitro que permita observar el efecto citopático en cultivo de células Vero (células de riñón de mono verde), CHO (células de ovario de hamster chino) o HeLa (células de carcinoma cérvico-uterino) (2,55). Para ello se requiere sembrar la cepa pura aislada del caso de diarrea en medio de Craig e incubar 24 h a 37ºC, posteriormente por filtración se separan las bacterias del sobrenadante que contiene la toxina, y se adicionan 20µl del sobrenadante a cultivos confluentes de células Vero. La toxina y las células se dejan en contacto hasta observar la aparición de un efecto citotónico (células alargadas) en el caso de toxina LT o bien efecto citotóxico (células redondas y muertas) debido a la toxina55,56 STX.
La caracterización y clasificación de cepas patógenas de E. coli se puede hacer con métodos de biología molecular, una de las herramientas de diagnóstico más recientes, tal es el caso del uso de sondas para la hibridación en fase sólida como es el "colony blot". Las sondas son fragmentos pequeños de DNA que contienen parte de los genes que codifican para algún factor de virulencia y pueden estar marcadas radioactivamente con 32P o no radiactivamente con biotina o digoxigenina y se pueden usar en ensayos sensibles y específicos para detectar cepas patógenas de interés clínico.57 Actualmente para E. coli hay sondas específicas para la toxina termolábil (LT) y termoestable (ST) del grupo ETEC, para la fimbria Bfp de EPEC, para el locus asociado con invasividad (ial) de EIEC y para la enterohemolisina (hlyA) de EHEC, entre otros factores de patogenicidad característicos de las bacterias.
El colony blot es la transferencia del DNA de una colonia de bacterias a una fase sólida que puede ser nylon, papel filtro o nitrocelulosa, para su posterior hibridación. Para esto las colonias puras, previamente aisladas de pacientes con diarrea, se inoculan directamente en forma ordenada sobre una placa de agar Luria y se incuban 4 h a 37 °C. Después de este tiempo se coloca la membrana de nylon sobre la superficie de la placa con las colonias en crecimiento y se incuba toda la noche.55
Las bacterias se rompen sobre la membrana y el DNA se desnaturaliza al colocar ésta en una solución de hidróxido de sodio para posteriormente fijarle el DNA. La hibridación se realiza con una sonda marcada con digoxigenina, la cual es un fragmento de un gene de virulencia específico, y se pone de manifiesto empleando un anticuerpo antidigoxigenina conjugado a la enzima fosfatasa alcalina, cuyo sustrato es el BCIP, y el NBT es el cromógeno que desarrolla un color negro o café cuando se incuba la membrana a temperatura ambiente y en la oscuridad.56,58
Otro método es la reacción de polimerización en cadena (PCR), una hibridación en fase líquida, en donde la hibridación se realiza entre el DAN blanco presente en la muestra y el iniciador, que es una secuencia conocida de un fragmento específico de un gene involucrado en la patogenicidad de cepas de E. coli.56,57 La muestra puede ser una cepa pura aislada de muestra clínica obtenida de un caso aislado de diarrea o bien de brotes, de viajeros, de peregrinos o durante desastres naturales. También puede ser materia fecal o alimentos implicados en brotes en los que se necesita confirmar la presencia de E. coli. La cepa se siembra por estría y se incuba durante 24 h; a continuación se resuspenden cinco colonias de bacterias en 0.2 ml de agua y se somete a ebullición para desnaturalizar el DNA. De la suspensión se toma una alícuota para el tubo donde se realiza la PCR. Los reactivos necesarios para la PCR de cada muestra son adenina, timina, citocina, guanina, MgCl2, iniciadores y enzima Taq polimerasa.
La síntesis in vitro del fragmento de DNA o amplificación se efectúa mediante cambios de temperatura en tres fases: una de desnaturalizacion, una de hibridación y fase de alargamiento, cuyas condiciones serán diferentes en función del gene que se desea amplificar. Al finalizar la amplificación se toman alícuotas y se someten a electroforesis en gel de agarosa para observar la presencia del producto amplificado. En esta prueba se requiere una área destinada a la preparación de las muestras para PCR y otra sólo para productos de PCR, ya que se debe evitar posibles contaminaciones con productos de PCR que puedan interferir en el resultado de la prueba y dar falsos positivos.59,60
El cuadro V presenta algunas secuencias de iniciadores y sondas empleadas en el diagnóstico.4,61
En el InDRE desde 1993 se empezó la estandarización de métodos de biología molecular como "colony blot" y PCR los cuales desde 1996 se emplean con fines diagnósticos y como apoyo a la vigilancia epidemiológica, para la caracterización de E. coli patógena aislada de casos de diarrea y cuyos resultados han sido presentados en diversas reuniones científicas.62
Los resultados obtenidos indican que el grupo más frecuente es el enterotoxigénico (48%), seguido por el enteroinvasivo (9%), el enteropatógeno (4%) y el enterohemorrágico (1%), del cual sólo se han aislado cepas no O157:H7. Además, se encontró que la presencia de E. coli es mayor durante los meses húmedos y calurosos y afecta principalmente a niños menores de cinco años. Actualmente se realiza la estandarización del método para la identificación del grupo enteroagregativo.63,64
La electroforesis de campo pulsado (pulsed-field gel electrophoresis PFGE) es un método de biología molecular que cada vez se emplea más con fines epidemiológicos, debido a que separan largos fragmentos de DNA que se obtienen al digerir DNA genómico con enzimas de restricción que realizan cortes poco frecuentes. Los patrones de bandas generados por PFGE se han usado en el análisis de microorganismos como Pseudomonas spp, Mycobacterium spp, Campylobacter spp y E. coli, provenientes de casos aislados y brotes, para establecer o descartar su posible relación filogenética. Por esta razón es necesario contar con una colección de cepas, en este caso de E. coli, que nos permita realizar en México el análisis, y determinar la relación que hay entre las clonas patógenas circulantes de este microorganismo, así como conocer su importancia epidemiológica.65 Para estos fines hay equipos comerciales (Kits), de la marca Bio-Rad para E. coli O157:H7 que pueden ayudar en el análisis.
Las muestras o cepas enviadas a un laboratorio deben remitirse con la siguiente información: fuente de aislamiento (humano o tipo de alimento), en caso de ser de origen humano indicar la edad y sexo del paciente, la localidad, si se trata de un caso o contacto, qué alimentos ingirió tres días previos al inicio de la diarrea, si viajó o asistió a un evento social, el tipo de diarrea (acuosa, con moco, con sangre), número de evacuaciones y durante cuántos días, si hubo deshidratación, si se dio tratamiento y cuál fue; todo esto con fines epidemiológicos y poder detectar oportunamente la presencia de un brote o de cepas de E. coli del serotipo O157:H7 para las cuales la vigilancia epidemiológica debe ser permanente, sobre todo en regiones fronterizas y turísticas.
En conclusión, cuando el laboratorio reporte el aislamiento de E. coli como patógeno, en un cuadro de diarrea, se debe tener presente su importancia como agente causal de cuadros graves de diarrea principalmente en niños menores de cinco años y no sólo considerarla como una bacteria de flora normal.
Referencias
1. Boop CA, Brenner FW, Wells JG, Strockbine N. Escherichia, Shigella, and Salmonella. En: Manual of clinical microbiology. Murray PR, E Jo Baron, MA Pfaller, FC Tenover, RH Yolken. 7thed Washington, D.C. E d. ASM. Press. 1999:459-474. [ Links ]
2. Eslava C, Mateo J, Cravioto A. Cepas de Escherichia coli relacionadas con la diarrea. En: diagnóstico de laboratorio de infecciones gastrointestinales. Giono S, Escobar A, Valdespino JL. Secretaria de Salud. México, 1994: 251. [ Links ]
3. Farmer JJ III. Enterobacteriaceae: Introduction and identification. En: Manual of clinical microbiology. 6ª ed . Washington, D.C. ASM Press1995: 440. [ Links ]
4. Nataro JP, Kaper JB. Diarrheagenic Escherichia coli. Clin Microbiol Rev 1998;11:142-201. [ Links ]
5. World Health Organization. Zoonotic non-O157 shiga toxin producing Escherichia coli (STEC). Report of a WHO scintific workin group meetin. 1998. [ Links ]
6. Cassels FJ, Wolf MK. Colonization factors of diarrheagenic E. coli and their intestinal receptors. J Ind Microbiol 1995;15:214-226. [ Links ]
7. Gutiérrez-Cázarez Z, Qadri F, Albert MJ, Giron JA. Identification of enterotoxigenic Escherichia coli harboring longus type IV pilus gene by DNA amplification. J Clin Microbiol 2000;38:1767-1771. [ Links ]
8. Mc Veigh A, Fasano A, Scott DA, Jelacic S, Moseley SL, Robertson DC et al. IS1414 an Escherichia coli insertion sequence with a heat-stable enterotoxin gene embedded in a transposase-like gene. Infect Immun 2000;68:5710-5715. [ Links ]
9. Sears CL, Kapper JB. Enteric bacterial toxins: Mechanisms of action and linkage to intestinal secretion. Microbiol Revs 1996;60:167-215. [ Links ]
10. Flores-Abuxapquí JJ, Suárez-Itoil GJ, Heredia-Navarrete MR, Puc-Franco MA, Franco-Monsreal J. Frequency of enterotoxigenic Eschericia coli in infants during the first three months of life. Arch Med Res 1994; 25:303-307. [ Links ]
11. Riley LW, Remis RS, Helgerson SD, McGee HB, Wells JG, Davis BR et al. Hemorrhagic colitis associated with a rare Escherichia coli serotype. N Engl J Med 1983;308:681-685. [ Links ]
12. Karmali MA, Steele BT, Petric M, Lim C. Sporadic cases of haemolytic uremic syndrome associated with fecal cytotoxin and cytotoxin- producing Escherichia coli in stools. Lancet 1983;1:619-620. [ Links ]
13. Konowalchuk J, Speirs JI, Stavric S. Vero response to a cytotoxin of Escherichia coli. Infect Immun 1977;18:775-779. [ Links ]
14. O´Brien A, GD LaVeck, MR Thompson, Formal SB. 1982. Production of Shigella dysenteriae type 1-like cytotoxin by Escherichia coli. J Infect Dis 1977;146:763-769. [ Links ]
15. O´Brien AD, Holmes RK. Shiga and Shiga-like toxins. Microbiol Revs 1987;51:206-220. [ Links ]
16. Cravioto A, Vázquez V, Soria A, Navarro A, Ortiz M. Producción de citotoxina tipo shiga (SLT)1 en cepas de Escherichia coli aisladas de niños con diarrea en una comunidad rural. Bol Med Hosp Infant Mex 1988; 45: 206-210. [ Links ]
17. Donnenberg MSS, Tzipori S, McKee ML, O´Brien AD, Alroy J, Kaper JB. The role of the eae gene of enterohemorrhagic Escherichia coli in intimate attachment in vitro and in a porcine model. J Clin Invest 1993;92:1418-1424. [ Links ]
18. Schmidt H, Beutin L, Karch H. Molecular analysis of the plasmid-encoded hemolysin of Eschericia coli O157:H7 strain 933. Infect Immun 1995;63:1055-1063. [ Links ]
19. Tarr PI. Escherichia coli O157:H7 clinical, diagnostic and epidemiological aspects of human infection. Clin Infect Dis 1995;20:1-10. [ Links ]
20. Mattar S, Vásquez E. Escherichia coli O157:H7 infection in Colombia. Emerging Infec Dis 1998;4:126-127. [ Links ]
21. Mattar S, Vásquez E. Importancia de la vigilancia de un brote de Escherichia coli. Bol Oficina Sanitaria Panamericana 1996;120:523. [ Links ]
22. Kobayashi M, Sasaki T, Saito N, Tamura K, Suzuki K, Watanabe H et al. Houseflies: Not simple mechanical vector of enteroheorrhagic Escherichia coli O157:H7. Am J Trop Med Hyg 1999;61:625-629. [ Links ]
23. Fields PI, Blom K, Jeanette H, Helsel LO, Feng P, Swaminathan B. Molecular characterization of the gene encoding H antigen in Escherichia coli of a PCR-restriction fragment length polymorphism test for identification of E. coli O157:H7 and O157:NM. J Clin Microbiol 1997;35:1066-1070. [ Links ]
24. Preston MA, Johnsonson W, Khakhria, Borczyk A. Epidemiologic subtyping of Escherichia coli serogroup O157 strains isolated in Ontario by phage typing and pulsed-field gel electrophoresis. J Clin Microbiol 2000; 38:2366-2368. [ Links ]
25. Fratamico PM, Sackitey SK, Wiedmann M, Deng MY. Detection of Escherichia coli O157:H7 by multiplex PCR. J Clin Microbiol 1995;33:2188-2191. [ Links ]
26. Rico-Martínez MG. Biología molecular en la patogenia de Shigella sp y Escherichia coli enteroinvasiva. Rev Latinoam Microbiol 1995;37:367-385. [ Links ]
27. Halet T, Sansonetti P, Schad P, Austin S, Formal SB. Characterization of virulence plasmids and plasmid associated outer membrane proteins in Shigella flexneri, Shigella sonnei and Escherichia coli. Infect Immun 1983;40: 340-350. [ Links ]
28. Sethabutr O, Venkatesan M, Yam-Pang LW, Smoak BL, Sang WK et al. Detection of PCR products of the ipaH gene from Shigella and enteroinvasive Escherichia coli by enzyme linked immunosorbent assay. Diagn. Micbrobiol Infect Dis 2000;37:11-16 [ Links ]
29. Snyder JD, Wells JG, Yashuk J, Puhr N, Blake PA. Outbreak of invasive Escherichia coli gastroenteritis on a cruise ship. Am J Trop Med Hyg 1984; 33:281-284. [ Links ]
30. Wood LV, Morris JG, Small PL, Sethabutr O, Toledo MR, Trabulsi L et al. Comparison of DNA probes and the Sereny test for identification of invasive Shigella and Escherichia coli strains. J Clin Microbiol 1986;24: 498-500. [ Links ]
31. Sasakawa C, Kamata K, Sakai T, Murayama SY, Makino S, Yoshikawa M. Molecular alteration of the 140 megadalton plasmid associated with loss of virulence and congo red binding activity in Shigella flexneri. Infect Immun 1986;51:470-475. [ Links ]
32. Frankel G, Riley L, Girón J, Valmassoi J, Friedmann A, Strocknine N et al. Detection of Shigella in feces using DNA amplification 1990;161:1252-1256. [ Links ]
33. Knutton S, Lloyd DR, Mc Neish AS. Adhesion of enteropathogenic Escherichia coli to human intestinal enterocytes and cultured human intestinal mucosa. Infect Immun 1987;55:69-77. [ Links ]
34. Girón JA, Ho ASY, Schoolnik GK. An inducible bundle-forming pilus of enropathogenic Eschericia coli. Science 1991;254:710-713. [ Links ]
35. Donnenberg MS, Girón JA, Kaper JB. A plasmid-encoded type IV fimbrial gene of enteropathogenic Eschericia coli associated with localized adherence. Mol Microbiol 1992;6:3427-3437. [ Links ]
36. Cravioto A, Gross RJ, Scotland SM, Rowe B. An adhesive factor found in strains of Escherichia coli belonging to the traditional infantile enteropathogenic serotypes. Curr Microbiol 1979;3:95-99. [ Links ]
37. Rosa AC, Mariano AT, Pereira AM, Tibana A, Gómez TA, Andrade JR. Enteropathogenicity markers in Escherichia coli isolated from infants with acute diarrhoea and healthy controls in Rio de Janeiro, Brazil. J Med Microbiol 1998;47:781-790 [ Links ]
38. Vial PA, Robins R, Lior H, Prado V, Kaper JB, Nataro JP et al. Characterization of enteroadherente-aggregative Escherichia coli a putative agent of diarrheal disease. J Infect Dis 1988;158:70-79. [ Links ]
39. Nataro JP, Deng Y, Maneval DR, German AL, Martin WC, Leviene MM. Aggregative adherence fimbriae I of enteroaggregative Escherichia coli mediate adherence to Hep-2 cells and hemagglutination of human eritrocytes. Infeact Immun 1992;60:2297-2304. [ Links ]
40. Czeczulin JR, Balepur S, Hicks S, Philips A, Hall R, Kothary MH et al. Aggregative adherence fimbria II, a second fimbrial antigen mediating aggregative adherence in enteroaggregative Escherichia coli. Infect Immun 1997;65:4135-4145 [ Links ]
41. Savarino SJ, Fasano A, Robertson DC, Levine MM. Enteroaggregative Escherichia coli elaborate a heat-stable enterotoxin demonstarable in an in vitro intestinal model. J Clin Invest 1991;87:1450-1455. [ Links ]
42. Savarino SJ, McVeigh A, Watson J, Molina J, Cravioto A, Echeverria P et al. Enteroaggregative Escherichia coli heat-stable enterotoxinis not restricted to enteroaggregative E. coli. J Infect Dis 1996;173:1019-1022. [ Links ]
43. Eslava C, Navarro-García F, Czeczulin JR, Henderson IR, Cravioto A, Nataro JP. Pet an autotransporter enterotoxin from enteroaggregative Escherichia coli. Infect Immun 1998;66:3155-3163. [ Links ]
44. Villaseca JM, Navarro-García F, Mendoza-Hernández G, Nataro JP, Cravioto A, Eslava C. Pet toxin form enteroaggregative Escherichia coli produces cellular damage associated with fodrin disruption. Infect and Immun 2000;68:5920-5927. [ Links ]
45. Henderson Ir, Czeczulin J, Eslava C, Noriega F, Nataro JP. Characterization of pic, a secreted protease of Shigella flexneri and enteroaggregative Escherichia coli. Infect Immun 1999;67:5587-5596 [ Links ]
46. Cobeljic M, Milijkovic-Selimovic B, Paunovic-Todosijevic D, Velickovic Z, Lepsanovic Z, Savic D et al. Enteroaggregative Escherichia coli associated with an outbreak of diarrhoea in a neonatal nursery ward. Epidemiol Infect 1996;117:11-16. [ Links ]
47. Cravioto A, Tello A, Navarro A, Ruiz J, Villafan H, Uribe F et al. Association of Escherichia coli Hep-2 adherence patterns with type and duration of diarrhoea. Lancet 1991;337:262-264. [ Links ]
48. Baudry B, Savarino SJ, Vial P, Kaper JB, Levine MM. A sensitive and specific DNA probe to identify enteroaggregative Escherichia coli, a recently discovered diarrheal pathogen. J Infect Dis 1990;161:1249-1251. [ Links ]
49. Schmidt H, Knop C, Franke S, Aleksic S, Heeseman J, Karch H. Development of PCR for screening of enteroaggregative Escherichia coli. J Clin Microbiol 1995;33:701-705. [ Links ]
50. Bilge SS, Clausen CR, Lau W, Moseley SL. Molecular characterization of a fimbrial adhesin, F1845, mediating diffuse adherence of diarrhea-associated Escherichia coli to Hep-2 cells. J Bacteriol 1989;171:4281-4289. [ Links ]
51. Benz I, MA Schmidt. 1992. Isolation and serologic characterization of AIDA-I, the adhesin mediating the diffuse adherence phenotype of the diarrhea associated Escherichia coli strain 2787 (O126:H27). Infect Immun 1996;60:13-18. [ Links ]
52. Poitrineau P, Forestier C, Meyer M, Jallat C, Rich C, Malpuech G et al. Retrospective case-control study of diffusely adhering Escherichia coli and clinical features in children with diarrhea. J Clin Microbiol 1995; 33:1961-1962. [ Links ]
53. Ojeda A, Prado V, Martínez J, Arellano C, Borczyk A, Johnson W et al. Sorbitol-negative phenotype among enterohemorrhagic Escherichia coli strains of different serotypes and from different sources. J Clin Microbiol 1995;33:2199-2201. [ Links ]
54. Ammon A, Petersen LR, Kar ch H. A large outbreak of hemolytic uremic syndrome caused by an unusual sorbitol-fermenting strain of Escherichia coli O157:H7. J Infect Dis 1999;179:1274-1277. [ Links ]
55. Rodríguez-Angeles G. Exotoxinas. En: Diagnóstico de laboratorio de infecciones gastrointestinales. Giono S, Escobar A y Valdespino JL, ed. México, Secretaría de Salud, 1994:369. [ Links ]
56. Giono S, Rodríguez-Angeles G, Rodríguez MJ, Valdespino JL. Identificación de enterotoxinas y citotoxinas de E. coli por cultivo de células Vero e hibridación en fase sólida (Colony blot). Rev Latinoam Microbiol 1994;36: 231-241. [ Links ]
57. Tenover FC. Diagnostic deoxyribonucleic acid probes for infectious diseases. Clin Microbiol Revs 1988;1:82-101. [ Links ]
58. Begum D, Strockbine NA, Sowerr EG, Jackson MP. Evaluation of a technique for identification of Shiga-like toxin-producing Escherichia coli by using polymerase chain reaction and digoxigenin-labeled probes. J Clin Microbiol 1993;31:3153-3156. [ Links ]
59. Rodríguez-Angeles G, Giono S, Valdespino JL. Efecto citotónico y citotóxico de la toxina colérica en células Vero y su relación con PCR. Rev Latinoam Microbiol 1994;36:263-271. [ Links ]
60. Olive DM. Detection of enterotoxigenic Escherichia coli after polymerase chain reaction amplification with a thermostable DNA polymerase. J Clin Microbiol 1989;24:261-265. [ Links ]
61. Rademaker CMA, Wolfhagen MJHM, Jansze M, Oteman M, Fluit AC, Glerum JH et al. Digoxigenin labelled DNA probes for rapid detection of enterotoxigenic and Vero cytotoxin producing Escherichia coli in faecal samples. J Microbiol Meth 1992;15:121-127. [ Links ]
62. Rodríguez G, Rodríguez MJ, Giono S, Gómez M, Valdespino JL. Identificación de cepas de Escherichia coli productoras de toxina termolábil (LT) en células Vero y colony blot. Memorias del V Congreso Nacional de Investigación en Salud Pública. II Congreso regional de la Asociación Internacional de Epidemiología 1994:106. [ Links ]
63. Rodríguez G. Estudio de cepas de Escherichia coli aisladas en México. Memorias IV Jornadas científico culturales de Químicos Clínicos. Instituto Mexicano del Seguro Social; 1998 noviembre 25; México, D.F., México. [ Links ]
64. Rodríguez G, Rincón MG, Cortés-Tenorio LJ, Moreno A. Escherichia coli causante de diarrea en México, identificada por hibridación en fase sólida colony blot. Enfermedades Infecciosas y Microbiologia, 2001. 21 supl: S116. [ Links ]
65. Arbeit RD, Arthur M, DunnR, Kim C, Selander RK, Goldstein R. Resolution of recent evolutionary divergence among Escherichia coli from related lineages: The application of pulsed field electrophoresis to molecular epidemiology. J Infect Dis 161:230-235. [ Links ]
(1) Instituto de Diagnóstico y Referencia Epidemiológicos. México, D.F., México
Fecha de recibido: 5 de marzo de 2002 • Fecha de aprobado: 17 de abril de 2002
Solicitud de sobretiros: Maestra Martha Guadalupe Rodríguez-Angeles. Laboratorio de Bacteriología Molecular, departamento de Biología Molecular, Instituto de Diagnóstico y Referencia Epidemiológicos. Prolongación Carpio 470, Colonia Santo Tomás, Delegación Miguel Hidalgo, 11340 México D.F., México.
Correo electrónico: magra@mail.telecom.ipn.mx , magraa_1999@yahoo.es














