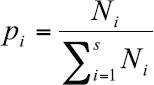Introducción
Los consorcios microbianos (CM) fotosintéticos constituidos por cianobacterias (Cyanophyta) tienen una distribución muy amplia en la naturaleza, tanto en ambientes acuáticos como terrestres. Los CM resultan clave en los ciclos biogeoquímicos, lo que les confiere importancia ecológica dada la gran cantidad de estrategias metabólicas que pueden desarrollar (Grewe & Pulz, 2012). Desde el punto de vista tecnológico, los CM se han utilizado para la remoción de metales pesados y de contaminantes orgánicos (Muñoz et al., 2006) presentes en aguas residuales, para la obtención de biodiésel (Hena et al., 2015) y de energía eléctrica (Gajda et al., 2015), y para la conversión de carbono y generación de biogás (Vasseur et al., 2012), han demostrado mayor eficiencia de síntesis de productos y conversión de nutrientes a menor costo, comparado con la utilización de cultivos puros (Muñoz et al., 2006; Vasseur et al., 2012; Hena et al., 2015). Otras ventajas del empleo de consorcios microbianos es su producción a gran escala, dado que los cultivos puros no son con frecuencia, económicamente factibles debido a los altos costos de mantenimiento, sustratos, esterilización, y recuperación de biomasa, entre otros factores (Höffner & Barton, 2014). Muchos microorganismos en ambientes naturales no viven aislados, sino que forman parte de consorcios que tienen una dinámica poblacional interna. La competencia por los recursos y la cooperación entre los microorganismos que integran un CM pueden determinar el éxito de su desempeño biotecnológico (Subashchandrabose et al., 2011).
Debido a la importancia de los consorcios microbianos resulta de interés identificar los miembros que los conforman, así como su aportación para el funcionamiento global de éstos (Gupta et al., 2013). Para la identificación de los organismos que componen un CM, se han utilizado métodos polifásicos, que combinan la caracterización de genotipos y su expresión fenotípica, porque, en ocasiones, la identificación basada sólo en métodos de taxonomía tradicional, resulta complicada debido a la alta variabilidad fenotípica entre morfotipos similares, observados en diferentes condiciones ambientales (Loza et al., 2013). La complementariedad de ambos procedimientos es de suma importancia, ya que permite realizar un análisis exhaustivo de los microorganismos que integran un CM.
En las últimas dos décadas se han empleado métodos polifásicos para identificar la diversidad de consorcios microbianos, aprovechando las ventajas que ofrecen tanto este método como el de taxonomía tradicional (Thacker & Paul, 2004; Loza et al., 2013; Muñoz-Martin et al., 2014; Krohn-Molt et al., 2013).
En estudios previos de nuestro grupo de trabajo, se aisló un CM fotosintético a partir de muestras tomadas de suelo, tallo y agua de anegamiento de un campo arrocero de Alpuyeca, Morelos, México. Posteriormente, el CM se propagó en diferentes volúmenes empleando un medio mineral sin fuente de nitrógeno, hasta establecer el cultivo en un fotobiorreactor tipo columna de burbuja de 11 L; se evaluó su producción de biomasa y capacidad de fijación de nitrógeno durante 8 resiembras en cultivo por lote, cada una de ellas a los 14 días, utilizando una intensidad de luz de 80 μmol fotones m-2 s-1 y una aireación de 11 L min-1 (1 vvm). La cantidad promedio de biomasa en base seca fue de 248 ± 54 mg L-1, y la actividad de nitrogenasa, de 5,188.5 ± 33.7 nmol etileno g-1 peso seco h-1 (Hernández-Melchor, 2009). Con base en los resultados mencionados, el objetivo del presente estudio fue llevar a cabo la identificación morfológica y filogenética de los microorganismos que integran dicho CM, con el propósito de establecer, de manera teórica, la probable interacción entre sus componentes y de explorar su posible aplicación biotecnológica.
Materiales y métodos
Recolección de la muestra. El material biológico utilizado provino de una cinética de cultivo por lote en un fotobiorreactor (FBR) tipo columna de burbuja con un volumen de operación de 11 L, diámetro de 0.23 m y altura de 0.34 m, empleando el medio BG110 (Rippka et al., 1979). El experimento duró 14 días bajo condiciones controladas de luz (80 μmol fotones m-2 s -1), con un fotoperiodo 12/12 (luz/oscuridad), temperatura de 21°C y aireación de 11 L min-1. Al final del cultivo, se tomó una muestra homogénea de 50 mL para llevar a cabo los análisis morfológico y filogenético del consorcio.
Peso seco de biomasa. Los días 0, 2, 5, 7, 9, 12 y 14 se tomaron alícuotas de 10 mL de cultivo y se filtraron por medio de membranas de celulosa de 5 μm de diámetro de poro (marca Millipore®), las cuales posteriormente, se secaron en horno a 70 ºC por 24 h (Tredici et al., 1991).
Actividad de nitrogenasa (ensayo de reducción de acetileno). Se determinó por el método propuesto por Venkataraman (1981), modificando el tiempo de incubación, de 48 a 120 h. Se colocaron 10 mL de cultivo en botellas serológicas de 25 mL, que después fueron selladas herméticamente con una engargoladora; posteriormente, se les reemplazó 1 mL de aire del espacio vacío por 1 mL de acetileno (1 % helium balance). Las botellas se incubaron por 5 días a 21 ± 2 ºC, con una iluminación de 80 μmol fotones m−2 s−1 y fotoperiodo de 12/12 h. Se determinó la cantidad de etileno producido en un cromatógrafo de gases marca Varian CP-3380 FID, bajo las siguientes condiciones: T columna = 50ºC, T inyector = 200ºC, T detector = 250 ºC, Columna: Carbowax 1540/Porapak Q 1/8”x 6 ft, acarreador: gas N, 15 psi. Cada determinación se hizo por triplicado los días 0, 2, 5, 7, 9, 12 y 14 del cultivo. Los resultados fueron expresados en nmoles etileno g-1 peso seco h-1.
Determinación de nitratos y amonio. Al día 14, se tomó una alícuota homogénea de 50 mL del cultivo en el FBR, se centrifugó a 3500 rpm por 20 min y se utilizó el sobrenadante para las determinaciones de nitratos por el método de la brucina-ácido sulfámico (Eaton et al., 1992) y de amonio por el método del fenato No.132 C (Eaton et al., 1992).
Diversidad morfológica. El estudio de los organismos presentes en el CM se hizo mediante observaciones al microscopio óptico, de las muestras tomadas al final del cultivo por lote.
Para la identificación morfológica de cianobacterias y organismos eucariotes que componen el CM, se utilizaron muestras frescas y para las diatomeas preparaciones fijas con Naphrax ® (Rushforth et al., 1984). Todas las muestras se examinaron y fotografiaron con un microscopio óptico BX51 (Olympus Corporation, Tokyo, Japan) y una cámara digital marca Nikon DXM1200. La identificación de las especies de diatomeas se hizo de acuerdo con (Krammer y Lange, 1986, 1988, 1991a, 1991b) y las clorofíceas, de acuerdo a Wehr y Sheath (2003).
Extracción de ADN. Para el análisis filogenético de la muestra colectada se centrifugaron tres alícuotas en un tubo Eppendorff de 5 mL a 3000 rpm para formar un pellet de aproximadamente 0.12 g de peso húmedo; posteriormente la muestra se trató con acetona para eliminar los pigmentos del cultivo que pudieran causar interferencias durante la extracción de ADN. Para la extracción del ADN metagenómico, se utilizó un kit comercial (QuickGene DNA tissue kit S, modelo FujiFilm QuickGene-810 / QuickGene-Mini80). La eficiencia de la extracción se cuantificó empleando alícuotas de 2 µL en un espectrofotómetro modelo NanoDrop Lite, marca Thermo Scientific.
Amplificación por PCR, construcción de una librería 16S rDNA y secuenciación. Para identificar los componentes bacterianos del CM, se amplificó un fragmento de ADN de la región codificante para el rRNA de la subunidad pequeña del ribosoma (16S rDNA). El 16S rDNA del ADN metagenómico fue amplificado por PCR, utilizando los primers específicos bacterianos 46F (5’-GCC TAA CAC ATG CAA GTC-3’) (Yu & Morrison, 2004) y 1540R (5’-AAG GAG GTG ATC CAG CCGCA-3’) (Edwards et al., 1989).
La mezcla de reacción (25 µL) contenía 100 ng de ADN, 0.5 µM de cada primer, 200 µM de dNTP, 2.5 mM MgCl2, y 1 U de Taq ADN polimerasa en el buffer de PCR proporcionado por el fabricante (Invitrogen Life Technologies, San Paulo, Brasil). Las condiciones de amplificación incluyeron un paso de desnaturalización a 93 ºC por 10 min, seguido por 25 ciclos a 93 ºC por 1 min, a 57 ºC por 1 min, a 72 ºC por 2 min, y un paso final a 72 ºC por 10 min. La reacción de amplificación de ADN se hizo en un termociclador programable (Touchgene Gradient, modelo FTGRAD2D, Techne Duxfort, Cambridge, Reino Unido). Los amplicones esperados fueron de aproximadamente 1,500 pb. Los amplicones PCR fueron clonados directamente en el vector pCR 2.1-TOPO, utilizando el TOPO TA Cloning Kit (Invitrogen Life Technologies, Carlsbad, CA, EEUU). Las colonias blancas se seleccionaron en placas con LB que contenían 40 µg mL-1 de X-Gal (Invitrogen Life Technologies, Carlsbad, CA, EEUU) y 30 µg mL-1 de kanamicina (Invitrogen Life Technologies, Carlsbad, CA, EEUU). Los plásmidos recombinantes se aislaron a partir de cultivos de una noche por lisis alcalina. Las secuencias del 16S rDNA se obtuvieron con un analizador de ADN 3730X (Applied Biosystems, Foster City, CA, EEUU) utilizando los primers M13.
Identificación molecular y análisis filogenético. Se realizó una asignación taxonómica de las secuencias analizadas, utilizando la distancia evolutiva con las secuencias de microorganismos reportadas en bases de datos. Todas las secuencias fueron analizadas para quimeras utilizando Chimera7 Check, además del programa de análisis online de la base de datos RDP-II. Las secuencias se sometieron a una búsqueda BLAST (Altschul et al., 1997) para determinar la identidad de las clonas. Se seleccionaron secuencias relacionadas del NCBI Taxonomy Homepage (TaxBrowser) y un análisis de alineamiento múltiple utilizando el programa Clustal X. La ponderación de la transversión/transición se realizó utilizando el modelo Tamura-Nei (Tamura & Nei, 1993), y se estimó el número de sustitución de bases entre cada par de secuencias utilizando el programa Mega V. 4. Los datos anteriores sirvieron para construir el árbol filogenético utilizando el método de Neighbor-joining, y para el análisis de distancia se utilizó el modelo Tamura-Nei; se ensayaron 1,000 bootstrap para respaldar las ramas internas (Valenzuela et al., 2009). Se tomaron en consideración las secuencias que diferían <2% para pertenecer al mismo filotipo (Rosselló & Amann, 2001).
Para identificar los organismos procariotes que conforman el CM, se utilizó el gen 16S rDNA como marcador molecular. La relación de las secuencias del gen 16S rDNA de 44 clonas obtenidas del CM con secuencias obtenidas del GenBank se hizo por medio de un análisis filogenético, para asignar por similitud el género y la especie que representa cada una de ellas. Se construyeron dos árboles a partir de 44 clonas (ca. 980 pb) utilizando el método de Neighbor-joining y Pyrococcus abyssi (AY099167) como grupo externo.
Número de acceso de las secuencias de nucleótidos. Las secuencias parciales 16S rDNA empleadas en este estudio, se localizan en el GeneBank con el número de acceso KC979066-KC979109.
Abundancia relativa. La abundancia relativa de cada componente microbiano en el CM se determinó a partir de su identificación morfológica y filogenética y se calculó de acuerdo con la siguiente ecuación (Franco et al., 2001):
Donde:
Pi |
= abundancia relativa de las especies i |
S |
= número total de especies en la comunidad |
Ni |
= número de individuos de la misma especie i |
En el análisis morfológico se tomaron en cuenta todas las fotografías tomadas a las microalgas verdes y cianobacterias presentes en el CM, además del número de repeticiones (N i ) de cada uno de los cuatro géneros registrados. Se siguió el mismo procedimiento para las diatomeas, cuyo total de fotografías fue S=81, y el número de repeticiones (N i ) de cada uno de los siete géneros encontrados. El análisis filogenético consideró el total de clonas analizadas (S=44) y el número de repeticiones (N i ) de los 13 géneros identificados.
Resultados
Biomasa y actividad de nitrogenasa. En la Figura 1 se presenta la cantidad máxima de biomasa en base seca al final del experimento (día 14), misma que fue de 0.305 g L-1, con una productividad de 0.015437 g L-1 d-1. También es posible observar que el CM presentó una fase de adaptación de 7 días, con una disminución de la cantidad de biomasa cercana al 5%. El crecimiento exponencial de la biomasa se presentó del día 8 al 14. En la misma figura se aprecia que la actividad de nitrogenasa tuvo un comportamiento oscilante: al día 7 disminuyó, y al final del experimento incrementó hasta 10,294 nmoles etileno g-1 peso seco h-1.
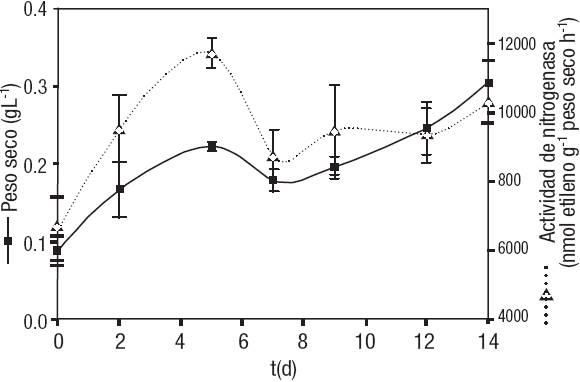
Figura 1: Evolución temporal de la biomasa (g L-1) y la actividad de nitrogenasa de un consorcio microbiano.
La concentración final de amonio y nitratos fue de 2.09 mg L-1 y 0.45 mg L-1, respectivamente.
Diversidad morfológica y abundancia relativa. El análisis microscópico de los morfotipos del CM registró un total de 11 taxones (Tabla 1); dos géneros fueron de cianobacterias filamentosas: Anabaena oscillarioides Bory de Saint-Vincent ex Bornet y Flahault (Figs. 2A-B) y Leptolyngbya sp. (Fig. 2C), además de nueve taxones unicelulares integrados por las clorofitas Monoraphidium sp. (Figs. 2D-E), Chlorella sp. (Fig. 2F), y las diatomeas Cyclotella meneghiniana Kützing (Fig. 2I), Melosira varians C. Agardh (Fig. 2H), Cocconeis placentula Ehrenberg (Fig. 2J), Achnanthidium chlidanos (Hohn et Hellerman) Novelo,Tavera et Ibarra (Fig.2M), Navicula radiosa Kützing ( Fig. 2K), Fragilaria ulna (Nitzsch) Lange-Bertalot (Fig. 2L) y Nitzschia sp. (Fig. 2G). En la Figura 3 se muestra la abundancia relativa de los organismos que comprenden el CM; se analizaron los datos microscópicos, donde se observan cinco especies mayoritarias: la cianobacteria filamentosa A. oscillarioides, la microalga verde Monoraphidium sp. y las diatomeas Cocconeis placentula, Fragilaria ulna y Nitzchia sp. (la más abundante).
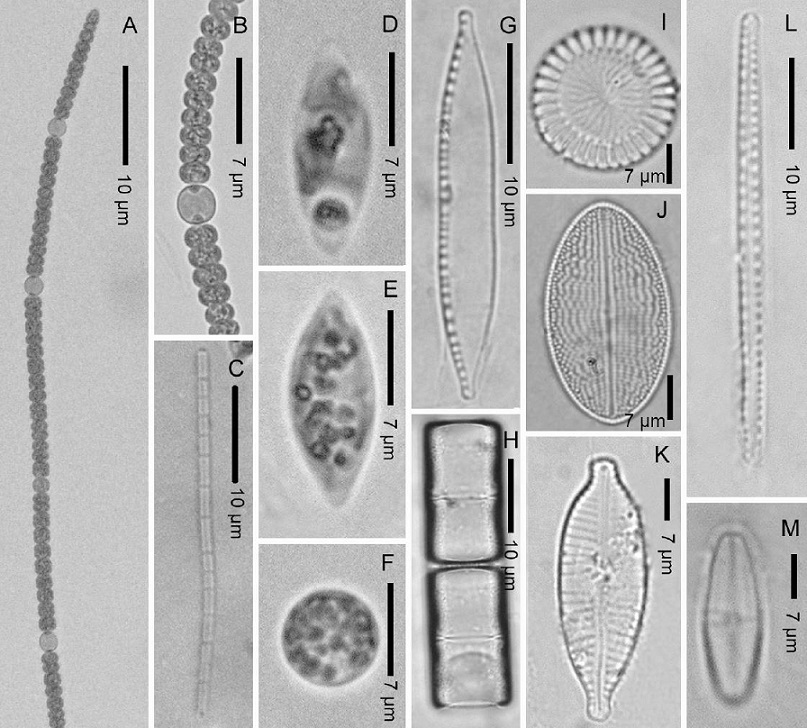
Figuras 2A-M: Microfotografías de los morfotipos de cianobacterias, microalgas verdes y diatomeas en el consorcio microbiano estudiado. A) Anabaena oscillarioides Bory de Saint-Vincent ex Bornet y Flahault, filamento y heterocistos intercalados. B) Anabaena sp., heterocisto y células vegetativas con abundante granulación. C) Leptolyngbya sp. D) Monoraphidium sp., célula vegetativa. E) Monoraphidium sp., vacuolas o lípidos celulares. F) Chlorella sp. G) Nitzchia sp. H) Melosira varians C. Agardh. I) Cyclotella meneghiniana Kützing. J) Cocconeis placentula Ehrenberg. K) Navicula radiosa Kützing. L) Fragilaria ulna (Nitzsch) Lange-Bertalot. M) Achnanthidium chlidanos (Hohn et Hellerman) Novelo, Tavera et Ibarra.
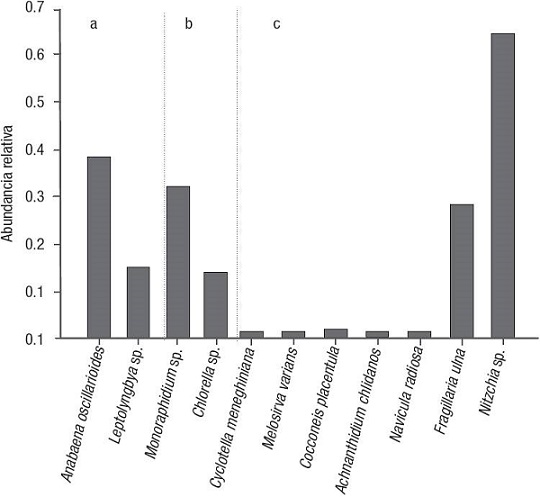
Figura 3: Abundancia relativa de cada taxón encontrado en el consorcio microbiano a través del análisis morfológico. a = cianobacterias, b = microalgas verdes, c = diatomeas.
Análisis filogenético y abundancia relativa. Se construyeron dos árboles filogenéticos a partir de 44 clonas (ca. 980 bp) del gen 16S rDNA y secuencias obtenidas del GenBank. Los resultados de los árboles filogenéticos mostraron la presencia de cinco clados principales con una similitud del 95.48 al 99.69 %. El clado V presentó la mayor abundancia relativa dentro del CM (Fig. 4).
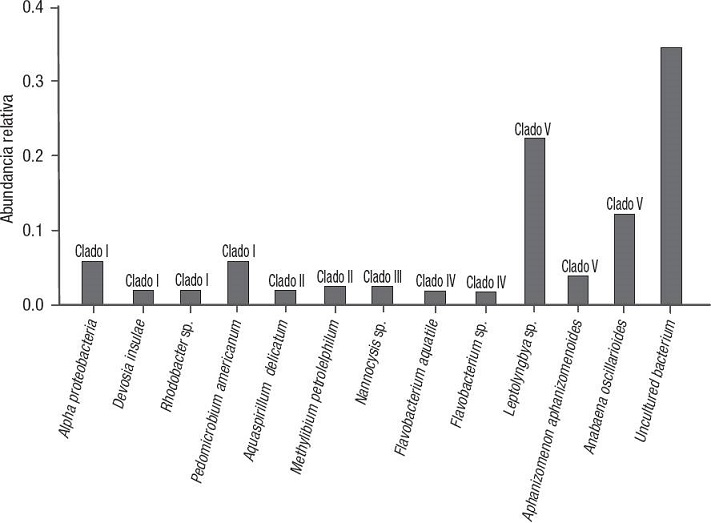
Figura 4: Abundancia relativa del consorcio microbiano a través del análisis filogenético de las secuencias amplificadas con el 16S rDNA.
El clado I incluyó 13 secuencias, de las cuales a siete de ellas se asignaron cuatro taxones de la clase α-proteobacteria: Rhodobacter sp., Devosia insulae Yoon, Kang, Park et Oh, Pedomicrobium americanum Gebers et Belse y una Alpha-proteobacteria no determinada. Las seis secuencias restantes se presentaron como organismos no cultivables (Figs. 5-6).
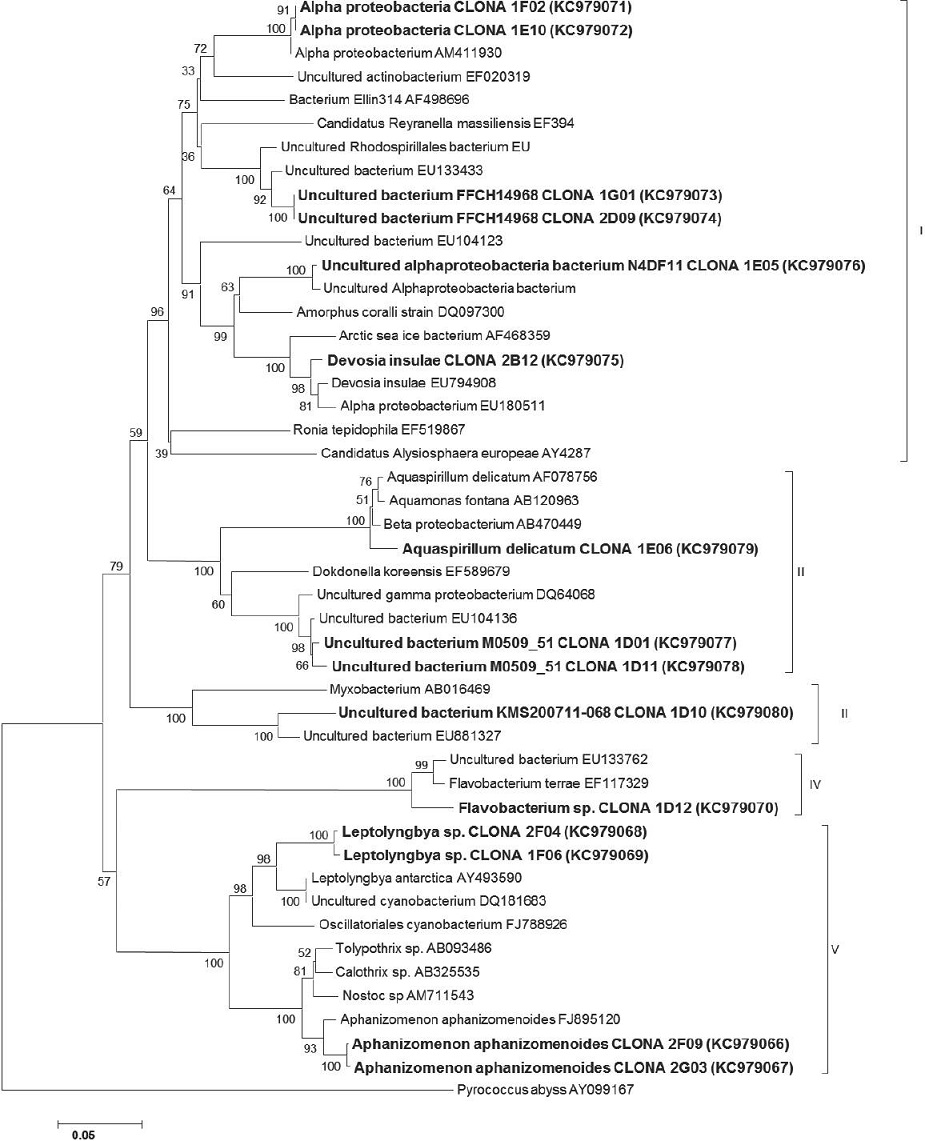
Figura 5: Relación filogenética entre las secuencias directas 16S rDNA obtenidas del consorcio microbiano. El árbol fue construido con secuencias relacionadas obtenidas de la base de datos del NCBI utilizando el algoritmo Neighbor-joining. Pyrococcus abyssi (AY099167) fue utilizado como grupo externo. Los números antes de las ramas representan el porcentaje de repeticiones “bootstrap” basadas en 1000 árboles.
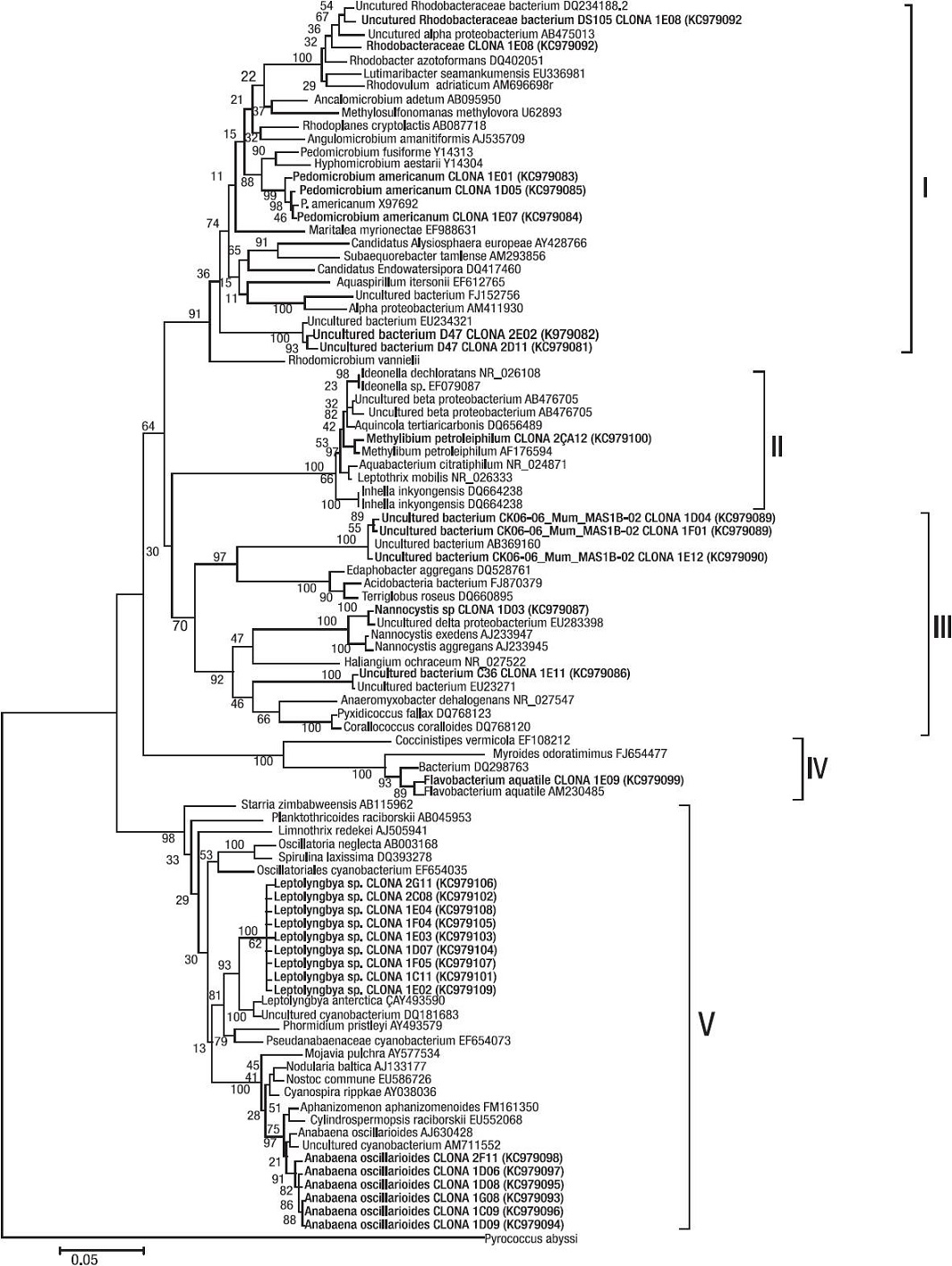
Figura 6: Relación filogenética entre las secuencias reversas 16S rDNA obteni das del consorcio microbiano. El árbol fue construido con secuencias relacio nadas obtenidas de la base de datos del NCBI utilizando el algoritmo Neighbor-joining. Pyrococcus abyssi (AY099167) fue utilizado como grupo externo. Los números antes de las ramas representan el porcentaje de repeticiones “bootstrap” basadas en 1000 árboles.
El clado II agrupó cinco secuencias relacionadas con dos géneros de la clase β-proteobacteria, con una similitud del 97.75 al 99.49%: Curvibacter delicatus (Leifson) Ding et Yokota ( = Aquaspirillum delicatum (Leifson) Hylemon et al.) y Methylibium petroleiphilum Nahatsu, Hristova, Haneda, Meng, Hanson, Scow et Kamagata. Las tres secuencias restantes se presentaron como organismos no cultivables (Figs. 5-6).
El clado III relacionó cinco secuencias de la clase δ-proteobacteria, con una similitud de 96.72%, una de ellas, afín al género Nannocystis sp.; las cuatro restantes se asociaron a organismos no cultivables (Fig. 6).
El clado IV reunió dos secuencias de la familia Flavobacteriaceae, con una similitud de 95.39 y 98.66% del género Flavobacterium F. sp. y F. aquatile (Frankland et Frankland) Begey et al. 1923 (Figs. 5-6).
En el clado V se encontraron 19 secuencias pertenecientes a los órdenes Oscillatoriales y Nostocales, con una similitud del 95.09 al 97.74%. Los géneros asociados a estas secuencias son: Aphanizomenon aphanizomenoides (Forti) Hortobágyi et Komárek, Leptolyngbya sp. y Anabaena oscillarioides Bory ex Bornet et Flahault (Figs. 5-6).
Discusión
El CM en cultivo por lote en el FBR de 11 L mostró una capacidad de fijación de nitrógeno cinco veces mayor a la reportado por Kannaiyan et al. (1997) para las especies Anabaena variabilis Sao (2678 nmoles etileno g-1 peso seco h-1) y Nostoc muscorum DOH (2 237 nmoles etileno g-1 peso seco h-1) presentes en su cultivo. La importancia de estas cianobacterias (Anabaena y Nostoc) en cultivos agrícolas radica en su capacidad para establecer simbiosis con plantas, y para fijar nitrógeno en el suelo, por lo que su biomasa ha sido ampliamente utilizada como biofertilizante en países tropicales (Kannaiyan, 2002). De acuerdo con lo anterior, podemos atribuir parte de la capacidad biológica de fijación de nitrógeno del CM a la presencia de tres cianobacterias filamentosas fijadoras de nitrógeno dentro del cultivo: Anabaena oscillarioides, Aphanizomenon aphanizomenoides y Leptolyngbya sp., cianobacterias que fueron identificadas mediante los análisis morfológico y filogenético.
Los análisis morfológico y filogenético mostraron elevada abundancia relativa de las cianobacterias Anabaena oscillarioides, Aphanizomenon aphanizomenoides y Leptolyngbya sp., y de las bacterias Rhodobacter sp. y Devosia insulae (Figs. 3-4, respectivamente), las cuales han sido reportadas por Rivas et al. (2002) y Li et al. (2010) como fijadoras de nitrógeno, por lo que es posible atribuir la elevada eficiencia de fijación de nitrógeno del CM a la presencia de estas especies.
A partir del análisis morfológico fue posible identificar a los géneros Monoraphidium sp. y Chlorella sp. como parte del CM. De acuerdo con reportes en literatura, estas especies tienen la capacidad de proveer a las plantas de diversos elementos constitutivos, como el fósforo; cuando se adicionan al suelo como biofertilizantes (Megharaj et al.,1992) y cuando se utilizan como inoculantes, aumentan la fertilidad de suelos áridos (Trejo et al., 2012). Por lo anterior, y considerando la alta abundancia relativa de las cianobacterias fijadoras de nitrógeno que constituyen al CM, es de esperarse el desempeño exitoso de éste como biofertilizante. Además, los géneros de diatomeas identificados pueden contribuir a los ciclos biogeoquímicos del carbono, nitrógeno, silicio y hierro; por lo tanto, tienen un efecto positivo en el ambiente y en otros componentes del CM (Amin et al., 2012). Adicionalmente, las diatomeas tienen la capacidad para producir partículas transparentes de exopolisacárido, que ayudan a formar una matriz de diatomeas agregadas y bacterias heterotróficas que mejoran la eficiencia de los consorcios microbianos (Gärdes et al., 2011).
La diversidad tan amplia de microorganismos que conforman el CM facilita la interacción de diferentes especies de organismos eucariotes y procariotes, relacionados con ese microcosmos particular.
Bauer et al. (2008) realizaron estudios similares al presente trabajo y destacaron la importancia de la identificación e interacción de los organismos que integran los consorcios microbianos. Uno de sus hallazgos más importantes, fue la presencia de cianobacterias y bacterias heterotróficas, las cuales establecen relaciones simbióticas entre sí. De manera similar, las diatomeas se asocian frecuentemente con bacterias epífitas en lagos de agua dulce (Paerl, 1990). La importancia de la amplia diversidad de microorganismos que integran un consorcio microbiano en un sistema natural o artificial, como es el caso de los fotobiorreactores, radica en que existe un intercambio de metabolitos y factores de crecimiento conforme transcurre el tiempo (Pearl & Pinckney, 1996). Estas asociaciones (cianobacterias-bacterias-diatomeas) pueden tener diferentes aplicaciones ambientales y biotecnológicas (Wartiainen et al., 2008).
El análisis filogenético permitió identificar 12 organismos procariotes (Figs. 5-6), los cuales se encuentran representados en cinco clados. El grupo más abundante de clonas analizadas se encuentra en el clado V y representa a las cianobacterias fijadoras de nitrógeno. El segundo grupo predominante fue el de las proteobacterias de las clases α, β y δ, representada en los clados I a III. De los géneros agrupados en el clado I podemos mencionar a Pedomicrobium americanum; a esta especie, estudiada por Tebo et al. (2010)), se le relaciona con la capacidad de oxidar y acumular manganeso en un polisacárido extracelular.
Aquaspirillum delicatum y Methylibium petroleiphilum, agrupadas en el clado II, tienen la capacidad de regular metabólicamente la fijación y el uso de amonio en el medio de cultivo (Pot et al., 2006; Cheng et al., 2011). Esto resulta importante porque la nitrogenasa puede inhibirse por concentraciones de amonio superiores a 18 mg L-1 y de nitrato superiores a 1,240 mg L-1 (Almon & Boger, 1988; Li et al., 2010).
En el clado III se encuentra el género Nannocystis sp., representado por varias especies que tienen la capacidad de formar un polisacárido extracelular al crecer en concentraciones de NaCl superiores a 7% (Zhang et al., 2002).
Pedomicrobium americanum y Nannocystis sp. son especies importantes para la agregación de los integrantes del CM, ya que contribuyen a la formación de una matriz extracelular de polisacáridos, que lo protege contra organismos autóctonos del suelo donde se llegase a adicionar como biofertilizante.
El clado IV contiene secuencias identificadas como Flavobacterium sp. y Flavobacterium aquatile, las cuales se han utilizado como inoculantes en plantas de trigo y en ambientes acuáticos. Las especies de la clase Flavobacteria tienen un papel importante en la mineralización de diversos tipos de materia orgánica, como carbohidratos, aminoácidos, proteínas y polisacáridos (Bernardet & Bowman, 2006), y como miembros del CM pueden participar en la degradación de metabolitos, facilitando su asimilación a los demás integrantes del CM.
Con base en los resultados obtenidos en el presente estudio, aproximadamente una tercera parte de las clonas analizadas tuvo similitud con organismos no cultivables, característica que define al CM como unidad funcional. Haber identificado aproximadamente 30% de microorganismos no cultivables en el CM pudo deberse al empleo de métodos polifásicos, que favorecen la identificación de la diversidad microbiana; sin embargo, aún existen limitaciones para identificar filogenéticamente a un gran número de ellos debido a que las bacterias cultivables identificadas, y cuyas secuencias han sido depositadas en bases de datos, representan aproximadamente el 0.1% de los microorganismos presentes en el ambiente (Loza et al., 2013).
Con base en la información obtenida en el análisis morfológico, aunada a la del análisis filogenético, fue posible identificar cianobacterias filamentosas fijadoras de nitrógeno y a proteobacterias como principales organismos que conforman el CM; esto permite asegurar la existencia de una interacción entre ellas y la de un intercambio de metabolitos entre todos los organismos que conforman el CM, lo que les permite sobrevivir en diferentes ambientes. En consorcios aislados en campos de arroz localizados en diferentes regiones del mundo (China, Japón e India), se ha reportado un tipo de interacción cianobacteria-bacteria. Dicha interacción se estudió por medio de un método polifásico para identificar consorcios presentes en suelo, granos de arroz, rizósfera, paja de arroz, etcétera. (Prasanna et al., 2009; Reyna-Velarde et al., 2012).
Por lo anterior, se puede concluir que la identificación de cianobacterias, bacterias, microalgas verdes y diatomeas que conforman el CM resultó esencial para comprender su crecimiento en el fotobiorreactor, así como para vislumbrar su posible aplicación biotecnológica como biofertilizante. La concentración de nitratos y amonio detectada en este estudio, es muy inferior a los niveles que causan inhibición de la actividad de nitrogenasa (Almon y Boger, 1988; Li et al., 2010), por lo que la capacidad de fijación de nitrógeno del CM no se vio afectada.











 nueva página del texto (beta)
nueva página del texto (beta)