INTRODUCCIÓN
México se considera el país con mayor diversidad de especies del género Pinus, al contar con 42 % de las especies que existen en el mundo (Sánchez-González, 2008). Una de las especies ampliamente distribuidas es Pinus cembroides Zucc., calificado como el pino piñonero (Pinus cembroides) de mayor importancia económica en el país (Luna-Cavazos et al., 2008); además, está incluido en la lista de especies prioritarias con fines de conservación, reforestación y restauración (CONABIO, 2011). Caracterizar la diversidad genética es útil en la definición de estrategias de mejora genética, conservación y manejo de los recursos genéticos (Furnier, 2004; Loo, 2011).
Actualmente se utilizan los análisis genéticos con marcadores moleculares para estimar el polimorfismo presente en el ADN, debido a su abundancia y a que no son afectados por el ambiente (Valadez-Moctezuma et al., 2001). Los ISSR (Inter-secuencias simples repetidas) son marcadores dominantes, altamente polimórficos, de bajo costo, no requieren información sobre la secuencia de la especie en cuestión y son reproducibles (Rocha et al., 2014).
Las coníferas tienen varios mecanismos que promueven el apareamiento cruzado, lo que genera un flujo genético eficiente entre poblaciones de una especie (Delgado et al., 1999). En cuanto a la estructura genética, las especies de pino que se distribuyen como poblaciones aisladas dispersas tienen una mayor diversidad genética entre las poblaciones, mientras que los pinos que tienen usualmente una distribución continua presentan menos diversidad entre las poblaciones, a pesar de tener amplia distribución, lo que es atribuible a un flujo genético importante (Hamrick et al., 1992, Ledig et al., 2001). Según Ledig y Conkle (1983), se espera que P. cembroides tenga altos niveles de diversidad genética, dadas las características biológicas de la especie (leñosa, de amplia distribución y alógama). Por otro lado, se ha observado que los pinos piñoneros muestran elevada diferenciación genética entre poblaciones (Fst≥ 0.15), asociado probablemente con bajas densidades, dispersión de semillas por aves y resistencia a sequía (Delgado et al., 2002).
Se tiene información de diversidad genética en ADN nuclear en poblaciones del norte del país de P. cembroides mediante AFLP (Wehenkel, 2015, Com. Pers.)1, quien encontró hasta 98 % de polimorfismo. Aunque se ha estudiado la diversidad genética medida a nivel de cloroplasto en poblaciones del norte del país, no hay reportes publicados (Cuenca, 2002, Com. Pers.)2; por lo que el presente trabajo pretende complementar la información genética de la especie, al evaluar la diversidad genética presente en ADN nuclear en poblaciones del centro del país.
La información sobre la magnitud y distribución de la diversidad genética entre poblaciones de las especies es fundamental para proponer estrategias de conservación y programas de manejo sustentable a largo plazo (Frankham et al., 2002). En estos programas se deben incluir poblaciones con componentes genéticos representativos y únicos para disminuir problemas relacionados con los efectos de endogamia, exogamia, flujo génico reducido y mutaciones deletéreas, que influyen en la habilidad de las especies para su evolución (Frankham et al., 2002). Estos efectos pueden conducir a la extinción de especies que crecen en poblaciones pequeñas y aisladas y en las cuales se esperaría una diversidad genética baja por la acción de la deriva genética y la endogamia.
Considerando lo anterior, el objetivo del presente trabajo fue evaluar la diversidad y estructura genética de poblaciones de P. cembroides de la región central de México mediante marcadores ISSR y determinar la relación de las distancias genéticas con las distancias geográficas. Las hipótesis panteadas fueron que se esperaba encontrar una alta diversidad genética en P. cembroides, distribuida principalmente dentro de las poblaciones debido a la amplia distribución de la especie, polinización cruzada, ciclo de vida largo y la dispersión por animales; además, que las poblaciones geográficamente cercanas presentarán mayor relación genética que las lejanas, debido a un mayor flujo de genes entre las mismas.
MATERIALES Y MÉTODOS
Recolecta de material vegetal
Se estudiaron ocho poblaciones de Pinus cembroides de la región central de México (Cuadro 1). Se recolectaron hojas jóvenes de 20 árboles seleccionados al azar en cada población, dejando una distancia de por lo menos 30 m entre ellos para evitar recolectar material de árboles emparentados. Se tomaron muestras de 10 g de hoja de cada uno de los individuos, éstas se depositaron en bolsas plásticas con cierre hermético y se mantuvieron sobre una capa de hielo hasta depositarlas en un ultracongelador (Thermo Scientific ULT2586, Waltham, Massachusetts, U.S.A.) a -80 ºC durante una semana.
Cuadro 1 Ubicación geográfica, temperatura y precipitación media anual de las localidades de procedencia de las poblaciones de Pinus cembroides estudiadas en la región central de México.
| Localidad | Municipio, Estado | Provincia | Latitud | Longitud | Altitud (msnm) | Temp. media anual (ºC) | Precip. Media anual (mm) |
|---|---|---|---|---|---|---|---|
| Fisiográfica† | |||||||
| La Amapola | San Luis Potosí, SLP | Mesa del Centro | 22º 08’ 23.3” | 101º 08’39.0” | 2369 | 17.9 | 341.0 |
| Bledos | Villa de Reyes, SLP | Mesa del Centro | 21º 48’ 34.7” | 101º 08’ 16.7” | 1960 | 19.5 | 360.0 |
| Ojo de Agua | San Felipe, Gto | Mesa del Centro | 21º 39’ 05.2” | 101º 11’ 55.0” | 1840 | 16.7 | 473.4 |
| El Cubo | San Felipe, Gto | Mesa del Centro | 21º 29’ 56.5” | 101º 05’ 20.5” | 2073 | 16.7 | 473.4 |
| Los Juárez | Cadereyta, Qro | Sierra Madre Oriental | 20º 51’ 58.3” | 99º 38’ 39.5” | 2699 | 17.3 | 506.0 |
| El Pinalito | Cardonal, Hgo | Sierra Madre Oriental | 20º 39’ 19.1” | 99º 06’ 0.3” | 2178 | 16.0 | 430.0 |
| La Florida | Cardonal, Hgo | Sierra Madre Oriental | 20º 31’ 26.4” | 98º 58’ 57.4” | 1933 | 16.0 | 430.0 |
| Santiago de Anaya | Santiago de Anaya, Hgo | Sierra Madre Oriental | 20º 26’ 14.7” | 98º 58’ 6.4” | 2038 | 16.0 | 550.0 |
†La Provincia Fisiográfica corresponde a la clasificación del INEGI (2008).
Extracción y calidad del ADN
Para la extracción de ADN se utilizó el método CTAB 2X con 1 % de polivinilpirrolidona (PVP). La calidad del ADN se confirmó mediante electroforesis en gel de agarosa al 0.8 % (p/v), que se corrió en amortiguador Tris-Acetato-EDTA (TAE 0.5 X) (Tris-Base, ácido acético y EDTA 0.5 M, pH 8.0). El gel fue teñido con bromuro de etidio al 0.1 % (p/v) y posteriormente se visualizó en un transiluminador de luz ultravioleta y un fotodocumentador KODAK EDAS-290. Se definió al ADN de calidad al obtener una banda uniforme y de alto peso molecular. En cada una de las muestras se cuantificó la cantidad de ADN con un espectrofotómetro marca GENESYS 10uv. El ADN se diluyó a una concentración de 10 ng µL-1 para ser utilizado en las reacciones de PCR.
Amplificación de fragmentos
Los componentes de reacción fueron los siguientes: 4 µL de ADN (10 ng µL-1), 0.3 µL de la enzima Taq ADN polimerasa, 10 µL de dNTPs (500 µM), 2.5 µL de amortiguador (10X), 2 µL de MgCl2 (50 mM), 3 µL de iniciador y 3.2 µL de H2O bidestilada. Las reacciones de PCR se realizaron en un termociclador Techne TC-512 (Bibby Scientific, Vernon Hills, Illinois, U.S.A.). Las condiciones de las reacciones fueron: a) un ciclo de desnaturalización inicial a 94 ºC durante 3 min; b) 30 ciclos con temperaturas de 94 ºC durante 1 min, 50 ºC por 1 min, 72 ºC por 2 min; y c) un ciclo de extensión final a 72 ºC durante 10 min. Se probaron 14 iniciadores ISSR, de los cuales se eligieron los nueve que generaron la mayor cantidad de bandas (Cuadro 2).
Cuadro 2 Iniciadores utilizados para el análisis de poblaciones de Pinus cembroides de la región central de México.
| Nombre | Secuencia (5’ - 3’) | Número de bandas | % de bandas polimórficas | Temperatura de alineamiento (ºC) |
|---|---|---|---|---|
| 7939 | AGGAGGAGGAGGAGGAGG | 15 | 80.0 | 50 |
| 7941 | GAGAGAGAGAGAGAGAC | 21 | 71.4 | 50 |
| 7944 | AGAGAGAGAGAGAGAGG | 14 | 50.0 | 50 |
| 7949 | GAGAGAGAGAGAGAGA | 18 | 77.7 | 50 |
| 7955 | AGAGAGAGAGAGAGAGA | 18 | 80.0 | 50 |
| A8 | AGAGAGAGAGAGAGAGT | 15 | 86.6 | 50 |
| P1 | GAGCAACAACAACAACAA | 19 | 78.9 | 50 |
| P3 | AGAGAGAGAGAGAGAGTG | 17 | 54.5 | 50 |
| P5 | AGAGAGAGAGAGAGAG | 17 | 82.3 | 50 |
| Promedio | 17.1 | 73.4 | 50 |
Electroforesis y obtención de datos
Los productos amplificados de PCR se separaron mediante electroforesis en geles de agarosa a una concentración de 1.2 %. Los geles se corrieron en amortiguador TAE 0.5X, a 120 V. El teñido y la documentación del gel se hicieron de la misma manera en que se verificó la calidad del ADN.
Diversidad genética
A partir de los patrones de bandeo en los geles se generó una matriz binaria con 1 y 0, donde el 1 indicó la presencia de una banda de ADN en una muestra y el 0 la ausencia de dicha banda. Para el análisis intra e interpoblacional se usó el programa Popgene 1.31 (Yeh et al., 1999) con la inclusión de todos los individuos de cada población. Los parámetros estimados fueron el porcentaje de loci polimórficos (frecuencia del alelo más común ≤ 0.95) de acuerdo con Hedrick (2011) y el índice de diversidad génica de Nei o heterocigosidad promedio esperada He (Nei, 1987).
Diferenciación genética
La distancia genética (DN) se estimó con base en lo señalado por Nei (1975). DN toma valores de cero cuando las poblaciones tienen frecuencias alélicas idénticas y tiende a uno en poblaciones que no comparten ningún alelo (Hedrick, 2005).
El coeficiente de diferenciación genética entre poblaciones (GST) se calculó con base en Nei (1975). Este estimador es altamente dependiente de la cantidad de variación que hay dentro de las poblaciones y del total de la población.
Tasa de migración y estructura genética
El número de individuos migrantes por generación (Nm) se obtuvo de acuerdo con McDermott y McDonald (1993) y para describir la estructura genética se realizó un análisis de varianza molecular (AMOVA) mediante el programa GenAlEx versión 6.5 (Peakall y Smouse, 2012).
Análisis de agrupamiento
A partir de la matriz binaria se obtuvo una matriz de distancias entre los individuos muestreados, misma que sirvió para realizar un análisis de coordenadas principales. El método que se utilizó para dicho análisis fue el de covarianza-estandarizada, con 1000 permutaciones, mediante el programa GenAlEx versión 6.5 (Peakall y Smouse, 2012).
RESULTADOS Y DISCUSIÓN
Diversidad Genética
Los nueve iniciadores que se utilizaron generaron un total de 154 bandas, dando como resultado un promedio de 17 bandas por iniciador. El número de bandas que se generó por iniciador fue variable, desde 14 con el iniciador 7944, hasta 21 con el iniciador 7941. El polimorfismo detectado por iniciador varió de 50 a 86.6 %, donde el iniciador A8 fue el que mostró un mayor porcentaje de loci polimórficos (Cuadro 2).
A nivel de especie se encontró alto polimorfismo (88.3 %), tal como se esperaba dadas las características de la especie y los iniciadores utilizados. En otras especies como Pinus tabuliformis f. shekanensis Carr. (Liu et al., 2015) y Pinus roxburghii Roxb. Ex. Lamb (Parasharami y Thengane, 2012) se encontraron de igual forma, altas tasas de polimorfismo (74.1 y 100 %, respectivamente) con el mismo tipo de marcadores. El polimorfismo en P. cembroides encontrado en el presente trabajo es mayor que el reportado para otros pinos piñoneros, como 65 % en Pinus culminicola Andresen et Beaman (Favela, 2010) con marcadores RAPD (también dominantes y polimórficos). Probablemente las diferencias estén relacionadas con el hecho de que en dicho trabajo se evaluaron sólo dos poblaciones.
A nivel poblacional, con los parámetros estimados se identificó un nivel de diversidad genética de moderado a alto en las ocho poblaciones que se evaluaron (Cuadro 3). Estos parámetros fueron más altos en las poblaciones de la Sierra Madre Oriental; por ejemplo, la amplitud de polimorfismo de las poblaciones que se distribuyen en la Sierra Madre Oriental fue de 61.0 a 63.9 %, mientras que dicho intervalo para las poblaciones de la Mesa Central fue de 45.5 a 61.7 %. La diversidad génica varió de 0.22 a 0.24 para las poblaciones de la Sierra Madre Oriental, y de 0.18 a 0.22 en las de la Mesa Central. Con el porcentaje de polimorfismo y la diversidad génica se encontró un patrón con la existencia de valores ligeramente más altos en las poblaciones de la Sierra Madre Oriental que en las de la Mesa del Centro (Cuadro 3).
Cuadro 3 Parámetros de diversidad genética observados en las poblaciones de Pinus cembroides en la región central de México.
| Localidad | % de polimorfismo | Diversidad génica (He) |
|---|---|---|
| El Pinalito | 63.9 | 0.24 |
| Los Juárez | 63.2 | 0.22 |
| Santiago de Anaya | 61.7 | 0.23 |
| La Florida | 61.0 | 0.23 |
| Promedio región Sierra Madre Oriental | 62.4 | 0.23 |
| La Amapola | 61.7 | 0.22 |
| Ojo de Agua | 58.8 | 0.21 |
| Bledos | 58.0 | 0.21 |
| El Cubo | 45.5 | 0.18 |
| Promedio Región Mesa Central | 56.0 | 0.20 |
| Promedio General | 59.28 | 0.22 |
La diversidad génica (He) promedio que se detectó mediante ISSR fue alta (0.22) de acuerdo con el criterio de Lowe et al. (2004). Al ser P. cembroides una conífera, se esperan altos niveles de diversidad genética, según lo señalado por Ledig y Conkle (1983) y Delgado et al. (1999), quienes sostienen que estos niveles de diversidad genética se deben entre otras cosas, a que poseen varios mecanismos que promueven el apareamiento cruzado, lo que podría compensar los efectos de la fragmentación, característica de los piñonares en México (Ledig, 1998).
En comparación con lo encontrado para P. cembroides (He = 0.59) por Cuenca (2002, Com. Pers.)2 con microsatélites de cloroplasto, la diversidad encontrada en el presente estudio con marcadores ISSR fue menor. Los microsatélites en general revelan más alto nivel de polimorfismo que los ISSR y además son codominantes (Höglund, 2009). Con AFLP, marcador dominante, Wehenkel (2015, Com. Pers.)1 evaluó las características genéticas de P. cembroides en unidades productoras de germoplasma en Chihuahua, donde encontró hasta 98 % de polimorfismo, lo que se puede atribuir a que los AFLP son capaces de detectar una mayor cantidad de variantes que los ISSR.
Análisis de agrupamiento de los individuos
Las poblaciones no se agruparon por región, sino que se concentraron en tres grupos de acuerdo con sus relaciones genéticas (Figura 1). Uno de los grupos lo conformaron las poblaciones de La Florida y El Pinalito. Los individuos se traslaparon, por lo que se puede inferir que estas poblaciones están muy relacionadas genéticamente, e incluso, por la cercanía geográfica, es probable que tengan un mismo origen. El grupo más numeroso estuvo conformado por poblaciones de las dos provincias fisiográficas (Bledos, Los Juárez, Santiago, Ojo de Agua y El Cubo), en el cual se observa sobreposición de los individuos, lo que evidencia también la estrecha relación genética entre estas poblaciones.
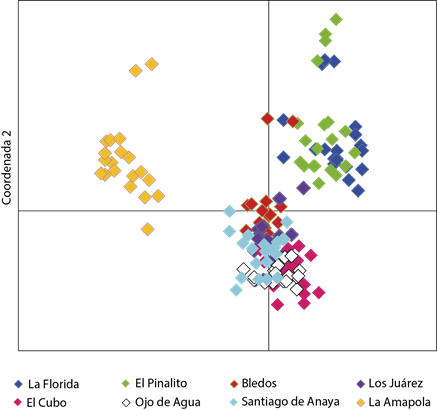
Figura 1 Gráfico construido con las dos primeras coordenadas derivadas del análisis de coordenadas principales con ISSR de ocho poblaciones de Pinus cembroides de la región central de México.
La población de La Amapola fue la más alejada genéticamente del resto. Un patrón semejante fue identificado por Cuenca (2002, Com. Pers.)2 para Pinus johannis Rob., quien reportó que la población de La Amapola S.L.P. fue la más distante genéticamente con respecto a otras dos poblaciones evaluadas, aun cuando una de las poblaciones fue muy cercana geográficamente, por lo que se puede inferir que existe en esta localidad alguna barrera que restringe el intercambio genético entre las poblaciones, o que la población se originó por un evento demográfico extremo que tuvo un efecto genético de cuello de botella.
Estructura y diferenciación genética
Los resultados del análisis de varianza molecular (AMOVA) (Cuadro 4) indicaron que la mayor parte de la variación genética se encuentra dentro de las poblaciones (74 %) y sólo 26 % entre las mismas. Esto concuerda con lo planteado por Hamrick et al. (1992), quienes sugieren que las especies leñosas con amplia distribución, sistemas de apareamiento cruzado y dispersión por animales tienen la mayor parte de la diversidad genética dentro de las poblaciones, pero menor variación entre las poblaciones. Este patrón es consistente también con la mayoría en los resultados reportados en otros trabajos de coníferas (Delgado et al., 2002; Flores-Rentería et al., 2013), y específicamente para el género Pinus como P. pinceana Gordon (Aguirre-Limón et al., 2017), P. culminicola (Favela, 2010), P. maximartinezii Rzedowski (Ledig et al., 2001), P. johannis (García-Gómez et al., 2014), P. greggii Engelm ex Parl (Paraguirre et al., 2002) y P. ayacahuite Ehremb ex. Schlecht (Castro-Félix et al., 2006). Lo anterior es válido para caracteres neutrales o casi neutrales a la selección, dado que autores como Rehfeldt (1985, 1986) indican importante diferenciación genética entre poblaciones de coníferas a lo largo de gradientes ambientales para caracteres cuantitativos con valor adaptativo como resistencia a heladas, potencial de crecimiento, dinámica y temporalidad de la elongación de la yema principal y otros.
Cuadro 4 Componentes del análisis de varianza molecular (AMOVA) para las poblaciones de Pinus cembroides de la región central de México.
| Fuente de variación | gl | SC | CM | CV | % |
|---|---|---|---|---|---|
| Entre poblaciones | 7 | 923.657 | 131.951 | 5.839 | 26 % ** |
| Dentro de poblaciones | 150 | 2497.128 | 16.648 | 16.648 | 74 % ** |
| Total | 157 | 3420.785 | 22.487 | 100 % |
gl: grados de libertad, SC: suma de cuadrados, CM: cuadrados Medios, CV: componentes de varianza, **: P ≤ 0.001. %: porcentaje de contribución a la variación.
En cuanto a la diferenciación genética existente entre las poblaciones de P. cembroides según las regiones, los valores de GST que se encontraron para cada región indican que las poblaciones están diferenciadas (0.25 para la Sierra Madre Oriental y 0.22 para la Mesa Central) (White et al., 2007). Al respecto, Ledig et al. (2000) afirman que los niveles elevados de diferenciación genética de las coníferas en México y América Central se deben a que estas poblaciones se encuentran fragmentadas, lo que puede ocasionar una reducción de los niveles de flujo genético y por ende haber favorecido, ya sea, la deriva genética en las poblaciones aisladas de menor tamaño o la diferenciación por la presión de selección por factores ambientales. Lo anterior es apoyado también por Delgado et al. (2002), quienes atribuyen la alta diferenciación entre las poblaciones de los pinos piñoneros de América del Norte a que éstas presentan bajas densidades y están relativamente aisladas.
Aunado a lo anterior, las poblaciones que se evaluaron se ubican en la parte discontinua de la distribución de P. cembroides, lo que implica que las poblaciones están separadas espacialmente y que el intercambio genético entre estas poblaciones se dificulta.
El número estimado de individuos migrantes por generación (Nm) entre las poblaciones de la Sierra Madre Oriental fue de 1.52 y de 1.75 en las poblaciones de la Mesa Central, lo que indica escaso flujo génico entre ellas (White et al., 2007). La distribución geográfica de P. cembroides no presenta un patrón continuo en todo el rango de su distribución (Romero et al., 1996). Las poblaciones que se incluyeron en este trabajo se localizan en el área discontinua de la distribución de la especie, lo que aunado a la posible fragmentación de las poblaciones podría explicar el reducido flujo génico entre ellas. El valor de Nm fue bajo en comparación con los valores estimados en otras especies de Pinus, como en P. johannis Rob. (Nm = 4) (García-Gómez et al., 2014) u otras consideradas por Ledig (1998), quien encontró valores altos de Nm en un rango de 4.6 a 17.2. P. johannis pertenece al mismo subgrupo (Haploxylon) que P. cembroides (Styles, 1998).
Se considera que si el Nm es alto (al menos igual a 4), el flujo génico supera los efectos de la deriva genética y evita la diferenciación local. Si Nm es pequeño (< 1), entonces se considera que la deriva actúa independientemente en cada una de las poblaciones (Eguiarte et al., 2010, Com. Pers.)3. De acuerdo con el valor de Nm que se encontró en este trabajo, el flujo génico supera ligeramente los efectos de la deriva genética en las poblaciones. Cuenca (2002, Com. Pers.)2 también encontró flujo génico restringido entre algunas de las poblaciones de P. cembroides que examinó; además, encontró también falta de equilibrio entre migración y deriva genética.
Distancia genética
Los valores de distancia genética de Nei que se encontraron entre las poblaciones de las dos regiones variaron de 0.075 a 0.206 (Cuadro 5). Las poblaciones más alejadas genéticamente fueron las de La Florida y La Amapola, lo que coincidió con que son las poblaciones geográficamente más lejanas; sin embargo, este patrón no se presentó para todas las poblaciones; por ejemplo, las de Santiago de Anaya y Los Juárez fueron las poblaciones más cercanas genéticamente, pero no son las poblaciones geográficamente más cercanas.
Cuadro 5 Distancia genética de Nei (bajo la diagonal) y distancia geográfica (km, sobre la diagonal) para las ocho poblaciones estudiadas de Pinus cembroides de la región central de México.
| Población | La Florida | El Pinalito | Juárez | Santiago | Bledos | El Cubo | Ojo de Agua | La Amapola |
|---|---|---|---|---|---|---|---|---|
| La Florida | 0 | 21.8 | 91.3 | 104.1 | 242.8 | 246.6 | 264.8 | 275.8 |
| El Pinalito | 0.113 | 0 | 70.5 | 88.5 | 227.5 | 228.7 | 246.0 | 255.5 |
| Juárez | 0.148 | 0.158 | 0 | 92.5 | 167.3 | 161.9 | 177.4 | 185.0 |
| Santiago | 0.159 | 0.168 | 0.077 | 0 | 253.3 | 238.9 | 248.8 | 242.7 |
| Bledos | 0.157 | 0.168 | 0.091 | 0.109 | 0 | 43.2 | 64.9 | 107.4 |
| El Cubo | 0.192 | 0.155 | 0.100 | 0.101 | 0.110 | 0 | 24.7 | 64.6 |
| Ojo de Agua | 0.194 | 0.196 | 0.091 | 0.090 | 0.114 | 0.092 | 0 | 43.5 |
| La Amapola | 0.235 | 0.233 | 0.156 | 0.138 | 0.147 | 0.186 | 0.187 | 0 |
De acuerdo con los valores de distancia genética, las poblaciones de La Florida y El Pinalito fueron las que se diferenciaron en mayor medida del resto de las poblaciones. El dendrograma que se generó a partir de la distancia genética de Nei (Figura 2) separó a las poblaciones en tres grupos, el primero integrado por las poblaciones de La Florida y El Pinalito, el segundo por la población de La Amapola y el tercero formado por el resto de las poblaciones, lo que concuerda con el arreglo de los individuos en el análisis de agrupamiento representado en la Figura 1.
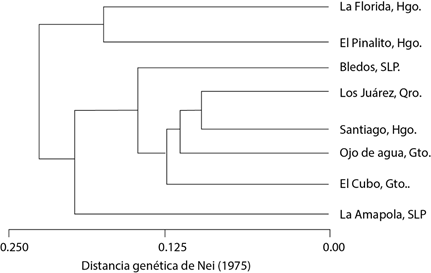
Figura 2 Dendrograma generado con base en la distancia genética de Nei (1975), mediante el método UPGMA para ocho poblaciones de Pinus cembroides de la región central de México.
Modelo de aislamiento por distancia
De acuerdo con la matriz de distancias geográficas y distancias genéticas (Cuadro 5), para algunas poblaciones con una menor distancia geográfica se tiene una menor distancia genética, como es el caso de las poblaciones de La Florida - El Pinalito, por citar un ejemplo, pero en otros casos no se tiene este comportamiento; por ejemplo, la población de Santiago de Anaya que es cercana a las de La Florida y El Pinalito muestra una mayor distancia genética con estas poblaciones que con las otras cuatro, que son más lejanas geográficamente.
La prueba de Mantel indicó que existe poca correlación entre distancia genética y distancia geográfica en las poblaciones de P. cembroides (r = 0.31) (Figura 3). Lo que sugiere que probablemente hay poblaciones cercanas con escaso flujo génico y otras más lejanas con niveles de flujo más altos. Al parecer, en las poblaciones de P. cembroides evaluadas el aislamiento por distancia no juega un papel importante en la diferenciación genética. En P. rzedowskii Madrigal et. Caballero (Delgado et al., 1999) y P. pinceana Gordon (Aguirre-Limón et al., 2017), ambos pinos piñoneros, no se encontró correlación entre estas variables. Por otro lado, para P. greggii Engelm. ex Parl, pino del otro subgénero de Pinus, Paraguirre et al. (2002) sí encontraron correlación significativa, aunque sólo para algunas de las poblaciones, lo que podría sugerir que son otros fenómenos los que determinan los patrones de variación genética entre las poblaciones evaluadas.
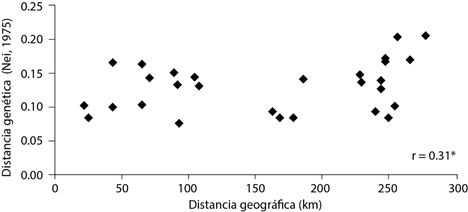
Figura 3 Relación entre las distancias geográficas y distancias genéticas para las poblaciones de Pinus cembroides. r: coeficiente de correlación de Pearson.
En cuanto a las aplicaciones que pueden tener los resultados de esta investigación en el al manejo o conservación de las poblaciones, se encontró que, por ejemplo, la población de La Amapola parece ser única en su constitución genética y merecería por tanto alguna protección especial.
Finalmente, los resultados del presente estudio indican que probablemente la fragmentación de las poblaciones genera una falta de continuidad en la distribución de la especie, lo que impide el flujo génico y, por tanto, facilita la diferenciación por deriva genética. Esto presenta un mayor reto para la conservación de la diversidad genética, ya que hace necesaria la conservación activa de un mayor número de poblaciones.
CONCLUSIONES
La diversidad genética encontrada en las poblaciones de P. cembroides fue alta, con los valores mayores en las poblaciones de en la Sierra Madre Oriental. La mayor parte de la diversidad genética se encontró dentro de las poblaciones y una menor parte entre poblaciones. Existe alta diferenciación genética entre las poblaciones y escaso flujo génico entre ellas. En las poblaciones evaluadas se determinó baja correlación entre la distancia geográfica y la distancia genética.











 nova página do texto(beta)
nova página do texto(beta)


