Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Salud Pública de México
versão impressa ISSN 0036-3634
Salud pública Méx vol.56 no.6 Cuernavaca Nov./Dez. 2014
Noticias
Noticias de salud ambiental ehp-spm
Environmental health news ehp-spm
Repensar qué Significa "Estéril"* El microbioma hospitalario
Cuando se inauguró el nuevo pabellón del hospital de la Universidad de Chicago, se veía inmaculado. Los pisos brillaban y las camillas de acero inoxidable resplandecían en el nuevo Centro de Atención y Descubrimiento. Incluso una vez que abrió sus puertas y se admitió a los primeros pacientes, las superficies seguían viéndose estériles en gran medida. Al parecer, las cosas estaban exactamente como deberían: tan libres de vida microbiana como fuera humanamente posible.
Los datos de Jack Gilberg contaban una historia diferente. Gilbert, microbiólogo ambiental del Laboratorio Nacional de Argonne, su equipo de estudiantes de posgrado y de posdoctorado y sus asistentes de investigación bajaban al hospital varias veces al día, aun antes de que éste se abriera al público. Armados con hisopos de algodón, enfocaban sus esfuerzos en los pisos de los quirófanos y de las salas de oncología. Cada uno de los miembros del equipo tomó muestras de los pisos, las camas, las sábanas, los lavabos, las computadoras, las estaciones del personal de enfermería, los respiraderos, etc. Si había algo que se pudiera nombrar, el equipo de Gilbert lo frotaba con un hisopo de algodón para obtener una pequeña muestra de los microbios que vivían allí.
Repitieron este proceso varias veces al día como parte del Proyecto del Microbioma Hospitalario, un esfuerzo con un costo de 850 000 dólares, financiado por la Fundación Alfred P. Sloan, para aprender más sobre la comunidad microbiana, o microbioma, de diversos ambientes hospitalarios: cómo se transfieren los microorganismos entre los seres humanos y las superficies y cómo se desarrollan los microbiomas con el paso del tiempo. Los investigadores consideran que pueden llegar a reducir las infecciones adquiridas en los hospitales comprendiendo a los diversos microorganismos que viven en los ambientes hospitalarios, identificando las características operativas de los edificios que influyen en estos microbiomas y realizando modificaciones a los ecosistemas interiores que ayuden a prevenir la propagación de los organismos patógenos. Una porción futura del estudio incluirá un análisis en profundidad del microbioma de una habitación del hospital del ejército en Alemania a lo largo de 16 meses.
"Cuando un organismo patógeno invade, no lo hace de manera aislada: lo hace en el contexto de miles de otras especies", dice Gilbert. "Muy pocos estudios han examinado el resto de las comunidades que existen en los hospitales".
Algunos investigadores están comenzando a ver los hospitales como ecosistemas en sí mismos. "Los científicos quieren estudiar el ecosistema del hospital para comprender qué microbios aparecen dónde", dice Jonathan Eisen, ecólogo microbiano de la Universidad de California en Davis.
Infecciones adquiridas en el hospital
En 2010 (el ultimo año sobre el cual se dispone de datos), 35.1 millones de estadounidenses pasaron por lo menos una noche en el hospital.1 Los Centros para el Control y la Prevención de Enfermedades estiman que 5% de los pacientes admitidos en los hospitales adquirirán una infección durante su estancia, lo que potencialmente conducirá a 99 000 muertes al año2 y tendrá un costo de 10 000 millones de dólares anuales.3
La adquisición de infecciones en los hospitales no es un fenómeno nuevo. Desde que las personas enfermas han buscado atención en los hospitales, ha existido el potencial de que se propaguen las enfermedades infecciosas. Con el advenimiento de la penicilina y de otros antibióticos, las preocupaciones sobre la transmisión de enfermedades disminuyeron, dado que los médicos creían contar con una bala mágica para combatir cualquier enfermedad que una persona pudiera contraer.4 El incremento de las bacterias resistentes a los antibióticos ha cambiado este modo de pensar.
Hoy en día, las infecciones resistentes a los antibióticos no muestran señales de parar, como tampoco lo hacen las enfermedades adquiridas en los hospitales.3 Históricamente, se ha culpado de estas infecciones a la presencia de bacterias nocivas y se ha visto como la solución la aplicación de normas y procedimientos de control de infecciones cada vez más rigurosos.5 Según una nueva hipótesis, las infecciones adquiridas en los hospitales no obedecen a la existencia de microbios nocivos sino a la ausencia de especies útiles.
Bajo las luces brillantes y sobre las camillas de acero inoxidable habita una extensa comunidad de microorganismos, la mayoría de los cuales son inocuos, y muchos de ellos pueden ser incluso benéficos.6 Algunos investigadores consideran que los microbiomas hospitalarios forman una parte clave del "sistema inmune" de los hospitales y en algunos casos pueden contribuir a proteger a los pacientes contra las enfermedades infecciosas.
"Durante los últimos 150 años, literalmente hemos tratado sólo de matar bacterias. Ahora hay una multitud de evidencias que sugieren que este enfoque de matar todo no está funcionando", dice Gilbert. "Ahora estamos tratando de entender si tal vez, sólo tal vez, cultivar bacterias no patógenas en las superficies de los hospitales, nos conduciría a un ambiente hospitalario más saludable".
Investigaciones sobre el microbioma humano han mostrado que el uso de antibióticos puede afectar al conjunto habitual de microbios que viven dentro y en la superficie de nuestro cuerpo.7 Los continuos intentos de esterilizar los hospitales pueden funcionar a un nivel similar: el uso de antibióticos de amplio espectro, blanqueadores y desinfectantes para manos puede eliminar algunos de los organismos patógenos nocivos, pero también provoca un fuerte impacto en las hordas de microorganismos no patógenos.
La eliminación de estos microbios comensales reduce la competencia, haciendo a los hospitales potencialmente más propicios para la proliferación de las especies patógenas, menciona Eisen. "Quizás algunos intentos de esterilización no sean útiles a la larga porque desalojen ecosistemas que luego quedarán expuestos a ser recolonizados por organismos patógenos y no sólo por bacterias normales y aburridas", dice.
"La gran mayoría de estos microbios apenas están sobreviviendo", dice el ecologista microbiano James Meadow, un académico con postdoctorado en el Centro de Biología y Medio Ambiente Construido de la Universidad de Oregón. "El medio ambiente construido parece más bien ser una sala de espera en la que estas bacterias potencialmente nocivas permanecen hasta que haya mejores condiciones. Muy pocas de ellas la están pasando bien". La eliminación de otros microbios puede sacar a los organismos patógenos destructivos de la sala de espera y ponerlos en acción.
Repensar qué significa "estéril"
Históricamente, los microbiólogos han estudiado las bacterias en cultivo puro, cultivando una especie a la vez. Si bien esto les ha permitido generar grandes cantidades de microbios de manera relativamente rápida, el método tiene varias limitaciones.
Muchos de los microbios que se encuentran en el ambiente son difíciles, si no imposibles, de cultivar de esta forma.8 Esto ha hecho que los microbiólogos subestimen drásticamente el número de los microbios que constituyen a los microbiomas humanos.9 Asimismo, debido a la importante amenaza a la salud pública que representan las enfermedades infecciosas, los investigadores prefirieron concentrar sus esfuerzos en las bacterias nocivas y no en las especies neutrales o benéficas.10
Hace apenas 50 años, cuando mucho de lo que sabían los científicos sobre los microbios estaba relacionado con organismos patógenos, tenía sentido eliminar de las superficies su delgada capa de microbios. Pero ese no es el caso hoy en día. A partir de finales de la década de 1990 y principios de los años 2000, la tecnología de secuenciación genética comenzó una serie de mejoras exponenciales,11 las cuales redujeron tanto el tiempo como el costo de secuenciar genes.12 Esto le permitió a Eisen, a Gilbert y a otros mejorar los métodos liderados por el ecologista microbiano Norman R. Pace en la década de 1980 para buscar en el medio ambiente microorganismos que no pueden cultivarse en el laboratorio; estos estudios mostraron que nuestro mundo está repleto de microbios.13 Inspecciones del medio ambiente revelaron que las bacterias cultivables, como las especies de Streptococcus y Staphylococcus, representan sólo una pequeña parte de los microbios que encontramos diariamente.14
Los diferentes tipos de bacterias contienen una versión única del gen 16S ARN ribosómico que actúa como huella digital. Al tomar una muestra de la superficie y después secuenciar los diferentes genes 16S ARN ribosómico presentes en esa muestra, los científicos pueden descubrir de manera rápida, barata y sencilla los tipos de bacterias presentes en cualquier lugar.15 Investigadores han descubierto así que la biodiversidad presente en una mesa, un barandal de cama o un pedazo de suelo puede competir con la que se encuentra en cualquier selva amazónica.
"Es muy difícil desalojar a todos los microbios de un ecosistema en particular", dice Eisen. En una reseña publicada para la revista Genome Biology, Gilbert y su coautor Scott Kelly escribieron que "probablemente exista un microbio capaz de sobrevivir en casi cualquier superficie o condición [del medio ambiente construido]".16 En pocas palabras, la esterilidad no existe.
Los microbios entre nosotros
Científicos han descubierto que lejos de ser una capa homogénea de vida unicelular, los microbios de los edificios varían ampliamente dependiendo de las condiciones ambientales y de las personas que habiten los cuartos. Algunos miembros de los microbiomas de interior son precisamente lo que los científicos habrían esperado. Estudios anteriores han mostrado que las bacterias que colonizan las piscinas de terapia17 y los grifos de las regaderas18 de los hospitales son micobacterias y proteobacterias que aman la humedad y residen en la tierra. Otro estudio descubrió que los grifos de las regaderas también estaban poblados por organismos patógenos oportunistas que son significativamente diferentes de los microbios encontrados en las habitaciones de los pacientes. Estas bacterias tienden a formar biopelículas, colonias persistentes de microbios que prefieren ambientes húmedos ricos en fósforo y pueden ser casi imposibles de matar.19
Las regaderas no son las únicas áreas hospitalarias idóneas para el desarrollo de un conjunto único de microbios. Cuando los investigadores de la Universidad de Colorado en Boulder analizaron los microbios presentes en los catéteres de Foley, una fuente común de infecciones adquiridas en hospitales,20 descubrieron que las bacterias presentes en el exterior del catéter eran considerablemente diferentes de las encontradas en el interior.21 En un estudio australiano, los catéteres arteriales de las unidades de cuidados intensivos mostraron un patrón similar.22
Sin embargo, la mayoría de los microbios presentes en el ambiente hospitalario llegan por medio de los humanos, ya sea que estén en las suelas de nuestros zapatos, en nuestros celulares o incluso en nuestros cuerpos. Así como el aura permanente de mugre que rodea al personaje Pigpen (o "Cochino") en la caricatura de "Carlitos y Snoopy", los seres humanos estamos rodeados de una nube de microbios.23 "Los humanos esparcimos microbios por donde sea que vamos", dice Gilbert.
Cada vez que tocamos un objeto, transferimos millones de microbios de nuestro cuerpo al medio ambiente.24 Ya que los tipos de microbios disponibles para ser transferidos varían de una persona a otra25 y de una parte del cuerpo a otra,26 es probable que diferentes superficies alberguen diferentes especies. Objetos tales como los teclados de computadora, los interruptores de luz y los dispensadores de jabón son continuamente re-infestados de microbios provenientes de nuestras manos cada vez que los tocamos.23,27,28 En los sanitarios, por otro lado, prevalecen los microorganismos asociados con los tractos gastrointestinales y urogenitales.
Un análisis detallado del microbioma en un salón de clases reveló que los tipos de especies encontrados variaban dependiendo del tipo de contacto humano que recibía cada superficie de la cual se tomaba una muestra.29 Los investigadores descubrieron que las sillas presentaban una preponderancia de microbios de los tractos gastrointestinales y urogenitales, así como de la piel. En los pisos y las paredes predominan las especies del exterior que probablemente se traen en los zapatos o se introducen a través del sistema de ventilación.30
Los resultados preliminares no publicados del Proyecto del Microbioma Hospitalario de Gilbert muestran que, dentro de las horas siguientes al ingreso de un nuevo paciente, los microbios en un cuarto cambiaron para reflejar la composición del último residente. "En cuestión de horas, el microbioma de la nueva persona se convirtió en la fuerza dominante en ese cuarto", dice.
También se ha demostrado el proceso opuesto, de acuerdo con investigaciones que analizaron los microbios encontrados en la unidad de cuidados intensivos neonatales (UCIN) del Hospital de Mujeres Magee del Centro Médico de la Universidad de Pittsburgh.31 La misma cantidad de microbios que se encuentran en el medio ambiente construido están asociados con los seres humanos; ahora los investigadores saben que los humanos pueden adquirir muchos de sus microbios a partir de su medio ambiente. Brandon Brooks, un ecologista microbiano de la Universidad de California en Berkeley y primer autor del estudio sobre la UCIN, dice que estudiar el microbioma de interior no es sólo cuestión de realizar un censo microbiano. La enterocolitis necrotizante, una infección vinculada con múltiples especies bacterianas, es excepcionalmente mortal en los bebés que presentan bajo peso al nacer.32 El saber cómo fue que los bebés en la UCIN adquirieron estos microbios podría ayudar a los médicos a prevenir brotes.
Si bien los recién nacidos no están completamente estériles, en ese momento tienen un microbioma que es mucho menos diverso que en cualquier otra etapa de sus vidas.33 Tenemos que empezar desde cero, lo cual hace que las señales que observamos sean diferentes", explica Brooks.
Brooks y sus colegas recopilaron muestras fecales de los recién nacidos en la UCIN cada tres días. Por cada muestra fecal, también recopilaban 33 muestras ambientales de los alrededores de la UCIN. Las principales especies de bacterias que encontraron en los intestinos de los bebés (Staphylococcus epidermidis, Klebsiella pneumoniae, Bacteroides fragilis y Escherichia coli) se encontraron por toda la UCIN, lo que implica que el ambiente hospitalario pudo haber sido la fuente de estos microbios.31 "El siguiente paso es probar más estrategias progresivas de regulación ambiental para ver si podemos cultivar un ecosistema que sea benéfico para sus ocupantes", dice Brooks.
¿Una bocanada de aire fresco?
Ordenar los factores que influyen en la composición de las comunidades microbianas de interiores podría ayudar a los científicos a identificar espacios y objetos con alto riesgo de portar organismos patógenos. Estos investigadores también creen que pequeñas modificaciones al diseño de un edificio, como alterar los sistemas de ventilación y humedad, podrían ayudar a reducir el número de organismos patógenos en el ambiente interior. Los arquitectos están viendo estas modificaciones en el contexto no sólo de los hospitales sino también de otras áreas públicas.
El sistema de calefacción, ventilación y aire acondicionado (en inglés, HAVC) en un edificio puede ser una gran fuente de microorganismos en los interiores, dado que muchos microbios viajan a través del aire.34,35 Susannah Tringe, una microbióloga del Instituto Conjunto del Genoma del Departamento de Energía y sus colegas tomaron muestras de bacterias en los climatizadores de dos centros comerciales urbanos densamente poblados. Identificaron hasta 300 especies distintas, muchas de las cuales estaban asociadas con el microbioma humano y eran considerablemente diferentes de las halladas en el aire exterior.36
"De hecho, hay mucho que se puede hacer para limitar la transmisión por aire, y ahí es donde los cambios al ambiente construido probablemente pueden tener un impacto más grande", dice Brent Stephens, un ingeniero arquitecto del Instituto de Tecnología de Illinois, quien no estuvo involucrado en el estudio Tringe.
El diseño arquitectónico de un espacio puede afectar a los microbios que se encuentran allí, de acuerdo con la investigación realizada por Meadow. Muchos de los microbios en espacios cerrados llegaron a través del sistema de HVAC,35 y la composición de la comunidad microbiana variaba dependiendo de si el sistema introducía o no aire del exterior. Los científicos también descubrieron que los cuartos que estaban físicamente cerca uno del otro eran propensos a tener composiciones microbianas similares, lo mismo que los cuartos con altos niveles de circulación humana.37
Un estudio del microbioma del Hospital Providence Milwaukie, en Oregón, mostró que las muestras de aire del interior contenían un porcentaje más grande de bacterias relacionadas con organismos patógenos potenciales que las muestras del exterior. En los cuartos que tenían los índices más altos de corrientes de aire y humedad se hallaron menos bacterias asociadas con los humanos y menos organismos potencialmente patógenos.38
Sin embargo, mejorar la salud ambiental de los hospitales no depende de simples sumas y restas científicas. Entender cómo se transmiten los organismos patógenos de un lugar a otro y de una persona a otra significa comprender los ecosistemas microbianos que viven en los hospitales. Tal vez entender esto parezca ser un cambio muy grande, dice Meadow, pero ha revolucionado la manera en que los científicos piensan sobre los hospitales y los microbios que viven ahí. "Si perturbamos una cosa al moverla o al esterilizarla, necesitamos entender qué más puede cambiar", dice.
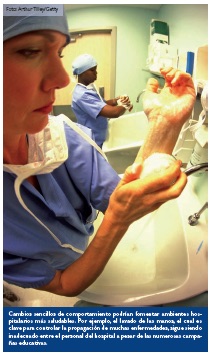
Los arquitectos e ingenieros ambientales por igual están empezando a pensar en los microbios al momento de diseñar los nuevos hospitales y modernizar los antiguos. Algunos de los cambios pueden ser simples, como no ubicar sanitarios al lado de áreas donde se preparan alimentos, para prevenir que el microbioma del baño emigre a la cocina. Meadow formula la hipótesis de que instalar ventanas que puedan abrirse hacia el exterior también podría ayudar a sembrar una comunidad microbiana diferente en el hospital.39 También es necesario que estos cambios ambientales se vean acompañados de cambios de comportamiento, dice Eisen, como mejorar el lavado de las manos entre el personal del hospital, el cual sigue siendo mediocre a pesar de las numerosas campañas.40
Cambiar el ambiente hospitalario para prevenir infecciones parece una idea innovadora nacida de la secuencia genética de alto rendimiento y otros avances de la biología moderna, pero Meadow observa que puede tratarse de una idea antigua cuyo momento ha llegado.41 "En el siglo XIX, Florence Nightingale sabía que los pacientes se sentían mejor con una ventana abierta", dice. "Desde hace tiempo hemos sabido que el sólo abrir una ventana puede cambiar drásticamente los microbios que están en el aire a nuestro alrededor y esto puede influir en nuestra salud a largo plazo".
Carrie Arnold
es una escritora científica independiente que radica en Virginia.
Sus trabajos han aparecido en revistas como Scientific American,
Discover, New Scientist y Smithsonian, entre otras.
Referencias
1. NCHS. National Hospital Discharge Survey, 2010. Atlanta, GA:National Center for Health Statistics, U.S. Centers for Disease Control and Prevention (2010). Disponible en: http://www.cdc.gov/nchs/data/nhds/1general/2010gen1_agesexalos.pdf [consultado el 29 de mayo de 2014] [ Links ].
2. Klevens RM, et al. Estimating health care-associated infections and deaths in U.S. hospitals, 2002. Public Health Rep 122(2):160–166 (2007); http://www.ncbi.nlm.nih.gov/pubmed/17357358. [ Links ]
3. Zimlichman E, et al. Health care–associated infections: a meta-analysis of costs and financial impact on the US health care system. JAMA Intern Med 173(22):2039–2046 (2013); doi: 10.1001/jamainternmed.2013.9763. [ Links ]
4. Kohn LT, et al., eds. To Err is Human: Building a Safer Health System. Washington, DC: Institute of Medicine [Instituto de medicina], National Academy Press (1999). Disponible en: http://goo.gl/cw95gd [consultado el 29 de mayo de 2014] [ Links ].
5. Ducel G, et al., eds. Prevention of Hospital-Acquired Infections: A Practical Guide, 2ª ed. Ginebra, Suiza: World Health Organization [Organización Mundial de la Salud] (2002). Disponible en: http://goo.gl/L3Nys2 [consultado el 29 de mayo de 2014] [ Links ].
6. Rintala H, et al. Diversity and seasonal dynamics of bacterial community in indoor environment. BMC Microbiol 8:56 (2008); doi: 10.1186/1471-2180-8-56. [ Links ]
7. Jakobsson HE, et al. Short-term antibiotic treatment has differing long-term impacts on the human throat and gut microbiome. PLoS ONE 5(3):e9836 (2010); doi: 10.1371/journal.pone.0009836. [ Links ]
8. Kowalchuk GA, et al. Finding the needles in the metagenome haystack. Microbial Ecol 53(3)475–485 (2007); doi: 10.1007/s00248-006-9201-2. [ Links ]
9. Li K, et al. Analyses of the microbial diversity across the human microbiome. PLoS ONE 7(6):e32118 (2012); doi: 10.1371/journal.pone.0032118. [ Links ]
10. Dethlefsen L, et al. An ecological and evolutionary perspective on human–microbe mutualism and disease. Nature 449(7164):811–818 (2007); doi: 10.1038/nature06245. [ Links ]
11. Knight R, et al. Unlocking the potential of metagenomics through replicated experimental design. Nat Biotechnol 30(6):513–520 (2012); doi: 10.1038/nbt.2235. [ Links ]
12. Caulfield T, et al. Reflections on the cost of "low-cost" whole genome sequencing: framing the health policy debate. PLoS Biol 11(11): e1001699 (2013); doi: 10.1371/journal.pbio.1001699. [ Links ]
13. Bunge J, et al. Estimating the number of species in microbial diversity studies. Annu Rev Stat Appl 1:427–445 (2014); doi: 10.1146/annurev-statistics-022513-115654. [ Links ]
14. Chitsaz H, et al. Efficient de novo assembly of single-cell bacterial genomes from short-read data sets. Nat Biotechnol 29(10):915–921 (2011); doi: 10.1038/nbt.1966. [ Links ]
15. Hugenholtz P, et al. Impact of culture-independent studies on the emerging phylogenetic view of bacterial diversity. J Bacteriol 180(18):4765–4774 (1998); http://jb.asm.org/content/180/18/4765.short. [ Links ]
16. Kelley ST, Gilbert JA. Studying the microbiology of the indoor environment. Genome Biol 14(2):202 (2013); doi: 10.1186/gb-2013-14-2-202. [ Links ]
17. Angenent LT, et al. Molecular identification of potential pathogens in water and air of a hospital therapy pool. Proc Natl Acad Sci USA 102(13):4860–4865 (2005); doi: 10.1073/pnas.0501235102. [ Links ]
18. Perkins SD, et al. Potentially pathogenic bacteria in shower water and air of a stem cell transplant unit. Appl Environ Microbiol 75(16):5363–5372 (2009); doi: 10.1128/AEM.00658-09. [ Links ]
19. Feazel LM, et al. Opportunistic pathogens enriched in showerhead biofilms. Proc Natl Acad Sci USA 106(38):16393–16399 (2009); doi: 10.1073/pnas.0908446106. [ Links ]
20. Maki DG, Tambyah PA. Engineering out the risk for infection with urinary catheters. Emerg Infect Dis 7(2):342–347 (2001); http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2631699/. [ Links ]
21. Frank DN, et al. Culture-independent microbiological analysis of Foley urinary catheter biofilms. PLoS ONE 4(11):e7811 (2009); doi: 10.1371/journal.pone.0007811. [ Links ]
22. Zhang L, et al. Microbiological pattern of arterial catheters in the intensive care unit. BMC Microbiol 10:266 (2010); doi: 10.1186/1471-2180-10-266. [ Links ]
23. Hewitt KM, et al. Office space bacterial abundance and diversity in three metropolitan areas. PLoS ONE 7(5):e37849 (2012); doi: 10.1371/journal.pone.0037849. [ Links ]
24. Dawson P, et al. Residence time and food contact time effects on transfer of Salmonella typhimurium from tile, wood and carpet: testing the five-second rule. J Appl Microbiol 102(4):945–953 (2007); doi: 10.1111/j.1365-2672.2006.03171.x. [ Links ]
25. Grice EA, et al. A diversity profile of the human skin microbiota. Genome Res 18(7):1043–1050 (2008); doi: 10.1101/gr.075549.107. [ Links ]
26. Grice EA, et al. Topographical and temporal diversity of the human skin microbiome. Science 324(5931):1190–1192 (2009); doi: 10.1126/science.1171700. [ Links ]
27. Flores GE, et al. Microbial biogeography of public restroom surfaces. PLoS ONE 6(11):e28132 (2011); doi: 10.1371/journal.pone.0028132. [ Links ]
28. Fierer N, et al. Forensic identification using skin bacterial communities. Proc Natl Acad Sci USA 107(14):6477–6481 (2010); doi: 10.1073/pnas.1000162107. [ Links ]
29. Meadow JF, et al. Bacterial communities on classroom surfaces vary with human contact. Microbiome 2(1):7 (2014); doi: 10.1186/2049-2618-2-7. [ Links ]
30. Fleischer M, et al. Microbiological control of airborne contamination in hospitals. Indoor Built Environ 15(1):53–56 (2006); doi: 10.1177/1420326X06062230. [ Links ]
31. Brooks B, et al. Microbes in the neonatal intensive care unit resemble those found in the gut of premature infants. Microbiome 2(1):1 (2014); doi: 10.1186/2049-2618-2-1. [ Links ]
32. Morowitz MJ, et al. Redefining the role of intestinal microbes in the pathogenesis of necrotizing enterocolitis. Pediatrics 125(4):777–785 (2010); doi: 10.1542/peds.2009-3149. [ Links ]
33. Funkhouser L, Bordenstein S. Mom knows best: the universality of maternal microbial transmission. PLoS Biol 11(8):e1001631 (2013); doi: 10.1371/journal.pbio.1001631. [ Links ]
34. Gaüzère C, et al. 'Core species' in three sources of indoor air belonging to the human micro-environment to the exclusion of outdoor air. Sci Total Environ 485–486:508–517 (2014); doi: 10.1016/j.scitotenv.2014.03.117. [ Links ]
35. Meadow JF, et al. Indoor airborne bacterial communities are influenced by ventilation, occupancy, and outdoor air source. Indoor Air 24(1):41–48 (2014); doi: 10.1111/ina.12047. [ Links ]
36. Tringe SG, et al. The airborne metagenome in an indoor urban environment. PLoS ONE 3(4):e1862 (2008); doi: 10.1371/journal.pone.0001862. [ Links ]
37. Kembel SW, et al. Architectural design drives the biogeography of indoor bacterial communities. PLoS ONE 9(1):e87093 (2014); doi: 10.1371/journal.pone.0087093. [ Links ]
38. Kembel SW, et al. Architectural design influences the diversity and structure of the built environment microbiome. ISME J 6(8):1469–1479 (2012); doi: 10.1038/ismej.2011.211. [ Links ]
39. Berg G, et al. Beneficial effects of plant-associated microbes on indoor microbiomes and human health? Frontiers Microbiol 5:15 (2014); doi: 10.3389/fmicb.2014.00015. [ Links ]
40. Lund S, et al. Reality of glove use and handwashing in a community hospital. Am J Infect Control 22(6):352–357 (1994); doi: 10.1016/0196-6553(94)90034-5. [ Links ]
41. Gilkeson CA, et al. Measurement of ventilation and airborne infection risk in large naturally ventilated hospital wards. Build Environ 65:35–48 (2013); doi: 10.1016/j.buildenv.2013.03.006 [ Links ]
Contiendas en el ámbito de la investigación**
Iniciativas para mejorar la reproducibilidad de los hallazgos de los estudios
Los avances en la ciencia dependen de que los investigadores sean capaces de reproducir los hallazgos de sus pares, proporcionando así una plataforma sólida a partir de la cual continuar avanzando con nuevas líneas de investigación científica. Sin embargo, por diversas razones, la irreproducibilidad parece ser un problema cada vez mayor en el ámbito de la investigación experimental. Actualmente, las agencias y revistas de investigación que proveen fondos están elaborando directrices para garantizar que los estudios que se publiquen estén bien diseñados y bien reportados, y que sean más capaces de generar resultados reproducibles.
Los reportes de los medios han señalado varios casos atroces de irreproducibilidad, incluyendo ciertos reportes que publicó la revista Nature hace algunos meses, según los cuales las células madres se vuelven pluripotentes si se las sumerge en una solución ligeramente ácida.1,2 Estos hallazgos fueron muy criticados cuando los científicos no pudieron reproducir los resultados en sus propios laboratorios.3 Posteriormente, el primer autor estuvo de acuerdo en retractarse de esos reportes.4 En otro ejemplo ampliamente publicitado, la empresa biofarmacéutica Amgen afirmó poder reproducir únicamente seis de los 53 estudios considerados como parteaguas en la investigación básica sobre el cáncer, pese a haber contado con la estrecha cooperación de los científicos originales a fin de asegurarse de que se utilizaran los mismos protocolos experimentales.5
Los expertos culpan del problema de la irreproducibilidad a diversos factores, entre los que se incluye el reportaje inadecuado de los métodos utilizados en los estudios de investigación que se publican. Evidencias cada vez mayores vinculan las descripciones deficientes de los métodos con el exceso de supuestos hallazgos;6-10 sin embargo, en otros casos, las conclusiones incorrectas se pueden atribuir a un mal diseño del estudio. En particular, la aleatorización y los cegamientos inadecuados pueden introducir sesgos que impiden que el estudio pueda someter sus tesis a pruebas precisas.11
La irreproductibilidad es particularmente problemática cuando atañe a hallazgos que conducen a ensayos clínicos en sujetos humanos o a regulaciones y políticas que pudieran afectar a la salud pública. Los investigadores documentaron uno de estos casos en el Instituto Nacional de Trastornos Neurológicos y Accidentes Cerebrovasculares (en inglés, NINDS), al descubrir que se había inscrito a pacientes con esclerosis lateral amiotrófica en un ensayo clínico basado en datos preclínicos inadecuados.
Según la directora del NINDS, Story Landis, los pacientes no estaban teniendo los resultados esperados con el tratamiento de prueba, que consistía en un antibiótico de amplio espectro llamado minociclina. Cuando los investigadores del NINDS examinaron con más atención los estudios preclínicos sobre los que se habían basado para realizar el ensayo, encontraron que los autores no había reportado si los estudios eran aleatorizados o a doble ciego. Es más, para el trabajo se había utilizado un pequeño número de animales. "Esa fue una señal que nos hizo despertar", dice Landis. "Los ensayos clínicos en sujetos humanos deben estar basados en hallazgos preclínicos sólidos".
Nuevas iniciativas
En un nivel básico, la "reproducibilidad" se refiere a la capacidad de los científicos para generar resultados comparables a aquéllos reportados en estudios previos. Así, la reproducibilidad difiere de la replicación, o sea, de la generación de los mismos resultados precisos usando los mismos métodos experimentales que usaron los investigadores originales. Estos términos suelen utilizarse de manera intercambiable, pero Shai Silberberg, director del programa NINDS, argumenta que la replicación es más un ideal que una meta práctica.
"Esto es porque en la práctica siempre existirán variables que no podemos controlar, lo cual hace imposible la replicación genuina", dice Silberberg. Por ejemplo, los investigadores no pueden usar los mismos animales dos veces, explica, y el uso de un grupo diferente de animales –aun cuando sean de la misma edad, sexo y cepa– introduce variabilidad en las condiciones del experimento.
En enero de 2014, los directores de los Institutos Nacionales de Salud (en inglés, NIH) anunciaron nuevas iniciativas para enfrentar el problema de la irreproducibilidad en la investigación.12 Lawrence Tabak, director adjunto principal de los NIH, dice que la incapacidad de reproducir los resultados revisados por pares puede obstruir el camino del avance científico. "Necesitamos construir sobre las bases firmes de descubrimientos previos para conseguir avanzar en la investigación", dice. "Y eso es aplicable a todas las ciencias, no sólo a lo que hacemos en el NIH".
Las iniciativas descritas en un comentario publicado en Nature que Tabak escribió en colaboración con el director del NIH, Francis Collins,12 incluyen el entrenamiento en diseño experimental e implementación de listados de verificación para garantizar que los aspirantes al financiamiento aborden de forma adecuada la aleatorización, el cegamiento y los métodos estadísticos apropiados. El NIH también explora el desarrollo de un índice de descubrimiento de datos que brinde acceso a datos primarios inéditos. Dicho acceso permitirá a los investigadores identificar aquellos casos en los que la irreproducibilidad sea resultado de errores en el análisis de datos o del uso de métodos analíticos inapropiados. Por último, un programa piloto llamado PubMed Commons14 ofrece un foro abierto para que los investigadores discutan artículos catalogados en PubMed.
Por lo que se refiere a las revistas, el Grupo Editorial Nature, Science y Science Translational Medicine han anunciado cada uno sus propias medidas para enfrentar la reproducibilidad. "Estamos en posición de ser parte de la solución", dice Marcia McNutt, directora en jefe de Science. Según McNutt, quienes financian pueden abordar la reproducibilidad antes de que comiencen los experimentos, mientras que los editores pueden incrementar la transparencia en cómo fueron realizados, alentando a los autores a describir con suficiente detalle los métodos estadísticos y de laboratorio utilizados. De ese modo, otros científicos pueden determinar su propio nivel de confianza en los resultados y confirmar los resultados reportados.
"Preguntamos a los autores: '¿Realizaron suficientes pruebas?, ¿Hubo imparcialidad al formar los grupos y controles?, ¿Tenían un plan preexperimental para abordar los valores atípicos, o cambiaron las reglas al vuelo?'", explica McNutt. Ahora los críticos y editores de Science señalan trabajos que muestran una transparencia ejemplar con el propósito de desarrollar directrices adicionales de reproducibilidad más adelante este año.15
Los estudios preclínicos bajo escrutinio
Las iniciativas tanto del NIH como de las editoriales están enfocadas desde el principio en las investigaciones experimentales preclínicas. Esto es en parte porque la intervención humana suele estar basada en datos de investigaciones preclínicas en animales, y también porque de manera rutinaria se realizan esfuerzos rigurosos para limitar los sesgos y fortalecer la confianza en los hallazgos científicos en los ensayos clínicos en sujetos humanos.12
McNutt agrega que la preocupación por la reproducibilidad preclínica también ha llevado a normas de consenso comunitarias derivadas de un taller convocado por el NINDS en junio de 2012, al cual asistieron alrededor de 50 personas interesadas del ámbito académico, editoriales, grupos defensores y organismos financieros, así como de la industria farmacéutica.16 Los participantes coincidieron ampliamente en que las deficiencias en la declaración de los métodos y el diseño experimental deficiente van de la mano, y en que se requieren recomendaciones para hacer frente a ese problema.16
Según Silberberg, el solo hecho de que los métodos no se describan de manera adecuada en un artículo no significa que el experimento no haya sido bien realizado. Pero los investigadores necesitan prestar más atención a los diseños experimentales, dice Landis, al igual que a los métodos estadísticos que utilizan, especialmente para grupos de datos complejos provenientes de investigaciones de alto rendimiento.
Cuando se hizo el taller del NINDS, ya existían directrices para la rendición de informes de investigación, en particular, las de ARRIVE (Investigación en animales: presentación de informes de experimentación in vivo, por sus siglas en inglés) desarrolladas por investigadores británicos y publicadas en 2010.17 Las directrices ARRIVE promueven mejores métodos para rendir informes revisados por pares y muchos organismos que financian investigaciones y editoriales (incluyendo a EHP) las han puesto en práctica. Sin embargo, de acuerdo con Silberberg, las directrices son detalladas y exhaustivas al grado de volverse difíciles de manejar. "Existen elementos muy importantes en ellas y otros que no lo son tanto", dice. "Si se pretende imponer la forma de redactar la sinopsis y el título, se perderá público".
Los participantes del taller del NINDS idearon un grupo de recomendaciones más restringidas que fueron publicadas el siguiente octubre en Nature.16 Estas recomendaciones exigen a los investigadores que, como mínimo, reporten cómo se determinaron los tamaños de las muestras, si los animales fueron aleatorizados durante el estudio y en qué forma, si se realizó un cegamiento de los investigadores respecto al tratamiento, y cómo se manejaron los datos.
Cada uno de estos factores es crucial para un buen diseño experimental. Por ejemplo, al asignar animales al azar a los grupos de tratamiento o de control y luego cegarse a los resultados, los investigadores limitan el potencial de introducir elementos de confusión que influyan en los resultados del estudio, dice Jim Berger, profesor de estadísticas de Duke University. De igual forma, es necesario que las muestras tengan tamaños apropiados para asegurar que los hallazgos sean estadísticamente viables.
El valor de los listados de verificación
A raíz del taller de NINDS, el grupo editorial Nature anunció una iniciativa de reproducibilidad en abril de 2013.18 Con ello, Nature levantó las restricciones de duración para el reporte de métodos de los suplementos en línea. Además, tanto los autores como revisores ahora deben completar listados de verificación del diseño experimental. Por último, la revista contrató especialistas en estadística para apoyar con la revisión y brindó un mecanismo mediante el cual el público puede acceder a los datos en bruto con los que se generaron los cuadros y las gráficas publicados. Las recomendaciones del NINDS también fueron citadas por McNutt como la fuente que inspiró la nueva iniciativa de reproducibilidad de Science.15
Las iniciativas de reproducibilidad del NIH todavía están en proceso, enfatiza Tabak, pero sitúan el entrenamiento a la vanguardia. Nuevos módulos de entrenamiento orientado a los aprendices de intramuros cubrirán cuestiones básicas de diseño experimental, y el NIH producirá cortometrajes sobre temas clave como aleatorización, el cegamiento, y las diferencias en las respuestas de los animales basadas en el género, mismos que, según afirma Silberberg, estarán disponibles dentro y fuera de los institutos.
Aparte de los módulos de enseñanza, el NIH está considerando cómo incorporar los listados a la evaluación de solicitudes de subvención. Se espera que los listados cubran las características estándar del diseño experimental (por ejemplo, aleatorización, cegamiento y estadísticas) pero Silberberg dice que también existe la intención deliberada de "no dañar", o en otras palabras, evitar suprimir la creatividad.
Silberberg distingue cuidadosamente entre la investigación de ensayos de hipótesis (que tiene estrictos requisitos metodológicos) y la investigación para generar hipótesis (que no los tiene). "Uno no debería estar obligado a ceñirse a reglas estrictas si se está haciendo estudios exploratorios", dice. "No queremos que nuestros revisores sean demasiado cerrados si la investigación tiene un potencial fascinante".
Varios centros e institutos del NIH ahora están desarrollando y poniendo a prueba listados basados en diversas propuestas de investigación.19 Con la información de esos pilotos, dice Tabak, los dirigentes del NIH decidirán más adelante en este mismo año cuáles adoptar en todo el Organismo, cuáles asignar a institutos y centros particulares y cuáles descartar.
La estrategia del NIEHS
El NIEHS, entre tanto, ha desarrollado un marco para la revisión sistemática de estudios publicados, el cual usa en sus valoraciones de peligros potenciales. Estas valoraciones son realizadas por revisores de la Oficina de Evaluación de la Salud y de Traslación (en inglés, OHAT) dentro del Programa Nacional de Toxicología del NIEHS (en inglés, NTP). Éstos llevan a cabo revisiones técnicas para identificar el potencial de daño ocasionado por las substancias que se encuentran en el medio ambiente. Basar estas revisiones en estudios de baja calidad puede llevar a conclusiones falsas y, por tanto, a políticas que podrían ser demasiado severas o insuficientes en términos de la minimización de riesgos.
A partir de 2011, la OHAT comenzó a explorar las revisiones sistemáticas como un medio para depurar estudios deficientes, en particular sobre distintos efectos del cáncer en la salud. Pero mientras que el método quedó bien establecido en la medicina clínica, no se lo adaptó a la toma de decisiones en el ámbito de la salud ambiental, que se basa en datos de fuentes más diversas, incluídos estudios de epidemiología y toxicología animal, además de estudios mecánicos con sistemas in vitro. Por lo tanto, la OHAT trabajó con expertos técnicos para encontrar la manera de modificar las revisiones sistemáticas para sus propios fines, en relación con lo cual publicó un marco tentativo de siete pasos en 2013.20
Ese marco ya está completo21 y se está implementando. Es posible que un revisor llegue a una conclusión equivocada usando la revisión sistematizada, pero habrá una ventana transparente que muestre cómo llegó a esa decisión. De acuerdo con John Bucher, Director Asociado del NTP, el marco hace posible agregar datos diversos de forma consistente. "Estamos tratando de crear un rastro de juicios sobre estudios individuales que pueda seguirse para explicar nuestra confianza en la evidencia en general", dice.
Sin embargo, aun los estudios mejor ejecutados no pueden controlar cada una de las variables capaces de afectar la reproducibilidad. En algunos casos, los reactivos experimentales son por sí mismos altamente vulnerables al cambio. Las líneas de células epiteliales, por ejemplo, son altamente susceptibles a los leves cambios de su microentorno, e incluso los científicos más expertos pueden introducir cambios que afecten los resultados experimentales sin percatarse, escribió Mina Bisell, una investigadora del cáncer en el Laboratorio Nacional de Lawrence Berkeley, en un comentario sobre la reproducibilidad en 2013.22
Rick Woychik, Subdirector del NIEHS y expresidente del Laboratorio Jackson, señala que en sólo 10 a 20 generaciones, las cepas de ratones reproducidas con propósitos específicos de investigación pueden desarrollar diferencias genéticas capaces de afectar su respuesta a sustancias químicos y farmacéuticas en el ambiente. Es más, dice: es de importancia crítica que los investigadores comprendan exactamente qué trasfondo genético están utilizando porque éste puede influir profundamente en el resultado de un experimento.
"Algunas cepas endogámicas de ratones son hipertensas, otras no. Algunas son altamente sensibles a fármacos como el acetaminofén y otras no lo son. La lista de rasgos variables no tiene fin", explica Woychik. "De forma más particular, un gen desactivado puede tener un fenotipo distinto en distintos trasfondos genéticos.
De modo que, si el trasfondo genético de la cepa no es controlado con cuidado entre dos laboratorios que estén estudiando, por ejemplo, el mismo gen desactivado, no se puede esperar obtener el mismo resultado".
Bucher añade que el NIEHS ha estado supervisando la deriva genética en sus colonias de roedores durante 30 años. De ser necesario, dice, en algunos casos es posible restaurar algunas líneas genéticamente definidas a partir de embriones de ratones criopreservados. Además, los nuevos e innovadores paneles de referencia sobre ratones conocidos como la Collaborative Cross (Cruz Colaborativa) y Diversity Outbred (Diversidad Exogámica)24 están ganando adeptos dentro de la NIEHS. "Estos dos paneles de referencia reflejan de manera más precisa la variabilidad fenotípica que existe en la población humana que una sola cepa endogámica", dice Woychik.
No habrá una bala de plata
"No habrá una bala de plata que solucione el problema de la reproducibilidad", dice Paula Stephan, profesora de la Universidad Estatal de Georgia, quien en 2011 reportó que algunos países –entre los que sobresalen China, Turquía y Corea del Sur– ofrecen incentivos monetarios de hasta un equivalente a 7.5% del salario anual de la facultad por publicar en revistas prestigiosas.25 Agrega: "La carga de revisión ha incrementado de forma dramática, y la calidad de los trabajos enviados está declinando".
Pero, dado que están aumentando los casos de irreproducibilidad, "ahora es el momento de sensibilizar la conciencia de los interesados", dice Tabak. "Tenemos que subrayar que los NIH no pueden resolver el problema por sí solos. Necesitamos que todas las partes interesadas –incluyendo a los editores de revistas, revisores, y miembros del sistema universitario– trabajen juntas para enfrentarlo".
Charles W. Schmidt,
MS, Maestro en Ciencias de Portland, Maine,
es un galardonado escritor científico;
ha escrito para las revistas Discover, Science y Nature Medicine.
Referencias
1. Obokata H, et al. Stimulus-triggered fate conversion of somatic cells into pluripotency. Nature 505(7485):641–647 (2014); doi: 10.1038/nature12968. [ Links ]
2. Obokata H, et al. Bidirectional developmental potential in reprogrammed cells with acquired pluripotency. Nature 505(7485):676–680 (2014); doi: 10.1038/nature12969. [ Links ]
3. Cyranoski D. Acid-bath stem-cell study under investigation. Nature News (17 February 2014); doi: 10.1038/nature.2014.14738. [ Links ]
4. Japan researcher agrees to withdraw disputed stem cell paper. Reuters (4 June 2014). Disponible en: http://goo.gl/3RIynL [consultado el 24 de junio de 2014] [ Links ].
5. Begley CG, Ellis LM. Drug development: raise standards for preclinical cancer research. Nature 483(7391):531–533 (2012); doi: 10.1038/483531a. [ Links ]
6. Macleod MR, et al. Evidence for the efficacy of NXY-059 in experimental focal cerebral ischaemia is confounded by study quality. Stroke 39(10):2824–2829 (2008); doi: 10.1161/STROKEAHA.108.515957. [ Links ]
7. Bebarta V, et al. Emergency medicine animal research: does use of randomization and blinding affect the results? Acad Emerg Med 10(6):684–687 (2003); doi: 10.1111/j.1553-2712.2003.tb00056.x. [ Links ]
8. Crossley NA, et al. Empirical evidence of bias in the design of experimental stroke studies—a metaepidemiologic approach. Stroke 39(3):929–934 (2008); doi: 10.1161/STROKEAHA.107.498725. [ Links ]
9. Rooke ED, et al. Dopamine agonists in animal models of Parkinson's disease: a systematic review and meta-analysis. Parkinsonism Relat Disord 17(5):313–320 (2011); doi: 10.1016/j.parkreldis.2011.02.010. [ Links ]
10. Vesterinen HM, et al. Improving the translational hit of experimental treatments in multiple sclerosis. Mult Scler J 16(9):1044–1055 (2010); doi: 10.1177/1352458510379612. [ Links ]
11. Krauth D, et al. Instruments for assessing risk of bias and other methodological criteria of published animal studies: a systematic review. Environ Health Perspect 121(9):985–992 (2013); doi: 10.1289/ehp.1206389. [ Links ]
12. Collins FS, Tabak LA. Policy: NIH plans to enhance reproducibility. Nature 505(7485):612–613 (2014); doi: 10.1038/505612a. [ Links ]
13. NIH. Development of an NIH BD2K Data Discovery Index Coordination Consortium (U24) [beca de investigación]. Bethesda, MD:National Institutes of Health [Institutos Nacionales de Salud] (13 de diciembre de 2013). Disponible en: http://grants.nih.gov/grants/guide/rfa-files/RFA-HL-14-031.html [consultado el 24 de junio de 2014] [ Links ].
14. NCBI. PubMed Commons [página web]. Bethesda, MD:National Center for Biotechnology Information, U.S. National Library of Medicine (2014). Disponible en: http://www.ncbi.nlm.nih.gov/pubmedcommons/ [consultada el 24 de junio de 2014] [ Links ].
15. McNutt M. Reproducibility. Science 343(6168):229 (2014); doi: 10.1126/science.1250475. [ Links ]
16. Landis SC, et al. A call for transparent reporting to optimize the predictive value of preclinical research. Nature 490(7419):187–191 (2012); doi: 10.1038/nature11556. [ Links ]
17. Kilkenny C, et al. Improving bioscience research reporting: the ARRIVE guidelines for reporting animal research. PLoS Biol 8(6):e1000412 (2010); doi: 10.1371/journal.pbio.1000412. [ Links ]
18. Announcement: reducing our irreproducibility. Nature 496(7446):398 (2013); doi: 10.1038/496398a. [ Links ]
19. NINR. Chronic Wounds: Advancing the Science from Prevention to Healing (R01) [beca de investigación]. Bethesda, MD:National Institute of Nursing Research, National Institutes of Health [Instituto Nacional de Investigación de Enfermería, Institutos Nacionales de Salud] (6 de mayo de 2014). Disponible en: http://grants.nih.gov/grants/guide/rfa-files/RFA-NR-15-001.html [consultado el 24 de junio de 2014]. La Sección IV(2) incluye un ejemplo de una lista de verificación. [ Links ]
20. NTP. Draft OHAT Approach for Systematic Review and Evidence Integration for Literature-based Health Assessments—Febrero de 2013. Research Triangle Park, NC:National Toxicology Program, National Institute of Environmental Health Sciences, National Institutes of Health [Programa de Toxicología del Instituto Nacional de Ciencias de la Salud Ambiental, Institutos Nacionales de Salud] (26 de febrero de 2013). Disponible en: http://goo.gl/85a20D [consultado el 24 de junio de 2014] [ Links ].
21. Rooney AA, et al. Systematic review and evidence integration for literature-based environmental health science assessments. Environ Health Perspect 122(7):711–718 (2014); doi: 10.1289/ehp.1307972. [ Links ]
22. Bissell M. Reproducibility: the risks of the replication drive. Nature 503(7476):333–334 (2013); doi: 10.1038/503333a. [ Links ]
23. The Churchill Group. The Collaborative Cross (CC) [página web]. Bar Harbor, ME:The Churchill Group, The Jackson Laboratory (2014). Disponible en: http://churchill.jax.org/research/cc/ccresources.shtml [consultada el 24 de junio de 2014] [ Links ].
24. The Churchill Group. The Diversity Outbred (DO) [página web]. Bar Harbor, ME:The Churchill Group, The Jackson Laboratory (2014). Disponible en: http://churchill.jax.org/research/cc/doresources.shtml [consultada el 24 de junio de 2014] [ Links ].
25. Franzoni C, et al. Changing incentives to publish. Science 333(6043):702–703 (2011); doi: 10.1126/science.1197286. [ Links ]
Notas
* Publicado originalmente en Environmental Health Perspectives, volumen 122, número 7, julio 2014, páginas A182-A187.
** Publicado originalmente en Environmental Health Perspectives, volumen 122, número 7, julio 2014, páginas A188-A191.