INTRODUCCIÓN
El labio y paladar hendido (LPH) es una patología de distribución mundial y se considera la más frecuente de las malformaciones congénitas.1 Su frecuencia varía entre las poblaciones debido a su fondo genético y a cuestiones ambientales.
Los tratamientos que reciben los pacientes con LPH pueden representar erogaciones financieras hasta de 200,000 dólares, debido a que se requieren manejos multidisciplinarios para atender tales malformaciones.2-4 El LPH es manejado principalmente con cirugías reconstructivas y procedimientos odontológicos; sin embargo, existe una serie de trastornos a largo plazo que representan un problema de salud traducidos en una alta incidencia de patologías mentales, así como un incremento de la probabilidad de muertes a temprana edad.4,5 El LPH también está asociado con una elevada probabilidad de presentar distintos tipos de cáncer (en cerebro, mama, colon) no sólo en los afectados de LPH, sino también en sus familiares.6-9 Las complicaciones derivadas de LPH en etapas tempranas de la vida tienen consecuencias devastadoras en países del tercer mundo, donde el acceso a los tratamientos médicos es difícil o inexistente.10
El LPH clínicamente es definido como un desarrollo embriológico craneofacial anormal. Los pacientes con estos rasgos clínicos presentan, además, otitis infecciosas recurrentes, trastornos del habla, de la masticación, de la deglución, así como alteraciones en la estética facial.11 Como ya se mencionó, el tratamiento de estos pacientes está a cargo de un equipo multidisciplinario, incluyendo el profiláctico: Otorrinolaringología, Cirugía Plástica y Maxilofacial, Ortodoncia, Terapia del lenguaje, Pediatría, Enfermería, Consejo Genético, Audiometría, Psicología o Trabajo Social, por enumerar algunos. Esto representa tratamientos muy complejos y a largo plazo, la mayoría en países del tercer mundo. De allí que es fundamental entender los mecanismos biológicos que son alterados durante etapas del desarrollo de los pacientes con LPH. Recientemente, han existido estudios acerca de las causas de dicha patología, entre ellas exposición ambiental a ciertos tóxicos, elevado riesgo a exposición al tabaco, alcohol o uso de ciertas drogas durante el embarazo, etcétera.12 Sin embargo, la etiología de la patología sigue siendo desconocida13 y se sabe aun menos de los posibles genes involucrados con los procesos patobiológicos del LPH.
Los estudios de asociación de genoma completo (GWAS) reportados en la literatura han arrojado alguna luz sobre los genes posiblemente involucrados en estos procesos.3,14-16 Los genes que muestran reproducibilidad en GWAS realizados por diversos grupos de investigación son: IRF6,17 MAFB,18 FGFR2,19 MSX1,20 VAX1,21 SOX9,22,23 ABCA4,18 BMP4,24 y SUMO1.25-27
Sin embargo, estos estudios reportados fueron hechos en poblaciones con fondos genéticos homogéneos. En el presente estudio se determina la frecuencia de diversos PNU localizados en los genes ABCA4, BMP4, MSX1, SUMO1, VAX1 e IRF6 en una población cerrada de la ciudad de Querétaro, México, con un fondo genético mixto.
MATERIAL Y MÉTODOS
Extracción del ADN total humano. El estudio fue aprobado por el Comité de Ética de la Facultad de Medicina de la Universidad Autónoma de Querétaro. Se seleccionaron sujetos adultos con un rango de edad entre 19-60 años. Tanto los padres como los abuelos de los voluntarios son nacidos y residentes de la ciudad de Querétaro por lo menos en tres generaciones. Los sujetos voluntarios que contaban con un padre nacido en la localidad y el otro de un estado o una ciudad distinta no fueron incluidos en el estudio. Ninguno de los voluntarios presentaron diagnóstico de maloclusión familiar. Otros datos tales como estilo de vida, talla, peso y otras patologías (enfermedades crónico-degenerativas o cáncer) fueron registradas, pero no consideradas para este trabajo. A los sujetos de estudio se les dio a firmar una hoja de consentimiento informado, siguiendo los lineamientos del Comité de Ética de la Facultad de Medicina de la Universidad Autónoma de Querétaro. Las muestras de ADN fueron obtenidas a partir de células epiteliales bucales tanto de los voluntarios sanos como de ambos padres como se describe en Aidar y Line, 2007. En este estudio se utilizaron 93 tríadas (279 individuos) para probar cada PNU.
Selección de PNU: PNU reportados para los genes ABCA4, BMP4, MSX1, SUMO1, VAX1 e IRF6 fueron localizados en la base de datos del NCBI (https://www. ncbi.nlm.nih.gov/snp/). Los PNU analizados en este trabajo fueron seleccionados bajo uno de los siguientes criterios: 1) que causen un cambio en el marco de lectura (missense), 2) que no tengan un efecto en el marco de lectura (synonimous), 3) que se encuentren localizados en zonas regulatorias (i.e. splicing, 3’UTR, 5’UTR). En el caso del la secuencia genómica del gen IRF6 se escogieron aquéllos que se encontraran esparcidos a lo largo de la secuencia genómica (Cuadros I y II). En todos los casos el cambio de nucleótido tiene que causar una modificación en el sitio de reconocimiento de una endonucleasa y este sitio de restricción no debe estar repetido en la secuencia que rodea al PNU en al menos 50 pb de los extremos 5’ y 3’. Para el análisis de sitios únicos de restricción factibles de ser analizados por RFLP para cada uno de los PNU aquí reportados se utilizó el programa NEB cutter v2 software (http://nc2.neb.com/NEBcutter2/).
PCR. Las reacciones en cadena de la polimerasa (PCR) se llevaron a cabo utilizando el reactivo GoTaq® MasterMix (Promega) de acuerdo a las especifi caciones del fabricante. Del ADN total de cada individuo se utilizaron 500 ng como templado para dicha reacción en un volumen total de 20 μL. Los cebadores (primers) fueron diseñados utilizando el programa http://biotools.umassmed.edu/bioapps/primer3_www.cgiy sintetizados por IDT technologies. Las secuencias de cada par de cebadores se encuentran localizadas en el cuadro I.
La reacción fue sujeta a los siguientes ciclos: 95 oC por cinco minutos, 30 ciclos de 95 oC por 30 segundos, temperatura de anclaje (annealing) de cada par de cebadores (primer annealing) por 30 segundos y extensión a 72 oC por 30 segundos. Al final de los 30 ciclos, se añadió un paso de extensión a 72 oC por 10 minutos. La Tm utilizada para el anclaje de los cebadores fue basada en la determinada por IDT. La amplificación fue llevada a cabo utilizando un termociclador marca Artik (Thermo-Fisher) en un volumen fonal de reacción 20 μL. Diez μL de los productos amplificados fueron incubados con su enzima de restricción siguiendo las especificaciones del fabricante (Cuadro II), en un volumen total de 20 μL. Todas las endonucleasas fueron adquiridas del fabricante NEB. Los productos de PCR sin enzima y con enzima de restricción fueron visualizados por electroforesis en cámara submarina en un gel de agarosa al 3% teñido con bromuro de etidio utilizando como búffer de corrimiento TBE al 0.5%. De acuerdo al patrón de restricción, el alelo para cada PNU fue determinado. Todos los reactivos químicos fueron adquiridos de Sigma Aldrich. Resultados representativos de este análisis se muestran en las figuras 1 y 2.
Cuadro I Frecuencias encontradas en muestras de individuos sanos de la ciudad de Santiago de Querétaro, Querétaro, México, para los PNU localizados en la secuencia genómica de los genes MAFB, FGFR2, MSX1, VAX1, SOX9, ABCA4, BMP4 y SUMO1. ADN de 93 tríadas (hijo/a, madre y padre) rodeando al PNU seleccionado fueron amplificadas por PCR de punto fi nal y restringidas con la correspondiente endonucleasa.
| PNU | Gen | Tipo de cambio | Cambio de aminoácido | Alelo 1 | Alelo 2 | Enzima | Alelo encontrado (279/279) | Genotipo (279/279) | Frecuencia reportada& |
| rs377311148 | ABCA4 | Miss | L (P | A | G | TspRI | G | GG | A = 1.0 |
| rs145066457 | BMP4 | Syn | L (L | A | T | BfaI | T | TT | T = 1.0 |
| rs149960650 | MSX1 | Syn | L (L | A | G | BfaI | G | GG | G = 0.996 A = 0.004 |
| rs143825095 | SUMOI | Syn | H (H | A | G | Mnl I | A | AA | A = 1.0 |
| rs200364078 | VAX1 | Intr | Ninguno | C | T | BfaI | T | TT | N/A |
& Frecuencia reportada en la base de datos del NCBI, Miss = Cambio de sentido; Syn = Cambio sinónimo; Intr = PNU localizado en la secuencia intrónica.
Cuadro II Frecuencias encontradas en muestras de individuos sanos de la ciudad de Santiago de Querétaro, Querétaro, México, para los PNU localizados en la secuencia genómica del gen IRF6. ADN de 93 tríadas (hijo/a, madre y padre) rodeando al PNU seleccionado fueron amplificadas por PCR de punto final y restringidas con la correspondiente endonucleasa.
| PNU | Gen | Tipo de cambio | Cambio de aminoácido | Alelo 1 | Alelo 2 | Enzima | Alelo encontrado (279/279) | Genotipo (279/279) | Frecuencia reportada& |
| rs141555342 | IRF6 | Syn | P (P | C | T | MspI | T | TT | T = 0.999983 C = 0.000016 |
| rs377332433 | IRF6 | Miss | P (L | A | G | Msp I | G | GG | G = 0.999950 A = 0.000049 |
| rs527840168 | IRF6 | Intr | N/A | C | T | MspI | C | CC | N/A |
| rs539406879 | IRF6 | 3’ UTR | N/A | C | T | MspI | T | TT | N/A |
| rs752367933 | IRF6 | Intr | N/A | C | G | BfaI | G | GG | N/A |
| rs767916600 | IRF6 | Miss | Q (R | C | T | MnlI | T | TT | T = 0.999991 C = 0.000008 |
| rs774631697 | IRF6 | Intr | N/A | A | G | MspI | A | AA | N/A |
& Frecuencia reportada en la base de datos del NCBI. Miss = Cambio de sentido; Syn = Cambio sinónimo; Intr = PNU localizado en la secuencia intrónica.
RESULTADOS Y DISCUSIÓN
El LPH representa la malformación congénita más frecuente en el mundo con una tasa aproximada de uno por cada 90 nacidos vivos. Estudios de asociación genética han revelado genes que se asocian con la aparición de LPH tales como: MAFB, FGFR2, MSX1, VAX1, SOX9, ABCA4, BMP4, SUMO1 e IRF6. Con base en ello decidimos probar si un marcador genético es adecuado para ser empleado en estudios de asociación. Las frecuencias encontradas para cada PNU seleccionado para el presente trabajo son mostrados en los cuadros I y III. En el cuadro I observamos que los PNU localizados en los genes ABCA4, BMP4, MSX1, SUMO1 y VAX1 son compartidos en el 100% de la población (279 de 279). Además, los PNU elegidos para ABCA4, BMP4, MSX1 y SUMO1 son concordantes con los resultados reportados en la base de datos de la NCBI PNU. En este trabajo se reporta por primera vez en la literatura la frecuencia de VAX1 en una población.
Debido a que ningún PNU resultó útil para ser utilizado como marcador genético para estudios de asociación en ninguno de estos genes, decidimos enfocar nuestra atención en IRF6. Estudiar el gen IRF6 como pieza clave en etiopatogenia del LPH es de gran interés, basados en dos reportes recientes, en los cuales, de manera independiente y en dos poblaciones diferentes, se demostró su capacidad de ser reproducible como marcador genético asociado a LPH. Su función durante el desarrollo embrionario es de gran importancia, porque han sido reportadas mutaciones en este gen, dando origen a fenocopias que semejan la patología no sindrómica de LPH. El producto del gen IRF6 es una proteína llamada factor regulador del interferón 6, la cual es un factor regulador perteneciente a la familia SMIR. Esta proteína se caracteriza por una estructura altamente conservada para reconocer las secuencias promotoras del ADN. Dicho gen se localiza en la región 1q32-q41, la cual se considera una zona crítica asociada con el síndrome de Van der Woude (VWS1). Asimismo, diferentes mutaciones del gen IRF6 dan como resultado fenotipos del VWS1, transmitiéndose de forma autosómica dominante con penetrancia incompleta y con rasgos clínicos de hoyuelos en labio inferior y/o LPH. Este síndrome representa la forma más frecuente de LPH y de malformaciones sindrómicas del labio. Los marcadores genéticos han sido descritos cerca del gen IRF6 para poblaciones homogéneas, como es el caso de Querétaro, así como en otras poblaciones de México. Las variantes alélicas que fueron halladas en nuestro trabajo son mostradas en el cuadro III.
Cuadro III Secuencias de los cebadores utilizados para la reacción en cadena de la polimerasa.
| Gen | PNU ID | Cebador izquierdo | Cebador derecho |
| ABCA4 | rs377311148 | TAACCCTCCCAGCTTTGGAC | AAGCTGCTCATTGTCTTCCC |
| BMP4 | rs145066457 | TCGGCTAATCCTGACATGCT | ACACGACTACTGGACACGAG |
| MSX1 | rs149960650 | CTCCGCAAACACAAGACGAA | GTAGTCTCTTTGCCTTGGCG |
| SUMO1 | rs143825095 | GCAGTTAAGCAGGGTACAAGA | TCTCCTTAGGATAGCAGTGAGA |
| VAX1 | rs200364078 | CCACAGCCTCTTCAGCCT | TTGTTGGTCCGGGAGTAAGG |
| IRF6 | rs141555342 | CAGCTCTCCTGGGTTTGAAG | GTCTTCCTCAGGGCCTCTTT |
| IRF6 | rs377332433 | GCTGTATAGGTGCCTGGGG | CCCCTGTCTGCTCACCATAT |
| IRF6 | rs539406879 | TTCCTCTGACATCATTCTGAAAA | AAGTGCCAAGAAATGCCAAG |
| IRF6 | rs527840168 | AAGCGGGAATGTAACGTGTG | AATAGACACTGGGGCCTCCT |
| IRF6 | rs767916600 | TTTGTTTTCTGGGTCCTTCC | CTGCGCTGTGCTCTCAATAA |
| IRF6 | rs774631697 | ACTTCTCAAGCCCCACAATG | ATGCTGCCCTACAAAACACC |
| IRF6 | rs752367933 | CTGCTCCCCAGTCCTATCAC | TCTCCTTTCCCATCATCAGG |
PCR = Reacciones en cadena de la polimerasa.
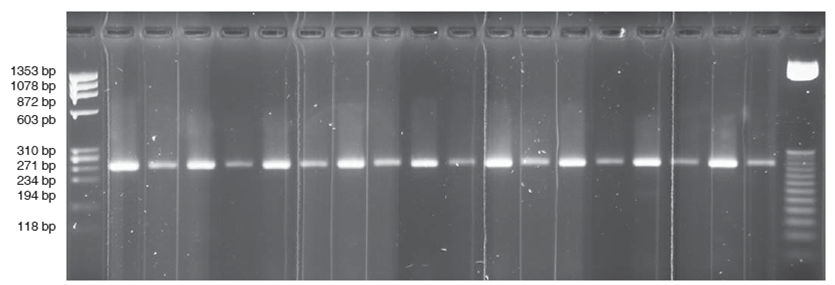
Figura 1 Muestra representativa del análisis por RFLP del PNU rs143825095 (A/G) ubicado en la secuencia codificante del gen SUMO1. El producto de 237pb obtenido por PCR (carriles 2, 4, 6, 8, 10, 12, 14, 16 y 18) obtenido de cada individuo sano fue sujeto a restricción con la endonucleasa MnlI por 1 hora a 37 oC (carriles 3, 5, 7, 9, 11, 13, 15, 17 y 19). Las muestras fueron cargadas en un gel de agarosa al 3% teñido con bromuro de etidio. Como marcadores de peso molecular se utilizó ADN del fago Phi 174 restringidos con la endonucleasa HaeIII (carril 1) y el marcador de 25 pb de NEB (carril 20). El gel se corrió por electroforesis en cámara submarina por una hora (material y métodos).

Figura 2 Muestra representativa del análisis por RFLP del PNU rs27840168 (C/T) ubicado en la secuencia intrónica del gen IRF6. El producto de 220bp obtenido por PCR (carriles 2, 4, 6, 8, 10, 12, 14, 16, 18, 20 y 22) obtenido de cada individuo sano fue sujeto a restricción con la endonucleasa MspI por 1 hora a 37 oC (carriles 3, 5, 7, 9, 11, 13, 15, 17, 19, 21 y 23). Las muestras fueron cargadas en un gel de agarosa al 3% teñido con bromuro de etidio. Como marcador de peso molecular se utilizó ADN del fago Phi 174 restringidos con la endonucleasa HaeIII (carriles 1 y 24). El gel se corrió por electroforesis submarina por una hora (material y métodos).
Los resultados del genotipo de la población analizada muestran que ninguno de estos PNU muestran las características necesarias para ser considerados como marcadores genéticos, debido a que se les encontró en el 100% de la población estudiada. Dos de los PNU: (rs767916600, rs141555342) concuerdan con lo reportado en la literatura, sin embargo las frecuencias de los PNU rs539406879, rs527840168, rs774631697, rs752367933 son descritas por primera vez provenientes de una población de Querétaro, México.
CONCLUSIÓN
Después de realizar el análisis RFLP de varios PNU ubicados en la región genómica de los genes ABCA4, BMP4, MSX1, SUMO1, VAX1 e IRF6, podemos concluir que cuando menos para dichos marcadores existe una homogeneidad genética en la población analizada, lo cual la convierte en la población ideal para realizar más estudios en ella. Aun cuando ninguno de los PNU reportados en el estudio son de utilidad para futuros posibles estudios de asociación, se plantea estudiar un conjunto de 36 PNU candidatos a análisis RFLP en la población sana que se encuentren distribuidos a lo largo de la secuencia genómica de IRF6, a fin de encontrar uno que llene las características de ser marcador molecular para estudios de asociación genética.
Financiamiento: El estudio fue financiado con los programas PRODEP-SEP 2016.











 nueva página del texto (beta)
nueva página del texto (beta)


