Introducción
Guarianthe skinneri (Bateman) Dressler & W.E. Higgins es una orquídea nativa del sureste de México, con una distribución natural desde México hasta Panamá (Bertolini et al., 2016). En la región del Soconusco (Chiapas, México) se conoce con el nombre común de Candelaria, debido a que la época de floración de la planta coincide con la fecha de la fiesta de la Virgen de la Candelaria y se usa para adornar los altares ceremoniales. Esto también ocurre en Guatemala, donde miles de plantas son utilizadas anualmente para una festividad similar, principalmente en el municipio de Jacaltenango (Archila, obs. pers., 2016). Desde 1939, G. skinneri es oficialmente la flor nacional de Costa Rica, con el nombre común de Guaria Morada (Dobles, 1945).
En México, esta especie se ha registrado históricamente sólo en el estado de Chiapas, con una abundancia baja en la Reserva de la Biosfera El Triunfo y también en la Reserva de la Biosfera Volcán Tacaná (Damon, 2010). Actualmente resulta difícil verla de forma natural en los alrededores de la ciudad de Tapachula (Chiapas, México). De hecho, las poblaciones G. skinneri en el Soconusco se han reducido drásticamente por la extracción intensiva e ilegal de sus individuos, y por la disminución de su hábitat natural (Jiménez-Bautista, 2009; Damon, 2013). Por estas razones, G. skinneri está registrada como especie amenazada en la Norma Oficial Mexicana NOM-059-SEMARNAT-2010 (SEMARNAT, 2010).
Para diseñar planes adecuados de manejo de poblaciones para uso ornamental, y posibles programas de reintroducción, es importante entender la diversidad genética de las poblaciones presentes para incrementar nuestra capacidad de predicción a amplia escala y tomar decisiones de conservación bajo consideraciones genéticas y ecológicas (Broadhurst et al., 2017). Manejar el mayor nivel de variación en la formación artificial de poblaciones incrementa la probabilidad de que los esfuerzos de conservación tengan resultados favorables a largo plazo (decenas o cientos de generaciones) (Hamrick et al., 1991; Oyama, 1993). En este mismo sentido, la definición de escenarios de distribución histórica y potencial es conocimiento que fortalece cualquier medida de conservación de orquídeas, ya que ayuda a reconocer aquellas áreas que reúnen condiciones ambientales para el desarrollo de sus poblaciones (Bertolini et al., 2016).
Diversos estudios genéticos indican que las especies de la familia Orchidaceae mantienen poblaciones con cierto grado de aislamiento genético, debido a probables barreras geográficas o ecológicas, así como a diferentes presiones selectivas (Ávila-Díaz y Oyama, 2007; Pinheiro, 2012; Quiant et al., 2013). En especies como G. skinneri, los factores antropogénicos pueden tener un efecto de gran magnitud en sus niveles y patrones de diversidad, como se ha señalado para otros grupos de plantas (Vargas et al., 2006; Ávila-Díaz y Oyama, 2007; Piñero et al., 2008). Las estimaciones de la diversidad genética de poblaciones silvestres o en cautiverio pueden ser determinadas mediante herramientas moleculares que a su vez se basan en la variación nucleotídica de secuencias de ADN relativamente cortas o fragmentos de bajo número de pares de bases (pb), o en la variación del tamaño de proteínas, producto del ADN de los individuos. A partir de secuencias, expresiones proteícas o fragmentos, se obtienen las frecuencias genotípicas y alélicas, y con base en éstas se pueden obtener parámetros, acordes al tipo de marcador seleccionado, de diversidad genética y niveles de diferenciación entre poblaciones que pueden estar correlacionados con factores ecológicos de las especies. Por ejemplo, Ávila-Díaz y Oyama (2007), con base en las frecuencias alélicas enzimáticas, detectaron en Laelia speciosa (Kunth) Schltr. una alta homocigosidad (Fis=0.216), y un polimorfismo alto (74%) en comparación con otras familias vegetales como Asteraceae, Cucurbitaceae, Chenopodiaceae y Onagraceae, entre otras (Hamrick y Godt, 1990). Vargas et al. (2006) encontraron valores significativos de diferenciación (Fst=0.370) entre ocho poblaciones de Myrmecophila christinae Carnevali & Gómez-Juárez var. christinae de la Península de Yucatán, México. Qian et al. (2013) reportaron considerables niveles de diferenciación de las poblaciones de Calanthe tsoongiana Tang & F.T. Wang, una orquídea terrestre rara y endémica, nativa de China, con base en marcadores de ISSR (Inter-Simple Sequence Repeat) (Gst=0.55), lo que indicó un reducido flujo genético entre sus poblaciones (Nm=0.408). Así mismo, Pinheiro et al. (2012) encontraron niveles altos de diferenciación entre las poblaciones de Cattleya labiata Lindl. (aproximadamente 15%).
Por lo anterior, la propagación con fines de reintroducción o de manejo de poblaciones para servicio ornamental debe considerar el origen y niveles de diversidad genética apropiados para los ambientes en consideración. En este sentido, determinar si las plantas de G. skinneri conservadas en los patios particulares de Tapachula (Chiapas) tienen niveles de diversidad similares a los de las poblaciones silvestres, en este caso de Guatemala, puede ser una forma de evaluar su potencial para ser empleadas como fuente de germoplasma, ya que pese a los esfuerzos dirigidos a la ubicación de poblaciones en la región del Soconusco (Chiapas), no se encontraron individuos de la especie (Bertolini, obs. pers., 2016), y por lo tanto el estudio se realizó sólo con muestras de la zona urbana de Tapachula y de áreas silvestres de Guatemala.
Los objetivos de nuestro estudio fueron: (1) evaluar la diversidad genética de la población urbana de G. skinneri conservada en los patios particulares de Tapachula y de poblaciones silvestres de Guatemala, para determinar su potencial como fuente de propágulos de uso ornamental y su posible reintroducción cuando se tenga certeza del origen de los individuos, y (2) estimar la estructura genética de las poblaciones estudiadas y si el nivel de diferenciación está directamente relacionada con la distancia geográfica que las separa. La información generada es útil para valorar si las poblaciones urbanas de Tapachula son aptas para ser empleadas como fuente de germoplasma, para el diseño de cruzas que generen propágulos con una suficiente variabilidad genética, e idónea para establecer un manejo sustentable de plantas útiles para ornato, y con ello contribuir a la reducción de la extracción clandestina de las poblaciones silvestres remanentes. También puede llegar a diseñarse un programa de reintroducción en el medio natural una vez que se conozca su procedencia y se valore su adaptabilidad al sitio destino.
Materiales y Métodos
Muestreo
Se colectaron 30 pseudobulbos con hojas a partir de individuos distintos y sanos de Guarianthe skinneri, procedentes de domicilios particulares de la ciudad de Tapachula (Chiapas, México), procurando que fuesen suficientemente distantes entre sí (1000 m de distancia mínima) (Cuadro 1), para disminuir la probabilidad de que fueran individuos emparentados. Este conjunto de individuos se consideró como una población. En Guatemala, se eligieron cinco localidades (Escuintla, Huehuetenango, Jutiapa, Mazatenango, Santa Rosa) para la colecta del material vegetal, considerando el conjunto de individuos de cada localidad como una población distinta (Cuadro 1). En total para Guatemala se obtuvieron 40 pseudobulbos con hoja.
Cuadro 1: Ubicación geográfica, altitud y ambiente general de los sitios de colecta de pseudobulbos y hojas de Guarianthe skineri (Bateman) Dressler & W.E. Higgins. * Variables contemporáneas (current 1950-2000) de Worldclim (Hijmans et al., 2005). ** bosque seco subtropical (bs-S), bosque muy húmedo (bmh-S), bosque húmedo subtropical (bh-S). Número de individuos=N.
| Localidad | N | Latitud (° sexagesimales) | Longitud (° sexagesimales) | Altitud (m s.n.m.) | Temp. promedio anual (°C)* | Ambiente** | |
|---|---|---|---|---|---|---|---|
| México | Guatemala | ||||||
| Tapachula | - | 30 | 14°53'28.34"N | 92°16'15.74"O | 135 | 26.7 | urbano |
| - | Escuintla | 9 | 14°16'35.94"N | 90°48'47.80"O | 248 | 26.6 | bh-S |
| - | Huehuetenango | 15 | 15°17'56.91"N | 91°28'28.20"O | 1834 | 16.9 | bs-S |
| - | Jutiapa | 5 | 14°10'11.62"N | 89°54'47.07"O | 847 | 24.4 | bmh-S |
| - | Mazatenango | 5 | 14°31'40.45"N | 91°30'12.25"O | 386 | 26.2 | bh-S |
| - | Santa Rosa | 6 | 14°10'55.33"N | 90°21'45.24"O | 596 | 24.9 | bmh-S |
Para la selección e individuación de poblaciones silvestres de la especie objeto de estudio en Guatemala, se escogieron los sitios de colecta con base en el patrón de distribución del nicho ambiental reportado por Bertolini et al. (2016), en áreas con alta probabilidad de presencia de nicho ambiental (Fig. 1).
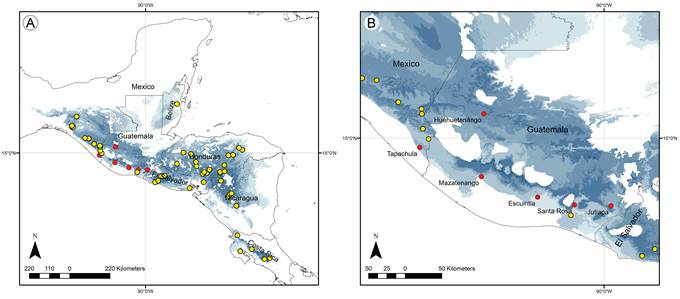
Figura 1: Registros históricos de Guarianthe skinneri (Bateman) Dressler & W.E. Higgins, a lo largo de Centroamérica (color amarillo). Modelación de su nicho ambiental (modelo logístico, gradiente de azul) y elección de los sitios de colecta (color rojo), coherentemente con la distribución del nicho ambiental potencial de la especie (fuente: Bertolini et al., 2016). Al azul más oscuro corresponden probabilidades más altas que se presenten las condiciones ambientales óptimas. A. distribución global; B. distribución local relativa a las colectas.
Una vez seleccionadas las localidades, constatada in situ la presencia de hábitat favorable y de poblaciones de la especie, previo permiso de la comunidad rural, se llevó a cabo la colecta con procedimientos de disección de un pseudobulbo con hoja, con el menor daño físico posible a las plantas madres, dejando esa última en su forofíto original.
Extracción y amplificación de ADN
El ADN genómico se extrajo de fragmentos de hojas frescas, con el kit de extracción DNeasy Plant Mini Kit de QIAGEN® (QIAGEN Inc., Valencia, CA, USA) y con el método convencional de extracción en plantas CTAB 2Xmercaptoetanol (Doyle y Doyle, 1987) con modificaciones.
Se ensayó la amplificación de ADN nuclear ribosomal (ITS, internal transcibed spacer): ITS 1 (TCCGTAGGTGAACCTGCGG) -2 (GCTGCGTTCTTCATCGATGC), e ITS 3 (GCATCGATGAAGAACGCAGC) -4 (TCCTCCGCTTATTGATATGC) para todas las muestras (White et al., 1990). Se eligió este marcador debido a que es una región del ADN nuclear, por lo que su herencia es biparental y casi universal para plantas, es de bajo costo y relativamente rápido de obtener (Álvarez y Wendel, 2003). El protocolo de amplificación constó de una desnaturalización inicial de 95 ºC por 3 min, seguida por 35 ciclos con una desnaturalización de 95 ºC por 30 s, una temperatura de alineamiento de 57 ºC para el ITS 1-2, 52 ºC para el ITS 3-4 por 30 s y una extensión de 72 ºC por 2 min; la extensión final se dio a 72 ºC por 10 min (White et al., 1990).
Cada reacción tuvo un volumen final de 39 μl, de los cuales 33 μl fueron de solución Master Mix (PROMEGA), 2 μl de cada primer a 2 mM, y 2 μl de ADN a 20 ng/μl. Los productos de las reacciones de PCR fueron visualizados mediante electroforesis en geles de agarosa a 2%, teñidos con bromuro de etidio y observados con luz UV con la ayuda de un marcador control (Ladder) de 100 pares de bases (pb) y el software Kodak 1D Image Analysis v. 3.6 (Eastman Kodak Company, 1998-2002).
Se realizó la secuenciación de cada muestra amplificada en Macrogen Inc. (Seúl, Corea del Sur), mediante el método de secuenciación por Electroforesis Capilar (Sanger), utilizando el kit de secuenciación ABI PRISM BigDye ®TM Terminator Cycle Sequencing Kit (Applied Biosystems, Foster city, EUA) y el secuenciador ABI PRISM 3730XL14-20139-001 Analyzer KB 1.4.0. 96 capillary type (Applied Biosystems, Foster city, EUA). La extracción y amplificación se realizó por dos ocasiones para corroborar las secuencias y disminuir errores de obtención del genotipo de cada individuo.
Análisis de diversidad y estructura genética
Una vez revisadas y editadas las secuencias se obtuvieron los estimadores de diversidad considerando un modelo de sustitución de bases de Kimura 2-parámetro (K2P), por ser el modelo evolutivo más simple para explicar la mayoría de los sitios polimórficos que son producto de la mutación; su fijación es esencialmente por deriva genética, y a su vez es una estimación de la divergencia entre pares de secuencias (Kimura, 1980; Hedrick, 2000). Las secuencias fueron editadas con el programa Chromas v. 2.4.4 (McCarthy, 1998-2016) y comparadas en la base internacional GenBank NCBI (National Resource for Molecular Biology information) para confirmar la veracidad de los resultados. El alineamiento se realizó con el programa Clustal_X v. 2.1 (Thompson et al., 1997). Los parámetros de diversidad genética se obtuvieron mediante el programa DNaSP v. 5 (Librado y Rozas, 2009) y fueron: sitios polimórficos (s), que corresponden a sitios en donde las secuencias difieren; diversidad de nucleótidos (π), que es el número de nucleótidos diferentes por sitio entre dos secuencias tomadas al azar; media de diferencias nucleotídicas (K), es el número total de posiciones segregantes en un grupo de secuencias; número de haplotipos (h); variación nucleotídica por secuencia (θ), que es la probabilidad de que dos muestras tomadas al azar difieran en una posición particular del genoma y diversidad haplotípica (Hd), que indica el polimorfismo dado por cambios nucleotídicos en la región analizada (Kimura, 1980; Nei y Gojobori, 1986; Castillo-Cobián, 2007; Arboleda, 2008).
Para evaluar la historia poblacional de cada especie se realizó la prueba de Tajima (1989) (D), la cual compara la diversidad nucleotídica (π) y la variación nucleotídica por secuencia (θ). Si D es negativa, θ posee un valor mayor que π, lo que indica la presencia de mutaciones deletéreas (Tajima, 1983). En cambio, si D resulta ser positiva quiere decir que π tiene un mayor valor que θ, lo que sugiere que algunos alelos se encuentran bajo selección positiva (por ejemplo, selección balanceadora), incrementando así sus frecuencias (Tajima, 1983). Si D es igual a cero se infiere que no existe diferencia entre ambos estimadores y por tanto se encuentran en equilibrio neutral (Tajima, 1989).
Para reconocer si hay estructura o diferenciación genética de las poblaciones estudiadas, se obtuvieron los índices de fijación equivalentes a Fst, Fis y Fit de Wright (1951). Fst es una estimación de la diferenciación entre poblaciones, Fis puede ser interpretado como el nivel de endogamia de los individuos con respecto a la población analizada, y el Fit endogamia de los individuos con respecto a la muestra total. Estos estimadores se obtuvieron a partir de un Análisis de Varianza Molecular (AMOVA), usando el software Arlequín v. 3.1 (Excoffier et al., 2005), con 1023 permutaciones. Con la finalidad de visualizar la posible estructuración de las poblaciones se realizó un análisis de agrupación de las poblaciones o localidades por el método de Neighbour Joining (NJ) y una red de haplotipos en el programa PAUP ver. 4.0a147 (Swofford, 2003) y en TCS ver. 1.21 (Clement et al., 2000), respectivamente. Para establecer la posible relación entre la estructura genética y la distancia geográfica que las separa, se hizo una correlación de la distancia genética Kimura 2-parámetro entre pares de poblaciones contra la distancia euclidiana (a partir de las coordenadas geográficas), equivalente a la distancia geográfica lineal entre poblaciones. Si la distancia geográfica influye en la diferenciación entre pares de poblaciones, entonces la distancia genética varía en función directa de la distancia geográfica que separa a las poblaciones (Slatkin, 1994). La correlación se hizo empleando el programa R 3.4.0 (Fox, 2005, 2007; R Core Team, 2017).
Resultados
Diversidad genética
De las 70 muestras colectados (30 de Tapachula y 40 de Guatemala) se logró extraer ADN de 25 muestras de Tapachula y 38 de Guatemala, se pudo amplificar la región ITS 1-2 y ITS 3-4 en 54 muestras (19 de Tapachula y 35 de Guatemala), por lo que el análisis genético se basó en 54 secuencias de 587 pb. Se encontraron 184 sitios polimórficos y 217 mutaciones. Se observaron en total 38 haplotipos. El número (H) de haplotipos varió entre 1 (Mazatenango, Guatemala) y 15 (Tapachula, México); en consecuencia, se obtuvo un intervalo amplio de diversidad haplotídica (Hd) que fue 0 para Mazatenango y 1 para Santa Rosa (Guatemala) que en cada individuo mostró una secuencia de ADN única (Cuadro 2). La diversidad con base en número promedio de sitios distintos o polimórficos (s) estuvo entre 0 y 165, que corresponde a Mazatenango y Tapachula, respectivamente. La diversidad nucleotídica (π) y el número promedio de diferencias nucleotídicas muestran la misma tendencia, evidenciando que Tapachula posee la mayor diversidad, seguida por Huehuetenango, Santa Rosa, Jutiapa, Escuintla y Mazatenango, con 0 de diversidad (Cuadro 2). Se obtuvieron valores negativos de D-Tajima no significativos para cada una de las poblaciones, lo que no permite descartar procesos evolutivos aleatorios ni selectivos (Cuadro 2).
Cuadro 2: Diversidad genética de poblaciones de Guarianthe skinneri (Bateman) Dressler & W.E. Higgins, de las localidades de Tapachula y Guatemala. Número de muestras=N, número de haplotipos=H, diversidad haplotípica=Hd, sitios polimórficos=s, diversidad de nucleótidos=π, media de diferencias nucleotídicas=K, prueba de Tajima=D. No significativo=NS.
| Localidad | N | H | Hd | s | π | K | D |
|---|---|---|---|---|---|---|---|
| Tapachula | 19 | 15 | 0.97 | 165 | 0.050 | 33.07 | -1.69NS |
| Escuintla | 4 | 2 | 0.50 | 1 | 0 | 0.5 | -0.61NS |
| Huehuetenango | 15 | 13 | 0.97 | 87 | 0.040 | 28.4 | -0.16NS |
| Jutiapa | 5 | 2 | 0.60 | 1 | 0.001 | 0.6 | 1.22NS |
| Mazatenango | 5 | 1 | 0 | 0 | 0 | 0 | - |
| Santa Rosa | 6 | 6 | 1 | 49 | 0.030 | 20.06 | -0.65NS |
Estructura genética
El Análisis de Varianza Molecular (AMOVA) mostró que la mayor variación genética se encuentra dentro de las poblaciones (91.71%), entre las poblaciones fue de 8.29% (Cuadro 3).
Cuadro 3: Análisis de varianza molecular (AMOVA) en Guarianthe skinneri (Bateman) Dressler & W.E. Higgins, con el modelo Kimura 2-parámetro.
| Fuente de variación | Suma de cuadrados | Componentes de varianza | % de variación | g.l. |
|---|---|---|---|---|
| Entre poblaciones | 110.42 | 1.143 | 8.29 | 5 |
| Dentro de las poblaciones | 607.21 | 12.65 | 91.71 | 48 |
| 717.62 | 13.79 |
El estimador de diferenciación genética reveló 8% de diferenciación entre poblaciones (Fst=0.083), así como niveles importantes de endogamia de los individuos con respecto a las subpoblaciones (Fis=0.145) o a la muestra total (Fit=0.099).
Se obtuvieron dos redes de haplotipos, una formada por sólo dos haplotipos y otra con 17 haplotipos. No se ubicaron en ninguna de las redes 10 haplotipos de Huehuetenango, ocho de Tapachula y tres de Santa Rosa (Fig. 2). El haplotipo basal de la red más grande (15 haplotipos) se colectó en Tapachula, de la cual se derivan los haplotipos observados en Jutiapa, Santa Rosa y Escuintla (Fig. 2), mientras que Mazatenango tuvo un solo haplotipo, el cual es el mismo que el basal de Tapachula. Al contrario, el análisis de agrupación por NJ mostró una relación genética entre las poblaciones de Escuintla, Jutiapa y Mazatenango, por un lado, y entre Huehuetenango y Santa Rosa por otro; Tapachula aparece de forma más distante genéticamente (Fig. 3). El análisis de correlación no evidenció una relación entre la distancia genética y geográfica (Fig. 4).
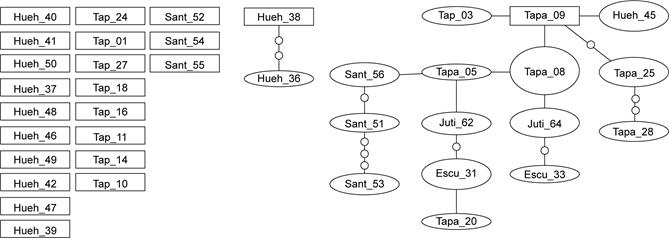
Figura 2: Red de haplotipos de Guarianthe skinneri (Bateman) Dressler & W.E. Higgins, colectados en Tapachula (México) y en las localidades Guatemaltecas de Escuintla, Huehuetenango, Jutiapa y Santa Rosa.
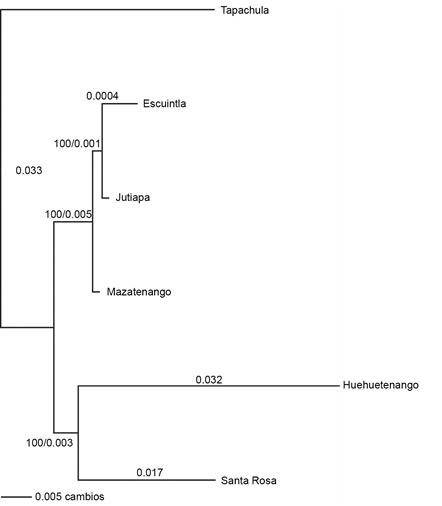
Figura 3: Resultado del análisis de agrupación con el método de Neighbour Joining (NJ) con base en las distancias genética Kimura 2-parámetro de poblaciones de Guarianthe skinneri (Bateman) Dressler & W.E. Higgins, de Chiapas (Tapachula, México) y Guatemala (Escuintla, Huehuetenango, Jutiapa, Mazatenango, Santa Rosa).
Discusión
Se observaron niveles de diversidad desde nulos hasta altos (π entre 0 y 0.05) en las poblaciones de Guarianthe skinneri. La estructura genética fue moderada (8% de divergencia entre poblaciones), debido a una probable reproducción endogámica en sus poblaciones silvestres, así como por su movilización para su utilización como planta de ornato en jardínes privados de Tapachula, y en festividades religiosas tanto en Chiapas como en Guatemala, lo que facilita el flujo de genes, de manera que la distancia geográfica y las barreras físicas entre poblaciones sólo tuvieron impacto entre las poblaciones de Tapachula, Huehuetenango y Mazatenango.
Los patrones de baja diversidad y una estructura de moderada a alta en las poblaciones silvestres de Guatemala son consistentes con lo observado en otras especies de orquídeas, como Calanthe tsoongiana (Qian et al., 2013). Es interesante que la población urbana de Tapachula presentara altos niveles de diversidad y que contenga el haplotipo basal de una de las redes de haplotipos; esto puede ser debido a que los individuos muestreados pueden tener un origen poblacional diverso. Este dato es alentador en el sentido de que la población urbana representa un reservorio de germoplasma y puede ser fuente de propágulos para programas de comercialización y/o cultivo en invernaderos de aquellos individuos, que incluyan las características más sobresalientes o de más calidad estética. Es claro que la diversidad evaluada de la población del centro urbano es un resultado que debe ser tomado con reserva, ya que se trata de una población artificial y probablemente es el resultado de colecta y/o comercio clandestino. Es posible que esta diversidad sea el efecto de colectas e introducciones de germoplasma procedente de diferentes áreas geográficas, en décadas diferentes. Guariante skinneri tiene un valor ceremonial en Mesoamérica. Considerando que el Soconusco es una región que ha sido habitada por la etnia mam, es posible que G. skinneri haya sido empleada ornamentalmente y para algún tipo ceremonial en épocas anteriores a la colonización española. Según de la Cerda (1940), los mames se constituyeron de poblaciones asentadas en la Huasteca potosina y veracruzana que llegaron a colonizar el Soconusco durante los siglos III y IV. Se sabe que las poblaciones prehispánicas en México apreciaban y empleaban las orquídeas como flores ceremoniales (Halbinger y Soto, 1997; Hágsater et al., 2005). Es probable, por lo tanto, que estas costumbres hayan trascendido la colonización y la evangelización católica realizada por los españoles durante la conquista y que permanezca hasta la actualidad con el empleo de flores de G. skinneri para la fiesta católica de la Virgen de la Candelaria, tanto en México (en Tuxtla Chico, a unos cuantos kilómetros de la ciudad de Tapachula) como en Guatemala. Este fenómeno antrópico explicaría en parte el porqué de una baja diferenciación entre las poblaciones y al mismo tiempo una alta diversidad en poblaciones artificiales como el caso de Tapachula. Además, la influencia del fenómeno migratorio actual entre los países de Centroamérica y México puede jugar un rol importante en la configuración de la diversidad y estructura genética de G. skinneri en la población artificial de la ciudad mencionada.
Asimismo, los datos muestran menos diversidad en las poblaciones de Guatemala que en la de Tapachula, al grado de observar un solo haplotipo en una población de Guatemala, lo cual probablemente puede ser el resultado del tamaño de muestra (García-Bautista et al., 2014) o que estas poblaciones se hayan originado a partir de uno o pocos genotipos. Aunque el estimador D-Tajima fue no significativo, de cualquier manera, se considera que la expansión demográfica de G. skinneri es reciente y que su diversidad genética responde a eventos principalmente aleatorios, sin un claro efecto de la selección natural (Tajima, 1983). Por lo anterior, se puede concluir que la diversidad y diferenciación entre poblaciones son aleatorias y asociadas en parte al manejo y uso de la flor. La reducción de las poblaciones por las modificaciones del hábitat o por la sustracción de individuos conduce a un riesgo de endogamia (Pinheiro et al., 2012) que es probable en G. skinneri, dado los valores de Fis y Fit observados. Además, la endogamia a su vez incrementa la probabilidad de expresión de genes deletéreos, con lo que la población se reduce en cada generación hasta llegar a un punto de riesgo de extinción. Es deseable un seguimiento de las poblaciones y estimaciones de la adecuación de los organismos muestreados para determinar apropiadamente el potencial evolutivo y viabilidad de las poblaciones.
Encontramos una baja diferenciación entre poblaciones que no pudo ser atribuida a la distancia geográfica. El nivel de diferenciación entre poblaciones de G. skinneri (8%), está por debajo de lo observado en otras poblaciones de orquídeas; por ejemplo, Brzosko et al. (2013) reportan una diferenciación genética de 6% en Goodyera repens (L.) R. Br.; en contraste, Pillon et al. (2007) reportan alta diferenciación genética significativa entre poblaciones de Liparis loeselii (L.) Rich. (Fst=0.382). El valor de Fst para G. skinneri sugiere un flujo genético que puede ser producto del movimiento realizado por el humano, al ser esta especie de interés como planta de ornato o también es posible que se trate de poblaciones formadas por los mismos genotipos, como es el caso de la población de Mazatenango que tuvo un solo haplotipo y éste se comparte con la muestra de Tapachula. El flujo génico encontrado en la orquídea Cymbidium sinense (Jacks.) Willd. fue de 2.4, e indica que el flujo de genes entre poblaciones que habitan las seis regiones que fueron muestreadas fue suficiente para evitar la diferenciación poblacional (Lu et al., 2011). La falta de una estructura con relación a la distancia que separa las poblaciones reveladas por el análisis de correlación parece común entre orquídeas. Qian et al. (2013) no observaron una correlación significativa entre las distancias genéticas y distancias geográficas en las poblaciones de colecta de la orquídea Calanthe tsoongiana. Asimismo, Smith et al. (2002) encontraron que la distancia genética entre poblaciones de Tipularia discolor (Pursh) Nutt. no se correlacionó significativamente con la distancia geográfica. La falta de correlación entre la distancia genética y la distancia geográfica en G. skinneri puede deberse a la dificultad de obtener un mayor número de poblaciones a lo largo de la escala geográfica considerada (la máxima distancia entre las poblaciones fue de 308.5 km).
Es posible que la estructura genética de G. skinneri observada tenga relación con la distribución geográfica, en tanto que las poblaciones de Guatemala tienen mayor semejanza genética en el análisis de NJ y Tapachula aparece como la más distante. Posiblemente si se plantean estudios con un mayor número de poblaciones y considerando todo el intervalo de distribución de la especie, se obtengan patrones más claros de estructura y sus causas, incluyendo las poblaciones que se conservan en jardines privados de ciudades en Guatemala y/o Costa Rica.
Una diferenciación escasa con bajos y moderados niveles de diversidad es un patrón que puede ser común para algunas orquídeas, atribuido a sus limitaciones de dispersión y reproducción (Pinheiro et al., 2012; Alamillo-Vásquez, 2013). Nuestros resultados sugieren que los factores antropogénicos pueden tener un efecto mayor sobre la diversidad y estructura genética de las poblaciones que los de tipo ecológico. La extracción indiscriminada de G. skinneri en las áreas silvestres de los alrededores de Tapachula ha reducido enormemente el número de individuos nativos, a tal grado que hoy en día es difícil observar poblaciones silvestres en los alrededores de la ciudad (Bertolini, obs. pers.). A pesar de la fuerte presión antrópica, la diversidad genética poseída por la población urbana de G. skinneri ha resultado ser altamente variada comparativamente con las poblaciones silvestres. Una resistencia similar se ha observado en poblaciones de Laelia speciosa en la cuenca de Cuitzeo en Michoacán (México), que pese a la extracción masiva mantiene un tamaño efectivo poblacional, diversidad alélica y una heterocigosidad moderada (Rojas-Méndez et al., 2017).
Conclusiones
Los resultados sugieren que, a pesar de la alta presión antrópica ejercida en la especie en cuestión, la comercialización ilegal de esta planta en la ciudad de Tapachula ha incrementado la diversidad genética en esta población. Este estudio ofrece una base sólida para sugerir una propagación in vitro de G. skinneri (Coutiño-Cortés et al., 2017), utilizando como individuos fuente a la población establecida en los jardines particulares de Tapachula, con fines de cultivo intensivo en invernaderos controlados y su uso sustentable, para bajar la presión antrópica sobre las poblaciones silvestres.
Cabe mencionar que este estudio no justifica ni pretende fomentar la colecta y venta ilegal G. skinneri, ni de otras orquídeas u otros organismos amenazados. En contraste, se resalta que los resultados señalan que hay posibilidades biológicas para la formación de invernaderos de cultivo para la especie, con una gran diversidad genética, para seleccionar a los mejores genotipos. Tapachula es un lugar con condiciones ambientales propias del nicho ambiental de G. skinneri, razón por la cual, a pesar del estrés de la colecta, las plantas han podido encontrar un ambiente favorable en los patios particulares y recuperar su vigor. Frente a este raro caso de éxito de conservación involuntario, hay decenas de otras especies de orquídeas que son traídas de altitudes mayores y por lo tanto de climas distintos, de tal manera que se conducen ineludiblemente a su muerte.











 nueva página del texto (beta)
nueva página del texto (beta)




