Introducción
El cáncer colorrectal (CCR) es el tercer tumor más prevalente en el mundo. La incidencia y la mortalidad del cáncer colorrectal en México ascienden a 10.6 y 5.4/100 000 habitantes, respectivamente.1 El CCR es una enfermedad compleja que abarca desde la transformación de la mucosa normal, hasta una neoplasia maligna en múltiples etapas que altera diversos mecanismos genéticos y epigenéticos de la vías de señalización.2 Estudios de ligamiento génico y de asociación del genoma completo (GWAS, genome-wide association studies) han identificado variantes comunes y raras relacionadas con más de 50 genes que confieren riesgo de CCR.3 Las variantes comunes pueden conferir diferentes magnitudes de riesgo en las distintas poblaciones debido a diferencias en frecuencias alélicas, factores ambientales, etcétera.4,5 Estudios en poblaciones europeas y asiáticas han demostrado asociaciones entre CCR y polimorfismos de nucleótido simple (SNP, single nucleotide polymorphism) implicados en la vía de señalización TGF-b/BMP, que involucran a TGFB1, SMAD7, BMP4, BMP2, GREM1, etcétera.6,7 Es importante comprender los efectos de estos SNP en CCR en poblaciones latinoamericanas.8
Realizamos un estudio de casos y controles de sujetos de tres ciudades mexicanas para analizar las asociaciones entre CCR y las variantes rs12953717, rs4939827, rs11874392, rs961253, rs4779584, rs11632715 y rs4444235, seleccionadas después de revisar los SNP relacionados con CCR en estudios de asociación del genoma completo en población española, debido a la contribución de este componente étnico en la población mexicana.7,9-12
Métodos
El estudio fue aprobado por los comités de ética de cada institución participante:
– Hospital Universitario, Universidad Autónoma de Nuevo León, HU-UANL (BI10-003).
– Instituto Mexicano del Seguro Social, IMSS (R-2012-785-032)
– Comisión Federal para la Protección contra Riesgos Sanitarios (CMN2012-001).
Se dispuso de 1000 muestras de ADN extraído de sangre periférica de pacientes con reporte patológico de CCR y de 1043 muestras de control. Las muestras de ADN fueron recolectadas entre diciembre de 2010 y agosto de 2012 en el Hospital de Oncología del Centro Médico Nacional Siglo XXI, IMSS, Ciudad de México (74.6 %); Unidad Médica de Alta Especialidad 25 del IMSS y HU-UANL en Monterrey, Nuevo León (22.7 %); Unidad de Medicina Familiar 16 y Unidad Médica de Alta Especialidad 71 del IMSS, así como Hospital Universitario de Torreón, Coahuila (2.7 %). Las muestras de control se reclutaron en Ciudad de México, Monterrey y Torreón (72.6, 25.9 y 1.5 %, respectivamente).
El cuestionario clínico incluyó datos de edad, sexo, patología, antecedentes familiares, datos socioeconómicos y factores de riesgo para estudiar variables externas y estratificar los resultados. Según los estándares de la Organización Mundial de la Salud, la obesidad se definió como índice de masa corporal > 30. Se excluyeron los casos sospechosos sin informes de patología, así como los casos y controles con antecedentes familiares de CCR, para controlar los efectos de variantes de alta penetrancia relacionadas con el tumor. Se incluyeron sujetos con antecedentes familiares de neoplasias no relacionados con CCR. Se obtuvo el consentimiento informado de todos los participantes.
El ADN genómico de las muestras de sangre periférica se aisló con el kit Wizard® (Promega, Madison, Wisconsin). Con la plataforma Sequenom® (San Diego, California) se realizó el genotipado de las variantes SNP de rs4444235 (cromosoma 14q22.2, SNP intergénico cerca del extremo 5’ de BMP4); rs4779584 y rs11632715 (cromosoma 15q13.3, SNP intergénicos entre SCG5 y GREM1); rs12953717, rs4939827 y rs11874392 (cromosoma 18q21, SNP intrónicos en SMAD7); y rs961253 (cromosoma 20p12.3, SNP intergénico cerca del extremo 5’ de BMP2).
El tamaño muestral se calculó considerando la frecuencia de alelos menores reportada en los controles, con lo cual se logró una potencia superior a 0.99 y un nivel de significación de 0.05. Se describieron las principales características demográficas de la muestra y se analizaron las diferencias significativas de las categorías de cada variable entre casos y controles mediante las pruebas chi cuadrada y ANOVA. La totalidad de los análisis y las representaciones gráficas se realizaron con el programa R versión 3.6.1.13
Se estudiaron desviaciones del equilibrio de Hardy-Weinberg en los controles. Se probaron las asociaciones entre CCR y las variantes rs4444235, rs4779584, rs11632715, rs4939827, rs12953717 y rs961253 mediante regresión logística univariada, y se estimaron los valores de razones de momios (RM). Para los modelos logísticos univariados se utilizó la condición caso/control como variable de respuesta (dicotómica) y cada uno de los polimorfismos como únicas variables explicativas.
Debido a la probable relevancia biológica para el fenotipo canceroso, también se realizaron análisis de regresión logística múltiple para cada SNP versus CCR y covariables, incluyendo en el modelo cada SNP (codificados bajo el modelo log-add) y las covariables seleccionadas (sexo, edad, obesidad, antecedentes no familiares de CCR, sedentarismo y ancestría). La significación se evaluó en función de los valores de p de dos colas después de la corrección por prueba de comparaciones múltiples de Bonferroni, considerando valores de p ≤ 0.05 como significativos en todos los análisis, los cuales se realizaron con el programa PLINK.14
La ancestría individual se evaluó utilizando el programa ADMIXTURE versión 1.3.1,15 usando un subconjunto de 31 000 SNP del microarreglo Affymetrix Genome-Wide Human SNP Array 6.0, con una muestra de 1712 sujetos (831 casos y 881 controles).8 Se establecieron tres poblaciones ancestrales para el análisis de la población mexicana.16 Estos análisis incluyeron muestras de ascendencia europea y africana de HAPMAP,17 así como muestras de nativos americanos.18 Se obtuvo la ascendencia individual de las muestras mexicanas y se incluyó el valor de la ascendencia europea como covariable en el análisis multivariado mencionado.
Resultados
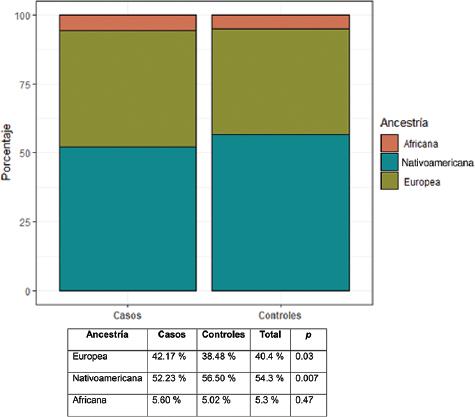
Todas las variantes mostraron equilibrio de Hardy-Weinberg, excepto la variante rs11874392 (gen SMAD7), que se descartó para los estudios posteriores. Los análisis bivariados de CCR y cada covariable de riesgo no genético mostraron asociaciones de CCR con el sexo (a favor de los varones), edad avanzada, obesidad, antecedentes familiares de cáncer no colorrectal, sedentarismo y ascendencia (Tabla 1 y Figura 1). Los análisis de ancestría mostraron una diferencia de proporciones de ascendencia europea entre casos y controles (42.2 y 38.4 %, respectivamente). Esa diferencia se complementó con una mayor ascendencia indoamericana en los controles (Figura 1). La ascendencia africana no mostró diferencias entre casos y controles. Estos resultados son consistentes con la mayor incidencia de CCR en europeos que en pobladores originarios de América, y la significación justificó incluir a la ancestría como covariable en los análisis multivariados.
Tabla 1 Asociaciones entre covariables no genéticas y cáncer colorrectal en población mexicana
| Variable | Afectados | No afectados | Total | RM | IC 95 % | p | |||
|---|---|---|---|---|---|---|---|---|---|
| n | % | n | % | n | % | ||||
| Sexo | |||||||||
| Hombre | 521 | 52.1 | 658 | 63.15 | 1179 | 57.74 | 0.6347 | 0.53-0.75 | < 0.0001 |
| Mujer | 479 | 47.9 | 384 | 36.8 | 863 | 42.26 | |||
| Edad (años) | 3.638 | 3.27-4.04 | < 0.0001 | ||||||
| < 20 | 6 | 0.6 | 76 | 7.29 | 82 | 4.02 | |||
| 21-30 | 24 | 2.4 | 373 | 35.8 | 397 | 19.44 | |||
| 31-40 | 94 | 9.4 | 291 | 27.93 | 385 | 18.85 | |||
| 41-50 | 195 | 19.5 | 198 | 19 | 393 | 19.25 | |||
| 51-60 | 336 | 33.6 | 84 | 8.06 | 420 | 20.57 | |||
| 60-70 | 343 | 34.3 | 18 | 1.73 | 361 | 17.68 | |||
| > 70 | 2 | 0.2 | 2 | 0.19 | 4 | 0.20 | |||
| Obesidad | 203 | 20.8 | 63 | 6.05 | 266 | 13.2 | 4.087 | 3.03-5.50 | < 0.0001 |
| Estilo de vida sedentario | 299 | 30.5 | 384 | 36.89 | 683 | 33.81 | 0.753 | 0.62-0.90 | 0.003 |
| Antecedentes familiares de cáncer no colorrectal | 561 | 50.7 | 342 | 33.7 | 903 | 42.02 | 2.024 | 1.69-2.42 | < 0.0001 |
Los análisis de asociación, poscorrección de Bonferroni, mostraron asociación entre CCR y las variantes rs4444235 (cercana a BMP4), rs4939827 y rs12953717 (variantes intrónicas de SMAD7). Las variantes restantes no mostraron asociación posterior a la corrección (Tabla 2). Después de ajustar las covariables, la regresión logística múltiple confirmó la asociación de CCR con las mismas variantes de BMP4 y SMAD7 (Tabla 3).
Tabla 2 Resultados de la regresión logística univariada entre polimorfismos de nucleótido simple y cáncer colorrectal en población mexicana
| Cromosoma | SNP | FAM Afectados | FAM No afectados | FAM Todos | A1 | A2 | RM | L95 | U95 | p* | p ajustada** |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 14q22.2 (BMP4) | rs4444235 | 0.272 | 0.224 | 0.2477 | T | C | 1.287 | 1.117 | 1.482 | 0.0004749 | 0.003799 |
| 15q13.3 (GREM1) | rs4779584 | 0.305 | 0.280 | 0.2925 | T | C | 1.135 | 0.9915 | 1.3 | 0.06638 | 0.531 |
| 15q13.3 (GREM1) | rs11632715 | 0.389 | 0.391 | 0.3905 | G | Un | 0.9854 | 0.8677 | 1.119 | 0.8212 | 1 |
| 18q21.1 (SMAD7) | rs4939827 | 0.298 | 0.242 | 0.27 | T | C | 1.318 | 1.148 | 1.514 | 8.94E-05 | 0.0007156 |
| 18q21.1 (SMAD7) | rs12953717 | 0.264 | 0.209 | 0.2365 | T | C | 1.346 | 1.165 | 1.554 | 5.352E-05 | 0.0004281 |
| 20p12.3 (BMP2) | rs961253 | 0.512 | 0.480 | 0.4963 | C | Un | 1.141 | 1.008 | 1.292 | 0.03768 | 0.3015 |
*Valores brutos.
**Valores ajustados por la prueba de Bonferroni.
A1: alelo 1; A2: alelo 2; C: citosina; FAM: frecuencia alélica menor; G: guanina; L95: límite inferior del intervalo de confianza de 95%; NO: no observado; RM: razón de momios;
SNP: single nucleotide polymorphism; T: timina; límite superior del intervalo de confianza de 95%.
Tabla 3 Análisis de regresión logística múltiple ajustando por sexo, edad, obesidad, estilo de vida sedentario, antecedentes familiares y ascendencia para el cáncer no colorrectal en población mexicana
| Cromosoma | SNP | A1 | RM | L95 | U95 | p | p ajustada |
|---|---|---|---|---|---|---|---|
| 14q22.2 (BMP4) | rs4444235 | T | 1.285 | 1.102 | 1.498 | 0.001344 | 0.01076 |
| 15q13.3 (GREM1) | rs4779584 | T | 1.145 | 0.9874 | 1.328 | 0.07311 | 0.5849 |
| 15q13.3 (GREM1) | rs11632715 | G | 0.9627 | 0.8368 | 1.108 | 0.5947 | 1 |
| 18q21.1 (SMAD7) | rs4939827 | T | 1.317 | 1.133 | 1.531 | 0.0003339 | 0.001882 |
| 18q21.1 (SMAD7) | rs12953717 | T | 1.342 | 1.147 | 1.57 | 0.0002353 | 0.002671 |
| 20p12.3 (BMP2) | rs961253 | C | 1.149 | 1.003 | 1.317 | 0.04584 | 0.3667 |
A1: alelo 1; C: citosina; G: guanina; L95: límite inferior del intervalo de confianza de 95%; RM: razón de momios; SNP: single nucleotide polymorphism; T: timina; U95: límite superior del intervalo de confianza de 95%.
Discusión
Este estudio replica las asociaciones de CCR con las variantes rs4939827 y rs12953717 localizadas en SMAD7 y la variante rs4444235 cercana a BMP4, y corrobora las asociaciones entre CCR y genes de la vía de señalización TGF-b/BMP reportada en poblaciones caucásicas y asiáticas, con lo cual se constata la relevancia biológica de estos polimorfismos comunes en la susceptibilidad a CCR.19,20 No se observaron asociaciones de CCR con otras variantes estudiadas (BMP2 y GREM1), posiblemente por las diferencias de frecuencias alélicas prevalentes en los mexicanos resultantes del mestizaje.21
Las variantes intrónicas de SMAD7 mostraron mayor relevancia. Los valores de asociación y de RM de rs12953717 son similares a los reportados por Ho et al., Slattery et al., Pittman et al. y Broderick et al., quienes establecieron que el alelo T, que disminuye la expresión del gen, es un alelo de riesgo de CCR.4,11,22,23 El alelo protector C fue más prevalente en la población analizada, a diferencia de las poblaciones caucásicas y de la base de datos 1000 Genomes Project.11,22,24-27 La variante T se asocia significativamente a tumores rectales (p = 0.002, RM = 4.85).26 Pittman et al. y Slattery et al. obtuvieron resultados similares cuando ajustaron sus estudios con las covariables edad, sexo y antecedentes familiares.11,22 También se ha observado que la asociación entre rs12953717 y el riesgo de CCR puede modificarse con el uso de medicamentos antiinflamatorios no esteroideos, el tabaquismo, el índice de masa corporal y el historial de pólipos.28 Respecto a la variante rs4939827 también se ha reportado un efecto genético considerable en cáncer rectal en portadores del alelo T.26,29 De nuevo, se observan diferencias en las frecuencias alélicas: el alelo protector C es más prevalente en la población mexicana que la reportada en 1000 Genomes Project.24
La proteína SMAD7 es un antagonista de la vía de señalización TGF-b que interfiere con el ensamblaje del complejo SMAD2/SMAD4 y afecta las respuestas inflamatorias dependientes de NF-kB.30,31 SMAD7 es un modulador de las acciones inflamatorias y tróficas del TGF-b en la mucosa intestinal y su expresión alterada por variantes intrónicas puede explicar su papel en el CCR.30,31
También hay asociación entre rs4444235 (locus BMP4) y CCR (Tablas 2 y 3). Los componentes de la señalización BMP involucrados en el riesgo de CCR podrían comprender otros genes como SMAD4, BMPR1A, GREM1, BMP2 y LAMA7.32 Existen variantes causales de CCR que no involucran la vía BMP/SMAD y que están relacionadas con los genes APC y CDH1 y algunos miembros de la vía Wnt.33
Para evaluar el papel de la ancestría en el riesgo de CCR, se incluyó la ascendencia europea como covariable (Tabla 3). Nuevamente, las variantes rs4444235, rs4939827 y 12953717 mostraron significación después de ajustar las covariables, lo que apoya la consistencia de las asociaciones encontradas.
Conclusiones
El estudio replica datos reportados para poblaciones caucásicas y asiáticas y refuerza la relevancia biológica de variantes comunes de SMAD7 y BMP4 en la susceptibilidad a CCR en una población mestiza. El análisis multivariado con covariables corroboró asociaciones que se encontraron en el análisis simple. Finalmente, el análisis multivariado mostró que las asociaciones de las variantes de SMAD7 y BMP4 con CCR son consistentes y confieren riesgo de CCR en nuestra población.











 nueva página del texto (beta)
nueva página del texto (beta)