INTRODUCCIÓN
La Campilobacteriosis Genital Bovina (CGB) es una enfermedad infecto contagiosa que afecta al ganado bovino. El género Campylobacter es el responsable de esta enfermedad. Son bacterias Gram negativas, de forma espiral, y móviles. Actualmente se reconocen 32 especies de Campylobacter (Chukwu et al., 2019) de los cuales Campylobacter fetus subsp. fetus (Cff) y Campylobacter fetus subsp. venerealis (Cfv) afectan el sistema reproductivo de los bovinos (Chiapparrone et al., 2014). La enfermedad es considerada de reporte obligatorio, incluída en la Lista B de las enfermedades de los animales terrestres según la Organización Mundial de Salud Animal (OMSA), antes OIE (Tshipamba et al., 2020). En rumiantes Cff se ha demostrado que el patógeno afecta el sistema entérico, especialmente el intestino (Li et al., 2022), por otro lado, los toros con Cfv son reservorio porque las bacterias viven y se adaptan en los quistes prepuciales, mientras que en las vacas las colonizaciones son en vagina, cuello uterino y útero (Cagnoli et al., 2020 y Lúcio et al., 2019) induciendo procesos proinflamatorios (Campos-Múzquez et al., 2021) debido a la internalización en células del epitelio endometrial (Campos-Múzquez et al., 2019) provocando abortos (Clune et al., 2022); C. fetus también puede afectar a humanos y se ha reportado induciendo infecciones sistémicas (Adhikari et al., 2022) endocarditis (Lynch et al., 2022) y meningitis (Fernández et al., 2022). El aislamiento bacteriológico y la utilización de pruebas bioquímicas juegan un papel importante para la identificación de C. fetus y la diferenciación entre Cff y Cfv, ya que se consideran el estándar de oro, sin embargo, la implementación de estas técnicas de cultivo es difícil, ya que Campylobacter requiere condiciones de cultivo in vitro más exigentes que otros géneros bacterianos, además, el cultivo es pobre en confiabilidad y reproducibilidad (Iraola et al., 2016). Para la identificación de Cfv se pueden utilizar diferentes métodos, como la reacción de antígeno- anticuerpos como Inmunoensayos Ligados a Enzimas (ELISA), Inmunofluorescencia Directa (ID) (Dorsch et al., 2022) y métodos moleculares como Electroforesis en Gel de Campo Pulsado (EGCP), Polimorfismo de Longitud de Fragmento Amplificado (PLFA), tipificación de secuencias multilocus y Reacción en Cadena de la Polimerasa (PCR). Actualmente las PCR son la prueba con la alternativa más prometedora para detectar de forma eficiente de Cfv a partir de muestras de campo (Chaban et al., 2012). El objetivo de este estudio fue detectar la presencia de C. fetus por la prueba de PCR en esmegma de toros utilizados como reprodutores de la zona centro del estado de Tamaulipas.
MATERIAL Y MÉTODOS
Muestreo
Se realizó un muestreo por conveniencia en sementales bovinos mayores de 2 años, de diferentes razas (Charolais, Beefmaster, Brangus rojo, Brahman) en Unidades de Producción Pecuaria (UPP) ubicadas en la zona centro de Tamaulipas, México. Se realizó de agosto a diciembre del 2020. Para la obtención de muestras prepuciales se utilizaron jeringas y cánulas desechables estériles para depositar 60 mL de Solución Ringer (PISA®) en el prepucio de cada semental; después se cerró el orificio prepucial y se realizó un masaje vigoroso durante 5 minutos en todo el prepucio, posteriormente se obtuvo el contenido del lavado (esmegma) por escurrimiento y se depositó en una bolsa plástica estéril con sellado hermético. Las muestras de semen se obtuvieron mediante electroeyaculación utilizando un equipo marca minitube®, en la estimulación para el eyaculado no se sobrepasó de 20 voltios por animal. Para la colecta del eyaculado se usó un estuche colector el cual sostiene un embudo de látex, en cuyo extremo se colocó un tubo graduado para recibir el semen, este tubo fue identificado antes de ser utilizado. Las muestras se mantuvieron en refrigeración (4°C) durante el transporte y hasta el procesamiento (Silveira et al., 2018).
Aislamiento de ADN
Las muestras colectadas fueron centrifugadas a 604 xG por 5 min. (Centurion Sc limited, UK) en tubos de plástico de 50 ml estériles, posteriormente se descartó el sobrenadante y se reconstituyo el sedimento con 0.2 ml de PBS 1X estéril. Para el aislamiento del ADN genómico, todas las muestras fueron tratadas con un kit comercial DNeasy sangre y tejido (QIAGEN® Alemania), siguiendo las instrucciones del fabricante. Una vez finalizado el proceso de obtención del ADN, los productos genómicos se mantuvieron en congelación (-20 ˚C) hasta su uso posterior. Para evaluar la purificación del ADN se realizó un estudio por espectrofotometría a razón de 260/280 nanómetros (JENWAY Genova, UK), y se consideraron valores ideales de pureza entre 1.8 a 2.0; las muestras de ADN que se encontraron en el rango fueron sometidas a la amplificación de ADN por PCR.
Amplificación de ADN y electroforesis
Se utilizó un PCR multiplex; los cebadores utilizados fueron nC1165g4F (AGGACACAAATGGTAACTGG) y nC1165g4R (GATTGTATAGCGACTTTGC) para detectar una región del gen cstA de Cfv con productos de amplificación de 233 pares de bases (pb); los cebadores MG3F (GGTAGCCGCAGCTAAGAT) y MG4R (TAGCTACAATAACGACAAC) detectan la región del gen virB11 Cff que amplifica a 764pb (Iraola et al., 2012 y Hum et al., 1997). Como control positivo de la reacción de PCR se utilizó ácido nucleico de una cepa de referencia ATCC de Cff 27374, posteriormente al realizar los ensayos de PCR se identificó una muestra (muestra 1) con amplificación sugestiva a Cfv. El resultado de amplificación fue secuenciado por el Instituto de Biotecnología de la UNAM. La secuencia se analizó en la base de datos de GenBank, a través de la herramienta en línea BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi). La reacción de PCR se realizó en 50 µL, (que contenía 5 µL de Buffer 10 X (Tris-HCl 100 mM pH 8.4, KCl 500 mM, gelatina 10 µg/ml, BSA 1.5 mg/ml, Tritón X 100 al 1% (Biotecmol, BIOTECMOL®, CDMX México), dNTPS 0.2 mM (InvitrogenTM, Life technologies; EUA), 25 µM de cada cebador, MgCl2 3.0 mM (BIOTECMOL ® CDMX, México), 5 U de ADN polimerasa Taq (Amplificasa® BIOTECMOL, CDMX, México), además de 300 ng de ADN, finalmente se ajustó con agua libre de nucleasas a 50 µL. Las condiciones de ciclado de PCR utilizadas fueron desnaturalización inicial por 7 min a 95 °C seguido de 35 ciclos de desnaturalización durante 30 seg a 94 °C, alineación 30 seg a 53 °C; y extensión por 1 min a 72 °C, y una extensión final de 2 min a 72 °C (Iraola et al., 2012). Los productos finales se analizaron por electroforesis en gel de agarosa al 3%, y luego se tiñeron con bromuro de etidio (promega) a una concentración de 0.5 µg/mL durante 30 min. Para la visualización e identificación de los productos de PCR se utilizó un transiluminador UV y se fotodocumentó en el sistema de formación de imágenes E-gel. Los productos de PCR se purificaron con el kit comercial PureLink™ PCR Purification Kit (Invitrogen, Carlsbad, EE. UU.). Algunos productos de PCR fueron secuenciados en el Instituto de Biotecnología UNAM, Ciudad de México, México. Los resultados de la secuenciación se analizaron en el BLASTn. Para la comparación de secuencias se utilizó la base de datos NCBI/GenBank (Altschul et al., 2012).
RESULTADOS
Se muestrearon 38 toros; se obtuvo una muestra de cada uno; 31 de lavado prepucial y 7 de semen. El 18.4% fueron muestras positivas (7/38) (Figura 1), de éstas, dos fueron de semen y cinco de lavado prepucial. De las 7 muestras positivas, 5 productos de PCR se sometieron a secuenciación. Todas las secuencias se compararon considerando la bacteria con número de acceso JF901335.1 en el Genbank.
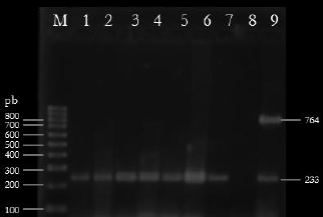
Figura 1 Gel de agarosa al 2.5% con los productos de PCR. Carril M: 100 mpb (BIOLINE, USA); carriles 1-9: Cfv positivos; carril 8: control negativo; carril 9: control positivo Cff 764 pb y Cfv 233 pb.
Las secuencias de nucleótidos de los amplicones obtenidos de muestras de campo por PCR fueron idénticas a las encontradas en la base de datos GenBank. El análisis mostró entre 99,17 y 97,87% de identidad con otras Cfv (Fig. 2). Estos resultados demostraron que Cfv está presente en hatos de la zona centro de Tamaulipas, y que Cfv juega un papel importante en la salud reproductiva del ganado bovino.
DISCUSIÓN
Está documentado que la CGB afecta a hatos bovinos; patología causada por Campylobacter fetus (C. fetus) subespecie venerealis (Cfv). La CGB es una de las principales causas de infertilidad y aborto en bovinos, ovinos y caprinos; y se ha confirmado la presencia en varios países del continente americano como Argentina, Brasil, Costa Rica, Jamaica y México. Sin embargo, en México, este patógeno ha sido poco estudiado en las zonas ganaderas. La detección de esta subespecie patógena en toros utilizados como sementales es de suma importancia para el control de esta enfermedad en los hatos, ya que son portadores asintomáticos; y, además de ser utilizados para la monta directa, el semen es frecuentemente utilizado para inseminación artificial, tanto en fresco como en pajillas conservadas en nitrógeno líquido. Aunado a los problemas de infertilidad, Cff se considera la principal causa de las alteraciones espermáticas debido a la capacidad que tienen estas bacterias de adherirse a los espermatozoides ocasionando daño en la membrana acrosomal así como también a la cromatina espermática, lo que disminuye la calidad del semen y consecuentemente el porcentaje de preñez, además de influir en la infertilidad en las hembras por la transmisión de las bacterias al tracto genital en la monta natural (Cagnoli et al., 2020).
El diagnóstico de esta enfermedad se puede realizar por diversos métodos como aislamiento bacteriológico, métodos serológicos como ELISA e IFD, y ensayos moleculares como PCR (Mshelia et al., 2010; Clune et al., 2022). El aislamiento bacteriológico es el método menos sensible, como lo demuestra Groff et al., en 2010, donde comparó este método con PCR, dando como resultado que este método molecular es 8.5 veces más sensible que el aislamiento bacteriológico tradicional. En este estudio se utilizó la PCR siguiendo las recomendaciones de otros investigadores; para la identificación de Cfv con el gen cstA y para Cff con el gen virB11 (Hum et al., 1997; Iraola et al., 2012), dado que los cebadores generados para la identificación de estos genes pueden detectar secuencias específicas de ADN de Cfv y Cff; con la PCR desarrollada permitió que se detectaran casos positivos de CGB en el área de estudio. Otro de los métodos que se ha utilizado es IFD; sin embargo, Ferreira en 2002 muestra que la técnica no distingue las subespecies de C. fetus.
En México, a partir de 1994 se exporta ganado bovino a los Estados Unidos de América, y el estado de Tamaulipas se ha caracterizado por tener uno de los mayores inventarios del país. En el ciclo 2020-2021 la exportación de ganado vacuno en pie fue de 30,896 cabezas a Estados Unidos según datos de la Secretaría de Agricultura y Desarrollo Rural (SADER). Considerando la necesidad de incrementar el número de animales para suplir la demanda de estos, es necesario contar con un diagnóstico zoosanitario de enfermedades que afectan parámetros reproductivos. El éxito del diagnóstico depende en gran medida de la toma de muestras; con base en informes previos informados por Silveira et al., 2018 y Delpiazzo et al., 2021.
La prevalencia de CGB es variable en diferentes áreas del mundo, y existen diversos factores que favorecen su transmisión (Hoque et al., 2021). En México poco se ha reportado la presencia de Campylobacter en bovinos; el único estudio encontrado en nuestro país fue realizado por Barajas en 2013 en zonas tropicales de México; mediante la prueba ELISA reveló una seroprevalencia de 21.3% en bovinos. El método de detección fue diferente y el muestreo fue menor; la frecuencia detectada en este estudio fue de 18.4%, lo que indica que la CGB está presente en los hatos bovinos de Tamaulipas. Cabe mencionar que el trabajo de Barajas se realizó con ganado del trópico húmedo del programa de extensionismo para ganaderos del Norte de Veracruz, estado colindante con Tamaulipas y con condiciones climáticas similares a la zona de estudio. El objetivo de este estudio fue detectar la presencia de C. fetus en toros de la zona centro del estado de Tamaulipas. De las 7 amplificaciones sugestivas de Cfv por PCR, 5 fueron evaluadas por secuenciación. Los resultados obtenidos en el análisis bioinformático confirman que la amplificación corresponde a una región del gen virB11 que es una secuencia de islas de patogenicidad presente solo en Cfv con 233 pb y ausente en Cff, lo que permite diferenciar estas dos subespecies por método molecular. Los resultados obtenidos en el presente trabajo son consistentes con lo publicado previamente por Iraola et al., 2016, por lo que la situación epidemiológica pudiera estar en riesgo por la diseminación de este patógeno por el uso de toros portadores con fines reproductivos.
CONCLUSION
En este estudio se detectó por PCR y se corroboró por secuenciación la presencia de Campylobacter fetus subsp. venerealis con una frecuencia del 18.4% de los toros analizados de la zona centro del estado de Tamaulipas, lo que representa el primer reporte en México de esta bacteria por métodos moleculares.











 texto en
texto en 




