Introducción
Xalapa, Veracruz, es una de las pocas ciudades de México que cuentan con un número significativo de áreas verdes urbanas y periurbanas (parques y jardines) dentro de la ciudad y sus alrededores (Capitanachi et al., 2004). El paisaje está formado por lomeríos e incluye parches de áreas perturbadas y preservadas de bosque de niebla mejor conocido como bosque mesófilo de montaña (Chacón et al., 1995). De acuerdo con Capitanachi et al. (2004), existen 112 áreas verdes en Xalapa. Sin embargo, solo siete de ellas (Santuario del Bosque de Niebla (SBN); Jardín Botánico Francisco Javier Clavijero (JBFJC); Lomas de San Roque; Parque Ecológico Cerro Macuiltépetl; Parque Natura; Parque Los Berros y Parque Los Tecajetes) se han mencionado en estudios taxonómicos sobre hongos, o sobre biodiversidad (Chacón et al., 1995; 2014; 2018(2019); Heredia et al., 2000; Chacón-Zapata y Tapia-Padilla, 2013; Bandala y Montoya, 2014; MacGregor-Fors et al., 2016; Tapia et al., 2017; Chacón-Zapata, 2019; Chacón y González, 2019). Referente a los hongos ascomicetos, hasta ahora solo se han citado algunos géneros y especies de Xylariales y Pezizales procedentes del SBN y del JBFJC (Chacón et al., 1995; Medel y Calonge, 2004; Medel et al., 2008; Chacón y González, 2019), por lo que el conocimiento sobre este grupo de hongos, incluyendo aquellos del orden Coronophorales, es todavía muy escaso.
El género Euacanthe Theiss. fue propuesto por Theissen (1917, en Subramanian y Sekar, 1990) para acomodar a Meliolopsis usambarensis Rehm. Sin embargo, Nannfeldt (1975) encontró que Sphaeria foveolata Berk. & M.A. Curtis era un nombre ya existente para este hongo, por lo que lo transfirió a Acanthonitschkea Speg., como Acanthonitschkea foveolata (Berk. & M.A. Curtis ex Berk.) Nannf. Posteriormente, Subramanian y Sekar (1990) reconocieron a Euacanthe y realizaron la combinación de A. foveolata, transfiriéndola a Euacanthe foveolata (Berk. & M.A. Curtis ex Berk.) Subram. & Sekar. No obstante, en la base de datos de Mycobank (Robert et al., 2005), se mantiene el registro de dos especies: E. usambarensis (Rhem) Theiss y E. foveolata, ambas adscritas a la familia Nitschkiaceae del orden Sordariales. En Index Fungorum (2021), Euacanthe es tratado como monotípico (E. usambarensis), toda vez que E. foveolata es considerada sinónimo de Acanthonitschkea foveolata. Sin embargo, Euacanthe se distingue de Acanthonitschkea por la presencia de cuerpos de expansión llamados quellkörper. Los cuerpos de quellkörper sirven para romper la pared del pseudotecio, propiciando con ello la apertura del ascoma y en consecuencia la liberación de las esporas (Mugambi y Huhndorf, 2010). La posición sistemática de Euacanthe ha sido tratada con caracteres morfológicos y moleculares. Estos últimos avalan sus cambios nomenclaturales y lo ubican actualmente como un género monotípico adscrito a la familia Scortechiniaceae, del orden Coronophorales (Huhndorf et al., 2004; Kirk et al., 2008; Mugambi y Huhndorf, 2010; Maharachchikumbura et al., 2016; Hyde et al., 2020), resolución que es aceptada en la presente contribución. Euacanthe se caracteriza por los ascomas superficiales colapsados, subículo presente con abundantes setas, ascas octosporadas y la presencia de cuerpos quellkörper (Subramanian y Sekar, 1990; Mugambi y Huhndorf, 2010).
Por otro lado, a pesar de la gran diversidad fúngica que ostenta México, el género Euacanthe no había sido registrado para el país, aunque sí otras especies adscritas a los Coronophorales, como Nitschkia grevillei (Rehm) Nannf. (Medel y Chacón, 1988), Acanthonitschkea argentinensis Speg. (San Martín González y Lavín, 1997), Calyculosphaeria calyculus (Mont.) Fitzp., C. tetraspora (Nannf.) F. San Martín, Scortechinia acanthostroma (Mont.) Sacc. & Berl., Spinulosphaeria thaxteri (Pat.) Sivan., Bertia didyma (Speg.) Mugambi & Huhndorf (como Thaxteria didyma (Speg.) Sacc.) y Fracchiaea heterogenea Sacc. (San Martín González, 1996; Raymundo et al., 2014). En esta investigación se describe una nueva especie de Euacanthe, con lo cual se eleva a dos, el número de especies descritas para este género de Coronophorales.
Materiales y Métodos
Recolección de ejemplares
El presente trabajo se basó en la revisión y estudio de ejemplares previamente depositados en la colección de hongos del herbario XAL, del Instituto de Ecología, A.C. (INECOL), y en material fresco recolectado entre 2013 y 2015. Las muestras estudiadas (G. Guzmán 24105 (XAL), 32780 (XAL), S. Chacón 4935 (XAL) y Tapia 2797 (XAL)) proceden de cuatro áreas verdes urbanas y periurbanas localizadas en la ciudad de Xalapa (Parque Ecológico “Cerro Macuiltépetl”, “Jardín de casa particular”, “Santuario del Bosque de Niebla” y “Zoncuantla”) (Fig. 1). Cada sitio se separa de los demás por una distancia entre 0.5 y 5 km, la altitud entre sitios varía de 1300 a 1586 m s.n.m., la precipitación promedio anual es de 1590 mm (Zolá, 1987). El clima es templado húmedo con lluvias la mayor parte del año; la temperatura promedio anual varía de 16 a 18 ºC. Algunos sitios incluyen parches conservados de bosque mesófilo de montaña con presencia de especies vegetales con afinidad templada de géneros como Carpinus L., Clethra L., Platanus L. y Quercus L., además de otros árboles y arbustos del sotobosque con afinidad tropical (Muñiz-Castro et al., 2006, Toledo-Aceves et al., 2014).
Estudio morfológico
Para la descripción de los ascomas se utilizó un microscopio estereoscópico (Zeiss Stemi DRC, Jena, Alemania). Además, se hicieron cortes a navaja de los ascomas y se colocaron en agua destilada e hidróxido de potasio (KOH) al 3% para su observación en el microscopio de campo claro (Zeiss 470801, Jena, Alemania), donde se tomaron medidas de ascas, ascosporas, setas, hifas del subículo, espesor del peridio y cuerpos quellkörper. Para una descripción más detallada se realizaron dibujos y se tomaron fotografías de algunas de las estructuras microscópicas más relevantes. Los ejemplares estudiados están depositados en la colección de hongos del herbario XAL.
Extracción de ADN, amplificación y secuenciación
Para la extracción de ADN, se separó un ascoma de los cuatro ejemplares usados para este estudio (G. Guzmán 24105 (XAL), 32780 (XAL), S. Chacón 4935 (XAL) y Tapia 2797 (XAL)). Cada ascoma se lavó repetidas veces con agua destilada antes de macerarse en un disruptor tisular (TissueLyser II, Qiagen, Valencia, EUA) por 45 segundos. La extracción de ADN se llevó a cabo utilizando el Kit NucleoSpin Plant II (Macherey-Nagel, Bethlehem, PA, EUA), siguiendo el protocolo del fabricante.
Se amplificaron tres regiones de genes nucleares, el 28S (codifica la subunidad grande del DNA ribosomal), el rpb2 (codifica la segunda subunidad grande de la RNA polimerasa II) y el tef1( (codifica el factor de elongación 1 alfa). Las reacciones de amplificación se realizaron en un termociclador Eppendorf (Mastercycler proS, Hamburgo, Alemania). La mezcla de reacción consistió en un volumen total de 25 µl que contenía entre 25-50 ng de ADN, 5 µl de buffer 5×, 0.2 mM dNTPs, 1.6 µM de ambos cebadores (directo e inverso), 2 mM MgCl2, 0.2 U de polimerasa Go Taq flexi DNA (Promega, Madison, Wisconsin, EUA) y agua destilada. Las condiciones de la amplificación se describen en el Cuadro 1.
Cuadro 1: Oligonucleótidos usados y condiciones de las reacciones de PCR que se usaron para amplificar Euacanthe renispora S. Chacón.
| Gen | Nombre | Referencia | Secuencia (5´-3´) | Hibridación y extensión de la reacción |
|---|---|---|---|---|
| 28S | LR0R | Vilgalys y Hester, 1990 | ACCCGCTGAACTTAAGC | 51 °C, 45 s |
| LR5 | TCCTGAGGGAAACTTCG | 72 °C, 1 min | ||
| rpb2 | RPB2-980F | Reeb et al., 2004 | TGYCCIGCIGARACICCHGARGG | 57 °C, 1 min |
| fRPB2-7cR | Liu et al., 1999 | CCCATRGCTTGYTTRCCCAT | 72 °C, 2 min | |
| tef1α | 983F | Sung et al., 2007 | GCYCCYGGHCAYCGTGAYTTYAT | 60 °C, 30 s |
| EF1-GY-R | Diseñado por el autor (DG) | TCRACGGAYTTGACTTCAGTGGT | 72 °C, 1 min |
Los productos de amplificación se corrieron en un gel de agarosa al 1.5% y se cortaron para su purificación con el kit GenElute Gel (Sigma-Aldrich, St. Louis, Missouri, EUA). La secuenciación se realizó con el kit ABI PRISM BigDye Terminator v. 3.1 (Applied Biosystems, Foster City, EUA), de acuerdo con las instrucciones del proveedor. Los productos de PCR se purificaron con isopropanol y se procesaron con el equipo ABI 310 Genetic Analyzer (Applied Biosystems, Foster City, California, EUA) del INECOL.
Las secuencias de las tres regiones génicas se editaron con el programa BioEdit v. 7.1.3.0 (Hall, 1999) y se compararon en la base de datos de nucleótidos (nr/nt) de GenBank (https://blast.ncbi.nlm.nih.gov/Blast.cgi) con el algoritmo blastn para conocer los taxa con afinidad en sus secuencias.
Después de identificar los taxones con mayor identidad, se descargaron todas las secuencias disponibles en GenBank del orden Coronophorales. Se ensamblaron tres matrices, una con secuencias del 28S; otra con las del rpb2 y la tercera con secuencias del tef1α. Estas matrices contenían secuencias principalmente de estudios realizados por Subramanian y Sekar (1990), Huhndorf et al. (2004) y Mugambi y Huhndorf (2010) dentro del orden Coronophorales (Cuadro 2). La alineación se realizó con Clustal W, dentro del programa BioEdit v. 7.1.3.0 (Hall, 1999).
Cuadro 2: Especies utilizadas en el análisis filogenético y número de acceso en GenBank. En negritas los números de acceso de Euacanthe renispora S. Chacón. 1/CBS: Central Bureau of Fungal Cultures; CPC: Culture collection of Pedro Crous; GKM: George K. Mugambi; IFRD: International Fungal Research & Development Centre; FMR: Facultat de Medicina i Ciències de la Salut; SMH: Sabine M. Huhndorf; ANM: Andrew N. Miller; F: Field Museum of Natural History; EA: National Museum of Kenya; MO: Missouri Botanical Garden.
| Especie1/(origen) | Números de acceso de GenBank | ||
|---|---|---|---|
| 28S | rpb2 | tef1α | |
| Acanthonitschkea argentinensis voucher SMH1395 | AY695259 | FJ968943 | FJ969042 |
| Acanthonitschkea tristis strain SMH4723 | FJ968949 | FJ969043 | |
| Bahusakala longispora CBS 544 84 from TYPE material | NG064138 | ||
| Bertia didyma strain SMH4256 | FJ968950 | ||
| Bertia didyma strain SMH4719 | FJ968958 | ||
| Bertia gigantospora strain GKM1136 | FJ969008 | ||
| Bertia moriformis | AY780151 | ||
| Bertia moriformis strain SMH5169 | FJ968951 | ||
| Bertia moriformis voucher SMH3344 | AY695261 | ||
| Bertia moriformis voucher SMH4320 | AY695260 | ||
| Bertia multiseptata strain SMH1153 | FJ968953 | ||
| Bertia multiseptata strain SMH3127 | FJ968952 | ||
| Bertia ngongensis EA GKM1239 from TYPE material | NG058634 | ||
| Bertia ngongensis strain GKM1239 | FJ968954 | ||
| Bertia orbis strain GKM1259 | FJ968959 | ||
| Bertia orbis strain GKM1271 | FJ968955 | FJ969009 | |
| Bertia orbis strain SMH4035 | FJ968960 | ||
| Bertia orbis strain SMH4557 | GQ184146 | ||
| Bertia orbis USJ SMH4557 from TYPE material | NG058636 | ||
| Bertia sinensis strain SMH4034 | FJ968961 | ||
| Bertia triseptata F SMH1525 from TYPE material | NG058635 | ||
| Bertia triseptata strain SMH1525 | FJ968957 | ||
| Bertia triseptata strain SMH4379 | FJ968956 | ||
| Bertia tropicalis F SMH1707 from TYPE material | NG058626 | ||
| Bertia tropicalis strain SMH3132 | FJ968962 | ||
| Bertia tropicalis strain SMH4046 | FJ968963 | ||
| Bertia tropicalis voucher SMH1707 | AY695262 | ||
| Bertia tropicalis voucher SMH3513 | AY695263 | ||
| Biciliospora velutina strain GKM1268 | FJ968964 | FJ968932 | FJ969018 |
| Chaetosphaerella fusca strain ANM53 | FJ968965 | ||
| Chaetosphaerella fusca strain ANM79 | FJ968966 | ||
| Chaetosphaerella fusca strain GKML124N | FJ968967 | FJ969002 | |
| Chaetosphaerella phaeostroma | AY346274 | AY780172 | |
| Chaetosphaerella phaeostroma strain SMH4585 | FJ969003 | ||
| Chaetosphaerella phaeostroma voucher SMH4257 | AY695264 | FJ968940 | FJ969004 |
| Coronophora myricoides IFRD 9201 from TYPE material | NG058972 | ||
| Coronophora myricoides strain IFRD 9201 | KX856174 | ||
| Coronophorella chaetomioides strain GKM1099 | FJ968969 | FJ968922 | FJ969034 |
| Coronophora gregaria strain ANM1555 | FJ968938 | FJ969007 | |
| Crassochaeta nigrita voucher SMH1667 | AY695265 | ||
| Crassochaeta nigrita voucher SMH2931 | AY695266 | ||
| Cryptosphaerella celata EA GKM1231 from TYPE material | NG058772 | ||
| Cryptosphaerella celata strain GKM1231 | FJ968975 | FJ968929 | FJ969035 |
| Cryptosphaerella costaricensis F MO2111 from TYPE material | NG058769 | ||
| Cryptosphaerella costaricensis strain MO2111 | FJ968971 | FJ969028 | |
| Cryptosphaerella cylindriformis EA GKM434N from TYPE material | NG058770 | ||
| Cryptosphaerella cylindriformis strain GKM1042 | FJ968973 | FJ968918 | FJ969032 |
| Cryptosphaerella cylindriformis strain GKM1075 | FJ968920 | FJ969030 | |
| Cryptosphaerella cylindriformis strain GKM1187 | GQ217531 | FJ968925 | FJ969033 |
| Cryptosphaerella cylindriformis strain GKM434N | FJ968972 | FJ968934 | FJ969031 |
| Cryptosphaerella elliptica F SMH4722 from TYPE material | NG058771 | ||
| Cryptosphaerella elliptica strain SMH4722 | FJ968974 | FJ968944 | FJ969029 |
| Cryptosphaerella globosa strain 396N | FJ968976 | ||
| Cryptosphaerella globosa strain GKM414N | FJ969037 | ||
| Cryptosphaerella globosa strain GKM437N | FJ969038 | ||
| Cryptosphaerella globosa strain GKM469N | FJ968977 | FJ968935 | |
| Cryptosphaerella globosa strain GKM471N | FJ969036 | ||
| Cryptosphaerella malindensis EA GKM1150 from TYPE material | NG058768 | ||
| Cryptosphaerella malindensis strain GKM1150 | FJ968970 | FJ968923 | FJ969027 |
| Euacanthe foveolata strain GKM1221 | FJ968978 | FJ968927 | FJ969026 |
| Euacanthe foveolata voucher SMH4408 | AY695267 | ||
| Euacanthe renispora S. Chacón | MW349938 | MW349939 | MW349940 |
| Fracchiaea broomeana strain SMH347 | FJ968979 | ||
| Fracchiaea broomeana strain GKM1071 | FJ968919 | FJ969040 | |
| Fracchiaea broomeana strain SMH347 | FJ969041 | ||
| Fracchiaea broomeana voucher SMH2809 | AY695268 | FJ968942 | FJ969039 |
| Fracchiaea lunata strain GKM1089 | FJ968921 | ||
| Fracchiaea lunata strain GKM1250 | FJ969005 | ||
| Gaillardiella pezizoides strain GKM1144 | FJ968980 | ||
| Gaillardiella pezizoides strain GKM1245 | FJ968981 | FJ969006 | |
| Nectria cinnabarina strain GJS 89 107 | AF543785 | ||
| Nectria cinnabarina strain GJS 91 111 | AF545567 | ||
| Neofracchiaea callista voucher SMH2689 | AY695269 | FJ968941 | FJ969020 |
| Neotrotteria pulchella strain GKM1255 | FJ968982 | ||
| Nitschkia calyculus strain SMH918 | FJ968983 | ||
| Nitschkia grevillei | AY346294 | ||
| Nitschkia menicoidea voucher SMH1523 | AY695270 | ||
| Nitschkia meniscoidea F SMH1523 from TYPE material | NG060247 | ||
| Nitschkia pezizoidea voucher SMH4409 | AY695271 | ||
| Nitschkia tetraspora strain GKML148N | FJ968987 | FJ968936 | FJ969011 |
| Nitschkia tetraspora strain GKML213N | FJ968985 | ||
| Nitschkia tetraspora strain SMH2469 2 | FJ968986 | ||
| Nitschkia tetraspora strain SMH4787 | FJ968984 | FJ969010 | |
| Parasympodiella elongata culture CBS 124768 | GQ303311 | ||
| Parasympodiella elongata culture CBS 522 93 | GQ303314 | ||
| Parasympodiella elongata culture CPC 13288 | GQ303312 | ||
| Parasympodiella elongata culture CPC 13498 | GQ303313 | ||
| Parasympodiella elongata from TYPE material | NG057829 | ||
| Parasympodiella elongata strain CBS 522 93 | MH874088 | ||
| Parasympodiella eucalypti culture CBS 124767 | GQ303315 | ||
| Parasympodiella eucalypti from TYPE material | NG057830 | ||
| Parasympodiella lauri CBS 138108 from TYPE material | NG058461 | ||
| Parasympodiella lauri strain FMR 13132 | KY853518 | ||
| Parasympodiella laxa culture CBS 102698 | GQ303316 | ||
| Parasympodiella longispora strain CBS 544 84 | MH873476 | ||
| Pseudocatenomycopsis rothmanniae strain CPC 22733 | KF777237 | ||
| Scortechinia acanthostroma strain GKML163N | FJ968991 | FJ969015 | |
| Scortechinia acanthostroma strain SMH1143 | FJ968988 | FJ968948 | FJ969012 |
| Scortechinia acanthostroma strain SMH1164 | FJ968989 | FJ968924 | FJ969014 |
| Scortechinia acanthostroma strain SMH5313 | FJ968990 | FJ969013 | |
| Scortechinia conferta voucher SMH2648 | AY695272 | ||
| Scortechinia diminuspora F SMH4763 from TYPE material | NG059922 | ||
| Scortechinia diminuspora strain SMH4763 | FJ968992 | ||
| Scortechiniella similis strain SMH2006 | FJ968994 | FJ968945 | FJ969019 |
| Scortechiniellopsis leonensis strain GKM1269 | FJ968993 | FJ968933 | FJ969021 |
| Spinulosphaeria nuda SMH 1952 from TYPE material | NG059923 | ||
| Spinulosphaeria nuda strain SMH1952 | FJ968995 | ||
| Tympanopsis confertula strain GKM1242 | FJ968997 | FJ968930 | FJ969023 |
| Tympanopsis confertula strain SMH1567 | FJ969001 | FJ968939 | FJ969025 |
| Tympanopsis confertula strain SMH4841 | FJ968998 | FJ968946 | FJ969024 |
| Tympanopsis confertula strain SMH2648 | FJ969022 | ||
| Tympanopsis uniseriata strain GKM1203 | FJ968999 | FJ968926 | FJ969016 |
| Tympanopsis uniseriata strain GKM1228 | FJ969000 | FJ968928 | FJ969017 |
| Nectria cinnabarina strain GJS 89 107 | AF543785 | ||
| Nectria cinnabarina strain GJS 91 111 | AF545567 | ||
| Nectriopsis violacea | AF193242 | ||
Análisis filogenéticos
Los análisis se realizaron con inferencia filogenética bayesiana (BPI) usando MrBayes v. 3.1.2 (Huelsenbeck y Ronquist, 2001; Ronquist y Huelsenbeck, 2003; Altekar et al., 2004). El modelo de sustitución se determinó con PartitionFinder 2 (Lanfear et al., 2017) para las tres matrices. Se corrieron cuatro cadenas de Markov (MCMC) independientes con dos millones de generaciones y 25% de “burn-in” cada 100 generaciones antes de calcular el árbol de consenso de mayoría y las probabilidades posteriores (PP) para los clados. Para enraizar el árbol se usaron especies de Hypocreales de acuerdo con Mugambi y Huhndorf (2010).
El apoyo estadístico de los nodos se determinó mediante un análisis de jackknife (JK) en PAUP v. 4 (Swofford, 2002) ejecutando 1000 réplicas con adición simple de secuencias, rearreglo de ramas TBR y 35% de caracteres eliminados. Adicionalmente, se realizó un “fast bootstrap” (BS) con 1000 réplicas y 10 búsquedas por réplica (Stamatakis, 2006; Boc et al., 2012) con el algoritmo “rapid hill-climbing”, usando RAxML enTrex-online (http://www.trex.uqam.ca/index.php?action=raxml). Los árboles filogenéticos se dibujaron con FigTree v. 1.4 (Rambaut, 2014).
Resultados
Tres de los cuatro ejemplares de la especie nueva (Euacanthe renispora) estaban contaminados con Aspergillus Micheli., por lo que solo fue posible generar secuencias para el ejemplar Tapia 2797 (XAL). La comparación con blastn indicó que la especie con mayor identidad fue Euacanthe foveolata (= Acanthonitschkea foveolata) del orden Coronophorales, con únicamente cuatro secuencias disponibles en GenBank; dos para el 28S y una para el rpb2 y tef1(, respectivamente. Para el orden existen solo 208 al 6 de noviembre del 2020, las que se usaron para conocer la posición de Euacanthe renispora dentro del orden. De las 208 secuencias, se seleccionaron los genes que tenían mayor representatividad de especies y se eliminaron las de taxones no identificados. Después de la alineación, la matriz del 28S consistió en 99 terminales y 947 caracteres; la del rpb2 en 32 terminales y 797 caracteres y la del tef1( en 44 terminales y 671 caracteres. El modelo de evolución de las secuencias para las tres matrices fue GTR (rates=invgamma, nst=6).
Los análisis filogenéticos con las matrices de los genes 28S y rpb2 mostraron que Euacanthe renispora forma un grupo monofilético con E. foveolata con valores de apoyo altos (Fig. 2, 3). Sin embargo, el análisis con la matriz del gen tef1( no agrupó a estas dos especies. Se observó que, en general, el tef1( tiene menor resolución filogenética que el 28S y el rpb2 (Fig. 3), pero evidenció que E. renispora es diferente al resto de las especies en ese clado, incluyendo un ejemplar de E. foveolata (Fig. 4). Adicionalmente, las cinco familias reconocidas de los Coronophorales (Mugambi y Huhndorf, 2010) se recuperaron monofiléticas en todos los análisis. Las excepciones fueron Nitschkia pezizoidea (Pat.) Kuntze (AY695271), que resultó parafilética, al igual que Fracchiaea broomeana (Berk.) Petch (AY695268, FJ968979), que se asocia a la familia Nitschkiaceae y Spinulosphaeria nuda Mugambi & Huhndorf (FJ968995, NG 059923), que se ha agrupado en la familia Bertiaceae (Subramanian y Sekar, 1990).
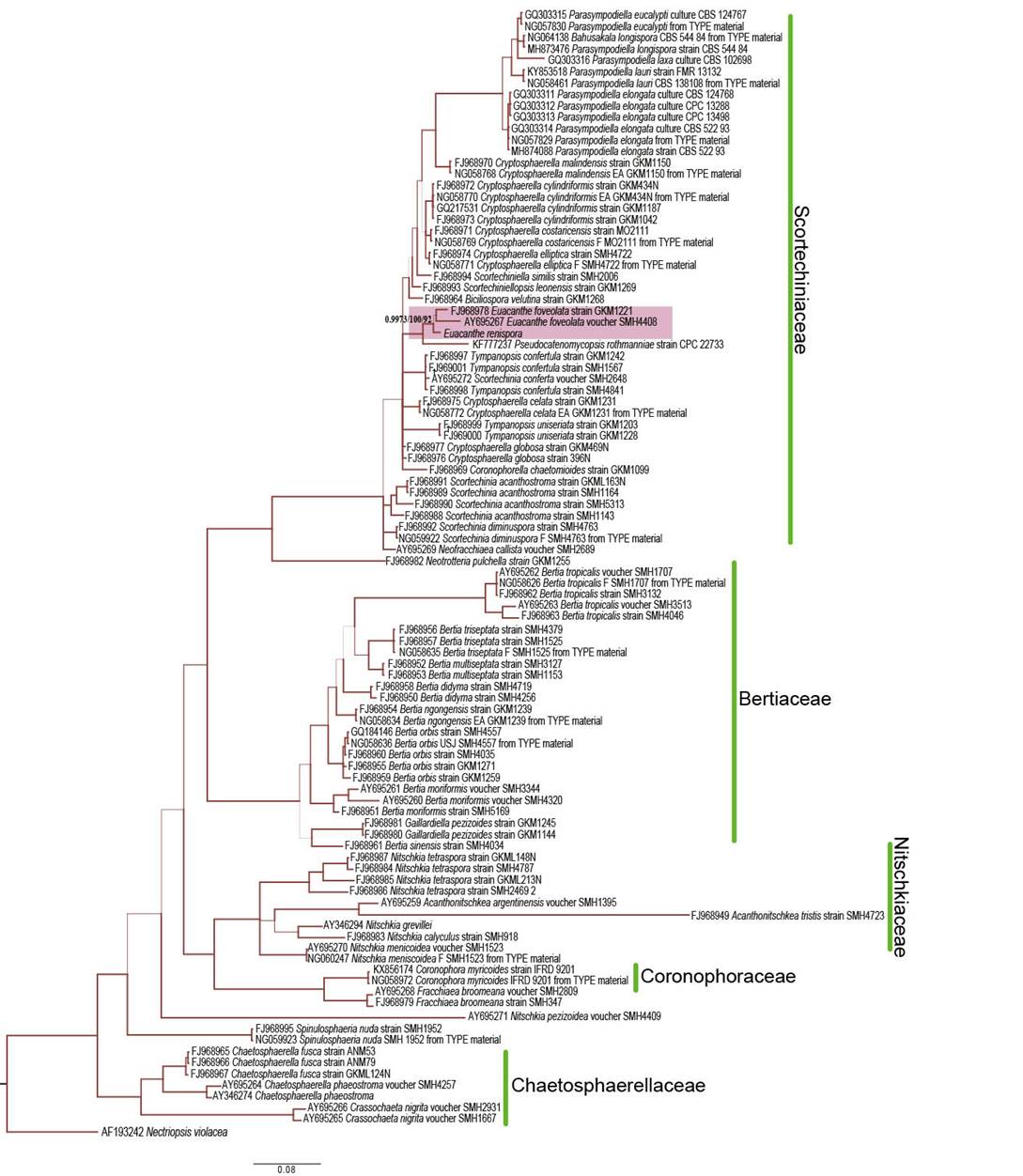
Figura 2: Filograma del árbol de consenso de mayoría bayesiana de 50%, resultado del análisis de las secuencias de una región del 28S. En el cuadro rosa se muestra la posición de Euacanthe renispora S. Chacón con los valores de apoyo (PP/JK/BS). El grosor de las ramas indica el nivel de apoyo de las PP. A la derecha del nombre de la especie el número de acceso a GenBank, a la izquierda su origen. CBS: Central Bureau of Fungal Cultures; CPC: Culture collection of Pedro Crous; GKM: George K. Mugambi; IFRD: International Fungal Research & Development Centre; FMR: Facultat de Medicina i Ciències de la Salut; SMH: Sabine M. Huhndorf; ANM: Andrew N. Miller; F: Field Museum of Natural History; EA: National Museum of Kenya; MO: Missouri Botanical Garden.
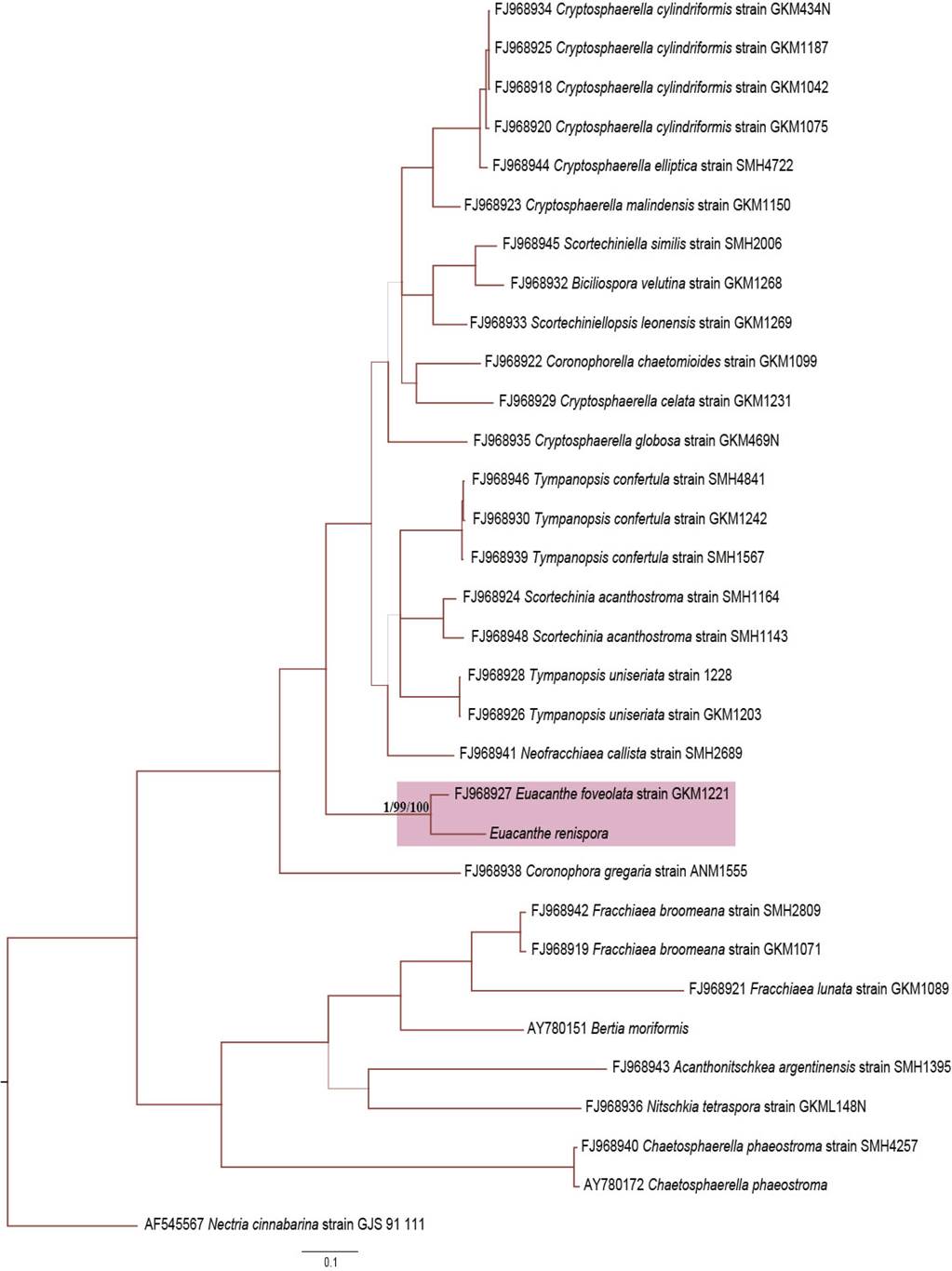
Figura 3: Filograma del árbol de consenso de mayoría bayesiana de 50% resultado del análisis de las secuencias de una región del rpb2. En el cuadro rosa se muestra la posición de Euacanthe renispora S. Chacón con los valores de apoyo (PP/JK/BS). El grosor de las ramas indica el nivel de apoyo de las PP. A la derecha del nombre de la especie el número de acceso a GenBank, a la izquierda su origen. GKM: George K. Mugambi; SMH: Sabine M. Huhndorf; ANM: Andrew N. Miller; GJS: Gary J. Samuels.

Figura 4: Filograma del árbol de consenso de mayoría bayesiana de 50% resultado del análisis de las secuencias de una región del tef1α. En el cuadro rosa se muestra la posición de Euacanthe renispora S. Chacón con los valores de apoyo (PP/JK/BS). El grosor de las ramas indica el nivel de apoyo de las PP. A la derecha del nombre de la especie el número de acceso a GenBank, a la izquierda su origen. GKM: George K. Mugambi; SMH: Sabine M. Huhndorf; ANM: Andrew N. Miller; MO: Missouri Botanical Garden; GJS: Gary J. Samuels.
Taxonomía
Euacanthe renispora S. Chacón, sp. nov. Figs. 5-7. MycoBank No.: MB 838214. TIPO: MÉXICO. Veracruz, municipio Xalapa, Parque ecológico “Cerro Macuiltépetl”, 1520 m, 19°32′54″N, 96°55′09″O, sobre ramas de troncos caídos. 17.III.2013, F. Tapia 2797 (holotipo: XAL!).
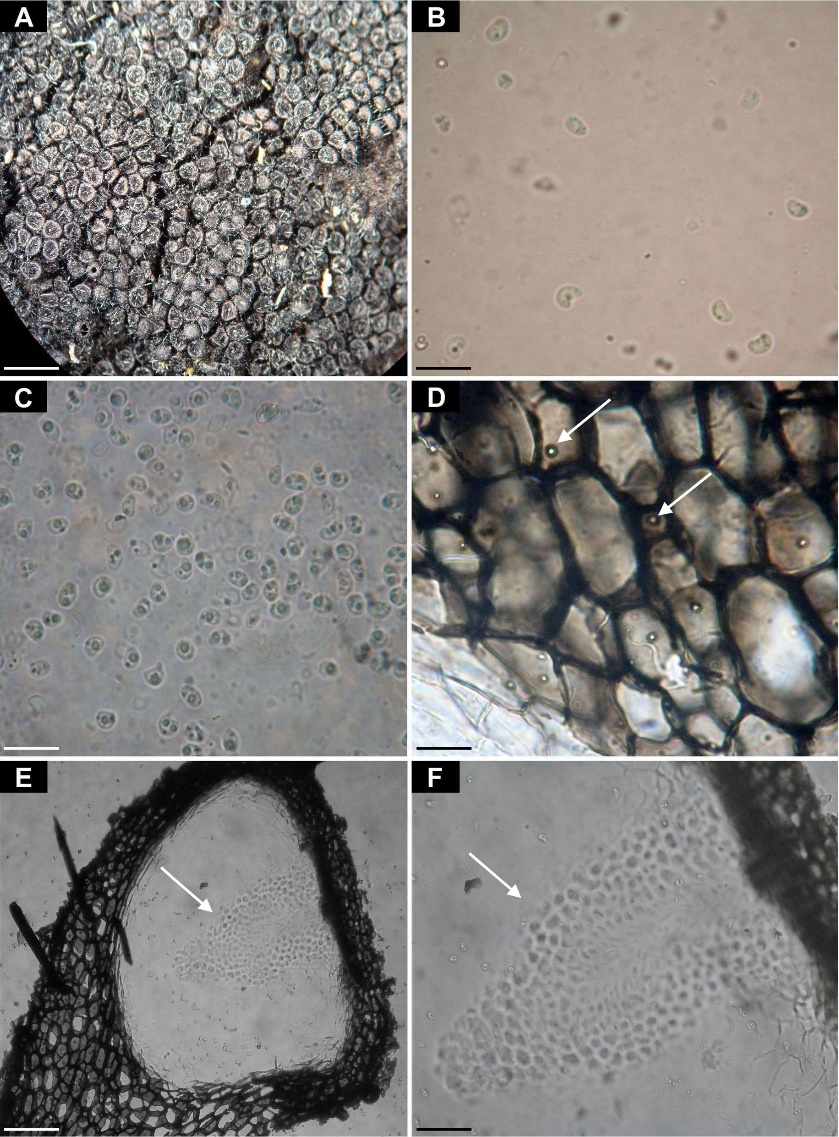
Figura 5: Euacanthe renispora S. Chacón. A. ascomas; B, C. esporas reniformes; D. células de la pared del ascoma, las flechas muestran los poros de Munk; E, F. sección longitudinal de un ascoma, las flechas muestran los cuerpos quellkörper. Escala, A=1.5 mm; B, C=14 µm; D=10 µm; E=90 µm; F=60 µm. Edición de imagen por Manuel Díaz.
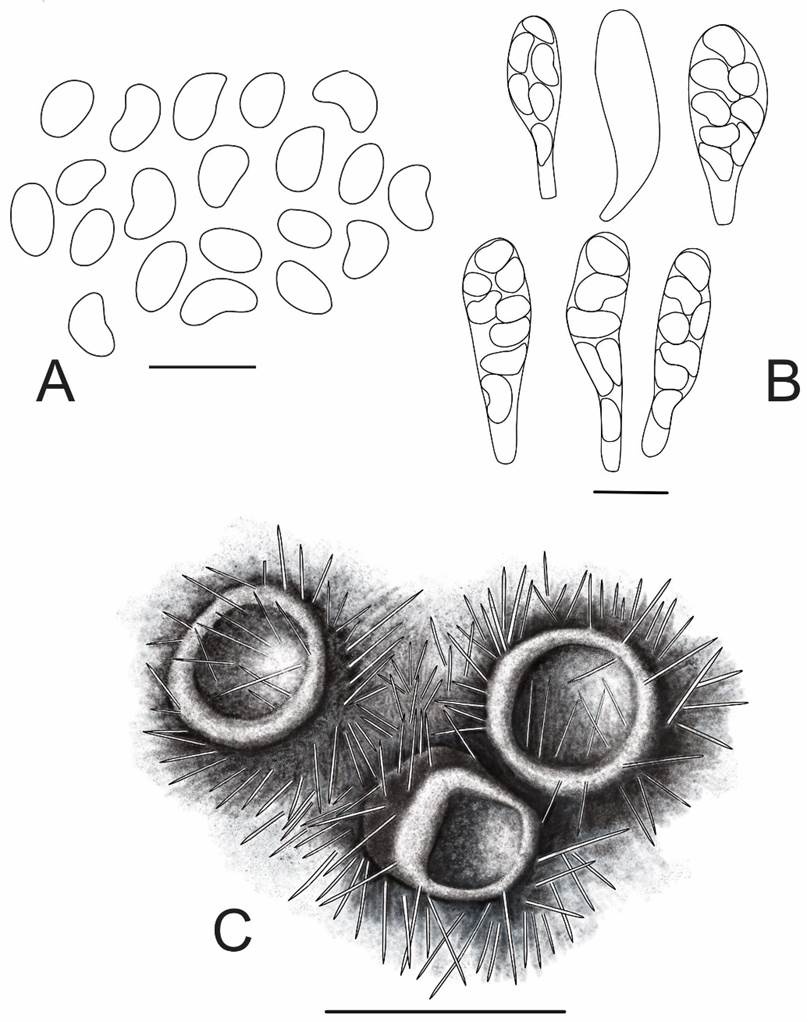
Figura 6: Euacanthe renispora S. Chacón. A. esporas; B. ascas con ascosporas; C. ascomas. Escala, A=7 µm; B=9 µm; C=0.75 mm. Dibujo por Manuel Escamilla.
Euacanthe renispora is closely related to Euacanthe foveolata (= Acanthonitschkea foveolata), but with smaller ascospores which are strictly reniform. Ascomata turbinate, collabent when dry, non-ostiolate, gregarious, seated on a dense subiculum developed on the surface of the bark, quellkörper bodies present. Asci 20-35 × 10-12 µm. Ascospores often reniform to occasionally obovate, hyaline, smooth, (4-)4.5-6(-6.5) × (3-)3.5-4 µm.
Ascoma carente de ostiolo, turbinado, colapsado cuando seco, gregario, asentado sobre un subículo denso, desarrollado sobre la superficie de la corteza, 500-600 µm de altura, 400-500 µm de ancho, superficie negra con abundantes setas; peridio 70-80 µm de ancho, constituido por células alargadas pseudoparenquimatosas formando cadenas, externamente de color oscuro a color marrón a marrón pálido al interior, 1-3 poros de Munk; hifas del subículo ramificadas, septadas, 5-8 µm diámetro, color café pálido; setas presentes en el ascoma y subículo 175-350 × 10-20 µm, marrón rojizo oscuro, anchas en la base, adelgazándose hacia la punta; quellkörper presentes; ascas 20-35 × 10-12 µm, claviformes con un pedúnculo corto, pared delgada, octosporadas, finalmente evanescentes; ascosporas reniformes a ocasionalmente aovadas, hialinas, lisas, (4-)4.5-6(-6.5) × (3-)3.5-4 µm; paráfisis no observadas.
Notas taxonómicas: Euacanthe renispora es parecida a E. foveolata, citado por Nannfeldt (1975) como Acanthonitschkea foveolata; sin embargo, esta última se diferencia de la primera porque presenta esporas ovoides o elipsoides subfusiformes, casi rectas y un poco más grandes (5-8 × 2-3 µm). Subramanian y Sekar (1990) citaron para E. foveolata esporas más pequeñas de 5-6.5 × 3.0-3.5 µm, elípticas a reniformes, características que lo asemejan al material mexicano. Sin embargo, debido a que el nombre válido para Euacanthe foveolata es Akanthonitschkea foveolata, la ausencia de cuerpos quellkörper en las especies de Acanthonitschkea las separan. Por otro lado, al revisar el material tipo de Acanthonitschkea amarkantakensisPatel, A.K. Pandey & R.C. Rajak, citada de India por Patel et al. (2003), se observó la presencia de cuerpos quellkörper, característica típica de Euacanthe y de otros géneros de Coronophorales, pero no de Acanthonitschkea (Subramanian y Sekar, 1990; Mugambi y Huhndorf, 2010).
Etimología: el nombre específico renispora hace referencia a las esporas de forma reniforme.
Ejemplares examinados: MÉXICO. Veracruz, municipio Coatepec, Zoncuantla, en el jardín de una casa, 19°29′59″N, 96°56′38″O, 1270 m, corteza de ramas caídas, 19.I.1999, G. Guzmán 32780 (XAL). Municipio Xalapa, jardín de una casa, 19°31′17″N, 96°56′19″O, sobre corteza de ramas caídas, 08.X.1983, G. Guzmán 24105 (XAL); Jardín Botánico Francisco Javier Clavijero, km 2.5 antigua carretera Xalapa - Coatepec, 19°29′00″N, 97°09′00″O, 1300 m, sobre corteza de ramas caídas, 07.IX.1995, S. Chacón 4935 (XAL).
Ejemplar examinado de Acanthonitschkea amarkantakensis: INDIA. Amarkantak, Madhya Pradesh, Shahdol, III.1991, sobre Psidium guajava L., leg. U.S. Patel, IMI 354054 (holotipo: K!).
Discusión y Conclusiones
Los estudios sobre hongos, y en particular de los ascomicetos, procedentes de áreas verdes urbanas (parques y jardines) de Xalapa son muy escasos; no obstante, en contribuciones recientes se reconocieron algunos nuevos registros y especies nuevas (Tapia et al., 2017; Chacón et al., 2018(2019); Chacón-Zapata, 2019; Chacón y González, 2019).
Hasta ahora, el género Euacanthe era considerado monotípico. Sin embargo, con la inclusión de E. renispora procedente de distintas localidades de Xalapa y alrededores, se sugiere una mayor distribución y abundancia de las especies en el bosque mesófilo de montaña de la región. Se observó variación en el tamaño y forma de las esporas en los cuatro ejemplares colectados. Los ejemplares Chacón 4935 (XAL) y Guzmán 32767 (XAL), 32780 (XAL) presentan esporas de 4.5-6(-6.5) × (3-)3.5-4 µm, ovoides, elipsoides o reniformes; mientras que en el ejemplar de Tapia 2797 (XAL) (Tipo) se encontraron esporas ligeramente más grandes, 5.5-7(-9) × 3.5-4 µm, y regularmente reniformes. A pesar de esto, todas las muestras mexicanas se incluyen como parte del material estudiado de Euacanthe renispora, constituyendo la primera especie de Euacanthe registrada en México y la segunda para el género. Sin embargo, para dilucidar la abundancia y diversidad de E. renispora en las áreas verdes urbanas y periurbanas de Xalapa, es necesario realizar nuevas recolecciones y hacer análisis adicionales. Particularmente porque, salvo para el ejemplar Tapia 2797 (XAL) (Tipo), de los tres ejemplares restantes no se lograron obtener secuencias puras, debido a que las secuencias de estas muestras resultaron estar contaminadas con Aspergillus sp.
La filogenia de los Coronophorales con las secuencias del 28S mostró algunas diferencias en la ubicación de los géneros a la obtenida por Mugambi y Huhndorf (2010), como la posición de las especies Nitschkia pezizoidea, Fracchiaea broomeana y Spinulosphaeria nuda. No obstante, Nitschkia G.H. Otth ex P. Karst. resultó en un clado monofilético junto con Acanthonitschkea en otro análisis, pero su relación no estuvo apoyada (Mugambi y Huhndorf, 2010). Por otro lado, el género Fracchiaea Sacc., aunque se ha asociado con la familia Nitschkiaceae, actualmente se considera polifilético; en nuestro análisis resultó formando un clado con dos ejemplares de la familia Coronophoraceae. Adicionalmente, la pertenencia del género Spinulosphaeria Sivan. dentro de los Coronophorales ha sido fuertemente debatida (Mugambi y Huhndorf, 2010). Por lo tanto, el análisis de estos taxa requiere un mayor escrutinio. Finalmente, debido a la presencia de cuerpos quellkörper en el material tipo de A. amarkantakensis, se sugiere a futuro transferir esta especie al género Euacanthe.











 nueva página del texto (beta)
nueva página del texto (beta)





