Desde hace mucho tiempo, los humanos han contado con las herramientas necesarias para hacer de este mundo un lugar mejor. Ahí donde el amor, la compasión, el altruismo y la justicia han fracasado, la manipulación genética no triunfará.
Gina Maranto, La búsqueda de la perfección: El camino para engendrar mejores seres humanos
Hoy en “Desde la trinchera de las ciencias básicas” reseñaré un tema que ha ganado tanta prominencia que seguramente en poco tiempo será objeto de un Premio Nobel; por lo pronto, acaba de ganar el Premio Príncipe de Asturias 2015 que otorga España (http://elpais.com/elpais/2015/05/28/ciencia/1432807151_067613.html). El tema al que me refiero tiene un críptico nombre derivado de sus orígenes bacterianos: clustered regularly interspaced short palindromic repeat (repeticiones palindrómicas cortas agrupadas a intervalos regulares) o, simplemente, CRISPR. Este nombre se refiere a un locus del cromosoma bacteriano en donde residen unos genes que están a la altura del fuego de Prometeo, pues con ellos se ha creado una poderosa herramienta que permite manipular el ácido desoxirribonucleico (ADN o DNA, por sus siglas en inglés) de cualquier organismo viviente sobre nuestro planeta. El nombre CRISPR identifica ahora a una técnica o herramienta con la que se logran cambios específicos en los genomas y que ha funcionado exitosamente en diversos organismos, incluyendo recientemente cigotos humanos1,2. El CRISPR tuvo sus orígenes en investigaciones de ciencia básica que tal vez a algunos les hubiesen parecido ociosas, oscuras e irrelevantes. Al estudiar ciertos genes bacterianos enigmáticos, se encontró que el locus CRISPR aloja los engranajes de un sistema de defensa que, funcionalmente hablando, equivale a la “inmunidad adquirida” en animales. Es decir, que los “sencillos” organismos procariontes también tienen manera de “inmunizarse” contra reinfecciones por bacteriófagos con los que ya habían estado en contacto3. Esta defensa está mediada por los genes del locus CRISPR, el cual está compuesto por el precursor de un ácido ribonucleico (ARN o RNA, por sus siglas en inglés) largo que es procesado en sus componentes maduros funcionales, los ARN cortos llamados crARN y tracrARN y por genes asociados “Cas” (CRISPR-associated).
El locus CRISPR funciona como un “archivo” de segmentos de ADN tomados del genoma de los fagos que previamente habían infectado a la bacteria. Los genes asociados (Cas) son fundamentales en su operación pues generan las proteínas involucradas en la creación del archivo. Sin embargo, la estrella del elenco es la Cas9 (CRISPR-associated 9), la endonucleasa que destruye al ADN del bacteriófago reinfectante, aunque ella por sí sola sea incapaz de actuar. La especificidad del sistema la proporcionan los crARN que reconocen, por apareamiento de bases, a las secuencias nucleotídicas del ADN de un bacteriófago reinfectante. Junto con el tracrARN, cada crARN apareado a su ADN blanco, forma un complejo con la Cas9 que corta al ADN blanco4,5.
La forma activa de la endonucleasa Cas9, solo existe cuando está en un complejo con el ADN, su crARN y un tracrARN, de ahí que no corta otros ADN de manera aleatoria. El esquema de la figura 1, enfatiza que los ácidos nucleicos de este complejo se hibridan por complementariedad de bases, lo cual explica la especificidad del sitio de corte. Este corte ocurre precisamente dentro de las bases apareadas en el híbrido ADN-ARN y en la hebra complementaria al ADN apareado. De ahí que en el complejo activo, la endonucleasa Cas9 (contorno azul en las figuras 1 y 2) corta ambas hebras del ADN blanco inhabilitando al fago reinfectante. Esta situación, que ocurre en la naturaleza, ha sido simplificada y adaptada para su uso en prácticamente cualquier célula viviente gracias al trabajo de Jennifer Doudna y Emmanuelle Charpentier 6, ganadoras del premio Princesa de Asturias. Ellas diseñaron y conjuntaron en un solo ARN, llamado sgARN (single guide RNA, o ARN guía), las funciones de los crARN y tracrARN.
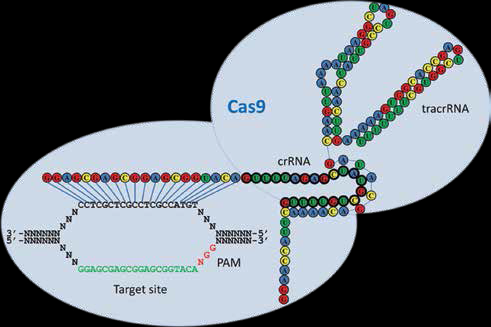
Figura 1 Esquema de un complejo natural del sistema CRISPR-Cas9 sobre un blanco de ADN (target site). Nótese que a pesar de que solo una hebra del ADN se aparea con el crARN, el corte del blanco ocurre en las 2 hebras. El tracrARN, que no es requerido para identificar al blanco, sirve para ensamblar al híbrido crARN-ADN en el sitio activo de la Cas9. (Tomada de Hwang WY, et al. Nature Biotech. 2013;31:227 9).
El sgARN dirige a la Cas9 hacia su blanco, con la misma precisión que lo harían los crARN y tracrARN de la bacteria4,6. Para obtener un sgARN con especificidad contra un blanco genómico (ADN) de elección, simplemente hay que diseñarlos con complementariedad (según las reglas de Watson y Crick) de bases (figura 2). Al introducir el sgARN y la Cas9 al núcleo de un eucarionte donde resida el ADN blanco, este será cortado en ambas hebras, generando un double stranded break (DSB). Usualmente este tipo de cortes son reparados, de manera imprecisa, por un sistema denominado non-homologous end-joining (NHEJ). La inherente falta de precisión del NHEJ ocasiona que, durante la reparación, el ADN sufra inserciones o deleciones de bases (indels) alrededor del DBS. De esta manera, el ADN blanco sufre mutaciones que, dependiendo de su naturaleza, ocasionan la pérdida de función del ADN blanco, por ejemplo, por la pérdida del marco de traducción en un exón codificante (ver ejemplo en figura 3).
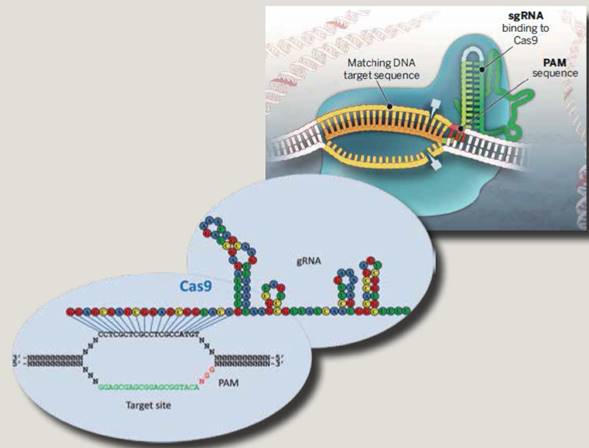
Figura 2 Esquemas que ilustran como un complejo del sistema CRISPR-Cas9 también puede formarse sobre un blanco de ADN (target site) usando un solo ARN, el sgARN o ARN guía, que en un solo ARN combina las 2 funciones del crARN y del tracrARN. Este descubrimiento simplificó el uso de CRISPR, pues hace posible apuntar a un blanco genómico introduciendo su sgARN complementario y a la Cas9 o a su mARN. (Tomadas Hwang WY, et al. Nature Biotech. 2013;31:227-9 y Doudna JA, Charpentier E. Science. 2014;346:1077).
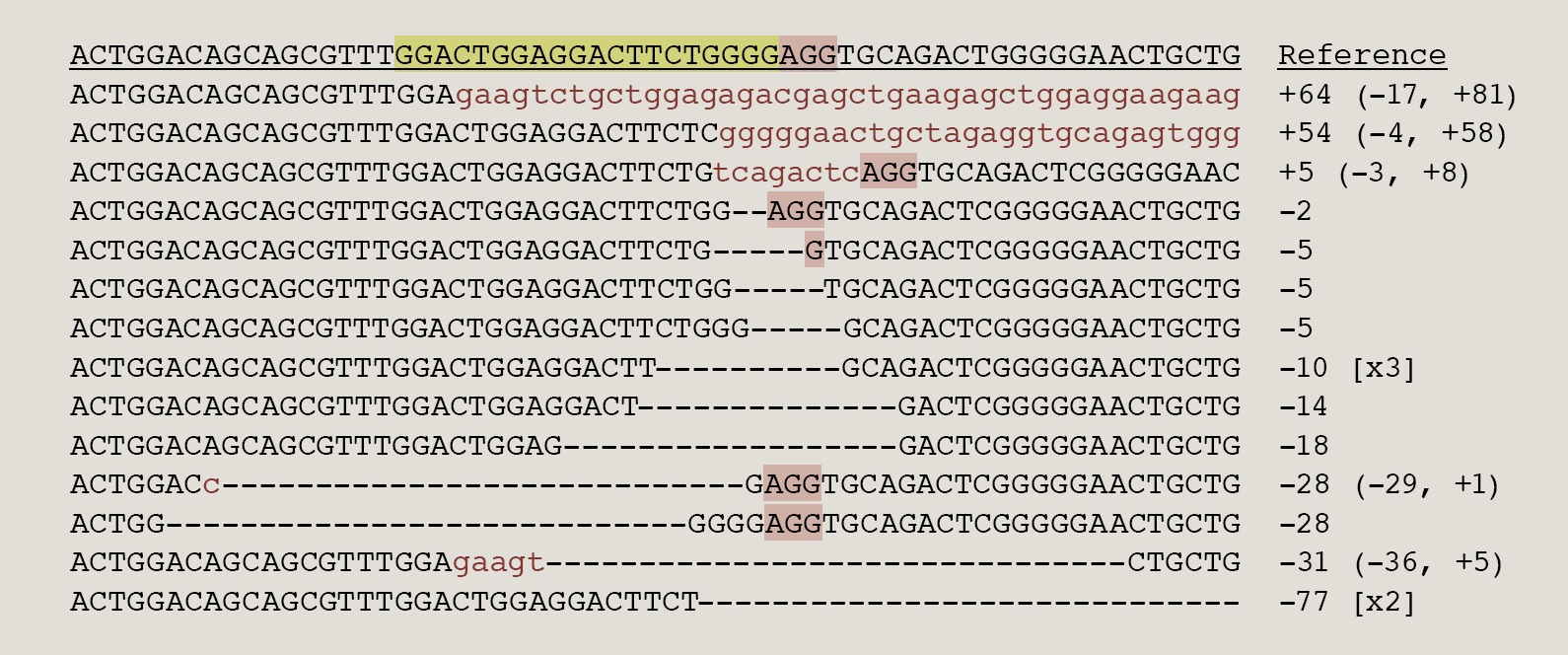
Figura 3 Las secuencias nucleotídicas alrededor del sitio blanco del sgARN para la tirosinasa usado en el experimento de la figura 4b, muestran los distintos tipos de indels (inserciones o deleciones) obtenidos. La línea superior (reference) es la secuencia silvestre y en sombreado gris y rosa se destaca la secuencia de apareamiento con el sgARN. Nótese la gran variedad de mutaciones afectan al gene silvestre, las bases rojas minúsculas son las inserciones y las deleciones se indican como guiones. (Tomado de Jao, et al. PNAS. 2013;110:13904 9).
Generar mutaciones tipo indel sobre un gene (ADN blanco) específico puede usarse para modificar alguna estructura o función celular en particular, es decir para modificar el fenotipo de un organismo. CRISPR-Cas ha demostrado funcionar de esta manera en todos los organismos en que ha sido probado. Un ejemplo “viejo”, publicado en agosto de 20137, demuestra a simple vista la inactivación del gene de la tirosinasa en el pez cebra. Este gene codifica a una enzima de la vía de síntesis de la melanina, el pigmento que le da al embrión de estos peces su patrón de manchas característico (figura 4a), de ahí que un pez carente de este gene debería ser albino.
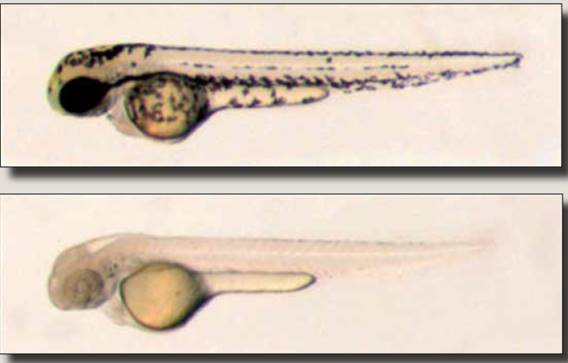
Figura 4 Embriones silvestres de pez cebra recién fertilizados (estadio embrionario de una célula) se inyectaron con un mARN que codifica a la endonucleasa Cas9 y con un sgARN específico para la tirosinasa (b) o con un sgARN control negativo (a). Las fotos muestran el aspecto que tenían a las 48 horas de desarrollo. Nótese, la falta de pigmentación en los embriones en que se mutó el gene de la tirosinasa. (Tomado de Jao, et al. PNAS. 2013;110:13904-9).
Este experimento se hizo co-inyectando embriones del pez cebra con un sgARN complementario al gene de tirosinasa junto con un mARN que se traduce en la proteína Cas9. Muchos de los embriones inyectados resultaron con algún grado de albinismo, en algunos de ellos casi completo, indicando que ambos alelos del gene fueron inactivados muy temprano durante la embriogénesis (figura 4b). Secuenciando genomas de diversos embriones sometidos a este tratamiento se obtuvo la confirmación de que este fenotipo se debió a la generación de mutaciones alrededor del ADN blanco del ARN guía (figura 3).
Este trabajo demostró la factibilidad de identificar y de modificar un sitio preciso de un genoma mediante el sistema CRISPR-Cas9. Considerando el gran tamaño que los genomas suelen tener y la facilidad con que se hizo, esto es un logro formidable. Si pensamos en que así como se llevó a cabo con el pez cebra, se puede hacer con cualquier organismo viviente, empezamos a comprender el gran poder de CRISPR-Cas.
Sin embargo, esto es solo es una de las muchas aplicaciones que los científicos ya han encontrado para CRISPR. En la figura 5 se ilustran varias modificaciones que se han hecho a la Cas9 para que, sin perder su capacidad de reconocer (junto con un sgARN) una secuencia blanco de ADN, hagan otras cosas sobre él. Por ejemplo, introduciendo mutaciones de un solo aminoácido, se han inactivado cada uno de los 2 sitios activos endonucleolíticos de la enzima silvestre, dejando versiones de Cas9 que solo cortan una hebra del ADN (figura 5a), ya sea la apareada al sgARN o la que queda libre. Estas Cas9 que solo cortan una cadena se denominan nickases es decir que no rompen la doble hélice, solo le introducen un nick (rompen un enlace fosfodiéster) en una hebra. Su utilidad reside en que, usadas en combinación, pueden crear dos nicks en relativa cercanía, pero en cadenas distintas (figura 5b). Esto redunda en mayor precisión y especificidad del sitio de corte, disminuyendo la posibilidad de cortar regiones del ADN errónea o aleatoriamente (cortes fuera del blanco [off-target]).
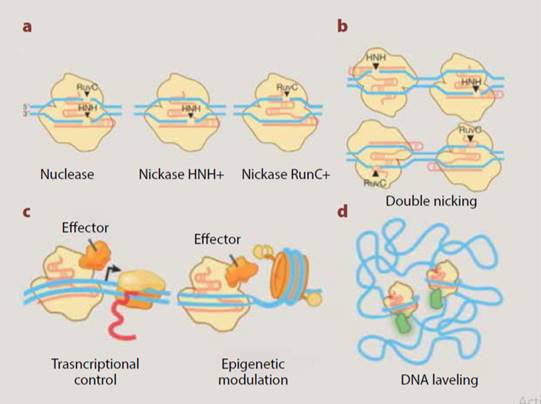
Figura 5 Gracias a distintas modificaciones de la Cas9, es posible usar el sistema CRISPR para aplicaciones limitadas solo por la imaginación. La mutación de aminoácidos específicos en Cas9 pueden inactivar 1 o 2 de los sitios de endonucleolíticos, creando “nickasas” que cortan una u otra hebra del ADN blanco (a). Una Cas9 con ambos sitios inactivados sería una enzima, que sin embargo se puede usar para dirigir a locus genómicos específicos reguladores transcripcionales o marcas fluorescentes (c y d, ver texto). (Tomado de Hsu, et al. Cell. 2014:157:1262-78).
Además de esta gran ventaja, al usar dos nickasas se genera un tipo de daño en el ADN que puede ser reparado por recombinación homóloga, un sistema que es mucho más preciso que el NHEJ que repara los DSB, pues usa el alelo intacto como molde para la reparación. Esto se ha aprovechado para insertar fragmentos de ADN exógenos en el sitio reparado, creando con ello versiones transgénicas del ADN blanco. Esta nueva manera de hacer organismos transgénicos es mucho mas fácil y eficiente que las que se han utilizado durante años y con técnicas anteriores.
Ahora imaginemos una Cas9 que tenga mutados sus 2 sitios nucleolíticos, esta versión estaría “muerta” respecto a su capacidad para romper el ADN, pero si se preserva su capacidad de unirse al sitio blanco (junto con el sgARN adecuado), entonces se puede usar para “marcar” o ubicar muy específicamente un sitio del genoma. Si esta “Cas9 muerta” se fusiona con una proteína reguladora de la transcripción (“efector” en la figura 5c) o con una proteína fluorescente (figura 5d), entonces se convierte, respectivamente, en un inédito factor transcripcional para activar o reprimir la expresión de genes específicos, o en un “faro” para ubicar por microscopía el locus blanco en el interior del núcleo celular.
Al llegar a este punto espero haber convencido al lector de que, gracias a la precisión del CRISPR-Cas, es posible implementar estrategias no solo para mutar ADN, sino también para lograr otras modificaciones tales como insertar o remover fragmentos completos de ADN en alelos específicos del genoma. Esto abre la puerta para que, en un futuro muy cercano, sea posible sintetizar genomas con secuencias específicas, distintas a las silvestres. Si esto se logra, tendremos la posibilidad de reparar genes mutantes causantes de enfermedades hereditarias que hoy son incurables. En ese caso, CRISPR representaría una verdadera “medicina molecular” que haría posibles todas las promesas, hasta ahora incumplidas, de la terapia génica.
Sin embargo, como cualquier otro fuego de Prometeo que la ciencia haya entregado al ser humano, no hay duda de que el poder casi “divino” del CRISPR podría usarse con fines positivos o negativos. Existe el riesgo fundado de que el CRISPR sea usado para hacer modificaciones que vayan más allá de la terapia génica, en un intento de hacer ingeniería genética con fines eugenésicos. Esto ha causado que muchos científicos hayan alertado al público sobre este uso éticamente reprobable y hayan propuesto una moratoria de la aplicación del CRISPR en humanos (http://www.nytimes.com/2015/03/20/science/biologists-call-for-halt-to-gene-editing-technique-in-humans.html?_r=0).
La decisión de cómo usar este portentoso descubrimiento es responsabilidad de la humanidad, idealmente toda ella de una manera informada y ética. Solo recordemos, parafraseando a Maranto, que si se trata de construir un mundo y una sociedad mejor, los humanos desde hace años tenemos ya los medios necesarios; “en donde el amor, la compasión, el altruismo y la justicia han fallado, la manipulación genética tampoco tendrá éxito”.











 nueva página del texto (beta)
nueva página del texto (beta)


