1. Introducción
El inicio de cinco décadas de desarrollo y uso de los antibióticos sintéticos denominados quinolonas está marcado por el descubrimiento del ácido nalidíxico, molécula derivada de la 1,8-naftiridina, y su introducción en 1967 para uso clínico en el tratamiento de infecciones del tracto urinario ocasionadas por bacterias Gram-negativas (5). Las quinolonas han sido el centro de un considerable interés, tanto científico como clínico, debido a que ofrecen muchos de los atributos de un antibiótico ideal: alta potencia, amplio espectro de acción, buena biodisponibilidad, formulaciones orales e intravenosas, altos niveles en suero, un amplio volumen de distribución y baja incidencia de efectos adversos (3). Como ha ocurrido con todos los antibióticos empleados en el tratamiento de infecciones bacterianas, en el caso de las quiniolonas también se han detectado microorganismos resistentes a estos antibióticos. Las bacterias han desarrollado variados e ingeniosos mecanismos de tolerancia a estas drogas, codificados tanto en genes cromosómicos como en genes de plásmidos. Los plásmidos son elementos genéticos extracromosómicos de ADN que están presentes en la mayoría de las bacterias. Los plásmidos con frecuencia se pueden transferir de una célula bacteriana a otra. Los plásmidos comúnmente poseen genes que codifican propiedades adaptativas que les permiten a las bacterias sobrevivir en condiciones ambientales adversas, como la presencia de antibióticos y de otros compuestos nocivos. En esta revisión se describirán brevemente los determinantes bacterianos de resistencia a quinolonas presentes en plásmidos.
2. Desarrollo de las quinolonas
A lo largo del tiempo se han hecho diversas modificaciones al núcleo naftiridina del ácido nalidíxico, considerado la estructura base (Fig. 1), las cuales han incrementado el espectro antimicrobiano y la potencia y mejorado la biodisponibilidad de los nuevos fármacos. Así, las quinolonas se consideran antibióticos eficaces y seguros para el tratamiento de infecciones bacterianas (2). Una de las primeras modificaciones a la estructura base fue la adición de un átomo de flúor en la posición 6 (Figura 1), la cual incrementó más de 10 veces su potencia; tal modificación estabiliza la unión del antibiótico con su sitio blanco al influir en la distribución de cargas de la molécula. Otras modificaciones se han realizado en las distintas posiciones del núcleo básico, ampliando el espectro de acción de las quinolonas hacia bacterias Gram positivas y microorganismos anaerobios (10) (Fig. 1). La Figura 2 muestra las etapas de desarrollo de las distintas quinolonas y las estructuras químicas de algunos de los antibióticos representativos del grupo.
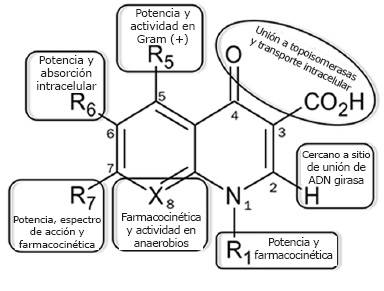
Figura 1. Estructura base de las quinolonas y funciones de los sustituyentes. Los grupos R representan las posiciones sustituidas comúnmente. Se indican los principales procesos en los que participan las modificaciones en cada posición. Las moléculas donde la posición 8 se sustituye por un átomo de carbono son quinolonas y cuando se sustituye por un átomo de nitrógeno son naftiridinas. Modificada de (2).
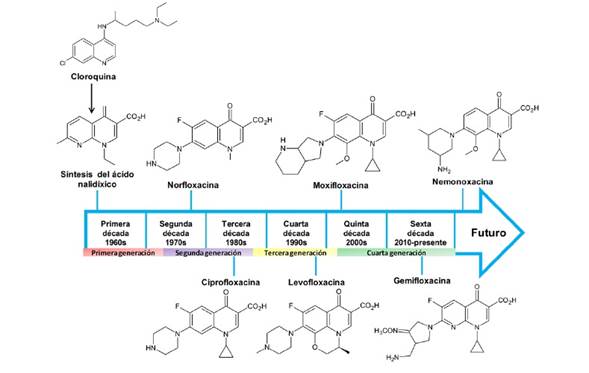
Figura 2. Etapas de desarrollo de las quinolonas. Se muestran las cuatro generaciones de quinolonas a través de casi seis décadas, desde la síntesis del ácido nalidíxico (1962) hasta el desarrollo de la nemonoxacina (2014). Se presentan las estructuras químicas de quinolonas representativas de las distintas etapas. Modificada de (5).
3. Mecanismo de acción de las quinolonas
Para ejercer sus efectos antimicrobianos, las quinolonas son primero transportadas al interior de la célula bacteriana mediante un proceso de difusión simple. El mecanismo de acción de estos antibióticos consiste en la inhibición de dos enzimas clave para la replicación, transcripción y reparación del ADN de la célula bacteriana: la ADN topoisomerasa II o ADN girasa y la ADN topoisomerasa IV (4). Estas enzimas pertenecen a la familia de topoiso-merasas tipo IIA y están compuestas cada una de dos subunidades: GyrA y GyrB para la ADN girasa, ParC y ParE para la topoisomerasa IV (Fig. 3). Ambas enzimas contribuyen al desenrollamiento del ADN que se requiere para que éste pueda ser procesado, pero funcionan de diferente manera: la girasa remueve superenrollamientos positivos y avanza delante de la horquilla de replicación, mientras que la topoisomerasa IV introduce superenrollamientos positivos avanzando detrás de la horquilla de replicación; esta enzima, además, participa en la segregación de los cromosomas posterior a la división celular (8).
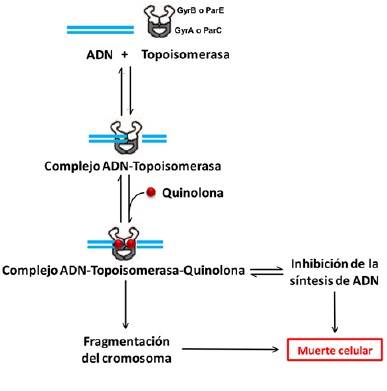
Figura 3. Mecanismo de acción de las quinolonas. Para relajar el ADN superenrollado y permitir que el ADN pueda ser procesado, las topoisomerasas introducen cortes en la molécula. La formación del complejo ADN-Topoisomerasa-Quinolona inhibe la replicación del ADN, impidiendo que los cortes en la doble hélice sean resellados, y el cromosoma se fragmenta; ambas situaciones pueden conducir a la muerte celular. Modificada de (4).
Las topoisomerasas tipo IIA introducen un par de cortes en la cadena sencilla de ADN, uniéndose covalentemente al extremo 5' formando el complejo ADN-topoisomerasa (Fig. 3), lo cual relaja el superenrollamiento de la doble hélice. Las quinolonas se unen al complejo, atrapando a la topoisomerasa en el ADN, lo que da como resultado la inhibición de la replicación. Este nuevo complejo ternario, ADN-Topoisomerasa-Quinolona (Fig. 3), bloquea el movimiento de la horquilla de replicación y de los complejos transcripcionales, inhibiendo el crecimiento bacteriano y causando eventualmente la muerte celular (9).
4. Resistencia bacteriana a quinolonas
Debido al origen sintético de las quinolonas, se consideró inicialmente que el único mecanismo de resistencia que las bacterias podrían adquirir serían las mutaciones en los genes que codifican a las proteínas blanco, las topoisomerasas tipo IIA, o a los transportadores de membrana, que expulsan compuestos tóxicos del citoplasma. En cuanto a la adquisición de resistencia mediante la transferencia de genes plasmídicos, se consideró que no sería posible, ya que el origen de los genes que confieren resistencia a antibióticos no-sintéticos generalmente radica en los propios microorganismos que los producen (7). En los últimos años se ha encontrado, sin embargo, que los sistemas de resistencia codificados en plásmidos representan una importante fuente de mecanismos bacterianos novedosos capaces de contrarrestar el efecto nocivo de las quinolonas. El análisis de los genes y proteínas relacionados con la resistencia a quinolonas sugiere que éstos surgieron mucho tiempo antes del uso de dichos antibióticos.
Los sistemas de resistencia a quinolonas pueden dividirse en dos grupos (1) (Fig. 4): i) los que están codificados en genes cromosómicos, que incluyen las modificaciones en los sitios blanco del antibiótico y los sistemas de expulsión; y ii) los codificados por genes presentes en plásmidos, que incluyen a las proteínas Qnr, la enzima aminoglucósido acetil transferasa modificada y los sistemas membranales de expulsión. Los sistemas plasmídicos se analizarán a continuación.
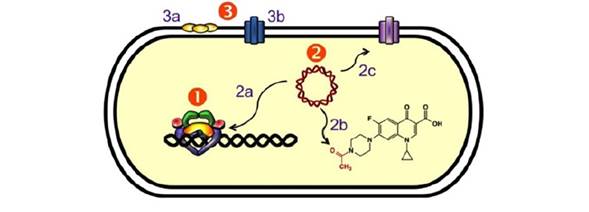
Figura 4. Sistemas de resistencia a quinolonas. Se representa una célula bacteriana mostrando los distintos sistemas de resistencia; los detalles se describen en el texto. (1) Resistencia mediada por mutaciones en los genes de las enzimas ADN girasa y topoisomerasa IV, que disminuyen la unión del antibiótico. (2) Resistencia mediada por genes plasmídicos: (2a) Proteínas Qnr (en amarillo), que inhiben la unión de la quinolona al complejo ADN-topoisomerasa; (2b) La enzima AAC(6')-Ib-cr, que acetila algunas quinolonas reduciendo su efectividad; (2c) Sistemas membranales de expulsión, que disminuyen la concentración intracelular de las quinolonas. (3) Resistencia mediada por genes cromosómicos: (3a) Disminución de la expresión de porinas, que limita el ingreso del antibiótico; (3b) Sistemas de expulsión. Modificada de (1).
5. Resistencia a quinolonas determinada por gener plasmídicos
5.1. Proteínas Qnr
Las proteínas Qnr se identificaron por primera vez en el plásmido pMG252, aislado de una cepa clínica de la enterobacteria Klebsiella pneumoniae resistente a ciprofloxacina. En esta bacteria se identificó una proteína de 219 aminoácidos, QnrA, que confiere resistencia a quinolonas (12). Las proteínas Qnr pertenecen a la familia de pentapéptidos repetidos, llamada así porque sus miembros contienen un motivo recurrente de cinco aminoácidos en tándem [Ser, Thr, Ala o Val] [Asp o Asn] [Leu o Phe] [Ser, Thr o Arg] [Gly] (12). Aunque se han identificado en los distintos genomas bacterianos más de 500 secuencias que contienen el motivo pentapéptido repetido, se desconoce la función de casi todas ellas. Las secuencias que codifican proteínas Qnr se encuentran ampliamente distribuidas en los genomas de enterobacterias. Además de QnrA, se han estudiado las proteínas QnrB, también de K. pneumoniae, y QnrS, de Shigella flexneri. Aunque se sabe que estas proteínas se unen al complejo ADN-Topoisomerasa, impidiendo la unión de las quinolonas (Fig. 4), no se conoce con detalle su mecanismo de acción.
5.2 Modificación enzimática de las quinolonas
Un sistema de modificación química de quinolonas, codificado en el plásmido pHSH10-2, se identificó en una cepa clínica de Escherichia coli resistente a ciprofloxacina (11). Mediante mutagénesis por transposición se encontró que pHSH10-2 codifica a la proteína AAC(6')-Ib-cr, cuya secuencia muestra similitud con las aminoglucósido acetil transferasas, enzimas que confieren resistencia a antibióticos del grupo de los aminoglucósidos por medio de su acetilación y consecuente inactivación. El análisis de la secuencia de esta enzima reveló que se encuentra modificada en los aminoácidos 102 (Trp ->Arg) y 179 (Asp -> Tyr), con respecto a las aminoglucósido acetil transferasas; la mutagénesis dirigida a los codones correspondientes mostró que, en ausencia de dichos cambios, la proteína no se asocia con la resistencia a quinolonas. Así, las mutaciones causaron un cambio drástico en la especificidad de sustrato en la enzima AAC(6')-Ib-cr. Mediante ensayos de acetilación con ciprofloxacina como sustrato, se confirmó que la enzima introduce un acetilo en el nitrógeno del grupo piperazina en la posición 6 del antibiótico (Fig. 1); la modificación reduce la afinidad de esta quinolona por las topoisomerasas (Fig. 4), originando la resistencia.
5.3 Sistemas de expulsión
A la fecha se han identificado dos plásmidos, pHPA y pOLA52, que confieren resistencia a quinolonas mediante sistemas membranales de expulsión, los cuales causan una disminución de la concentración intracelular del fármaco (Fig. 4).
El plásmido pHPA, aislado de una cepa clínica de E. coli codifica a la proteína de membrana interna QepA, la cual se agrupa en la superfamilia de transportadores MFS. Esta proteína confiere resistencia mediante la expulsión del citoplasma de quinolonas hidrofilicas como ciprofloxacina y norfloxacina (Fig. 2).
Por otra parte, el plásmido pOLA52, proveniente de una cepa de E. coli aislada de estiércol de cerdos, codifica a OqxA, una proteína de membrana interna perteneciente a la familia de transportadores RND, y a OqxB, una proteína del periplasma (6).
Las proteínas OqxAB confieren resistencia a ácido nalidíxico y ciprofloxacina; el hallazgo de que son capaces de expulsar al colorante bromuro de etidio, sugiere que la resistencia a quinolonas también está dada por su expulsión del citoplasma. Para conferir resistencia a quinolonas, OqxA y OqxB requieren de un componente adicional de la membrana externa, la proteína TolC, por lo que funcionan como un complejo tripartita de expulsión, OqxAB-TolC, similar a algunos sistemas codificados por genes cromosómicos (Fig. 5).
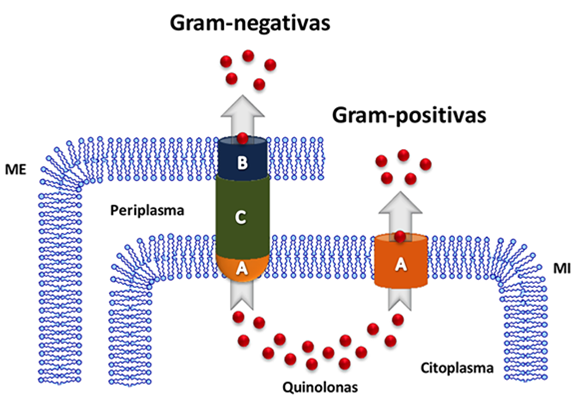
Figura 5. Sistemas bacterianos de expulsión. Se muestra esquemáticamente el funcionamiento de un complejo tripartita de expulsión. A) Proteína de la membrana interna (MI), que une a la quinolona (esferas de color rojo) en el citoplasma. B) proteína de la membrana externa (ME), que expulsa al antibiótico al exterior de la célula. C) Proteína del periplasma, que conecta a los dos componentes membranales. En la parte superior se señala el tipo de bacteria donde se encuentra cada sistema de expulsión.
6. Consideraciones finales
La historia de las quinolonas abarca más de 50 años, período en el cual se ha desarrollado una gran variedad de moléculas con actividad antimicrobiana. Sin embargo, de manera paralela al desarrollo de nuevas quinolonas, el número de bacterias resistentes ha ido en aumento. No obstante, la ciprofloxacina, con más de 30 años de uso, continúa siendo un fármaco de primera elección en el tratamiento de infecciones por enterobacterias.
A pesar de que se han descrito varios mecanismos de resistencia a quinolonas, la forma más común de resistencia es la causada por mutaciones especificas en los genes de la ADN girasa y de la topoisomerasa IV. Como se describió en este trabajo, los determinantes de resistencia codificados en plásmidos representan una fuente continua de mecanismos evolutivos de las bacterias que les permiten contrarrestar el efecto tóxico de las quinolonas.











 nueva página del texto (beta)
nueva página del texto (beta)


